2. 100071 北京,解放军总医院第五医学中心呼吸与危重症医学科;
3. 100071 北京,军事科学院军事医学研究院微生物流行病研究所,病原微生物安全国家重点实验室;
4. 100029 北京,北京化工大学生命科学与技术学院
2. Department of Respiratory and Critical Care Diseases, the Fifth Medical Center, PLA Chinese General Hospital, Beijing, 100071;
3. State Key Laboratory of Pathogen and Biosecurity, Institute of Microbiology and Epidemiology, Academy of Military Medical Sciences, Beijing, 100071;
4. College of Life Science and Technology, Beijing University of Chemical Technology, Beijing, 100029, China
金黄色葡萄球菌(Staphylococcus aureus)是一种机会致病菌,无症状地定植于全球约三分之一人口的皮肤和鼻孔[1-2]。金黄色葡萄球菌是医院和社区常见的感染细菌之一,尤其是在老年人中[3];近20年来,金黄色葡萄球菌的感染趋势有所增加[4]。据报道,由于金黄色葡萄球菌引起的细菌血症的死亡率高达15%~60%[5]。细菌耐药性的产生是导致更高发病率和死亡率的主要原因。
在20世纪40年代初期,第1种抗生素——青霉素(benzylpenicillin)问世,解决了葡萄球菌感染的问题,但是持续使用该试剂导致了耐青霉素酶(β-内酰胺酶)金黄色葡萄球菌的出现;随后甲氧西林开始应用,但1年后便出现新的耐药菌株,即耐甲氧西林金黄色葡萄球菌(MRSA);之后万古霉素、利奈唑胺和达普霉素作为对抗MRSA的新型药物,目前已有耐药菌株报道。这些耐药菌株不断增多,且在世界范围内流行,其中MRSA已成为医院获得性(HA)多药耐药(MDR)生物全球威胁的主要原因[6-7]。由金黄色葡萄球菌引起的心内膜炎、骨髓炎、肺炎及手术伤口和血管内装置感染代表了主要的公共卫生问题[8-9]。最近研究表明,在美国每年有超过80 000例侵袭性金黄色葡萄球菌感染和11 000例死于MRSA感染[10]。
噬菌体是专门侵染细菌的病毒,通常分为两类:①裂解性噬菌体,感染宿主菌并在细菌内复制裂解细胞释放其后代;②温和性噬菌体,可整合在细菌染色体中或作为染色体外质粒样遗传元件,与细菌一起复制。裂解性噬菌体具有较大的抗菌应用价值。由于全球金黄色葡萄球菌感染的严重性,活跃于自然界的噬菌体引起了科学家们广泛的关注,临床试验和动物模型也证明了金黄色葡萄球菌噬菌体可有效治疗金黄色葡萄球菌引起的感染,尤其是金黄色葡萄球菌的耐药菌菌株感染[11-13]。截止到2019年,目前已被分离并释放到公共数据库NCBI的金黄色葡萄球菌噬菌体有400余株(数据获取日期:2019年10月1日),但是已明确分类的噬菌体仅有54株,包括Staphylococcus virus G1 (19株), Staphylococcus phage 812 (17株), Staphylococcus virus 11 (8株), Staphylococcus virus Twort (6株), Staphylococcus virus 13 (4株)。2009年,RHOADS等[14]将金黄色葡萄球菌的噬菌体用于临床试验治疗慢性静脉性腿部溃疡,结果显示使用噬菌体治疗没有安全性问题。因此,金黄色葡萄球菌噬菌体可作为潜在的噬菌体治疗制剂。
本研究报道了一株裂解性金黄色葡萄球菌噬菌体,用透射电子显微镜观察其形态,研究噬菌体的生物特性,确定并表征了其基因组序列,为进一步研究金黄色葡萄球菌噬菌体在临床治疗中作为抗生素替代品的应用奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株来源与培养条件金黄色葡萄球菌株sa35分离自中国人民解放军总医院第五医学中心南院区检验科。使用16S rRNA引物扩增基因和解放军第五医学中心南院区检验科VITEK 2 Compact全自动药敏分析仪,进行初步的金黄色葡萄球菌菌种鉴定和耐药分析[15]。然后使用终浓度25%的甘油保存至军事医学科学院微生物流行病研究所-80 ℃细菌库中。金黄色葡萄球菌通常在LB(Luria-Bertani)培养基中37 ℃通风培养。
1.1.2 主要溶液配制LB液体培养基:氯化钠NaCl 10 g,酵母提取物Yeast extract 5 g,胰蛋白胨Tryptone 10 g,加水至1 L,于1×105 Pa灭菌20 min。LB固体培养基:氯化钠NaCl 10 g,酵母提取物Yeast extract 5 g,胰蛋白胨Tryptone 10 g,琼脂粉15 g,加水至1 L,1×105 Pa灭菌20 min。LB半固体培养基:氯化钠NaCl 10 g,酵母提取物Yeast extract 5 g,胰蛋白胨Tryptone 10 g,琼脂粉7.5 g,加水至1 L,1×105 Pa灭菌20 min。
1.1.3 试剂和仪器Taq DNA聚合酶(北京全式金有限公司);0.22 μm微孔过滤器(Pall Corporation,USA);酚:氯仿:异戊醇液(25 :24 :1)、DNA平衡酚(北京索莱宝科技有限公司);MiSeq测序仪(Illumina,USA);蛋白酶K、RNase、DNase(Thermo Fisher Scientific,USA);50 mL超速离心管、超速离心机Optima XE-100 Ultracentrifuge(Beckman,USA)。
1.2 噬菌体的分离噬菌体的分离纯化按照文献[16]所描述的步骤进行。筛选噬菌体的污水取自解放军总医院第五医学中心南院区污水站。将污水以12 000×g离心10 min,通过0.22 μm过滤器过滤。然后,将3 mL滤液与指数生长期[D(600) = 0.4~0.6]的1 mL sa35混合并加入3 mL 3×LB培养基。将细菌和污水滤液的混合物在37 ℃下震荡培养过夜以获得更多噬菌体,12 000×g离心10 min,然后100 μL上清液和500 μL指示菌指数生长期混合,混合物加入约5 mL上层琼脂(含0.7%琼脂,50 ℃水浴)中,倒入含有下层琼脂的LB平板上(双层法)[17],待上层琼脂凝固后移至37 ℃恒温培养箱培养过夜。然后,选择单个透明噬菌斑并在宿主细菌sa35中再扩增,通过选择单个透明噬斑进行5次纯化后获得纯的噬菌体菌株。分离的噬菌体保存在4 ℃备用。
1.3 噬菌体的纯化噬菌体的纯化使用蔗糖密度梯度离心法。简要步骤为:先在50 mL超速离心管中加入25~30 mL的上述步骤所获取的噬菌体裂解液,然后在离心管中加入30%蔗糖,加的时候用10 mL长针头从底部缓慢往上加入。使用Optima XE-100 Ultracentrifuge超速离心机,SWTi转子30 000 r/min 4 ℃离心2 h。去上清,用适量TEN(10 mmol/L Tris-Cl,1 mmol/L EDTA,100 mmol/L NaCl)溶液悬浮沉淀,获得了纯化的病毒,-20 ℃冻存备用。
1.4 噬菌体电镜噬菌体vB_SauH_IME522的形态进一步可视化通过透射电子显微镜(TEM)观察[18]。纳米材料负染制样:①将镀碳支持膜铜网放到封口膜上,滴一滴样品(大约30 μm)到支持膜上,停留5~10 min,用带尖的滤纸从边缘部分吸去多余的溶液,转移到滤纸上停留1 min左右。②再把干燥后的支持膜放到封口膜上,滴一滴醋酸双氧铀染液,染色90 s, 用带尖的滤纸吸去多余的染液,夹到滤纸上干燥3 h观察。在100 kV TEM(JEM-1200EX,Japan)下观察噬菌体的形态。
1.5 噬菌体最佳感染复数(multiplicity of infection, MOI)测定参照XING等[19]的方法略有改动。为了确定最佳MOI,根据感染比例,将噬菌体和宿主菌液分别按照MOI为100、10、1、0.1、0.01、0.001的拷贝数比例混合,各200 μL,加入新鲜的5 mL LB培养基中,37 ℃,220 r/min振荡培养6 h后,6 000×g离心15 min除去细胞沉淀。收集上清液,使用0.22 μm的滤膜过滤,通过双层平板法测定噬菌体的滴度。其中噬菌体滴度最高的MOI是最佳的感染复数[20]。每次独立实验重复3次。
1.6 噬菌体一步生长曲线(one-step growth)的测定参照ZHAO等[21]的方法略有改动。使用培养至指数生长中期的金黄色葡萄球菌菌株sa35[D(600)=0.2~0.6]并与噬菌体vB_SauH_IME522以0.01(106 :108)的MOI混合,将混合物置于37 ℃恒温培养箱孵育15 min,使噬菌体与细菌充分吸附,然后将混合物以10 000×g离心1 min,将含有宿主细菌的沉淀物用37 ℃新鲜LB洗涤2次。然后将混合物转移到15 mL LB培养基中并在37 ℃振荡培养。然后在感染后的前100 min每10 min收集样品1次,之后每20 min收集样品1次,直到240 min。使用双层琼脂测定噬菌体滴度,每次独立实验重复3次。
1.7 噬菌体裂解谱测定通过点板试验,将受试菌(共10株金黄色葡萄球菌)培养至D(600)约为0.5,取出500 μL铺双层平板,室温静置5 min,将5 μL噬菌体浓缩液滴于双层平板上,室温静置至噬菌体浓缩液被吸收后,倒置于37 ℃温箱中9 h后观察有无噬菌斑产生。
1.8 噬菌体基因组提取噬菌体基因组提取使用SDS/蛋白酶裂解方案[22]。取600 μL已纯化过滤的噬菌体裂解液,加入DNase Ⅰ(1 μg/mL)和RNase A(1 μg/mL)(Thermo Scientific,USA),将混合物充分涡旋并在37 ℃下温育过夜;80 ℃酶灭活20 min;加入裂解缓冲液(终浓度:0.5% SDS,20 mmol/L EDTA和50 μg/mL蛋白酶K)(Thermo Scientific,USA),将混合物在金属浴中56 ℃温育1 h;加入等体积的DNA平衡酚(北京索莱宝科技有限公司),充分涡旋振荡1 min,然后将混合物12 000×g离心10 min。转移上层水相到一个新的1.5 mL离心管中;加入相同体积的苯酚-氯仿-异戊醇(25 :24 :1)(Sigma),充分涡旋振荡1 min,12 000×g离心10 min,将上层水相再次转移至新的1.5 mL离心管中,加入等体积的异丙醇(国药化学试剂有限公司),-20 ℃放置4 h沉淀基因组DNA。然后将样品4 ℃条件下1 000×g离心20 min后收集沉淀物。用75%的乙醇(预冷)洗涤DNA沉淀,在室温下风干,然后用30 μL无核酸水重悬DNA沉淀,并储存在-20 ℃备用。
1.9 噬菌体的全基因组测序及生物信息学分析噬菌体vB_SauH_IME522的DNA全基因组测序使用的是Illumina Miseq测序平台。DNA文库构建使用NEBNextⓇ UltraTMⅡ DNA Library Prep Kit来完成。在去离子水中溶解100 ng的基因组DNA,总体积为50 μL,由Bioruptor超声系统打断,超声处理后的基因组片段使用接头修复。接头-片段产物使用PCR扩增,然后使用AMPure XP珠子纯化。在测序之前,使用Bioanalyser 2100(Agilent Technologies,USA)对文库进行了质量控制分析。然后使用Illumina Miseq(Diego,CA,USA)测序。基因组序列组装使用的是NewblerV3.0软件[23](Roche Applied Science)和CLC 3.0软件。使用RAST在线数据库[24](http://www.rast.nmpdr.org)进行开放阅读框(ORF)初步确定,所有的ORF使用NCBI(National Center for Biotechnology Information)的蛋白质的基本局部比对工具BLASTp(http://www.ncbi.nlm.nih.gov/BLAST/)手动验证。此外,进一步的基因组分析,使用tRNA scan-SE 2.0版(http://lowelab.ucsc.edu/tRNAs can-SE /)[25]预测tRNA,搜索基因组的原核生物启动子序列(http://www.fruit fly.org/seq tools /promo ter.html)和终止子序列(http://rna.igmor s.upsud.fr/toolbox/arnold/)。选择DNA聚合酶(ORF157)使用MEGA 6.0版[26] Neighbor joining的方法构建系统发育树。
2 结果 2.1 宿主菌的药敏性宿主菌的耐药性分析见表 1。结果显示,宿主菌sa35为多重耐药菌。
| 抗生素 | 最小抑菌浓度 /μg·mL-1 |
药敏性 |
| β-内酰胺酶Beta-Lactamase | POS | + |
| 头孢西丁筛查Cefoxitin Screen | POS | + |
| 氨苄青霉素Benzylpenicllin | ≥0.5 | R |
| 奥沙西林Oxacllin | ≥4 | R |
| 庆大霉素Gentamicin | ≤0.5 | S |
| 环丙沙星Ciprofloxacin | ≥8 | R |
| 左氧氟沙星Levofloxacin | ≥8 | R |
| 莫西沙星Moxifloxacin | ≥8 | R |
| 红霉素Erythromycin | ≥8 | R |
| 克林霉素Clindamycin | ≥8 | R |
| 奎奴普丁/达福普汀Quinupristin/Dalfopristin | 0.5 | S |
| 利奈利德Linezilid | 2 | S |
| 万古霉素Vancomycin | 1 | S |
| 替曲丁Tetracydine | ≥16 | R |
| 替吉西定Tigecydine | ≤0.12 | S |
| 呋喃妥因Nitrofurantoin | 32 | S |
| 利福平Rifampicin | ≥32 | R |
| 甲氧苄啶/磺胺甲恶唑Trimethoprim/sulfamethoxazole | ≥10 | S |
| S:敏感;I:中介度;R:耐药;POS:头孢西丁筛查实验阳性 | ||
2.2 噬菌体的分离与鉴定
从医院污水中分离到一株金黄色葡萄球菌噬菌体,并命名为vB_SauH_IME522。噬菌体经过5次单斑分离纯化之后,37 ℃下孵育12 h,在双层平板上,该噬菌体产生直径1~2 mm的斑块。电子显微照片(图 1)显示噬菌体具有典型的二十面体结构和收缩的尾部,其头部直径为(78±1)nm,尾部长度为(135±2)nm,根据目前的国际病毒分类委员会分类系统,噬菌体vB_SauH_IME522被划分为Herelleviridae科。

|
| 图 1 噬菌斑(A)及噬菌体vB_SauH_IME522(B)的显微观察 |
2.3 噬菌体最佳感染复数
噬菌体与宿主菌金黄色葡萄球菌菌株sa35按一定比例混合,当以MOI为0.01感染时,噬菌体vB_SauM_IHE522产生最大滴度(图 2),表明0.01是噬菌体vB_SauH_IME522的最佳MOI。

|
| 图 2 噬菌体vB_SauH_IME522最佳感染复数的测定 |
2.4 噬菌体的一步生长曲线
由图 3可知噬菌体vB_SauH_IME522的噬菌体与对数生长期的金黄色葡萄球菌混合培养20 min曲线走势无明显变化,即噬菌体的潜伏期(latent period)约为20 min,而后噬菌体在约200 min后其滴度趋于平稳,则噬菌体感染期的时间约为200 min,由此可得裂解量=裂解末期噬菌体滴度(8.97×109)/感染初期宿主菌浓度(1×108)=89 PFU/cell,即噬菌体vB_SauH_IME522感染宿主菌的裂解量约为89 PFU/cell。

|
| 图 3 噬菌体一步生长曲线 |
2.5 噬菌体裂解谱
通过点板实验测定裂解谱vB_SauH_IME522,结果显示,该噬菌体可以裂解10株金黄色葡萄球菌中的4株(sa1、sa3、sa6、sa9),其中在sa1、sa9细菌平板上可以形成较透亮的噬菌斑,而在sa3、sa6细菌平板上为圆形混浊噬菌斑(表 2)。这表明噬菌体具有一定的裂解谱,且对不同的细菌裂解能力不一。
| 金黄色葡萄球菌编号 | 裂解性 |
| sa1 | ++ |
| sa2 | - |
| sa3 | + |
| sa4 | - |
| sa5 | - |
| sa6 | + |
| sa7 | - |
| sa8 | - |
| sa9 | ++ |
| sa10 | - |
|
++:形成圆形透亮噬菌斑;+:形成圆形混浊的噬菌斑; -:没有观察到噬菌斑形成 | |
2.6 噬菌体的全基因组分析
从头组装噬菌体vB_SauH_IME522基因组获得全基因组序列。噬菌体vB_SauH_IME522具有线性dsDNA基因组,基因组全长为140 246 bp,G+C含量为30.2%,其核酸序列NCBI登录号为MN304941。基因组序列注释显示,噬菌体vB_SauH_IME522基因组中存在223个开放阅读框(ORF),4个tRNA基因,164个启动子序列(图 4A)和66个终止子序列(图 4B)。噬菌体vB_SauH_IME522编码的ORF中,23%的ORFs(52个ORFs)预测为编码序列(CDS),剩余171个ORFs注释为假设蛋白质或未知功能蛋白质(表 3)。ORF长度从102 bp到4 056 bp不等,平均ORF长度约570 bp。在233个ORF中,208个ORFs起始密码子为ATG,5个ORFs起始密码子为GTG,10个ORFs起始密码子为TTG;终止密码子中,163个ORF为TAA,49个ORF为TAG,11个ORF为TGA。

|
| 堆块的高度反映了序列在该位置的保守性;堆块内符号的高度描述了该位置的核苷酸碱基的相对频率 图 4 在噬菌体vB_SauH_IME522基因组中发现的启动子(A)和终止子(B)共有序列 |
噬菌体vB_SauH_IME522基因组与Herelleviridae家族病毒的相似性范围从61%~99%,观察到最高的相似性的是Staphylococcus phage HSA30(MG557618.1,99%),Staphylococcus phage VB_SavM_JYL01(MH159197.1,99%),Staphylococcus phage vB_SauM_LM12(MG721208.1,98%)和Staphylococcus phage SA3(MF001365.1,99%)。基因注释结果显示,噬菌体vB_SauH_IME522基因组含有7个抗生素抗性预测区域,其中6个为庆大霉素抗性预测区域,1个为克林霉素抗性预测区域。通过BlastN程序,各个抗性区域未比对到同源耐药基因。然而,在相似性最高的噬菌体基因组中并未预测到与噬菌体vB_SauH_IME522相似的耐药相关预测区域,即7个抗生素抗性预测区域均为噬菌体vB_SauH_IME522所独有。进一步的基因组分析,如图 5所示,在Mauve中对噬菌体基因组的分析表明它们是相关但彼此不同的。噬菌体vB_SauH_IME522基因组还编码4种tRNA,即Asp-tRNA,Phe-tRNA,Pseud-tRNA和Met-tRNA。编码它们的区域分别跨越核苷酸1 431~1 504,1 511~1 583,1 590~1 661和23 241~23 312。

|
| 相似性水平随曲线的高度而变化,并且块内的强度与平均核苷酸序列同一性成比例;块内部或外部的白色空间代表这些噬菌体的基因组之间的差异区域 图 5 噬菌体vB_SauH_IME522(A)与近源噬菌体Staphylococcus phage HSA30(B),Staphylococcus phage VB_SavM_JYL01(C),Staphylococcus phage vB_SauM_LM12(D)和Staphylococcus phage SA3(E)同源性分析 |
对噬菌体vB_SauH_IME522注释的所有功能蛋白质进行分类,该基因组主要涉及4个功能模块,包括DNA复制和调控(DNA连接酶,DNA聚合酶,DNA引物酶,DNA解旋酶,RNA聚合酶,整合宿主因子);代谢模块(推定的核糖核酸酶,重组核酸内切酶,重组核酸外切酶);噬菌体结构和包装蛋白质(主要尾纤维蛋白,推定的底板蛋白,尾管蛋白,尾鞘蛋白,主要衣壳蛋白,推定的大终止酶亚基,推定的门蛋白)和与宿主溶解有关的蛋白质(噬菌体溶素,穴蛋白)(表 3,图 6)。
| ORF序号 | 功能 | 启始 | 终止 | GenBank编号 | 一致性(%)(aa)a | E值 |
| 3 | putative holin | 2091 | 2594 | JQ686190.1 | 167/167(100%) | 7.00E-117 |
| 4 | lysin | 2594 | 4081 | KY779849.1 | 495/495(100%) | 0 |
| 6 | putative membrane protein | 4503 | 5297 | JQ686190.1 | 264/264(100%) | 0 |
| 7 | putative transglycosylase IsaA | 5494 | 6186 | JQ686190.1 | 230/230(100%) | 3.00E-168 |
| 9 | transcriptional regulator | 6525 | 6755 | NC_019726.1 | 75/75(100%) | 1.00E-46 |
| 12 | putative ribonuclease | 7590 | 8015 | JQ686190.1 | 141/141(100%) | 3.00E-95 |
| 16 | putative DNA ligase | 9733 | 10626 | NC_025426.1 | 297/297(100%) | 0 |
| 30 | putative membrane protein | 18381 | 18545 | JQ686190.1 | 54/54(100%) | 3.00E-29 |
| 42 | serine/threonine protein phosphatase | 23750 | 24451 | MH159197.1 | 233/233(100%) | 1.00E-169 |
| 78 | terminal repeat-encoded protein | 38325 | 38035 | NC_025426.1 | 96/96(100%) | 8.00E-62 |
| 95 | nicotinamide phosphoribosyl transferase | 45035 | 43566 | NC_025426.1 | 489/489(100%) | 0 |
| 96 | ribose-phosphate pyrophosphokinase | 45961 | 45053 | JQ686190.1 | 302/302(100%) | 0 |
| 98 | putative membrane protein | 46634 | 46344 | JQ686190.1 | 96/96(100%) | 1.00E-59 |
| 101 | putative membrane protein | 47246 | 47088 | JQ686190.1 | 52/52(100%) | 5.00E-24 |
| 116 | putative membrane protein | 52266 | 51856 | JQ686190.1 | 136/136(100%) | 3.00E-90 |
| 121 | putative membrane protein | 54305 | 54027 | JQ686190.1 | 92/92(100%) | 1.00E-56 |
| 124 | putative membrane protein | 55228 | 55046 | JQ686190.1 | 60/60(100%) | 5.00E-33 |
| 128 | putative membrane protein | 56661 | 56017 | LR215718.1 | 212/214(99%) | 4.00E-149 |
| 134 | putative membrane protein | 58770 | 58372 | JQ686190.1 | 130/132(98%) | 1.00E-89 |
| 145 | putative DNA repair exonuclease | 66218 | 64968 | JQ686190.1 | 416/416(100%) | 0 |
| 148 | putative major tail protein | 67552 | 67325 | JQ686190.1 | 74/75(99%) | 4.00E-44 |
| 149 | major tail protein | 68079 | 67567 | NC_019726.1 | 168/170(99%) | 8.00E-114 |
| 151 | RNA polymerase sigma factor | 69523 | 68861 | KY794642.1 | 220/220(100%) | 6.00E-157 |
| 153 | repair recombinase | 71123 | 69867 | LR215721.1 | 417/418(99%) | 0 |
| 157 | DNA polymerase A | 76473 | 73255 | LR215718.1 | 1063/1072(99%) | 0 |
| 158 | integration host factor | 76854 | 76549 | NC_025416.1 | 100/101(99%) | 2.00E-66 |
| 160 | thioredoxin-like protein | 77988 | 77668 | KY779849.1 | 106/106(100%) | 1.00E-69 |
| 162 | ribonucleoside-diphosphate beta subunit | 79368 | 78319 | NC_025426.1 | 349/349(100%) | 0 |
| 163 | ribonucleotide reductase large subunit | 81496 | 79382 | NC_025426.1 | 703/704(99%) | 0 |
| 164 | ribonucleotide reduction protein NrdI | 81903 | 81511 | NC_025416.1 | 129/130(99%) | 1.00E-88 |
| 168 | DNA primase | 84438 | 83371 | MH159197.1 | 355/355(100%) | 0 |
| 170 | recombination endonuclease | 86968 | 85049 | NC_025426.1 | 639/639(100%) | 0 |
| 172 | recombination exonuclease | 88370 | 87345 | NC_025426.1 | 341/341(100%) | 0 |
| 174 | helicase DnaB-like | 90310 | 88868 | NC_025426.1 | 480/480(100%) | 0 |
| 175 | putative Rep protein | 91916 | 90303 | NC_025426.1 | 537/537(100%) | 0 |
| 176 | DNA helicase | 93676 | 91928 | MH159197.1 | 582/582(100%) | 0 |
| 177 | putative tail fiber protein | 95142 | 93766 | NC_025426.1 | 458/458(100%) | 0 |
| 181 | adsorption-associated tail protein | 101125 | 97667 | NC_025426.1 | 1152/1152(100%) | 0 |
| 184 | putative tail protein | 105910 | 104864 | JQ686190.1 | 346/348(99%) | 0 |
| 185 | putative baseplate protein | 106629 | 105925 | JQ686190.1 | 233/234(99%) | 1.00E-171 |
| 188 | glycerophosphoryl diester phosphodiesterase | 110597 | 108051 | NC_025426.1 | 848/848(100%) | 0 |
| 190 | N-acetylmuramoyl-L-alanine amidase | 113924 | 111498 | MH159197.1 | 808/808(100%) | 0 |
| 192 | RNA polymerase | 118649 | 118113 | NC_025426.1 | 178/178(100%) | 7.00E-127 |
| 199 | tail tube protein | 121173 | 120745 | NC_023573.1 | 141/142(99%) | 2.00E-99 |
| 200 | tail sheath protein | 123009 | 121246 | NC_025426.1 | 587/587(100%) | 0 |
| 204 | capsid protein | 125606 | 124728 | NC_019726.1 | 291/292(99%) | 0 |
| 207 | putative capsid protein | 128320 | 126929 | JQ686190.1 | 463/463(100%) | 0 |
| 209 | prohead protease | 130187 | 129414 | KX011028.1 | 255/257(99%) | 0 |
| 210 | putative portal protein | 132071 | 130380 | JQ686190.1 | 563/563(100%) | 0 |
| 211 | putative portal protein | 132446 | 132075 | JQ686190.1 | 123/123(100%) | 2.00E-83 |
| 213 | putative membrane protein | 134119 | 132890 | JQ686190.1 | 409/409(100%) | 0 |
| 217 | putative terminase large subunit | 137428 | 135611 | JQ686190.1 | 605/605(100%) | 0 |
| tRNA-Asp-GTC | 1431 | 1504 | rna.1 | |||
| tRNA-Phe-GAA | 1511 | 1583 | rna.1 | |||
| tRNA-Pseudo-CCA | 1590 | 1661 | rna.1 | |||
| tRNA-Met-CAT | 23241 | 23312 | rna.1 | |||
| Gentamicin resistance predicted region | 10015 | 10029 | ||||
| Gentamicin resistance predicted region | 18792 | 18807 | ||||
| Gentamicin resistance predicted region | 40006 | 40020 | ||||
| Gentamicin resistance predicted region | 46557 | 46571 | ||||
| Gentamicin resistance predicted region | 61584 | 61599 | ||||
| Gentamicin resistance predicted region | 104138 | 104152 | ||||
| Clindamycin resistance predicted region | 134690 | 134704 | ||||
| a:BlastP搜索到比对百分比 | ||||||

|
| 不同颜色代表编码不同功能的蛋白质的ORF,红色代表裂解模块,青色代表复制和调节模块,绿色代表代谢模块,蓝色代表结构模块,紫色代表包装模块,假定的ORF和没有噬菌体相关功能的ORF用灰色显示;基因组图谱是使用CLC Genomics Workbench 3.0生成的 图 6 噬菌体vB_SauH_IME522基因图谱和功能注释 |
在DNA复制和调控模块中,ORF168预测为DNA引物酶,ORF176预测为DNA解旋酶,两者的主要功能是参与DNA的修复,重组和复制[27]。ORF158预测为宿主整合因子,其与Staphylococcus phage MCE-2014高度相似,同一性为99 % (100/101)。整合宿主因子(integration host factor,IHF)作为特异性位点重组蛋白质是在大肠杆菌中首次发现的[28]。λ噬菌体的位置特异性重组需要宿主编码的整合宿主因子的参与,IHF由α亚基(himA编码)和β亚基(hip编码)构成,结合于attP位点,在多种过程中起辅助因子的作用,包括复制,位点特异性重组和转录并调节噬菌体和细菌基因的表达[29-30]。噬菌体vB_SauH_IME522编码的ORF145(DNA修复核酸外切酶),ORF170(重组核酸外切酶),ORF172(重组核酸内切酶),这些酶通过水解宿主基因组DNA和RNA来产生用于噬菌体DNA合成的脱氧核糖核苷酸[31]。噬菌体vB_SauH_IME522编码的结构相关蛋白中,与噬菌体尾部相关的蛋白主要为ORF177(尾纤蛋白),ORF184(尾丝蛋白),ORF199(尾管蛋白),ORF200(尾鞘蛋白),它们主要参与了尾部组装及宿主裂解过程[32]。ORF204编码主要衣壳蛋白,与Staphylococcus phage JD007(NC_019726.1)具有高度的序列相似性,同一性为99% (291/292),E值为0。ORF204编码推定的门蛋白与Staphylococcus phage GH15(JQ686190.1)具有100%(563/563)同一性,其在包装过程中是必不可少的[33]。另外,注释结果显示预测出3个与宿主裂解相关的基因,ORF3编码孔蛋白,ORF4内溶素,ORF190编码N-乙酰基胞壁酰-L-丙氨酸酰胺酶。细胞内溶素和孔蛋白是噬菌体裂解宿主必需的两种蛋白[34]。
2.7 噬菌体vB_SauH_IME522的系统发生分析使用噬菌体vB_SauH_IME522中推定的DNA聚合酶(ORF157)及从ICIV数据库获得的其他同源蛋白序列构建了系统发生树。进化树结果都显示噬菌体vB_SauH_IME522与Staphylococcus phage JD007,Staphylococcus phage MCE2014,Staphylococcus phage K,Staphylococcus phage vB_SauM_LM12,Staphylococcus phage HSA30,Staphylococcus phage GH15,Staphylococcus phage VB_SavM_JYL01和Staphylococcus phage SA3最密切相关并且均属于Twortvirinae亚科的Kayvirus属(图 7)。
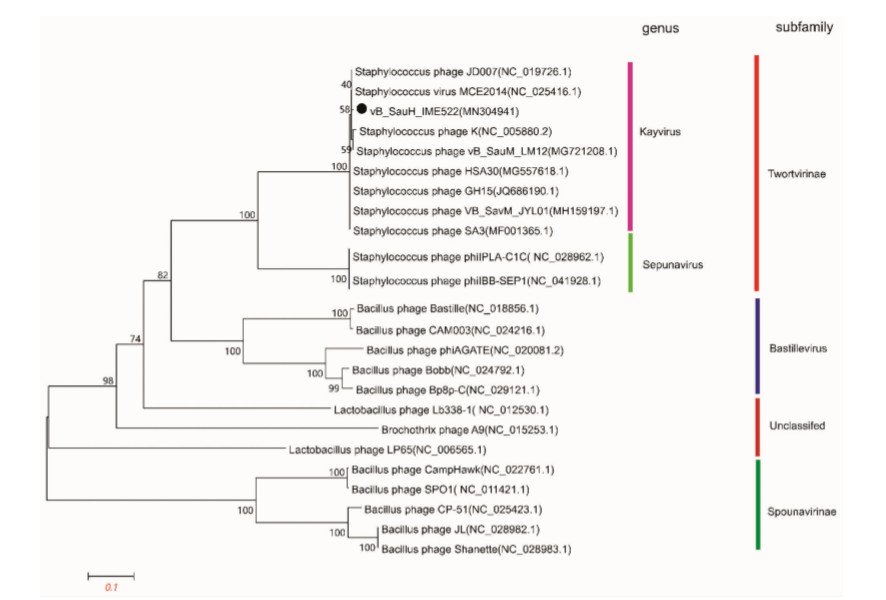
|
| 分支点上的数字:代表可信度, 数值越接近100,可信度越强;标尺:代表遗传距离;黑色圆点:vB_SauH_IME522 图 7 使用噬菌体DNA聚合酶(ORF157)制作的进化树 |
3 讨论
细菌感染是导致患者死亡的主要原因之一[35]。美国传染病学会,欧洲临床微生物学和传染病学会认为多重耐药病原体是人类最大的威胁之一[36-37]。随着细菌对抗生素敏感性的降低,国内外科学家们都在试图寻找抗生素的补充与替代产品。噬菌体作为细菌的病毒[38],在克服细菌耐药领域内潜力巨大,噬菌体疗法有望成为抗生素补充或替代策略[39-40]。
金黄色葡萄球菌是引起医院和社区获得性感染的重要病原体。目前,医师几乎没有其他治疗方法来治疗由甲氧西林引起的感染。耐甲氧西林金黄色葡萄球菌是一个日益严重的问题,对能够有效治疗的人类几种难以治疗的感染构成了重大威胁[41]。与MRSA相关的高死亡率已有报道。MRSA相关感染已成为全世界的严重问题,噬菌体疗法将有助于抵御这种威胁。
在噬菌体vB_SauH_IME522基因组中预测出了7个抗生素相关耐药区域。噬菌体是细菌适应不同环境的一个潜在遗传基因库,是细菌维持自身群落稳定的重要因素。例如,MODI等[42]为了探索抗生素治疗是否会增加噬菌体编码的抗药性基因,他们使用比较宏基因组方法探讨抗生素的作用在噬菌体中编码的扰动功能,以及验证抗生素治疗如何改变噬菌体-细菌生态网络。结果表明,在抗生素选择压力,口服相关浓度的环丙沙星(一种喹诺酮类药物,抑制DNA合成)或氨苄青霉素(抑制细胞壁的β-内酰胺合成)治疗年轻成年小鼠组(n=5),富含由环丙沙星或氨苄青霉素处理的小鼠噬菌体抗生素抗性基因与对照小鼠相比的读数显著增高,此外,在噬菌体中还发现了其他新的抗性基因。噬菌体可以贡献有利于宿主菌的基因[43-44],反过来也促进了自身的生存和传播。这种基因流表明噬菌体可能在微生物组适应环境压力中起重要作用[45-47]。尽管在一些噬菌体DNA上检测到耐药基因的存在,然而,相关研究表明[48],噬菌体基因组上很少携带耐药基因或者所携带的与目前已知的耐药基因虽然具有相似性的片段但没有活性,目前有关噬菌体耐药基因传播的证据还非常有限。噬菌体在耐药基因传播中是否发挥作用还存在争论,还没有一个完全公认的结论。为进一步验证噬菌体vB_SauH_IME522基因组上的耐药基因是否有意义,我们使用Center for Genomic Epidemiology网站(http://genomicepidemiology.org/)未预测到相关的耐药基因存在。但是,7个耐药基因相关预测片段中有两个片段在噬菌体vB_SauH_IME522基因组中是独立存在于ORF之外存在的,我们猜测很有可能是耐药基因的不完全插入导致的。对于这些片段的活性及具体功能机制,还需要更加深入的研究。
总之,本研究分离到一株金黄色葡萄球菌噬菌体,并对其进行了生物学特性研究。此外,通过全基因组测序得到其全长基因组序列,再通过生物信息学分析对其进行注释,并预测了噬菌体裂解相关基因,最后通过系统发生分析讨论了其分类学地位,为以后研究该噬菌体的基因功能及治疗耐药细菌感染奠定了基础[49]。
| [1] |
GRAHAM P L 3rd, LIN S X, LARSON E L. A US population-based survey of Staphylococcus aureus colonization[J]. Ann Intern Med, 2006, 144(5): 318-325. DOI:10.7326/0003-4819-144-5-200603070-00006 |
| [2] |
GORWITZ R J, KRUSZON-MORAN D, MCALLISTER S K, et al. Changes in the prevalence of nasal colonization with Staphylococcus aureus in the United States, 2001-2004[J]. J Infect Dis, 2008, 197(9): 1226-1234. DOI:10.1086/533494 |
| [3] |
KENGNE M, FOTSING O, NDOMGUE T, et al. Antibiotic susceptibility patterns of Staphylococcus aureus strains isolated at the yaounde central hospital, Cameroon: a retro prospective study[J]. Pan Afr Med J, 2019, 32: 103. DOI:10.11604/pamj.2019.32.103.15743 |
| [4] |
NNIS SYSTEM. National nosocomial infections surveillance (NNIS) system report, data summary from January 1990-May 1999, issued June 1999[J]. Am J Infect Control, 1999, 27(6): 520-532. DOI:10.1016/s0196-6553(99)70031-3 |
| [5] |
JULANDER I. Unfavourable prognostic factors in Staphylococcus aureus septicemia and endocarditis[J]. Scand J Infect Dis, 1985, 17(2): 179-187. DOI:10.3109/inf.1985.17.issue-2.09 |
| [6] |
SONG J H, HSUEH P R, CHUNG D R, et al. Spread of methicillin-resistant Staphylococcus aureus between the community and the hospitals in Asian countries: an ANSORP study[J]. J Antimicrob Chemother, 2011, 66(5): 1061-1069. DOI:10.1093/jac/dkr024 |
| [7] |
OTTER J A, FRENCH G L. Community-associated meticillin-resistant Staphylococcus aureus strains as a cause of healthcare-associated infection[J]. J Hosp Infect, 2011, 79(3): 189-193. DOI:10.1016/j.jhin.2011.04.028 |
| [8] |
MARRIS E. Bugs gain vital ground in their battle against drugs[J]. Nat Med, 2005, 11(5): 461. DOI:10.1038/nm0505-461b |
| [9] |
MILLER L G, PERDREAU-REMINGTON F, RIEG G, et al. Necrotizing fasciitis caused by community-associated methicillin-resistant Staphylococcus aureus in los angeles[J]. N Engl J Med, 2005, 352(14): 1445-1453. DOI:10.1056/NEJMoa042683 |
| [10] |
DANTES R, MU Y, BELFLOWER R, et al. National burden of invasive methicillin-resistant Staphylococcus aureus infections, United States, 2011[J]. JAMA Intern Med, 2013, 173(21): 1970-1978. DOI:10.1001/jamainternmed.2013.10423 |
| [11] |
O'FLAHERTY S, ROSS R P, MEANEY W, et al. Potential of the polyvalent anti-Staphylococcus bacteriophage K for control of antibiotic-resistant staphylococci from hospitals[J]. Appl Environ Microbiol, 2005, 71(4): 1836-1842. DOI:10.1128/aem.71.4.1836-1842.2005 |
| [12] |
CAPPARELLI R, NOCERINO N, LANZETTA R, et al. Bacteriophage-resistant Staphylococcus aureus mutant confers broad immunity against staphylococcal infection in mice[J]. PLoS ONE, 2010, 5(7): e11720. DOI:10.1371/journal.pone.0011720 |
| [13] |
GUPTA R, PRASAD Y. Efficacy of polyvalent bacteriophage P-27/HP to control multidrug resistant Staphylococcus aureus associated with human infections[J]. Curr Microbiol, 2011, 62(1): 255-260. DOI:10.1007/s00284-010-9699-x |
| [14] |
RHOADS D D, WOLCOTT R D, KUSKOWSKI M A, et al. Bacteriophage therapy of venous leg ulcers in humans: results of a phase Ⅰ safety trial[J]. J Wound Care, 2009, 18(6): 237-238, 240-243. DOI:10.12968/jowc.2009.18.6.42801 |
| [15] |
GREISEN K, LOEFFELHOLZ M, PUROHIT A, et al. PCR primers and probes for the 16S rRNA gene of most species of pathogenic Bacteria, including Bacteria found in cerebrospinal fluid[J]. J Clin Microbiol, 1994, 32(2): 335-351. DOI:10.1128/JCM.32.2.335-351.1994 |
| [16] |
MI L Y, LIU Y N, WANG C, et al. Identification of a lytic Pseudomonas aeruginosa phage depolymerase and its anti-biofilm effect and bactericidal contribution to serum[J]. Virus Genes, 2019, 55(3): 394-405. DOI:10.1007/s11262-019-01660-4 |
| [17] |
ANDERSON T F. Bacteriophages[J]. Annu Rev Microbiol, 1950, 4(1): 21-34. DOI:10.1146/annurev.mi.04.100150.000321 |
| [18] |
ACKERMANN H W. Basic phage electron microscopy[J]. Methods Mol Biol, 2009, 501: 113-126. DOI:10.1007/978-1-60327-164-6_12 |
| [19] |
XING S Z, MA T P, ZHANG X, et al. First complete genome sequence of a virulent bacteriophage infecting the opportunistic pathogen Serratia rubidaea[J]. Arch Virol, 2017, 162(7): 2021-2028. DOI:10.1007/s00705-017-3300-x |
| [20] |
DELBRVCK M. The growth of bacteriophage and lysis of the host[J]. J Gen Physiol, 1940, 23(5): 643-660. DOI:10.1085/jgp.23.5.643 |
| [21] |
ZHAO F Y, SUN H Z, ZHOU X Y, et al. Characterization and genome analysis of a novel bacteriophage vB_SpuP_Spp16 that infects Salmonella enterica serovar pullorum[J]. Virus Genes, 2019, 55(4): 532-540. DOI:10.1007/s11262-019-01664-0 |
| [22] |
LU S G, LE S, TAN Y L, et al. Genomic and proteomic analyses of the terminally redundant genome of the Pseudomonas aeruginosa phage PaP1: establishment of genus PaP1-like phages[J]. PLoS ONE, 2013, 8(5): e62933. DOI:10.1371/journal.pone.0062933 |
| [23] |
MARGULIES M, EGHOLM M, ALTMAN W E, et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature, 2005, 437(7057): 376-380. DOI:10.1038/nature03959 |
| [24] |
AZIZ R K, BARTELS D, BEST A A, et al. The RAST Server: rapid annotations using subsystems technology[J]. BMC Genomics, 2008, 9: 75. DOI:10.1186/1471-2164-9-75 |
| [25] |
LOWE T M, EDDY S R. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence[J]. Nucleic Acids Res, 1997, 25(5): 955-964. DOI:10.1093/nar/25.5.955 |
| [26] |
TAMURA K, STECHER G, PETERSON D, et al. MEGA6: molecular evolutionary genetics analysis version 6[J]. Mol Biol Evol, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [27] |
GUILLIAM T A, KEEN B A, BRISSETT N C, et al. Primase-polymerases are a functionally diverse superfamily of replication and repair enzymes[J]. Nucleic Acids Res, 2015, 43(14): 6651-6664. DOI:10.1093/nar/gkv625 |
| [28] |
CRAIG N L, NASH H A. E.coli integration host factor binds to specific sites in DNA[J]. Cell, 1984, 39(3 Pt 2): 707-716. DOI:10.1016/0092-8674(84)90478-1 |
| [29] |
GOOSEN N, VAN DE PUTTE P. The regulation oftranscription initiation by integration host factor[J]. Mol Microbiol, 1995, 16(1): 1-7. DOI:10.1111/j.1365-2958.1995.tb02386.x |
| [30] |
KRAWCZYK B, KUR J. Integration host factor, a histone-like Escherichia coli protein, binds to at least four sites of the DNA fragment containing the recA gene[J]. Acta Microbiol Pol, 1994, 43(2): 145-153. |
| [31] |
KROPINSKI A M, WADDELL T, MENG J C, et al. The host-range, genomics and proteomics of Escherichia coli O157: H7 bacteriophage rV5[J]. Virol J, 2013, 10: 76. DOI:10.1186/1743-422X-10-76 |
| [32] |
LOESSNER M J. Bacteriophage endolysins: current state of research and applications[J]. Curr Opin Microbiol, 2005, 8(4): 480-487. DOI:10.1016/j.mib.2005.06.002 |
| [33] |
ISIDRO A, HENRIQUES A O, TAVARES P. The portal protein plays essential roles at different steps of the SPP1 DNA packaging process[J]. Virology, 2004, 322(2): 253-263. DOI:10.1016/j.virol.2004.02.012 |
| [34] |
WANG I N, SMITH D L, YOUNG R. Holins: the protein clocks of bacteriophage infections[J]. Annu Rev Microbiol, 2000, 54: 799-825. DOI:10.1146/annurev.micro.54.1.799 |
| [35] |
HERON M. Deaths: leading causes for 2007[J]. Natl Vital Stat Rep, 2011, 59(8): 1-95. |
| [36] |
LAXMINARAYAN R, DUSE A, WATTAL C, et al. Antibiotic resistance-the need for global solutions[J]. Lancet Infect Dis, 2013, 13(12): 1057-1098. DOI:10.1016/S1473-3099(13)70318-9 |
| [37] |
MANGRAM A J, HORAN T C, PEARSON M L, et al. Guideline for prevention of surgical site infection, 1999[J]. Am J Infect Control, 1999, 27(2): 97-132. DOI:10.1016/S0196-6553(99)70088-X |
| [38] |
HAGENS S, LOESSNER M J. Bacteriophage for biocontrol of foodborne pathogens: calculations and considerations[J]. Curr Pharm Biotechnol, 2010, 11(1): 58-68. DOI:10.2174/138920110790725429 |
| [39] |
DOMINGO-CALAP P, GEORGEL P, BAHRAM S. Back to the future: bacteriophages as promising therapeutic tools[J]. HLA, 2016, 87(3): 133-140. DOI:10.1111/tan.12742 |
| [40] |
GÓRSKI A, MIEDZYBRODZKI R, WEBER-DABROWSKA B, et al. Phage therapy: combating infections with potential for evolving from merely a treatment for complications to targeting diseases[J]. Front Microbiol, 2016, 7: 1515. DOI:10.3389/fmicb.2016.01515 |
| [41] |
NIU H X, YEE R, CUI P, et al. Identification of agents active against methicillin-resistant Staphylococcus aureus USA300 from a clinical compound library[J]. Pathogens, 2017, 6(3): E44. DOI:10.3390/pathogens6030044 |
| [42] |
MODI S R, LEE H H, SPINA C S, et al. Antibiotic treatment expands the resistance reservoir and ecological network of the phage metagenome[J]. Nature, 2013, 499(7457): 219-222. DOI:10.1038/nature12212 |
| [43] |
CHEN J, NOVICK R P. Phage-mediated intergeneric transfer of toxin genes[J]. Science, 2009, 323(5910): 139-141. DOI:10.1126/science.1164783 |
| [44] |
OLIVER K M, DEGNAN P H, HUNTER M S, et al. Bacteriophages encode factors required for protection in a symbiotic mutualism[J]. Science, 2009, 325(5943): 992-994. DOI:10.1126/science.1174463 |
| [45] |
LINDELL D, JAFFE J D, JOHNSON Z I, et al. Photosynthesis genes in marine viruses yield proteins during host infection[J]. Nature, 2005, 438(7064): 86-89. DOI:10.1038/nature04111 |
| [46] |
BEUMER A, ROBINSON J B. A broad-host-range, generalized transducing phage (SN-T) acquires 16S rRNA genes from different genera of Bacteria[J]. Appl Environ Microbiol, 2005, 71(12): 8301-8304. DOI:10.1128/AEM.71.12.8301-8304.2005 |
| [47] |
BALCAZAR J L. Bacteriophages as vehicles for antibiotic resistance genes in the environment[J]. PLoS Pathog, 2014, 10(7): e1004219. DOI:10.1371/journal.ppat.1004219 |
| [48] |
ENAULT F, BRIET A, BOUTEILLE L, et al. Phages rarely encode antibiotic resistance genes: a cautionary tale for virome analyses[J]. ISME J, 2017, 11(1): 237-247. DOI:10.1038/ismej.2016.90 |
| [49] |
MAGANA M, IOANNIDIS A, MAGIORKINIS E, et al. Therapeutic options and emerging alternatives for multidrug resistant staphylococcal infections[J]. Curr Pharm Des, 2015, 21(16): 2058-2072. DOI:10.2174/1381612821666150310101851 |





