2. 400039 重庆,金凤实验室,渝粤病理科学研究中心;
3. 400038 重庆,陆军军医大学(第三军医大学)第一附属医院:输血科;
4. 400038 重庆,陆军军医大学(第三军医大学)第一附属医院:神经外科
2. Yu-Yue Pathology Scientific Research Center, Jinfeng Laboratory, Chongqing, 400039, China;
3. Department of Blood Transfusion, First Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400038;
4. Department of Neurosurgery, First Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400038
胶质母细胞瘤(glioblastoma,GBM)是常见的成人中枢神经系统原发恶性肿瘤,起病隐匿,进展快,易复发,患者中位生存期仅为14~16个月[1-2]。胶质瘤干细胞(glioma stem cells,GSCs)是促进胶质瘤发生和演进并导致肿瘤耐药及复发的关键因素[3-4]。业已证实,与非干性肿瘤细胞(non-stem tumor cells,NSTCs)相比,GSCs高表达CD133和CD15等干性标志物并具有自我更新能力和多向分化潜能[5]。GBM瘤内浸润的肿瘤相关巨噬细胞(tumor-associated macrophages,TAM)多由外周血单核细胞向瘤内募集而产生,其占比为GBM内全部免疫细胞的10%~50%[6]。外周单核细胞募集进入肿瘤后,会被肿瘤微环境中的各类因子诱导发生表型极化,呈现促炎活化(M1)表型或免疫抑制(M2)表型。业已证明,GBM内GSCs与TAM空间分布存在毗邻关系,提示GSCs与TAM可能密切相互作用。本课题组前期研究发现,阻断蛋白酪氨酸磷酸酶受体Z1型(protein tyrosine phosphatase receptor-type Z1,PTPRZ1)通路可显著抑制GSCs干性表型和肿瘤生长,提示PTPRZ1是介导GSCs恶性生物学表型的重要分子[5, 7]。
新近研究发现,GSCs主动塑造GBM免疫抑制性微环境,抑制瘤内浸润免疫细胞的抗肿瘤活性[7]。GBM中大量浸润的TAM多呈现M2表型,而M1表型受抑。M2表型TAM吞噬能力下降,抗原呈递功能减弱,抑制T细胞活化并诱导细胞毒性T细胞耗竭,促进GBM进展。但高表达PTPRZ1的GSCs如何调控TAM向M1或M2型极化及其对TAM吞噬能力的影响和具体机制仍不清楚。鉴于此,本研究拟通过生物信息学分析和细胞功能实验,阐明GSCs对诱导M2型TAM极化和TAM吞噬的影响和调控机制。
1 材料与方法 1.1 细胞、质粒、菌株和GBM组织样本人GBM组织样本来自陆军军医大学第一附属医院生物样本库。所有样本的使用通过患者和监护人知情同意并经过伦理审查委员会批准(KY2022138)。新鲜获得的GBM样本解离后用流式细胞术分选富集获取GSCs(CD15+/CD133+)和NSTCs(CD15-/CD133-)。人新鲜外周血白膜层来源于陆军军医大学第一附属医院血库并经伦理审查委员会批准(KY2022138)。小向导RNA(small guide RNA,sgRNA)原始质粒购自北京全式金公司。
1.2 主要试剂和耗材Ficoll-PaqueTM PLUS淋巴细胞分离液购自英国GE Healthcare公司;人CD14微珠(CD14 MicroBeads)购自德国Miltenyi公司;洛斯维·帕克纪念研究所(Roswell Park Memorial Institute,RPMI) 1640培养基、胎牛血清(fetal bovine serum,FBS)、Neurobasal培养基和B27补充剂购自美国Gibco公司;表皮生长因子和碱性成纤维细胞生长因子购自美国PeproTech公司;木瓜蛋白酶解离试剂盒购自美国Worthington公司;异硫氰酸荧光素(fluorescein isothiocyanate,FITC)缀合的抗人CD15流式抗体购自美国BD Biosciences公司;藻红蛋白(phycoerythrin,PE)缀合的抗人CD133流式抗体购自德国Miltenyi公司;Horizon Brilliant Violet 421TM缀合的抗人CD11b流式抗体、Horizon Brilliant Violet 510TM缀合的抗人CD163流式抗体和Fixable Viability Dye eFluor 780流式抗体购自美国BioLegend公司;抗人CCL20抗体购自英国Abcam公司;抗人趋化因子配体2(C-X-C motif chemokine ligand 2,CXCL2)抗体购自美国Thermo Fisher Scientific公司;抗人β肌动蛋白(β-actin)抗体购自德国Cell Signaling Technology公司;细胞膜红色荧光染色试剂盒(DiL kit)购自上海碧云天公司;Costar超低黏附6孔板和96孔板购自美国Coring公司;FlyCut NotⅠ、FlyCut XbaⅠ seamless cloning试剂盒、TransScript ® Ⅳ One-Step gDNA Removal and cDNA Synthesis SuperMix和TransScript ® Ⅱ Green One-step RT-qPCR SuperMix购自北京全式金生物公司;PCR引物序列设计来源于美国国家生物技术信息中心(national center for biotechnology information,NCBI)网站;引物由北京擎科生物公司合成;人CXCL2重组蛋白和CCL20重组蛋白购自美国MedChemExpress公司。
1.3 方法 1.3.1 TAM诱导分化将新鲜获得人外周血白膜层用蔗糖密度梯度离心,获得外周血单个核细胞(peripheral blood mononuclear cell,PBMC)。用人CD14微珠富集CD14阳性单核细胞,计数后以3×106/孔均匀铺于超低黏附6孔板中,使用含1%双抗、10% FBS的RPMI 1640为基础培养基并加入巨噬细胞集落刺激因子(macrophage colony stimulating factor,M-CSF)(20 ng/mL)诱导分化。分化至第4天换液,将培养基换为胶质瘤细胞培养上清与巨噬细胞培养基按照体积比1 ∶1混合的条件培养基,48 h后收集TAM,用RT-qPCR和流式细胞术检测M2表型TAM相关标记物CD163的表达。经上述方法培养分化的TAM用于后续实验。
1.3.2 GSCs分离和培养用木瓜蛋白酶系统将新鲜人GBM组织标本解离为单个细胞,置于神经干细胞培养基[Neurobasal培养基中加入B27补充剂、表皮生长因子(10 ng/mL)和碱性成纤维细胞生长因子(10 ng/mL)] 中,使用FITC-CD15流式抗体和PE-CD133流式抗体在4 ℃孵育细胞40 min,经BD FACSAria Ⅱ流式细胞仪分离富集获得GSCs(CD15+/CD133+)和NSTCs(CD15-/CD133-),在37 ℃和5% CO2的培养箱里培养并鉴定GSCs干性特征[5]。
1.3.3 稳定敲除PTPRZ1(PTPRZ1-KO)的GSCs细胞株构建利用NCBI数据库中查找人PTPRZ1基因组序列并利用zlab网站(https://www.zlab.bio/guide-design-resources)设计打靶序列(ACTAATGACTACCG-TGTCAG),选择综合评分较高者作为后续合成验证序列。通过酶切连接构建CRISPR/Cas9-PTPRZ1慢病毒载体,并包装为慢病毒。GSCs加入PTPRZ1-sgRNA慢病毒感染细胞48 h,通过流式细胞仪分选PTPRZ1-KO GSCs并检测基因敲除效率。未转染慢病毒的GSCs作为PTPRZ1-Control (PTPRZ1-Ctrl GSCs)。
1.3.4 转录组测序收集PTPRZ1-Ctrl GSCs组和PTPRZ1-KO GSCs组细胞沉淀,使用RNA提取试剂盒提取RNA。使用多聚胸腺嘧啶、T重复寡核苷酸[oligo(dT)]微珠纯化信使RNA(messenger RNA,mRNA),利用离子化方式打断为300 bp左右的片段,反转录合成双链互补DNA(complementary DNA,cDNA),双链末端修复及3′末端加poly(A)尾。使用特定测序接头连接DNA片段两端,高保真聚合酶扩增构建测序文库。基于Illumina测序平台测序,得到fastq数据。
1.3.5 吞噬实验TAM诱导分化至第6天,消化细胞以1×105/孔铺于超低黏附96孔板中培养16 h,将GSCs、NSTCs、PTPRZ1-Ctrl GSCs和PTPRZ1-KO GSCs用DiL kit染色5 min。按照TAM与肿瘤细胞比例为1 ∶1共培养4 h,使用BV421-CD11b流式抗体4 ℃染色30 min,使用BD LSRFortessaTM流式细胞仪检测TAM吞噬比例。
1.3.6 Western blot实验收集PTPRZ1-Ctrl GSCs组与PTPRZ1-KO GSCs组细胞沉淀并提取总蛋白。用BCA蛋白浓度测定试剂盒测定各组蛋白浓度,将浓度调平后加入等量体积样品进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamide gel electrophoresis,SDS-PAGE),后转移至聚偏二氟乙烯(polyvinylidene fluoride, PVDF) 膜上。5%脱脂奶粉封闭膜1 h,加入抗体稀释液配制好的抗人CCL20抗体、CXCL2抗体和β-actin抗体,4 ℃孵育16 h。次日加入辣根过氧化物酶标记二抗,37 ℃孵育1 h后,使用SuperSignalTM West Pico PLUS化学发光底物在ChemiDocTM MP成像系统(美国Bio-Rad公司)显影观察。利用Image J软件定量灰度值,根据内参蛋白β-actin计算CCL20和CXCL2蛋白的相对表达量。
1.3.7 RT-qPCR收集细胞沉淀使用RNA提取试剂盒按步骤提取后测浓度,使用反转录试剂盒按照说明书反转录获得cDNA。按照TransScriptⅡGreen One-Step qRT-PCR SuperMix配置反应体系20 μL,每组设置4个复孔,采用VeritiTM 96孔快速热循环仪(美国Applied Biosystems公司)完成反应,测定基因mRNA的表达水平。以β-actin为内参,用2-ΔΔCt方法计算基因的相对表达量。
| 基因 | 引物序列(5′-3′) |
| CXCL2 | 正向:GGCAGAAAGCTTGTCTCAACCC |
| 反向:CTCCTTCAGGAACAGCCACCAA | |
| CCL20 | 正向:TGCTGTACCAAGAGTTTGCTC |
| 反向:CGCACACAGACAACTTTTTCTTT | |
| ACTB | 正向:CTCCTCCGAGTCAACAGATTCA |
| 反向:CAACAGCTTCTGAGGTAGGGA |
1.3.8 流式细胞术检测TAM中CD163表达情况
将诱导分化至第4天的TAM加入人CXCL2重组蛋白(10 ng/mL)、CCL20重组蛋白(10 ng/mL)或相同体积含5%海藻糖的磷酸盐缓冲液(phosphate buffered saline,PBS)培养72 h。加入BV510-CD163抗体孵育30 min后,利用BD LSRFortessaTM流式细胞仪检测CD163表达水平。
1.3.9 生物信息学分析使用R 4.3.2软件中的limma包对基因表达综合数据库(gene expression omnibus, GEO)数据库中GSE54791数据集分析差异表达基因,筛选阈值参数设置为:P<0.05且|log2 FC|>2。癌症和肿瘤基因图谱(the cancer genome atlas,TCGA)-GBM数据来自在线数据库(https://portal.gdc.cancer.gov),利用R软件sur.cut函数获得最佳cut-off值后,将GBM样本分为靶基因高表达和低表达2组,利用tinyarray和patchwork包批量分析生存时间,筛选获得与GBM患者不良预后相关基因集。利用clusterProfiler包对TCGA数据库中PTPRZ1高表达组和低表达组的差异基因进行GSEA富集分析,筛选阈值参数设置为:P<0.05,获得吞噬功能相关信号通路的差异,利用GseaVis包对基因集富集分析(gene set enrichment Analysis,GSEA)富集结果进行可视化展示。人膜蛋白编码基因集来自在线数据库(https://www.proteinatlas.org)。使用Venn包将三者取交集。
1.4 统计学分析使用GraphPad Prism 8.0软件进行数据统计。使用x±s表示计量资料,使用两独立样本t检验进行两样本间统计学差异分析。若P<0.05则认为差异具有统计学意义。
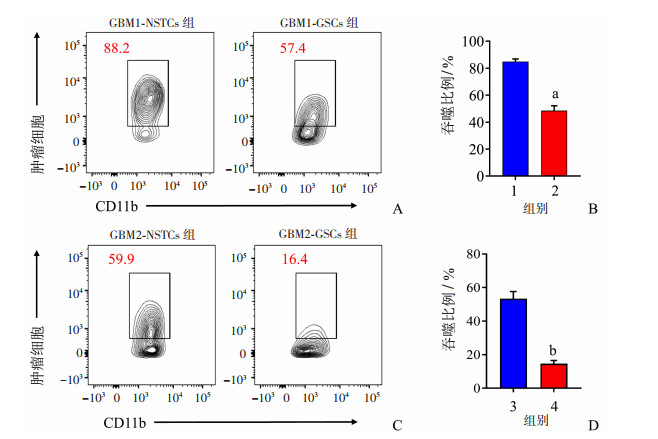
2 结果 2.1 GSCs抵抗TAM吞噬的能力强为比较GSCs和NSTCs抵抗TAM吞噬的差异,分别从2例人GBM样本中分选富集得到GSCs和NSTCs,并检测GSCs和NSTCs抵抗人PBMC来源TAM吞噬的能力。流式细胞术检测结果表明,GBM1和GBM2这2例原代GBM样本中GSCs抵抗TAM吞噬能力显著高于NSTCs组(GBM1:P<0.05,图 1A、B;GBM2:P<0.01,图 1C、D)。以上结果提示,GSCs抵抗TAM吞噬能力显著高于NSTCs。
|
|
1:GBM1-NSTCs组;2:GBM1-GSCs组;3:GBM2-NSTCs组;4:GBM2-GSCs组 A、B:流式细胞术检测TAM吞噬GBM1-GSCs与GBM1-NSTCs的能力与比例统计 a:P<0.05,与GBM1-NSTCs组比较;C、D:流式细胞术检测TAM吞噬GBM2-GSCs与GBM2-NSTCs的能力与比例统计 b:P<0.01,与GBM2-NSTCs组比较 图 1 TAM吞噬GSCs与NSTCs能力检测 (n=3,x±s) |
2.2 GSCs特异性高表达PTPRZ1且抑制吞噬通路
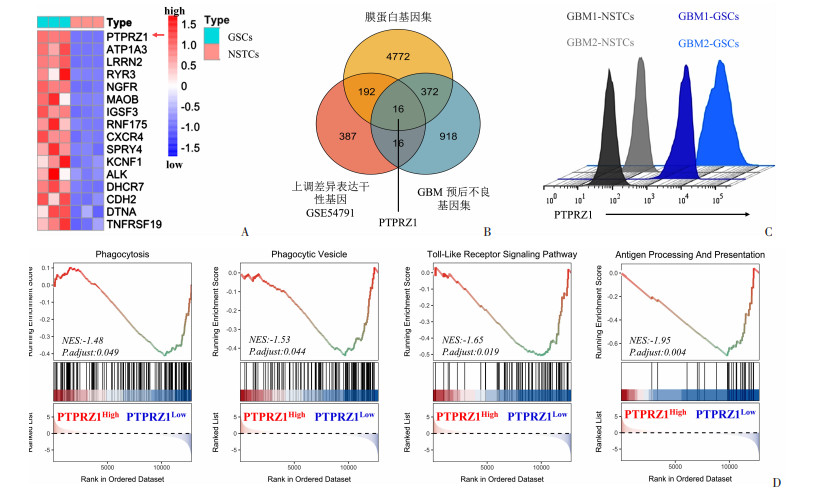
为进一步探究GSCs抵抗TAM吞噬的机制,本研究分析了GSCs与NSTCs的差异表达基因。GSE54791数据集分析结果显示,与NSTCs比较,GSCs高表达611个基因;其中PTPRZ1在GSCs中上调表达最明显(图 2A)。鉴于肿瘤细胞抵抗TAM吞噬提示肿瘤患者预后差且上述行为多与肿瘤细胞表达的膜蛋白有关,本研究将GSCs上调表达基因与TCGA-GBM数据库中预后不良基因集和编码人膜蛋白的基因数据集(https://www.proteinatlas.org/humanproteome/tissue/secretome)取交集,证实膜蛋白PTPRZ1在GSCs高表达提示GBM患者预后不良(图 2B)。流式细胞术检测结果也证实,与NSTCs相比,2例原代GSCs特异性高表达PTPRZ1(图 2C)。GSEA通路富集分析TCGA-GBM数据库结果提示,与PTPRZ1低表达(PTPRZ1Low) 的GBM样本比较,PTPRZ1高表达(PTPRZ1High)的GBM样本中与TAM吞噬功能相关的通路(细胞吞噬、吞噬小泡形成、Toll样受体信号、抗原加工呈递)显著抑制(P<0.05,图 2D)。以上结果提示,PTPRZ1高表达于GSCs且PTPRZ1高表达的GBM样本中吞噬功能相关通路受抑。
|
| A:胶质瘤干细胞与非干性肿瘤细胞差异基因热图;B:胶质瘤干细胞高表达基因集、GBM预后不良基因集与人膜蛋白基因集交集韦恩图;C:流式细胞术检测GBM1-GSCs与GBM1-NSTCs和GBM2-GSCs与GBM2-NSTCs细胞PTPRZ1表达;D:PTPRZ1高表达与PTPRZ1低表达GBM样本GSEA通路富集分析 图 2 GSCs和NSTCs中PTPRZ1表达检测及TCGA-GBM样本吞噬功能相关通路分析 |
2.3 敲除PTPRZ1后GSCs抵抗TAM吞噬能力下降
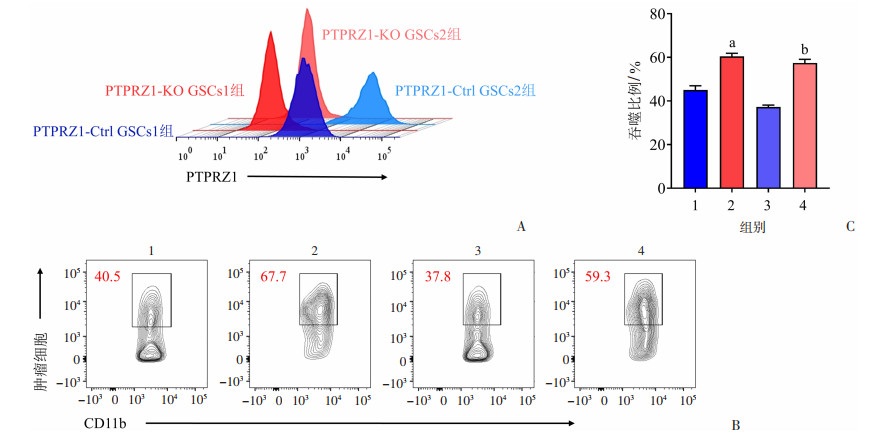
为进一步探究PTPRZ1是否为GSCs抵抗TAM吞噬能力中的关键分子,本研究成功构建了PTPRZ1-KO的GSCs并验证其抵抗TAM吞噬的能力。流式细胞术证实2株原代GSCs中PTPRZ1敲除效果(图 3A)。流式细胞术检测结果显示,与PTPRZ1-Ctrl GSCs1和GSCs2比较,对应PTPRZ1-KO GSCs1和GSCs2组抵抗TAM吞噬能力显著下降(P<0.01,图 3B、C)。以上结果提示,PTPRZ1高表达促进GSCs抵抗TAM吞噬。
|
|
1:PTPRZ1-Ctrl GSCs1组;2:PTPRZ1-KO GSCs1组;3:PTPRZ1-Ctrl GSCs2组;4:PTPRZ1-KO GSCs2组 A:流式细胞术检测PTPRZ1表达;B、C:流式细胞术检测2株PTPRZ1-Ctrl GSCs组与对应2株PTPRZ1-KO GSCs组细胞抵抗TAM吞噬的能力与比例统计 a:P<0.01,与PTPRZ1-Ctrl GSCs1组比较;b:P<0.01,与PTPRZ1-Ctrl GSCs2组比较 图 3 敲除PTPRZ1的GSCs抵抗TAM吞噬能力的检测 (n=3,x±s) |
2.4 PTPRZ1介导TAM向M2型极化,维持GSCs抗吞噬能力
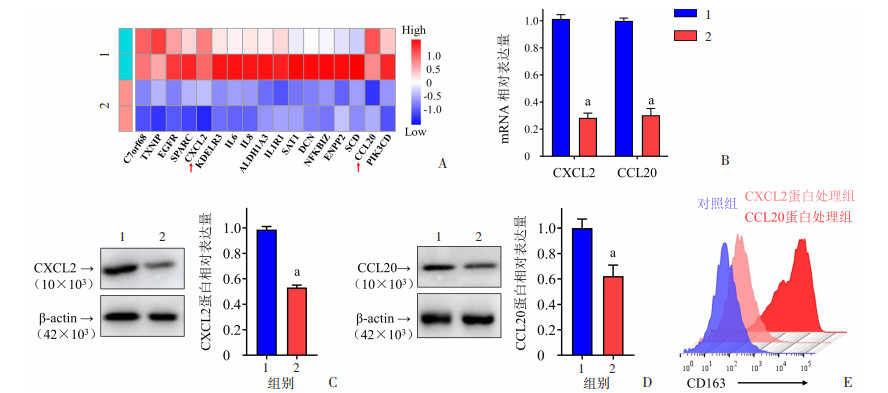
为进一步探索PTPRZ1维持GSCs抵抗TAM吞噬能力的机制,将PTPRZ1-KO GSCs与PTPRZ1-Ctrl GSCs进行转录组测序。数据分析显示:与PTPRZ1-Ctrl GSCs比较,PTPRZ1-KO GSCs下调表达252个基因,依据统计学差异显著性排序展示前17位差异表达基因(图 4A)。为了进一步验证上述结果,选取与M2型TAM表型极化相关的趋化因子CXCL2和CCL20作为候选验证分子进行验证。RT-qPCR结果提示,与PTPRZ1-Ctrl GSCs比较,PTPRZ1-KO GSCs的CXCL2和CCL20的转录水平显著下降(P<0.05,图 4B)。Western blot结果提示:与PTPRZ1-Ctrl GSCs比较,PTPRZ1-KO GSCs中CXCL2和CCL20蛋白表达水平明显下降(P<0.05,图 4C、D)。流式细胞检测结果显示:与对照组比较,CCL20重组蛋白处理TAM,可促进M2型标记物CD163表达水平升高,提示CCL20可促进TAM向M2型极化。然而,CXCL2重组蛋白处理并未观察到CD163表达升高(图 4E)。以上结果提示PTPRZ1高表达GSCs可能通过分泌CCL20诱导TAM向M2型极化,进而抑制TAM吞噬能力。
|
|
a:P<0.05,与PTPRZ1-Ctrl GSCs组比较;1:PTPRZ1-Ctrl GSCs组;2:PTPRZ1-KO GSCs组 A:2组差异基因热图 ↑:示候选分子;B:PCR检测CXCL2和CCL20 mRNA表达;C:Western blot检测CXCL2蛋白表达并定量分析;D:Western blot检测CCL20蛋白表达并定量分析;E:流式细胞术检测CXCL2和CCL20重组蛋白处理TAM后CD163表达 图 4 CCL20和CXCL2表达检测及其对TAM向M2型极化的影响 (n=3, x±s) |
3 讨论
TAM是GBM微环境中浸润数量最多的免疫细胞[9-10]。既往研究提示,GSCs与TAM存在密切相互作用并促进肿瘤发生发展。GSCs及其微环境产生多种细胞因子和趋化因子促进骨髓来源单核细胞向GBM募集,并进一步将单核细胞诱导为M2型TAM。例如,GSCs旁分泌的骨膜素(periostin,POSTN)、Wnt诱导信号蛋白1(Wnt1-inducible signaling pathway protein 1,WISP1)与TAM表面对应的整合素受体结合,募集TAM并诱导其向M2型极化,有效抑制其吞噬活性[11-12]。GSCs还可通过膜蛋白直接与TAM相互作用,诱导TAM表型塑造。此外,GSCs还可通过产生乳酸等代谢产物或释放外泌体,诱导TAM极化为M2型。本研究发现,PTPRZ1高表达GSCs诱导TAM向M2型极化的机制可能与其高表达趋化因子CCL20有关。PTPRZ1-KO GSCs中显著下调CCL20,且经CCL20重组蛋白刺激后的TAM呈现M2型极化。已有研究证实,多种实体肿瘤中的肿瘤干细胞大量分泌CCL20促进肿瘤进展[13]。肿瘤源性CCL20与免疫细胞表面受体C-C基序趋化因子受体6(C-C motif chemokine receptor 6,CCR6)结合促进肿瘤免疫抑制性微环境的形成[14-15]。此外,肿瘤细胞源性CCL20可诱导TAM向M2型极化,进而促进结直肠癌肺转移[16],与本研究发现一致。
鉴于GSCs和M2型TAM相互作用能促进肿瘤生长,阻断GSCs与M2型TAM的相互作用或可抑制肿瘤演进。既往研究发现,靶向阻断C-C基序趋化因子配体2(C-C motif chemokine ligand 2,CCL2)通路能抑制TAM募集[17]。利用特异性靶向CD206表达的纳米脂质体载体可有效杀伤M2型TAM[18]。靶向干预IL-6和IL-10等炎症因子可抑制M2型TAM极化,能抑制肿瘤生长并与化疗和放疗等常规治疗联合,提高抗肿瘤治疗效果。鉴于本研究发现CCL20能促进TAM向M2型极化且靶向CCL20的阻断抗体或抑制剂已被临床前研究证实可有效抑制结直肠癌内免疫抑制性细胞浸润[19],上述CCL20靶向药或可作为抑制胶质瘤内TAM向M2型极化的潜在免疫治疗药物。
GSCs主动逃逸TAM杀伤是肿瘤细胞存活和肿瘤演进的重要机制。既往研究提示,GSCs通过下调信号转导和转录激活因子1(signal transducer and activator of transcription 1,STAT1)的表达,抵抗Ⅰ型干扰素诱导的肿瘤杀伤效应。此外,GSCs高表达CD47等吞噬检查点抑制分子,其与TAM细胞上对应的受体信号调控蛋白α(signal regulatory protein alpha,SIRPα)等结合能抑制TAM吞噬,进而促进肿瘤细胞逃避TAM吞噬杀伤。阻断GSCs与TAM上的吞噬检查点分子可增强TAM吞噬能力[20]。本研究发现高表达PTPRZ1的GSCs抵抗TAM吞噬能力显著高于NSTCs,敲除PTPRZ1的GSCs抵抗TAM的吞噬能力显著下降,提示PTPRZ1或可作为吞噬免疫检查点分子。本课题组前期研发的PTPRZ1单克隆抗体能有效抑制GSCs自我更新和侵袭[6],但其能否用于靶向阻断GSCs抵抗TAM吞噬,效果和机制尚不明确。未来进一步研发靶向PTPRZ1的单克隆抗体或其他针对PTPRZ1的小分子抑制剂,或具有潜在的GSCs靶向免疫治疗价值。
| [1] |
TAN A C, ASHLEY D M, LÓPEZ G Y, et al. Management of glioblastoma: state of the art and future directions[J]. CA Cancer J Clin, 2020, 70(4): 299-312. DOI:10.3322/caac.21613 |
| [2] |
罗静远, 杨静, 李雪, 等. 2-脱氧葡萄糖修饰共载siPD-L1及替莫唑胺脂质纳米粒的脑靶向性研究[J]. 空军军医大学学报, 2024, 45(1): 73-79. LUO J Y, YANG J, LI X, et al. Brain targeting of 2-deoxyglucose modified lipid nanoparticles co-loaded with siPD-L1 and temozolomide[J]. J Air Force Med Univ, 2024, 45(1): 73-79. DOI:10.13276/j.issn.2097-1656.2024.01.015 |
| [3] |
GISINA A, KHOLODENKO I, KIM Y, et al. Glioma stem cells: novel data obtained by single-cell sequencing[J]. Int J Mol Sci, 2022, 23(22): 14224. DOI:10.3390/ijms232214224 |
| [4] |
BOYD N H, TRAN A N, BERNSTOCK J D, et al. Glioma stem cells and their roles within the hypoxic tumor microenvironment[J]. Theranostics, 2021, 11(2): 665-683. DOI:10.7150/thno.41692 |
| [5] |
SHI Y, PING Y F, ZHOU W C, et al. Tumour-associated macrophages secrete pleiotrophin to promote PTPRZ1 signalling in glioblastoma stem cells for tumour growth[J]. Nat Commun, 2017, 8: 15080. DOI:10.1038/ncomms15080 |
| [6] |
YIN W, PING Y F, LI F, et al. A map of the spatial distribution and tumour-associated macrophage states in glioblastoma and grade 4 IDH-mutant astrocytoma[J]. J Pathol, 2022, 258(2): 121-135. DOI:10.1002/path.5984 |
| [7] |
刘庆, 杨莹, 安乐乐, 等. PTPRZ1单克隆抗体对胶质瘤干细胞自我更新和侵袭的抑制作用[J]. 陆军军医大学学报, 2023, 45(12). LIU Q, YANG Y, AN L L, et al. Inhibiting and invasive self-renewal effect of PTPRZ1 monoclonal antibody on glioma stem cells[J]. J Army Medical University, 2023, 45(24): 2530-2536. DOI:10.16016/j.2097-0927.202308042 |
| [8] |
GANGOSO E, SOUTHGATE B, BRADLEY L, et al. Glioblastomas acquire myeloid-affiliated transcriptional programs via epigenetic immunoediting to elicit immune evasion[J]. Cell, 2021, 184(9): 2454-2470.e26. DOI:10.1016/j.cell.2021.03.023 |
| [9] |
GROVER A, SANSEVIERO E, TIMOSENKO E, et al. Myeloid-derived suppressor cells: a propitious road to clinic[J]. Cancer Discov, 2021, 11(11): 2693-2706. DOI:10.1158/2159-8290.CD-21-0764 |
| [10] |
WANG G Q, ZHONG K H, WANG Z, et al. Tumor-associated microglia and macrophages in glioblastoma: from basic insights to therapeutic opportunities[J]. Front Immunol, 2022, 13: 964898. DOI:10.3389/fimmu.2022.964898 |
| [11] |
ZHOU W C, KE S Q, HUANG Z, et al. Periostin secreted by glioblastoma stem cells recruits M2 tumour-associated macrophages and promotes malignant growth[J]. Nat Cell Biol, 2015, 17(2): 170-182. DOI:10.1038/ncb3090 |
| [12] |
TAO W W, CHU C W, ZHOU W C, et al. Dual role of WISP1 in maintaining glioma stem cells and tumor-supportive macrophages in glioblastoma[J]. Nat Commun, 2020, 11(1): 3015. DOI:10.1038/s41467-020-16827-z |
| [13] |
HIPPE A, BRAUN S A, OLÁH P, et al. EGFR/Ras-induced CCL20 production modulates the tumour microenvironment[J]. Br J Cancer, 2020, 123(6): 942-954. DOI:10.1038/s41416-020-0943-2 |
| [14] |
KADOMOTO S, IZUMI K, MIZOKAMI A. The CCL20-CCR6 axis in cancer progression[J]. Int J Mol Sci, 2020, 21(15): 5186. DOI:10.3390/ijms21155186 |
| [15] |
WANG D, YANG L, YU W N, et al. Colorectal cancer cell-derived CCL20 recruits regulatory T cells to promote chemoresistance via FOXO1/CEBPB/NF-κB signaling[J]. J Immunother Cancer, 2019, 7(1): 215. DOI:10.1186/s40425-019-0701-2 |
| [16] |
XU C C, FAN L N, LIN Y F, et al. Fusobacterium nucleatum promotes colorectal cancer metastasis through miR-1322/CCL20 axis and M2 polarization[J]. Gut Microbes, 2021, 13(1): 1980347. DOI:10.1080/19490976.2021.1980347 |
| [17] |
DONG D D, ZHANG G J, YANG J C, et al. The role of iron metabolism in cancer therapy focusing on tumor-associated macrophages[J]. J Cell Physiol, 2019, 234(6): 8028-8039. DOI:10.1002/jcp.27569 |
| [18] |
OVAIS M, GUO M Y, CHEN C Y. Tailoring nanomaterials for targeting tumor-associated macrophages[J]. Adv Mater, 2019, 31(19): e1808303. DOI:10.1002/adma.201808303 |
| [19] |
VAN VLERKEN-YSLA L E, RIOS-DORIA J, MOYNIHAN J, et al. Abstract 4779: targeting the CCL20-CCR6 axis as a novel opportunity to stimulataneously modulate cancer stem cells and the tumor-immune infiltrate by a dual anti-cancer mechanism[J]. Cancer Res, 2017, 77(13_Supplement): 4779. DOI:10.1158/1538-7445.am2017-4779 |
| [20] |
郑文静, 张瀚, 霍毅, 等. 巨噬细胞吞噬检查点阻断在抗肿瘤免疫治疗中的应用[J/OL]. 空军军医大学学报. [2024-03-05]. http://kns.cnki.net/kcms/detail/61.1526.R.20240129.1508.004.html. ZHENG W J, ZHANG H, HUO Y, et al. Application of macrophage phagocytic checkpoint blockade in anti-tumor immunotherapy[J]. J Air Force Med Univ, [2024-03-05]. http://kns.cnki.net/kcms/detail/61.1526.R.20240129.1508.004.html. |




