2. 400038 重庆,陆军军医大学(第三军医大学)第一附属医院全科医学科
2. Department of General Practice, First Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400038, China
最新癌症数据统计结果显示,肺癌患病率在恶性肿瘤中位居第2,病死率位居首位[1]。非小细胞肺癌(non-small cell lung cancer, NSCLC)是肺癌的主要组织学类型,占85%以上[2],具有高转移和高复发的特点,包括4个亚型:肺腺癌(lung adenocarcinoma, LUAD)、肺鳞状细胞癌、大细胞癌和神经内分泌癌,其中肺鳞状细胞癌约占NSCLC的30%[3]。虽然肺癌的治疗方式(包括手术、化疗、放疗、免疫治疗和靶向治疗等)在过去几十年中取得了显著进步[4],但肺鳞状细胞癌预后仍较差,且缺乏有效的靶向疗法[5-6]。缺乏特征性临床表现是肺鳞状细胞癌患者预后较差的原因,所以大部分晚期肺鳞状细胞癌患者错过了根治性手术的最佳时机,导致其5年总生存率仅为19%[7-8]。免疫检查点阻断(immune checkpoint blockade, ICB)疗法是癌症治疗的热点,然而其只对肺鳞状细胞癌的部分患者有效[9-10]。一旦肺鳞状细胞癌患者发生远处转移,手术、放化疗等主要治疗方式的效果就会受限。进一步探究肺鳞状细胞癌的发展机制,寻找可用于开发分子靶点的新型生物标志物至关重要。
碱性核蛋白1(basonuclin 1, BNC1)是特异性的转录因子,它参与调节角质细胞的分化。BNC1主要表达于人类分层上皮的基底角质细胞、人类睾丸、小鼠睾丸和卵巢生殖细胞以及小鼠单细胞胚胎中[11-13]。研究表明,BNC1在头颈部肿瘤中与TP63稳定结合促进上皮分化,在乳腺癌中受TGF-β调控而抑制上皮分化[14-15]。但在肺鳞状细胞癌中,BNC1所发挥的生物学作用尚未明了。因此,本研究通过生物信息学和组织芯片分析BNC1在肺鳞状细胞癌组织中的表达,并构建稳定敲低和稳定过表达的肺鳞状细胞癌细胞模型,并对BNC1促进肺鳞状细胞癌细胞迁移和侵袭的生物学作用进行探索,以期为肺鳞状细胞癌的治疗提供新的方式及靶点。
1 材料与方法 1.1 材料 1.1.1 细胞来源人肺鳞状细胞癌细胞系H226、SK-MES1均购自武汉普诺赛生物公司。
1.1.2 主要试剂RPMI1640培养基和胎牛血清购自美国Gibco公司;RNA提取试剂盒、反转录试剂盒以及PCR试剂均购自日本TaKaRa公司;Transwell小室以及基质胶购自美国康宁公司;小干扰RNA(si-RNA) 以及基因引物购自中国擎科公司;BNC1敲低慢病毒购自中国汉恒病毒公司;BNC1过表达慢病毒购自中国吉凯基因公司;BCA蛋白浓度测定试剂盒、RIPA蛋白裂解液、Western blot封闭液购自上海碧云天公司;兔来源β-actin抗体购自美国CST公司;兔来源的CDH1抗体购自中国华安生物抗体公司;兔来源BNC1抗体购自美国赛默飞世尔科技公司。
1.2 方法 1.2.1 在线数据库分析BNC1表达与临床的相关性利用公共数据库发现BNC1的mRNA表达水平与肺鳞状细胞癌的临床分期和生存率相关。首先通过癌症和正常组织基因表达剖面分析互动平台(Gene Expression Profiling Interactive Analysis,GEPIA)数据库(http://gepia.cancer-pku.cn/)发现肺鳞状细胞癌中肿瘤组织和癌旁组织的mRNA水平差异具有统计学意义,随后调查BNC1的mRNA水平与肺鳞状细胞癌肿瘤分期的关联,最后通过Kaplan-Meier Plotter(KMplot) 数据库(http://www.kmplot.com/)发现BNC1的表达与肺鳞状细胞癌患者的生存率密切相关。
1.2.2 免疫组织化学染色采用的组织芯片购自上海芯超生物科技有限公司(序列号: HLugS020PG02),包含肺鳞状细胞癌10例,癌组织及癌旁组织各1例,病理分级以Ⅱ级为主。在研究前已获得所有患者的书面知情同意书,本研究获得上海芯超生物科技有限公司伦理委员会的批准(SHYJS-CP-1910012)。组织切片用二甲苯脱蜡,梯度乙醇水化后,在100 ℃的柠檬酸盐缓冲液(pH=6)中孵育20 min以进行抗原修复,在3% H2O2中孵育25 min以淬灭内源性过氧化物酶,用正常山羊血清阻断10 min,然后与BNC1一抗(1 ∶150)在4 ℃孵育12 h,用PBS冲洗,室温下孵育2 h,然后用DAB辣根过氧化物酶显色试剂盒显色。用苏木精进行反染色后,载玻片用中性树胶装片。最后由2位独立的病理科医师,进行双盲阅片,评估染色结果。癌组织为实验组,癌旁组织为对照组。
1.2.3 细胞培养以及转染人肺鳞状细胞癌细胞H226培养于10%胎牛血清的RPMI1640培养基,人肺鳞状细胞癌细胞SK-MES1培养于10%胎牛血清的MEM培养基,将细胞放置于培养箱(37 ℃,5%CO2) 中培养。将呈对数生长期的H226细胞以3×105个/孔接种于6孔板中,第2天待细胞融合生长达50%~60%时进行转染,转染过程按照锐博转染试剂说明书进行,并同时转染阴性对照(si-NC),每组设3个复孔,转染浓度为50 nmol/L。将培养板置于培养箱中培养48 h,PBS洗3次后进行RNA提取验证敲低效率。H226的细胞实验中,si-BNC1为实验组,si-NC为对照组。
1.2.4 稳定细胞株的构建感染前按(0.5~1.0)×105/mL将生长状态良好的H226和SK-MES1细胞接种于T25瓶中,培养基体积为5 mL,37 ℃、5%CO2培养箱中培养24 h。根据测定的H226感染复数(multiplicity of infection, MOI)(10)和SK-MES1 MOI(50)对细胞进行慢病毒感染。12 h后将含病毒的培养基更换为含10% FBS的RPMI1640和MEM完全培养基。72 h后倒置荧光显微镜观察到绿色荧光蛋白(green fluorescent protein, GFP)的表达后,分别换为含嘌呤霉素(2 μg/mL)的RPMI1640和MEM完全培养基来筛选抗性细胞。而后每隔1 d更换为新鲜的含嘌呤霉素(2 μg/mL)的培养基以防止代谢产物影响细胞生长状态。当倒置荧光显微镜下观察到现存细胞中都有明亮的绿色荧光时,可将含嘌呤霉素的培养基换为完全培养基。待细胞长满后,将细胞转移至10 cm培养皿中培养,及时消化传代并收集细胞,以备后续实验所需。H226细胞的慢病毒感染实验中,sh-BNC1为实验组,sh-NC为对照组;SK-MES1的细胞实验中,BNC1-OE为实验组,Vector为对照组。
1.2.5 转录和RT-qPCR检测根据试剂盒说明书提取肺鳞状细胞癌细胞SK-MES1、H226的总RNA,并检测其浓度。使用TaKaRa反转录试剂盒将RNA反转录为cDNA,条件为37 ℃ 15 min,85 ℃ 5 s,然后使用TaKaRa的PCR试剂盒对cDNA进行qPCR实验,反应条件为:95 ℃ 30 s,95 ℃ 5 s,60 ℃ 30 s, 72 ℃ 30 s,×40个循环。引物序列见表 1,样品以GAPDH为内参,设置3个复孔,BNC1的相对表达量采用2-△△Ct法表示。
| 基因 | 引物序列(5′→3′) | 产物长度/bp |
| BNC1 | 上游:AATGCACACATGCACAGTGG | 154 |
| 下游:CAGAAGGTAAGCTGCACGGA | ||
| CDH1 | 上游:TAGAGAACGCATTGCCACATACA | 124 |
| 下游:GAATTCGGGCTTGTTGTCATTCT | ||
| CDH2 | 上游:TATGGATGAAACGCCGGGATAAA | 134 |
| 下游:GCTCAAGTCATAGTCCTGGTCTT | ||
| β-Catenin | 上游:CATCCCACTGGCCTCTGATAAAG | 127 |
| 下游:GCACGAACAAGCAACTGAACTAG | ||
| FN1 | 上游:GAGGGCAGAAGAGACAACATGAA | 117 |
| 下游:CCCTTCATTGGTTGTGCAGATTT | ||
| VIM | 上游:GGGAGAAATTGCAGGAGGAGATG | 209 |
| 下游:TTGGACATGCTGTTCCTGAATCT | ||
| CDC42 | 上游:AGATTACGACCGCTGAGTTATCC | 228 |
| 下游:AGTCTCTGGAGTGATAGGCTTCT | ||
| RAC1 | 上游:CCTTGTGAGTCCTGCATCATTTG | 158 |
| 下游:GAGTCAGCTTCTTCTCCTTCAGT | ||
| MMP-2 | 上游:ACCAGAATACCATCGAGACCATG | 137 |
| 下游:CAGATCAGGTGTGTAGCCAATGA | ||
| MMP-7 | 上游:TGCAGTGATGTATCCAACCTATG | 217 |
| 下游:TTGCTAAATGGAGTGGAGGAACA | ||
| RHOA | 上游:CTTCGGAATGATGAGCACACAAG | 96 |
| 下游:GCCAATCCTGTTTGCCATATCTC | ||
| GAPDH | 上游:GCACCGTCAAGGCTGAGAAC | 138 |
| 下游:TGGTGAAGACGCCAGTGGA |
1.2.6 划痕愈合实验检测细胞迁移
将si-BNC1的H226细胞和BNC1-OE的SK-MES1细胞以及各自阴性对照组细胞按4×104/孔接种到6孔板中,待生长至90%以上,用10 μL的无菌吸头在细胞单层上划出划痕。PBS洗涤2次后,加入含1%的血清培养基,在显微镜下拍照并记录0 h划痕距离。将细胞放回培养箱培养12~24 h,再次用显微镜拍照并记录各组细胞的划痕距离,根据划痕差值计算各组细胞的相对迁移率。
1.2.7 Transwell小室检测细胞迁移以及侵袭Transwell侵袭实验:将si-BNC1的H226细胞和BNC1-OE的SK-MES1细胞以及各自阴性对照组细胞于无血清培养基中用手动计数板对细胞进行计数并计算浓度,最后稀释成5×105/mL和7×105/mL,然后吸取其中的100 μL置于预先涂有Matrigel(美国康宁公司)的Transwell插入物(美国康宁公司)的上腔中。下腔注入补充了20%胎牛血清作为趋化吸引剂的培养基。37 ℃孵育24 h后,去除膜上表面的细胞,用含4%的多聚甲醛固定膜下表面的细胞,并用1%结晶紫染色。在显微镜下每孔随机取5个视野对入侵细胞进行成像和计数。
Transwell迁移实验:将si-BNC1的H226细胞和BNC1-OE的SK-MES1细胞以及各自阴性对照组细胞于无血清培养基中用手动计数板对细胞进行计数并计算浓度,最后稀释成5×105/mL和7×105/mL,然后吸取其中的100 μL置于Transwell插入物(美国康宁公司)的上腔中。下腔注入补充了20%胎牛血清作为趋化吸引剂的培养基。37 ℃孵育12 h后,去除膜上表面的细胞,用含4%的多聚甲醛固定膜下表面的细胞,并用1%结晶紫染色。在显微镜下每孔随机取5个视野对迁移细胞进行成像和计数。
1.2.8 Western blot首先使用RIPA裂解缓冲液(含1%蛋白酶和磷酸酶抑制剂)裂解各组细胞15 min,然后利用超声破碎仪将蛋白破碎。4 ℃、14 000×g离心15 min,收集上清液中的蛋白质样本,并用BCA法进行蛋白质定量。并加入5×的Loading Buffer在100 ℃下10 min让蛋白质变性。然后每个样本取20 μg,4%~20%的SDS-PAGE凝胶将样本分离等量蛋白质,转移到PVDF膜上。用5%脱脂牛奶阻断,并与特异性一抗在4 ℃下孵育过夜。第2天用TBST(100 mmol/L NaCl,10 mmol/L Tris-HCl,pH 7.5,0.1% Tween-20)冲洗3遍后,在室温下用与辣根过氧化物酶结合的第二抗体孵育膜1 h,利用化学发光法显影观察蛋白质。
1.3 统计学分析实验均独立重复3次。采用GraphPad 8.0进行统计分析和绘图。符合正态分布的2组间数据比较采用独立样本t检验,多组间数据比较采用单因素方差分析,以P < 0.05表示差异具有统计学意义。
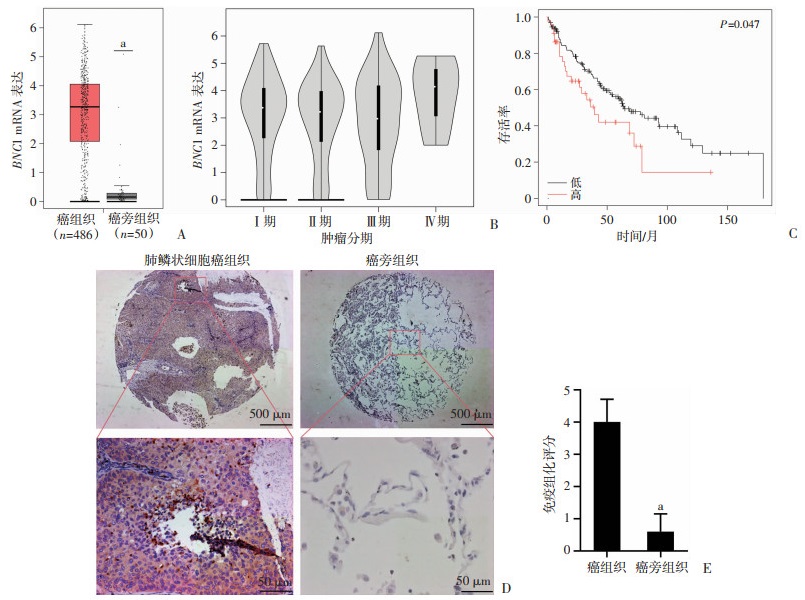
2 结果 2.1 BNC1与肺鳞状细胞癌的临床相关性GEPIA网站分析结果显示:BNC1的mRNA在肺鳞状细胞癌组织中的表达高于癌旁组织(图 1A)。进一步分析显示,BNC1 mRNA高表达的晚期肺鳞状细胞癌患者生存率较低(P=0.047,图 1B、C)。提示BNC1可能在肺鳞状细胞癌发挥促癌功能。采用肺鳞状细胞癌组织芯片进行免疫组化染色,结果显示:BNC1蛋白表达于胞核和胞质,阳性表达在癌组织区域(图 1D)。癌组织平均病理评分为4分,显著高于癌旁组织(P < 0.05,图 1E)。
|
|
a:P < 0.05,与癌组织比较 A:GEPIA网站中肺鳞状细胞癌组织和癌旁组织中BNC1的mRNA表达;B:GEPIA数据库中不同临床分期肺鳞状细胞癌患者的BNC1 mRNA表达;C:KMplot网站中BNC1 mRNA表达高低对肺鳞状细胞癌患者生存率的影响;D:BNC1蛋白在肺鳞状细胞癌组织和癌旁组织中的表达;E: 肺鳞状细胞癌组织和癌旁组织中BNC1蛋白的免疫组化评分 图 1 BNC1在肺鳞状细胞癌组织中的表达及对患者生存率的影响 |
2.2 BNC1敲低与BNC1过表达稳定细胞株的构建
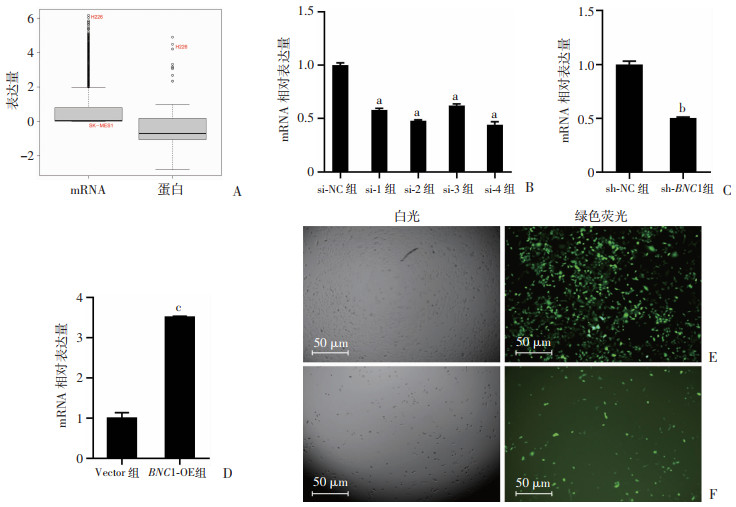
H226和SK-MES1均来源于男性肺鳞状细胞癌患者,由转移性胸腔积液分离而来。通过对癌症细胞系百科全书(Cancer Cell Line Encyclopedia,CCLE)数据库(https://depmap.org/)的探索,发现H226细胞的BNC1表达较SK-MES1细胞高(图 2A)。为了研究BNC1基因对肺鳞状细胞癌细胞生物学功能的影响,合成并设计针对BNC1基因的小干扰RNA。随后对合成的4条si-RNA进行H226细胞的干扰实验,通过RT-qPCR对干扰效率进行验证。结果发现,4条si-RNA都敲低H226细胞中的BNC1基因,其中si-4的干扰效率最佳(图 2B)。因此选择这条干扰序列和阴性干扰序列进行慢病毒包装并感染H226细胞(MOI=10),构建BNC1稳定敲低(sh-BNC1组)和阴性对照(sh-NC组)的H226细胞(图 2C、E);同时构建BNC1稳定过表达(BNC1-OE组)和阴性对照(Vector组)的SK-MES1的细胞(MOI=50),以进行后续相关实验(图 2D、F)。
|
|
a:P < 0.05,与si-NC组比较;b:P < 0.05,与sh-NC组比较;c:P < 0.05,与Vector组比较 A:CELL数据库中H226细胞与SK-MES1细胞的BNC1表达情况;B:4种si-RNA对H226细胞中BNC1的干扰效率验证;C:BNC1稳定敲低的H226细胞;D:BNC1稳定过表达的SK-MES1细胞;E:H226细胞的MOI;F:SK-MES1细胞的MOI 图 2 BNC1敲低与BNC1过表达稳定细胞株的构建 |
2.3 BNC1表达水平对肺鳞状细胞癌细胞迁移和侵袭能力的影响
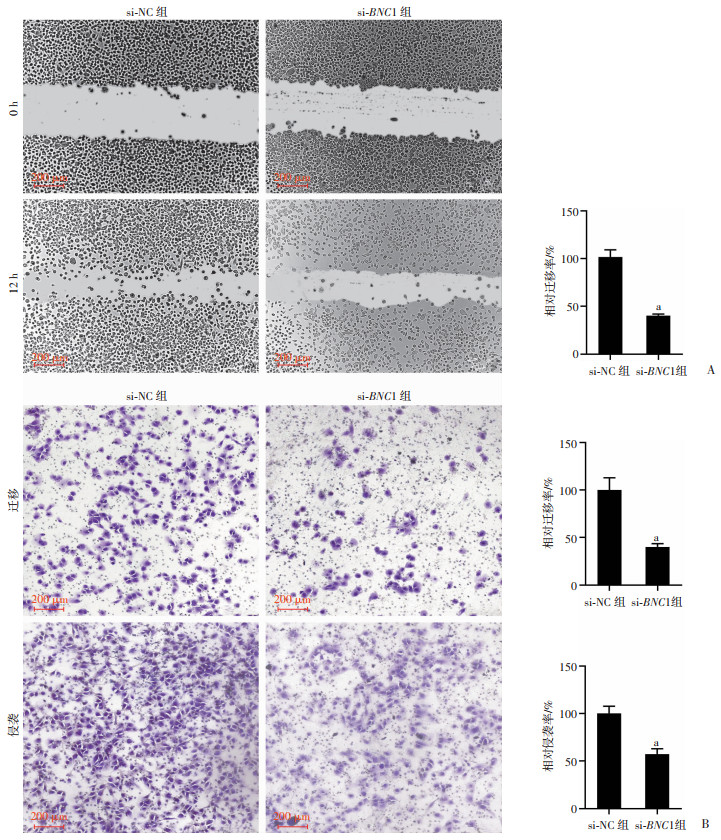
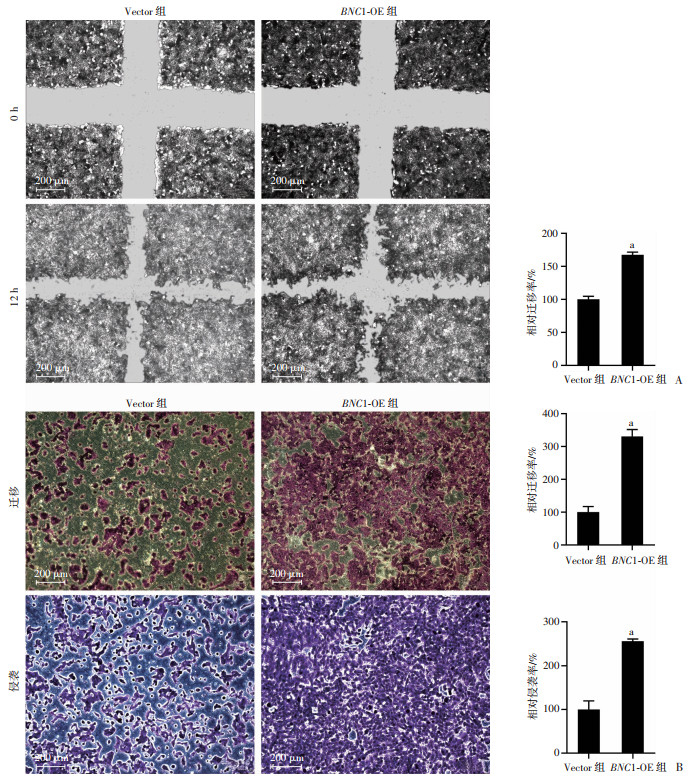
对si-BNC1和BNC1-OE的肺鳞状细胞癌细胞进行迁移和侵袭能力的检测。划痕愈合实验结果显示,si-BNC1组H226细胞迁移率明显低于对照组(P < 0.05,图 3A),提示BNC1敲低能够抑制肺鳞状细胞癌细胞的迁移能力;BNC1-OE组SK-MES1细胞的迁移率明显高于对照组(P < 0.05,图 4A),提示BNC1过表达能够增强肺鳞状细胞癌细胞的迁移能力。Transwell迁移与侵袭实验结果显示,在添加和不添加基质胶的小室中,si-BNC1组H226细胞穿过小室膜的数量均明显低于对照组(P < 0.05,图 3B),表明BNC1敲低能够抑制肺鳞状细胞癌细胞的迁移和侵袭;BNC1-OE组SK-MES1细胞穿过小室膜的数量均明显高于对照组(P < 0.05,图 4B)。表明BNC1过表达能够增强肺鳞状细胞癌细胞的迁移和侵袭。
|
|
a:P < 0.05,与si-NC组比较 A:划痕愈合实验检测H226细胞的迁移能力(左)及定量分析(右);B:Transwell实验检测H226细胞的迁移和侵袭能力(左)及定量分析(右) 图 3 BNC1敲低对肺鳞状细胞癌细胞迁移和侵袭能力的影响 |
|
|
a:P < 0.05,与Vector组比较 A:划痕愈合实验检测SK-MES1细胞的迁移能力(左)及定量分析(右);B:Transwell实验检测SK-MES1细胞的迁移和侵袭能力(左)及定量分析(右) 图 4 BNC1过表达对肺鳞状细胞癌细胞迁移和侵袭能力的影响 |
2.4 BNC1促进肺鳞状细胞癌细胞迁移和侵袭的下游分子机制
通过查阅文献筛选了10个跟迁移和侵袭现象密切相关的分子,并设计这10个分子的相关引物,提取si-BNC1和BNC1-OE以及各自阴性对照的肺鳞状细胞癌细胞株的RNA,通过RT-qPCR对10个基因进行了验证(图 5)。
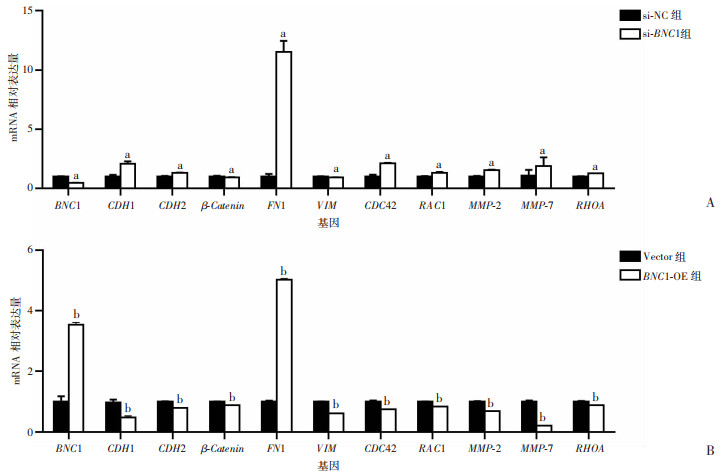
|
|
a:P < 0.05,与si-NC组比较;b: P < 0.05, 与Vector组比较 A:H226细胞BNC1敲低后检测mRNA水平的相对变化;B:SK-MES1细胞BNC1过表达后检测mRNA水平的相对变化 图 5 BNC1促进肺鳞状细胞癌细胞迁移和侵袭的下游分子机制 |
2.5 BNC1通过调控CDH1基因的表达影响肺鳞状细胞癌细胞的迁移和侵袭
通过BNC1过表达以及BNC1敲低细胞的mRNA水平交叉验证,只发现CDH1基因的变化符合细胞迁移与侵袭的规律,它所编码的蛋白在mRNA层面随着BNC1的敲低而升高,随BNC1的过表达而减低(P < 0.05,图 6A、B)。随后,提取sh-BNC1和sh-NC的H226细胞以及BNC1-OE和Vector的SK-MES1细胞,进行蛋白层面的验证,结果发现CDH1表达在蛋白层面随着BNC1的过表达而减低,随着BNC1的敲低而升高,与mRNA层面一致(P < 0.05,图 6C~F)。提示BNC1基因在肺鳞状细胞癌细胞中可以通过下调CDH1的表达影响迁移和侵袭能力。
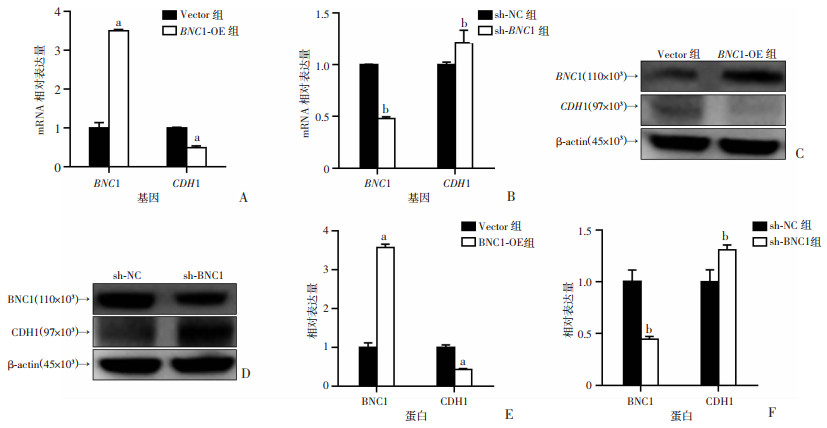
|
|
a:P < 0.05,与Vector组比较b:P < 0.05,与sh-NC组比较 A:在SK-MES1细胞中过表达BNC1后CDH1 mRNA水平的变化;B:在H226细胞中敲低BNC1后CDH1 mRNA水平的变化;C:在SK-MES1细胞中过表达BNC1后CDH1蛋白的表达变化;D:在H226细胞中敲低BNC1后CDH1蛋白的表达变化;E:SK-MES1细胞的Western半定量分析F:H226细胞的Western半定量分析 图 6 BNC1敲低和过表达对CDH1表达的影响 |
3 讨论
BNC1是一种锌指蛋白特异性转录因子,位于15号染色体上[16]。其首次在体外培养的人类表皮角质细胞中被发现,并可能在生殖细胞中发挥重要作用。随后更多的研究发现BNC1与多种肿瘤的发生密切相关,如头颈部鳞状细胞癌、乳腺癌、基底细胞癌等[17-18]。提示BNC1是1个致癌基因,但BNC1是否调控鳞癌细胞迁移与侵袭尚不清楚。
3.1 BNC1在肺鳞状细胞癌组织中高表达本研究首先通过GEPIA在线数据库分析发现:与癌旁组织比较,BNC1基因在肺鳞状细胞癌组织中高表达,且肺鳞状细胞癌晚期患者BNC1基因较早期阶段的肺鳞状细胞癌患者表达有升高的趋势,然后在KMplot数据库探索发现BNC1的表达与肺鳞状细胞癌患者的生存率呈负相关,最后肺鳞状细胞癌组织芯片免疫组化实验结果证实BNC1高表达于癌组织而低表达于癌旁组织。这些结果提示在肺鳞状细胞癌患者中,BNC1可能促进肺鳞状细胞癌的发生发展。
3.2 BNC1促进肺鳞状细胞癌细胞迁移与侵袭为探索BNC1对肺鳞状细胞癌发生发展的生物学作用,首先利用CCLE数据库筛选出BNC1低表达和高表达的肺鳞状细胞癌细胞系,对高表达BNC1的肺鳞状细胞癌细胞进行小干扰RNA的敲低筛选,构建稳定敲低的细胞株;同时用慢病毒载体构建BNC1过表达的细胞株。随后进行细胞的生物学功能验证,结果发现,BNC1敲低的肺鳞状细胞癌细胞迁移和侵袭能力减低,而BNC1过表达的肺鳞状细胞癌细胞迁移和侵袭能力增强,提示BNC1可能是促进肺鳞状细胞癌迁移和侵袭能力的癌基因。
3.3 BNC1下游分子机制的初步探索为探索BNC1可能作用的下游分子,分别提取si-BNC1和BNC1-OE肺鳞状细胞癌细胞的RNA进行RT-qPCR实验,查阅文献发现癌细胞主要通过PI3K/Akt、MAPK/Erk和Rho GTP酶信号通路进行侵袭及迁移。根据文献筛选10个信号分子进行验证,其中CDH1所编码的E-钙粘蛋白是一种细胞间黏附分子,存在于上皮细胞的黏附连接处,参与调节上皮细胞的细胞-细胞黏附、流动性和增殖机制,具有强大的侵袭抑制和限制细胞运动的作用[19-21]。CDH1基因突变及其相关蛋白E-Cadherin缺失会导致上皮-间质转化过程,即细胞失去细胞间黏附能力和顶端极性,最终促进肿瘤的转移[22]。另外CDH1基因可通过多种信号通路(如Wnt信号通路、EGFR/RAS/RAF/MEK信号通路、P-120/Rho/MAPK信号通路等)促进肿瘤的侵袭和转移[23]。CDH2/FN1/CDC42/RAC1/MMP-2/MMP-7是PI3K/Akt和MAPK/Erk信号通路中的重要信号分子,而β-Catenin是Wnt信号通路的关键分子,VIM/RAC1/RHOA/CDC42则是Rho GTP酶信号通路中的重要分子,所以选取这10个分子作为调控基因[24-28]。通过过表达和敲低BNC1基因,结果发现,CDH1基因表达符合生物学作用规律;虽然FN1基因的变化最为明显,但其表达水平都是升高的,不符合BNC1对FN1调控作用的经典规律,因此未深入探索;其他基因的表达不符合细胞迁移与侵袭的规律,也予以排除。对sh-BNC1以及BNC1-OE的肺鳞状细胞癌细胞进行蛋白层面的验证结果显示,CDH1基因随着BNC1的敲低而升高,BNC1的过表达而降低,与mRNA水平一致。所以,BNC1很可能通过下调CDH1基因的表达,从而导致肺鳞状细胞癌细胞之间的黏附减低,细胞的运动增加,最终促进肺鳞状细胞癌的迁移和侵袭。较少研究报道BNC1基因在肺鳞状细胞癌细胞中有调控细胞迁移和侵袭的功能,本研究报告了BNC1对CDH1的调控作用,但是否通过结合启动子位点,还是其他信号通路有待后续深入研究。
综上所述,本研究结果显示BNC1在肺鳞状细胞癌患者中发挥着重要的生物学作用,BNC1高表达于肺鳞状细胞癌组织而低表达于癌旁组织,且BNC1能够促进肺鳞状细胞癌细胞迁移和侵袭,下游分子筛选发现BNC1通过下调CDH1的表达影响肺鳞状细胞癌细胞的迁移和侵袭。本研究为肺鳞状细胞癌的诊断和治疗提供了新的分子标志物,后续研究将继续挖掘BNC1下调CDH1基因的具体机制以及BNC1在肺鳞状细胞癌中发挥的其他生物学功能及其机制,为肺鳞状细胞癌的诊治以及预后提供新的方案。
| [1] |
SIEGEL R L, MILLER K D, WAGLE N S, et al. Cancer statistics, 2023[J]. CA Cancer J Clin, 2023, 73: 17-48. |
| [2] |
PAN L, MO R, ZHU L H, et al. Time trend of mediastinal lymph node dissection in stage IA non-small cell lung cancer patient who undergo lobectomy: a retrospective study of surveillance, epidemiology, and end results (SEER) database[J]. J Cardiothorac Surg, 2020, 15(1): 207. |
| [3] |
XU F, LIN H, HE P, et al. A TP53-associated gene signature for prediction of prognosis and therapeutic responses in lung squamous cell carcinoma[J]. Oncoimmunology, 2020, 9(1): 1731943. |
| [4] |
THAI A A, SOLOMON B J, SEQUIST L V, et al. Lung cancer[J]. Lancet, 2021, 398(10299): 535-554. |
| [5] |
NING J, WANG F, ZHU K, et al. Characterizing the copy number variation of non-coding RNAs reveals potential therapeutic targets and prognostic markers of LUSC[J]. Front Genet, 2021, 12: 779155. |
| [6] |
HIRSCH F R, SCAGLIOTTI G V, MULSHINE J L, et al. Lung cancer: current therapies and new targeted treatments[J]. Lancet, 2017, 389(10066): 299-311. |
| [7] |
ZHAO J G, BAO W M, CAI W Y. Immune infiltration landscape in lung squamous cell carcinoma implications[J]. BioMed Res Int, 2020, 2020: 5981870. |
| [8] |
HERBST R S, MORGENSZTERN D, BOSHOFF C. The biology and management of non-small cell lung cancer[J]. Nature, 2018, 553(7689): 446-454. |
| [9] |
YUAN H, LIU J, ZHANG J. The current landscape of immune checkpoint blockade in metastatic lung squamous cell carcinoma[J]. Molecules, 2021, 26(5): 1392. |
| [10] |
CUI L, LI H, HUI W, et al. A deep learning-based framework for lung cancer survival analysis with biomarker interpretation[J]. BMC Bioinformatics, 2020, 21(1): 112. |
| [11] |
TEUMER J, TSENG H, GREEN H. The human basonuclin gene[J]. Gene, 1997, 188(1): 1-7. |
| [12] |
TSENG H, GREEN H. Association of basonuclin with ability of keratinocytes to multiply and with absence of terminal differentiation[J]. J Cell Biol, 1994, 126(2): 495-506. |
| [13] |
MAHONEY M G, TANG W, XIANG M M, et al. Translocation of the zinc finger protein basonuclin from the mouse germ cell nucleus to the midpiece of the spermatozoon during spermiogenesis[J]. Biol Reprod, 1998, 59(2): 388-394. |
| [14] |
GAO Y B, LI J Y, JIAO J, et al. Upregulation of Basonuclin1 is associated with p63-involved epithelial barrier impairment and type-2 helper T-cell inflammation in chronic rhinosinusitis with nasal polyps[J]. Int Arch Allergy Immunol, 2021, 182(11): 1046-1057. |
| [15] |
FEUERBORN A, MATHOW D, SRIVASTAVA P K, et al. Basonuclin-1 modulates epithelial plasticity and TGF-β1-induced loss of epithelial cell integrity[J]. Oncogene, 2015, 34(9): 1185-1195. |
| [16] |
ATABIEKOV I, HOBEIKA E, SHEIKH U, et al. The role of gene therapy in premature ovarian insufficiency management[J]. Biomedicines, 2018, 6(4): 102. |
| [17] |
GARCÍA-DÍEZ I, HERNÁNDEZ-MUÑOZ I, HERNÁNDEZ-RUIZ E, et al. Transcriptome and cytogenetic profiling analysis of matched in situ/invasive cutaneous squamous cell carcinomas from immunocompetent patients[J]. Genes Chromosom Cancer, 2019, 58(3): 164-174. |
| [18] |
GHEORGHE G, BUNGAU S, ILIE M, et al. Early diagnosis of pancreatic cancer: the key for survival[J]. Diagnostics, 2020, 10(11): 869. |
| [19] |
BISWAS K H. Molecular mobility-mediated regulation of E-cadherin adhesion[J]. Trends Biochem Sci, 2020, 45(2): 163-173. |
| [20] |
BÜCKER L, LEHMANN U. CDH1 (E-cadherin) gene methylation in human breast cancer: critical appraisal of a long and twisted story[J]. Cancers, 2022, 14(18): 4377. |
| [21] |
RÄTZE M A K, KOORMAN T, SIJNESAEL T, et al. Loss of E-cadherin leads to Id2-dependent inhibition of cell cycle progression in metastatic lobular breast cancer[J]. Oncogene, 2022, 41(21): 2932-2944. |
| [22] |
PETROVA Y I, SCHECTERSON L, GUMBINER B M. Roles for E-cadherin cell surface regulation in cancer[J]. Mol Biol Cell, 2016, 27(21): 3233-3244. |
| [23] |
SHENOY S. CDH1 (E-cadherin) mutation and gastric cancer: genetics, molecular mechanisms and guidelines for management[J]. Cancer Manag Res, 2019, 11: 10477-10486. |
| [24] |
KUNIDA K, TAKAGI N, AOKI K, et al. Decoding cellular deformation from pseudo-simultaneously observed Rho GTPase activities[J]. Cell Rep, 2023, 42(2): 112071. |
| [25] |
EBRAHIMI N, KHARAZMI K, GHANAATIAN M, et al. Role of the Wnt and GTPase pathways in breast cancer tumorigenesis and treatment[J]. Cytokine Growth Factor Rev, 2022, 67: 11-24. |
| [26] |
HE W Z, XU L L, DING J Y, et al. Co-targeting of ACK1 and KIT triggers additive anti-proliferative and-migration effects in imatinib-resistant gastrointestinal stromal tumors[J]. Biochim Biophys Acta Mol Basis Dis, 2023, 1869(5): 166690. |
| [27] |
JIANG Y F, LIU Y R, ZHANG Y Y, et al. MicroRNA-142-3P suppresses the progression of papillary thyroid carcinoma by targeting FN1 and inactivating FAK/ERK/PI3K signaling[J]. Cell Signal, 2023, 109: 110792. |
| [28] |
BENTON D, CHERNOFF J. RHOA drivers take alternate routes in gastric cancer[J]. Sci Signal, 2023, 16(816): eadk9171. |




