慢性肾脏病(chronic kidney disease, CKD)毋庸置疑已成为威胁全世界公共健康的主要疾病之一。流行病学调查结果显示,全球范围内CKD患病率高达11%~13%[1],患病人数超过8.5亿[2]。中国CKD患病率为10.8%,约1.2亿CKD患者[3]。CKD的3个主要不良后果是进展到终末期肾脏病(end-stage kidney disease, ESKD)、发生心血管疾病(cardiovascular disease, CVD)和死亡[4],大多数CKD患者未达到ESKD之前就已死亡[5]。CKD已成为全球范围内导致死亡的主要原因之一,预计到2040年,它将成为死亡的第五大原因[6]。因此,减少CKD患者死亡的发生是CKD干预的重要环节。
传统的观点认为估算的肾小球滤过率(estimated glomerular filtration rate, eGFR)降低、蛋白尿增加,合并高血压、糖尿病、肥胖等是并发CVD及CVD死亡的危险因素[7]。近些年,矿物质骨代谢异常(mineral and bone disorder, MBD)、蛋白质能量消耗(protein energy wasting, PEW)、成纤维细胞生长因子23(fibroblast growth factor 23,FGF23)过表达等新兴的危险因素在CKD进展及CVD并发症的发生过程中也起着非常重要的作用[8-10]。本课题组前期从基础研究、临床转化证实了Klotho蛋白可作为CKD患者预后的独立预测因子[11-12]。在CKD患者中,低血清Klotho水平预示着更快的CKD进展,更高的心血管事件和死亡风险[12]。因此,将这些新型的预后指标纳入临床评价中具有重要价值。
机器学习(machine learning,ML)作为人工智能(artificial intelligence, AI)的一个分支,它能利用计算机算法处理大量的复杂数据,通过对不同的变量之间的线性和非线性相互作用进行建模,从而产生更精确的预测模型,超越了传统统计方法[13]。在肾脏病学中,ML已被研发用于识别临床进展性疾病的高风险患者[14],血液透析患者的处方决策[15]、腹膜透析的感染病原体鉴定[16]等。因为CKD患者显示出较高的死亡风险,且与普通人群的危险预后指标不完全一致,基于普通人群中构建的预测全因死亡的风险预测模型在CKD患者中尚不适用,目前需要进一步构建针对CKD患者全因死亡的风险预测模型[17]。因此,本研究利用ML,整合简便易得的临床特征,构建并验证CKD1~5期非透析患者包含血清Klotho蛋白在内的全因死亡风险预测模型。
1 资料与方法 1.1 研究人群本研究采用回顾性队列研究设计方案,通过陆军军医大学第二附属医院伦理委员会批准(2018-研第006-02)。选择2012年2月7日至2019年10月18日于陆军军医大学第二附属医院肾内科住院确诊为CKD 1~5期的非透析患者456例,随访日期截至2022年2月1日。
1.2 纳入与排除标准纳入标准:①因各种原因导致的CKD就诊的患者且未进行任何肾脏替代治疗(血液透析、腹膜透析和肾移植);②年龄≥18岁。排除标准:①急性肾功能衰竭;②孕妇或哺乳期妇女;③有器官移植史需要长期服用免疫抑制剂;④近期有恶性肿瘤或其他未控制的活动性感染;⑤急性肾功能衰竭;⑥失访者;⑦基线数据缺失≥30%。
1.3 临床资料及实验室指标收集患者入院时的以下基线数据:包含人口学特征、既往吸烟史、既往患病史、血压、体质指数(BMI)、肾功、尿蛋白、肝功、血脂、血常规、T细胞亚群计数、补体、血清钙镁磷、PTH、炎症标志物。根据CKD-EPI公式计算eGFR[18]。
1.4 血清Klotho检测采集晨起空腹患者外周静脉血5 mL,静置30 min,900×g离心10 min,取血清分装,-80 ℃保存备用。ELISA检测血清Klotho蛋白水平,试剂盒由武汉华美生物工程有限公司提供。
1.5 终点事件的定义本研究的终点事件定义为各种原因引起的死亡。
1.6 统计学方法采用R4.3.2软件进行统计分析。对于计量资料,服从正态分布的采用x±s描述,组间比较采用独立样本t检验,不符合正态分布则采用M(P25,P75)进行描述,组间比较采用秩和检验;计数资料采用例数(%)进行描述,组间比较采用检验,不满足χ2检验条件采用Fisher确切概率法。定义检验水准α=0.05。以双侧P<0.05为差异有统计学意义。
1.7 机器学习对于少量缺失值,采用R语言“missRanger”包进行多重插补。所有研究对象经R语言sample函数按照7∶3比例随机划分为训练集和验证集。基于训练集进行预测模型构建:①基于单因素Cox回归分析,以结局事件为因变量,筛选出全因死亡可能的预测因子(P<0.05)。②进一步针对筛选出的变量,使用最小绝对收缩和选择算子(least absolute shrinkage and selection operator,Lasso)-Cox回归模型,采用10折交叉验证确定最优λ,选择标准为lambda.min。③对于筛选出的预测因子,进行多因素Cox回归分析(逐步法,双向)探索独立预测因子(P<0.05)。④基于独立预测因子构建预测模型列线图,对所构建列线图进行bootstrap抽样验证,绘制校准曲线以评估模型校准度,进一步通过时间依赖的受试者工作曲线(receiver operating characteristic, ROC)曲线分析,计算曲线下面积(area under the curve,AUC)、一致性指数(C指数)等指标,评估其鉴别效能。⑤将所构建的列线图模型在验证集数据中进行进一步验证。⑥根据列线图模型计算所有研究对象的列线图评分,在训练集中,以全因死亡为结局,根据列线图评分的最佳截断值将所有研究对象进行高、低风险分组,进一步绘制Kaplan-Meier曲线进行Logrank检验来验证。
2 结果 2.1 参与者的基线特征研究最终纳入400例非透析CKD1~5期非透析患者、47个变量及全因死亡1个终点事件进行分析。在平均6.62年的随访时间中,共有73例(18.25%)出现死亡。随机分为训练集及验证集,训练集280例,验证集120例。训练集有52例死亡,228例存活;验证集有21名死亡,99例存活,训练集和验证集不同指标之间基本无显著差异(P>0.05,表 1)。患者的人口统计学特征、合并症、实验室检测结果的基线情况详见表 1。
| 变量 | 总人群(n=400) | 训练集(n=280) | 验证集(n=120) | P |
| 死亡 | 73 (18.25) | 52 (18.57) | 21 (17.50) | 0.799 |
| 死亡时间/月 | 77.97 (57.11, 81.88) | 77.45 (52.95, 80.74) | 78.83 (60.91, 82.11) | 0.181 |
| 年龄/岁 | 47.00 (39.00, 58.00) | 47.00 (38.75, 57.00) | 48.00 (39.75, 60.00) | 0.429 |
| 男性 | 213 (53.25) | 142 (50.71) | 71 (59.17) | 0.121 |
| BMI/(kg/m2) | 23.28±3.39 | 23.25±3.32 | 23.35±3.56 | 0.790 |
| 吸烟史 | ||||
| 从不 | 299 (74.75) | 208 (74.29) | 91 (75.83) | 0.488 |
| 既往 | 44 (11.00) | 34 (12.14) | 10 (8.33) | |
| 现在 | 57 (14.25) | 38 (13.57) | 19 (15.83) | |
| 心血管疾病病史 | 74 (18.50) | 53 (18.93) | 21 (17.50) | 0.736 |
| 糖尿病病史 | 31 (7.75) | 21 (7.50) | 10 (8.33) | 0.775 |
| 高血压病史 | 112 (28.00) | 76 (27.14) | 36 (30.00) | 0.560 |
| 收缩压/mmHg | 138.00 (126.00, 152.25) | 140.00 (127.00, 154.25) | 135.00 (124.75, 150.00) | 0.059 |
| 舒张压/mmHg | 86.00 (78.00, 95.00) | 86.00 (78.00, 95.00) | 85.00 (77.00, 93.00) | 0.322 |
| eGFR/(mL/min·1.73 m2) | 16.86 (7.84, 43.15) | 15.83 (7.50, 41.59) | 23.35 (8.68, 46.79) | 0.135 |
| 尿素/(mmol/L) | 13.32 (8.26, 22.13) | 13.48 (8.83, 22.66) | 11.54 (7.22, 19.20) | 0.041 |
| 尿酸/(μmol/L) | 495.56±122.88 | 504.31±125.25 | 475.16±115.10 | 0.025 |
| 胱抑素C/(mg/L) | 3.18 (1.80, 4.59) | 3.33 (1.96, 4.65) | 2.67 (1.64, 4.41) | 0.073 |
| 视黄醇结合蛋白/(mg/L) | 73.00 (54.00, 98.00) | 74.00 (55.00, 98.00) | 72.00 (51.98, 99.25) | 0.775 |
| 24 h尿蛋白定量/g | 1.52 (0.70, 2.99) | 1.57 (0.80, 3.23) | 1.35 (0.52, 2.86) | 0.102 |
| 24 h尿微量白蛋白/mg | 1 016.25 (314.45, 2 260.10) | 1 052.00 (372.15, 2 298.58) | 845.00 (242.54, 2 216.01) | 0.160 |
| 尿微量白蛋白/肌酐/(mg/mmol) | 428.60 (133.50, 1 475.03) | 459.35 (140.12, 1 472.70) | 378.05 (128.50, 1 477.55) | 0.614 |
| 纤维蛋白原/(g/L) | 3.25 (2.77, 3.75) | 3.31 (2.82, 3.92) | 3.08 (2.57, 3.63) | 0.014 |
| 碱性磷酸酶/(U/L) | 65.00 (53.00, 85.00) | 66.00 (53.00, 87.25) | 64.00 (53.98, 79.50) | 0.288 |
| 总胆固醇/(mmol/L) | 4.57 (3.75, 5.45) | 4.59 (3.74, 5.65) | 4.44 (3.77, 5.16) | 0.234 |
| 甘油三酯/(mmol/L) | 1.52 (1.10, 2.14) | 1.50 (1.08, 2.11) | 1.58 (1.18, 2.23) | 0.358 |
| 高密度脂蛋白胆固醇/(mmol/L) | 1.08 (0.91, 1.31) | 1.10 (0.92, 1.35) | 1.05 (0.88, 1.27) | 0.203 |
| 低密度脂蛋白胆固醇/(mmol/L) | 2.76 (2.19, 3.40) | 2.80 (2.19, 3.47) | 2.75 (2.22, 3.18) | 0.212 |
| 白蛋白/(g/L) | 38.75 (34.00, 42.40) | 38.60 (34.00, 42.00) | 39.75 (34.00, 43.47) | 0.207 |
| 白细胞/(109/L) | 6.23 (5.15, 7.54) | 6.24 (5.08, 7.60) | 6.19 (5.42, 7.38) | 0.841 |
| 中性粒细胞/(109/L) | 4.16 (3.36, 5.33) | 4.21 (3.33, 5.35) | 4.08 (3.44, 5.18) | 0.890 |
| 淋巴细胞/(109/L) | 1.35 (0.99, 1.73) | 1.30 (0.96, 1.66) | 1.48 (1.02, 1.82) | 0.047 |
| 嗜酸性粒细胞/(109/L) | 0.12 (0.06, 0.22) | 0.12 (0.06, 0.23) | 0.12 (0.06, 0.21) | 0.935 |
| 单核细胞/(109/L) | 0.38 (0.28, 0.50) | 0.39 (0.28, 0.50) | 0.38 (0.28, 0.49) | 0.646 |
| 髓淋比 | 3.42 (2.61, 4.78) | 3.44 (2.64, 4.78) | 3.32 (2.44, 4.68) | 0.364 |
| 血红蛋白/(g/L) | 101.00 (81.00, 124.25) | 99.50 (81.00, 120.25) | 109.50 (81.75, 131.25) | 0.050 |
| 血小板/(109/L) | 169.00 (131.00, 219.00) | 172.00 (131.00, 219.25) | 163.00 (132.25, 212.75) | 0.553 |
| CD4 T细胞 | 452.00 (359.00, 585.00) | 446.00 (348.00, 572.00) | 468.00 (396.00, 604.00) | 0.094 |
| CD8 T细胞 | 400.00 (308.00, 558.00) | 398.00 (304.00, 564.00) | 408.00 (320.00, 540.00) | 0.444 |
| CD3 T细胞 | 888.00 (728.00, 1 209.00) | 876.00 (720.00, 1 208.00) | 944.00 (758.00, 1 213.00) | 0.178 |
| CD4/CD8 | 1.09 (0.85, 1.42) | 1.07 (0.86, 1.39) | 1.16 (0.83, 1.52) | 0.416 |
| 补体C3/(g/L) | 0.74 (0.64, 0.86) | 0.73 (0.63, 0.84) | 0.77 (0.67, 0.88) | 0.055 |
| 补体C4/(mg/dL) | 20.30 (17.00, 24.50) | 20.20 (16.60, 24.40) | 21.15 (17.80, 24.90) | 0.298 |
| 钙/(mmol/L) | 2.18 (2.06, 2.28) | 2.18 (2.06, 2.26) | 2.21 (2.09, 2.29) | 0.111 |
| 镁/(mmol/L) | 0.85 (0.77, 0.93) | 0.85 (0.77, 0.93) | 0.85 (0.76, 0.92) | 0.918 |
| 磷/(mmol/L) | 1.26 (1.08, 1.60) | 1.29 (1.08, 1.61) | 1.23 (1.07, 1.53) | 0.273 |
| 甲状旁腺激素/(pg/mL) | 112.00 (53.15, 265.25) | 123.10 (56.00, 265.25) | 95.15 (49.40, 251.25) | 0.279 |
| C反应蛋白/(mg/L) | 2.90 (1.90, 5.23) | 2.95 (2.00, 5.30) | 2.65 (1.80, 5.00) | 0.347 |
| IL-6/(pg/mL) | 3.15 (2.10, 5.23) | 3.33 (2.12, 5.81) | 2.90 (2.10, 4.32) | 0.068 |
| IL-8/(pg/mL) | 7.33 (5.00, 11.50) | 7.83 (5.00, 12.70) | 6.74 (5.00, 10.22) | 0.041 |
| TNF-α/(pg/mL) | 12.30 (8.90, 16.60) | 12.55 (9.20, 16.90) | 11.10 (8.78, 15.88) | 0.108 |
| Klotho蛋白/(pg/mL) | 684.65 (340.94, 1 079.07) | 653.51 (327.92, 1 117.75) | 692.26 (367.41, 1 000.00) | 0.978 |
2.2 单因素Cox回归分析结果
将训练集中的变量纳入单因素Cox回归分析结果显示,年龄、心血管疾病病史、胱抑素C、碱性磷酸酶、白蛋白、嗜酸性粒细胞、血红蛋白、补体C3、钙、C反应蛋白、肿瘤坏死因子-α(TNF-α)以及血清Klotho蛋白可能是全因死亡的预测因子(P<0.05,表 2)。
| 变量 | 回归系数 | 标准误差 | 统计量 | HR (95%CI) | P |
| 年龄 | 0.052 | 0.010 | 5.279 | 1.053 (1.033, 1.073) | <0.001 |
| 性别 | |||||
| 男性 | 0.000 | reference | |||
| 女性 | -0.027 | 0.277 | -0.097 | 0.974 (0.565, 1.677) | 0.923 |
| BMI | -0.064 | 0.043 | -1.483 | 0.938 (0.863, 1.021) | 0.138 |
| 吸烟史 | |||||
| 从不 | 0.000 | reference | |||
| 既往 | 0.377 | 0.390 | 0.966 | 1.458 (0.679, 3.131) | 0.334 |
| 现在 | 0.109 | 0.412 | 0.263 | 1.115 (0.497, 2.501) | 0.792 |
| 心血管疾病病史 | |||||
| 无 | 0.000 | reference | |||
| 有 | 0.689 | 0.301 | 2.291 | 1.991 (1.105, 3.588) | 0.022 |
| 糖尿病病史 | |||||
| 无 | 0.000 | reference | |||
| 有 | 0.386 | 0.471 | 0.819 | 1.471 (0.584, 3.705) | 0.413 |
| 高血压病史 | |||||
| 无 | 0.000 | reference | |||
| 有 | 0.272 | 0.296 | 0.921 | 1.313 (0.735, 2.344) | 0.357 |
| 收缩压 | 0.007 | 0.007 | 1.084 | 1.007 (0.994, 1.020) | 0.278 |
| 舒张压 | -0.016 | 0.010 | -1.626 | 0.984 (0.965, 1.003) | 0.104 |
| eGFR | -0.011 | 0.007 | -1.687 | 0.989 (0.977, 1.002) | 0.092 |
| 尿素 | 0.018 | 0.013 | 1.411 | 1.018 (0.993, 1.043) | 0.158 |
| 尿酸 | 0.001 | 0.001 | 0.661 | 1.001 (0.999, 1.003) | 0.509 |
| 胱抑素C | 0.196 | 0.083 | 2.373 | 1.217 (1.035, 1.432) | 0.018 |
| 视黄醇结合蛋白 | -0.002 | 0.005 | -0.430 | 0.998 (0.989, 1.007) | 0.667 |
| 24 h尿蛋白定量 | 0.026 | 0.050 | 0.517 | 1.026 (0.931, 1.131) | 0.605 |
| 24 h尿微量白蛋白 | 0.000 | 0.000 | 0.145 | 1.000 (1.000, 1.000) | 0.885 |
| 尿微量白蛋白/肌酐 | 0.000 | 0.000 | 0.808 | 1.000 (1.000, 1.000) | 0.419 |
| 纤维蛋白原 | 0.088 | 0.143 | 0.614 | 1.092 (0.825, 1.446) | 0.539 |
| 碱性磷酸酶 | 0.011 | 0.003 | 3.457 | 1.011 (1.005, 1.017) | 0.001 |
| 总胆固醇 | -0.044 | 0.074 | -0.588 | 0.957 (0.827, 1.108) | 0.557 |
| 甘油三酯 | -0.117 | 0.125 | -0.938 | 0.890 (0.697, 1.136) | 0.348 |
| 高密度脂蛋白胆固醇 | 0.204 | 0.337 | 0.605 | 1.226 (0.634, 2.373) | 0.545 |
| 低密度脂蛋白胆固醇 | -0.134 | 0.121 | -1.111 | 0.875 (0.690, 1.108) | 0.266 |
| 白蛋白 | -0.041 | 0.017 | -2.382 | 0.960 (0.928, 0.993) | 0.017 |
| 白细胞 | 0.028 | 0.060 | 0.472 | 1.029 (0.915, 1.156) | 0.637 |
| 中性粒细胞 | 0.031 | 0.070 | 0.444 | 1.032 (0.899, 1.184) | 0.657 |
| 淋巴细胞 | -0.197 | 0.247 | -0.798 | 0.821 (0.506, 1.332) | 0.425 |
| 嗜酸性粒细胞 | 1.425 | 0.470 | 3.033 | 4.160 (1.656, 10.451) | 0.002 |
| 单核细胞 | 0.718 | 0.646 | 1.111 | 2.050 (0.578, 7.276) | 0.267 |
| 髓淋比 | 0.044 | 0.035 | 1.245 | 1.045 (0.975, 1.120) | 0.213 |
| 血红蛋白 | -0.013 | 0.005 | -2.377 | 0.987 (0.977, 0.998) | 0.017 |
| 血小板 | 0.001 | 0.002 | 0.440 | 1.001 (0.997, 1.005) | 0.660 |
| CD4 T细胞 | -0.000 | 0.001 | -0.574 | 1.000 (0.998, 1.001) | 0.566 |
| CD8 T细胞 | -0.000 | 0.001 | -0.759 | 1.000 (0.998, 1.001) | 0.448 |
| CD3 T细胞 | -0.000 | 0.000 | -0.669 | 1.000 (0.999, 1.000) | 0.503 |
| CD4/CD8 | -0.056 | 0.333 | -0.169 | 0.945 (0.492, 1.815) | 0.866 |
| 补体C3 | -2.280 | 0.884 | -2.578 | 0.102 (0.018, 0.579) | 0.010 |
| 补体C4 | -0.042 | 0.024 | -1.731 | 0.959 (0.914, 1.006) | 0.083 |
| 钙 | -1.290 | 0.468 | -2.758 | 0.275 (0.110, 0.689) | 0.006 |
| 镁 | 0.014 | 0.925 | 0.016 | 1.015 (0.165, 6.222) | 0.988 |
| 磷 | 0.273 | 0.231 | 1.183 | 1.314 (0.836, 2.065) | 0.237 |
| 甲状旁腺激素 | 0.000 | 0.001 | 0.133 | 1.000 (0.999, 1.001) | 0.894 |
| C反应蛋白 | 0.013 | 0.005 | 2.562 | 1.013 (1.003, 1.023) | 0.010 |
| IL-6 | 0.000 | 0.005 | 0.071 | 1.000 (0.992, 1.009) | 0.943 |
| IL-8 | -0.000 | 0.000 | -0.221 | 1.000 (0.999, 1.001) | 0.825 |
| TNF-α | 0.025 | 0.007 | 3.610 | 1.025 (1.011, 1.039) | <0.001 |
| Klotho蛋白 | -0.001 | 0.000 | -2.310 | 0.999 (0.999, 1.000) | 0.021 |
2.3 Lasso-Cox模型对风险因子的筛选
考虑到变量之间的多重共线性,本研究将单因素Cox回归分析中P<0.05变量纳入Lasso-Cox回归模型中,在选择最优Lambda时采用100次10倍交叉验证确定最优λ(λ=0.014 3),选择标准为lambda.min(图 1),确定了年龄、胱抑素C、碱性磷酸酶、白蛋白、嗜酸性粒细胞、补体C3、钙、C反应蛋白、TNF-α以及血清Klotho蛋白共10个非零系数变量纳入多因素Cox逐步回归。
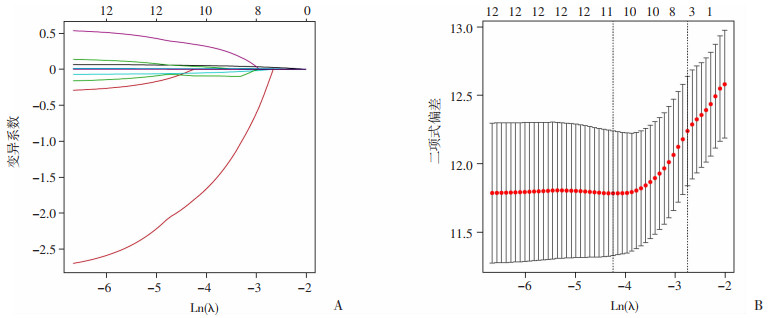
|
| A:LASSO回归变量变异系数与Ln(λ)的关系曲线图;B:LASSO回归二项式偏差与Ln(λ)的关系曲线图 图 1 Lasso-Cox回归对CKD患者全因死亡的风险因子筛选图 |
2.4 全因死亡风险预测模型的构建
本研究对Lasso-Cox模型筛选出的10个危险因子纳入多因素Cox回归分析(逐步法),筛选出4个独立预测因子(P<0.05),分别为年龄、白蛋白、补体C3及血清Klotho,其中年龄大是CKD患者全因死亡的危险因素,而白蛋白、补体C3及血清Klotho高是CKD患者全因死亡的保护因素(表 3),进一步我们将其均按数值进行了划分,进一步构建列线图(图 2)。
| 变量 | 回归系数 | 标准误差 | 统计量 | HR(95%CI) | P |
| 年龄 | 0.061 | 0.010 | 6.149 | 1.063 (1.042, 1.083) | <0.001 |
| 碱性磷酸酶 | 0.006 | 0.003 | 1.883 | 1.006 (1.000, 1.013) | 0.060 |
| 白蛋白 | -0.075 | 0.020 | -3.805 | 0.928 (0.892, 0.964) | <0.001 |
| 补体C3 | -2.790 | 0.892 | -3.128 | 0.061 (0.011, 0.353) | 0.002 |
| Klotho | -0.001 | 0.000 | -2.327 | 0.999 (0.999, 1.000) | 0.020 |

|
| 图 2 CKD患者全因死亡的风险预测列线图 |
图 2对各风险变量的组合评分合计后可以预测CKD患者1、3、5年生存率。例如,某CKD患者年龄为50岁,白蛋白为15 g/L,补体C3为0.5 g/L,血清Klotho蛋白为500 pg/mL,每个变量从坐标轴上的位置向上延伸出一条垂直线与“分数”坐标轴的交点对应数值分别为:50、78、67、55分,分值总和为250分,在“总得分”坐标轴上找到相应的点向下延伸出一条垂直线与“1、3、5年生存率”坐标轴的交点所对应的值0.72、0.35、0.20即为CKD患者预测的1、3、5年生存率。
2.5 CKD患者全因死亡风险预测模型的验证模型bootstrap抽样验证结果显示(图 3A、C):训练集和验证集在1、3、5年,模型预测的总生存概率与实际生存概率比较吻合,证明模型校准度较好。时间依赖性ROC曲线结果显示(图 3B、D):训练集和验证集1、3、5年AUC均>0.7,其中1年AUC>0.8。模型总体C指数在训练集、验证集中分别为0.755 (95%CI:0.685, 0.826)、0.720 (95%CI:0.614, 0.826),提示模型预测效能良好。模型在训练集和验证集中的敏感度、特异度、准确度详见表 4。

|
| A:在训练集中模型的校正曲线;B:在训练集中模型的ROC曲线;C:在验证集中模型的校正曲线;D:在验证集中模型的ROC曲线 图 3 CKD患者全因死亡风险预测模型的精度评估 |
| 组别 | 时间 | 敏感度 | 特异度 | 准确度 |
| 训练集 | 1年 | 0.818 | 0.903 | 0.900 |
| 3年 | 0.586 | 0.924 | 0.889 | |
| 5年 | 0.500 | 0.941 | 0.871 | |
| 验证集 | 1年 | 1.000 | 0.544 | 0.567 |
| 3年 | 0.667 | 0.870 | 0.850 | |
| 5年 | 0.750 | 0.692 | 0.700 |
本研究根据列线图模型计算了所有研究对象的列线图评分,按照列线图评分的最佳截断值(214.57)对训练集和验证集所有患者进行风险分层(低风险、高风险组),结果显示: 在训练集和验证集人群中,低风险组的生存率显著高于高风险组(P均<0.001, 图 4)。

|
| A:训练集Kaplan-Meier曲线;B:验证集Kaplan-Meier曲线 图 4 CKD患者生存分析曲线 |
3 讨论
本研究利用回顾性队列研究,运用机器学习的方式,成功地构建了CKD患者全因死亡的风险预测模型。多因素Cox逐步回归分析显示,碱性磷酸酶不是CKD患者全因死亡的独立预测因子,而年龄、白蛋白、补体C3及血清Klotho是CKD患者全因死亡的独立预测因子。1项回顾性研究报道,较高的血清碱性磷酸酶是男性血液透析患者全因死亡的独立危险因素[19],而对于非透析CKD患者而言尚无临床证据支撑,有待进一步探讨。低白蛋白血症与CKD患者的死亡风险增加有关,有研究已经证实了低白蛋白血症是CKD3~4期患者全因死亡的独立危险因素[20],与本研究的结果相一致。
既往多项研究表明,血清Klotho蛋白可以抑制左心室肥厚从而抑制尿毒症性心肌病的进展[11],延缓CKD相关的血栓形成和动脉粥样硬化[21],调节磷酸盐的摄取来抑制血管钙化[22],进一步临床研究证实了在CKD非透析患者中,较低的血清Klotho蛋白水平与心血管事件及全因死亡独立相关[12]。在本研究的模型中,不仅纳入了年龄等传统因素,还加入了血清Klotho蛋白这一新兴危险因子,提高了模型的预测价值,同时也再此证实了血清Klotho蛋白对CKD患者不良结局的预测作用。
1项调查CKD患者终末期肾病、心血管事件和死亡的风险预测模型的系统综述研究表示,需要进一步构建心血管事件和全因死亡的模型[17]。本研究研发的全因死亡预测模型具有令人满意的内部性能(ROC>0.75)。TRAN等[23]针对法国CKD4~5期非透析人群,比较了4种机器学习模型,选择了贝叶斯网络构建了包含7个变量的全因死亡风险预测模型,模型的性能较好(ROC>0.7),其中模型纳入了年龄、心血管疾病史、吸烟状况、铁蛋白、甲状旁腺激素、25羟维生素D及红细胞生成刺激剂。LANDRAY等[24]早已证实了CKD3~5期非透析人群发生死亡的高风险,利用传统的Cox回归构建了死亡风险预测模型,包括年龄、肌钙蛋白T、吸烟史等强有力危险因素,预测效能高(AUC=0.82)。
随着电子病历系统的广泛使用,AI越来越多地被应用于决策支持并优化个体治疗。比如,对于CKD患者,DEO等[25]基于循环蛋白质组构建了心血管并发症相关风险方程,其效能优于包含eGFR在内的传统临床风险模型;ZELNICK等[26]利用机器学习在CKD人群中构建了房颤的模型;SEGAL等[27]针对CKD1~4期患者利用XGBoost模型构建了终末期肾脏病风险预测模型,展示出优异的预测效能(C指数>0.9)。
本研究创新地将血清Klotho蛋白作为CKD预后核心预测因子,采用危险因素积分方法,通过ML建模的方式,建立AI预后预测模型,很大程度提高了CKD患者死亡预后的预测效能。此外,本研究纳入的相关风险指标全面且随访周期长,构建的全因死亡预测模型使用的参数在临床上很容易获得,因此使该模型更容易应用于基层卫生保健环境。
本研究也存在如下不足:首先,本研究的数据来自单中心的样本量,仅代表中国部分CKD人群,未考虑种族、地域的差异,需要在中国进行多中心的外部验证;然后,一些额外的预测变量,包括影像学指标(腹部彩超、血管彩超、心脏彩超等结果)、遗传因素、代谢、行为方式等也可能对死亡结局有影响,但在招募时未纳入分析;另外,目前血清Klotho蛋白这一指标尚未进行临床常规开展检测,期待进一步研发更有效的检测方式,加速其临床应用的转化。
综上所述,本研究基于ML成功构建并验证了CKD患者全因死亡风险预测工具。研究结果表明,基于ML构建的该模型是可行和有效的,对预测CKD患者全因死亡具有一定的指导意义。
| [1] |
HILL N R, FATOBA S T, OKE J L, et al. Global prevalence of chronic kidney disease—A systematic review and meta-analysis[J]. PLoS One, 2016, 11(7): e0158765. DOI:10.1371/journal.pone.0158765 |
| [2] |
JAGER K J, KOVESDY C, LANGHAM R, et al. A single number for advocacy and communication-worldwide more than 850 million individuals have kidney diseases[J]. Nephrol Dial Transplant, 2019, 34(11): 1803-1805. DOI:10.1093/ndt/gfz174 |
| [3] |
ZHANG L X, WANG F, WANG L, et al. Prevalence of chronic kidney disease in China: a cross-sectional survey[J]. Lancet, 2012, 379(9818): 815-822. DOI:10.1016/S0140-6736(12)60033-6 |
| [4] |
LEVEY A S, ECKARDT K U, TSUKAMOTO Y, et al. Definition and classification of chronic kidney disease: a position statement from kidney disease: improving global outcomes (KDIGO)[J]. Kidney Int, 2005, 67(6): 2089-2100. DOI:10.1111/j.1523-1755.2005.00365.x |
| [5] |
ZOCCALI C, MALLAMACI F, ADAMCZAK M, et al. Cardiovascular complications in chronic kidney disease: a review from the European Renal and Cardiovascular Medicine Working Group of the European Renal Association[J]. Cardiovasc Res, 2023, 119(11): 2017-2032. DOI:10.1093/cvr/cvad083 |
| [6] |
FOREMAN K J, MARQUEZ N, DOLGERT A, et al. Forecasting life expectancy, years of life lost, and all-cause and cause-specific mortality for 250 causes of death: reference and alternative scenarios for 2016-40 for 195 countries and territories[J]. Lancet, 2018, 392(10159): 2052-2090. DOI:10.1016/S0140-6736(18)31694-5 |
| [7] |
GANSEVOORT R T, CORREA-ROTTER R, HEMMELGARN B R, et al. Chronic kidney disease and cardiovascular risk: epidemiology, mechanisms, and prevention[J]. Lancet, 2013, 382(9889): 339-352. DOI:10.1016/S0140-6736(13)60595-4 |
| [8] |
NORRIS K C, OLABISI O, BARNETT M E, et al. The role of vitamin D and oxidative stress in chronic kidney disease[J]. Int J Environ Res Public Health, 2018, 15(12): 2701. DOI:10.3390/ijerph15122701 |
| [9] |
KOPPE L, FOUQUE D, KALANTAR-ZADEH K. Kidney cachexia or protein-energy wasting in chronic kidney disease: facts and numbers[J]. J Cachexia Sarcopenia Muscle, 2019, 10(3): 479-484. DOI:10.1002/jcsm.12421 |
| [10] |
EDMONSTON D, GRABNER A, WOLF M. FGF23 and klotho at the intersection of kidney and cardiovascular disease[J]. Nat Rev Cardiol, 2024, 21(1): 11-24. DOI:10.1038/s41569-023-00903-0 |
| [11] |
YANG K, WANG C, NIE L, et al. Klotho protects against indoxyl sulphate-induced myocardial hypertrophy[J]. J Am Soc Nephrol, 2015, 26(10): 2434-2446. DOI:10.1681/ASN.2014060543 |
| [12] |
YANG K, YANG J X, BI X J, et al. Serum klotho, cardiovascular events, and mortality in nondiabetic chronic kidney disease[J]. Cardiorenal Med, 2020, 10(3): 175-187. DOI:10.1159/000506380 |
| [13] |
THONGPRAYOON C, KAEWPUT W, CHOUDHURY A, et al. Is it time for machine learning algorithms to predict the risk of kidney failure in patients with chronic kidney disease?[J]. J Clin Med, 2021, 10(5): 1121. DOI:10.3390/jcm10051121 |
| [14] |
NIEL O, BOUSSARD C, BASTARD P. Artificial intelligence can predict GFR decline during the course of ADPKD[J]. Am J Kidney Dis, 2018, 71(6): 911-912. DOI:10.1053/j.ajkd.2018.01.051 |
| [15] |
BARBIERI C, MOLINA M, PONCE P, et al. An international observational study suggests that artificial intelligence for clinical decision support optimizes anemia management in hemodialysis patients[J]. Kidney Int, 2016, 90(2): 422-429. DOI:10.1016/j.kint.2016.03.036 |
| [16] |
ZHANG J J, FRIBERG I M, KIFT-MORGAN A, et al. Machine-learning algorithms define pathogen-specific local immune fingerprints in peritoneal dialysis patients with bacterial infections[J]. Kidney Int, 2017, 92(1): 179-191. DOI:10.1016/j.kint.2017.01.017 |
| [17] |
TANGRI N, KITSIOS G D, INKER L A, et al. Risk prediction models for patients with chronic kidney disease: a systematic review[J]. Ann Intern Med, 2013, 158(8): 596-603. DOI:10.7326/0003-4819-158-8-201304160-00004 |
| [18] |
LEVEY A S, STEVENS L A, SCHMID C H, et al. A new equation to estimate glomerular filtration rate[J]. Ann Intern Med, 2009, 150(9): 604-612. DOI:10.7326/0003-4819-150-9-200905050-00006 |
| [19] |
KOBAYASHI I, SHIDARA K, OKUNO S, et al. Higher serum bone alkaline phosphatase as a predictor of mortality in male hemodialysis patients[J]. Life Sci, 2012, 90(5-6): 212-218. DOI:10.1016/j.lfs.2011.11.006 |
| [20] |
MENON V, GREENE T, WANG X, et al. C-reactive protein and albumin as predictors of all-cause and cardiovascular mortality in chronic kidney disease[J]. Kidney Int, 2005, 68(2): 766-772. DOI:10.1111/j.1523-1755.2005.00455.x |
| [21] |
YANG K, DU C H, WANG X M, et al. Indoxyl sulfate induces platelet hyperactivity and contributes to chronic kidney disease-associated thrombosis in mice[J]. Blood, 2017, 129(19): 2667-2679. DOI:10.1182/blood-2016-10-744060 |
| [22] |
LIU L, LIU Y, ZHANG Y, et al. High phosphate-induced downregulation of PPARγ contributes to CKD-associated vascular calcification[J]. J Mol Cell Cardiol, 2018, 114: 264-275. DOI:10.1016/j.yjmcc.2017.11.021 |
| [23] |
TRAN N T D, BALEZEAUX M, GRANAL M, et al. Prediction of all-cause mortality for chronic kidney disease patients using four models of machine learning[J]. Nephrol Dial Transplant, 2023, 38(7): 1691-1699. DOI:10.1093/ndt/gfac316 |
| [24] |
LANDRAY M J, EMBERSON J R, BLACKWELL L, et al. Prediction of ESRD and death among people with CKD: the Chronic Renal Impairment in Birmingham (CRIB) prospective cohort study[J]. Am J Kidney Dis, 2010, 56(6): 1082-1094. DOI:10.1053/j.ajkd.2010.07.016 |
| [25] |
DEO R, DUBIN R F, REN Y, et al. Proteomic cardiovascular risk assessment in chronic kidney disease[J]. Eur Heart J, 2023, 44(23): 2095-2110. DOI:10.1093/eurheartj/ehad115 |
| [26] |
ZELNICK L R, SHLIPAK M G, SOLIMAN E Z, et al. Prediction of incident atrial fibrillation in chronic kidney disease: the chronic renal insufficiency cohort study[J]. Clin J Am Soc Nephrol, 2021, 16(7): 1015-1024. DOI:10.2215/CJN.01060121 |
| [27] |
SEGAL Z, KALIFA D, RADINSKY K, et al. Machine learning algorithm for early detection of end-stage renal disease[J]. BMC Nephrol, 2020, 21(1): 518. DOI:10.1186/s12882-020-02093-0 |






