2. 100850 北京,中国人民解放军军事科学院军事医学研究院生物信息中心;
3. 100853 北京,解放军总医院:第一医学中心心内科
2. Bioinformatics Centre, Academy of Military Medical Sciences, Academy of Military Sciences of Chinese PLA, Beijing, 100850, China;
3. Department of Cardiology, First Medical Center, Chinese PLA General Hospital, Beijing, 100853
乳腺癌是威胁我国乃至全球女性健康的第一大恶性肿瘤,其发病率和死亡率在女性肿瘤中位居前列[1-2]。目前乳腺癌的治疗方法较多,其中化疗、放疗和内分泌治疗是公认较有效的方法[3]。放射治疗是乳腺癌新辅助治疗、术中和术后治疗的重要组成部分,对疾病早期和中晚期均有较好的疗效[4-6]。
放射治疗通过电离辐射(ionizing radiation, IR)诱导细胞DNA损伤,抑制细胞增殖并促进其凋亡。理论上辐射对所有肿瘤细胞都有杀伤作用,然而每个细胞对辐射的敏感性不同,导致疗效不均一[7-8]。在辐射诱导下,部分肿瘤细胞通过复杂而精细的调节机制来应对出现的DNA损伤,包括激活DNA修复通路、延迟细胞周期、抵抗凋亡等,进而引起肿瘤的复发和转移,导致预后不佳[9-10]。RRM2B是一种关键的核糖核苷酸还原酶亚基,在DNA修复通路发挥重要作用[11]。既往对RRM2B的研究多集中在线粒体相关疾病,如先天性耳聋、线粒体神经胃肠道脑病等[12]。最新的研究表明,RRM2B能够影响肝癌、神经母细胞瘤、卵巢癌等肿瘤的发生发展及预后[13]。然而,RRM2B在乳腺癌及其在放疗中的DNA损伤修复作用仍然未知。
本研究通过对辐射处理ZR-75-1细胞和正常ZR-75-1细胞进行RNA测序分析对比,观察与DNA损伤修复相关基因中RRM2B的表达;采用TCGA-BRCA数据库分析RRM2B在乳腺癌组织中的表达及与患者总生存率的关系;在乳腺癌细胞系中敲低RRM2B再进行辐射处理,观察细胞DNA损伤、细胞凋亡及增殖的变化。该研究验证RRM2B在乳腺癌抵抗辐射损伤中的重要作用机制,为提高乳腺癌细胞放疗敏感性提供新的思路和方法。
1 材料与方法 1.1 细胞系和试剂人乳腺癌细胞系(ZR-75-1、MCF-7、BT-474、MDA-MB-231)、人胚肾细胞HEK-293T和人正常乳腺上皮细胞MCF-10A均购自武汉普诺赛生命科技有限公司,由实验室长期保存。DMEM培养基购自美国Gibco公司;胎牛血清购自美国HyClone公司;1%青-链霉素溶液、0.25%胰蛋白酶购自北京市阳光英锐生物科技有限公司;RRM2B多克隆抗体(18005-1-AP)、α-Tubulin多克隆抗体(1224-1-AP)、HRP偶联山羊抗兔IgG(SA00001-2)购自武汉三鹰公司;488偶联山羊抗兔IgG(AS053)购自武汉爱博泰克公司;γ-H2AX抗体(9718)购自中国CST公司;反转录试剂(R333 Vazyme)及qPCR试剂(Q712 Vazyme)购自南京诺唯赞生物科技股份有限公司;TRIzol Universal总RNA提取试剂盒购自中国天根生化科技有限公司;CCK-8试剂盒(40203ES60)购自中国翌圣生物科技股份有限公司;RRM2B shRNA及shNC由北京天一辉远公司合成;Hoechst 33342/PI双染试剂盒(CA1120)和Annexin V-FITC/PI凋亡检测试剂盒(CA1020)购自北京索莱宝科技有限公司;RNA测序相关试剂盒及Illumina NexSeq 550测序仪由军事医学研究院提供。
1.2 方法 1.2.1 细胞培养及辐射处理使用含有10%胎牛血清和1%青-链霉素溶液的DMEM完全培养基,在含5% CO2、37 ℃的恒温细胞孵箱中培养细胞。细胞消化使用0.25%胰蛋白酶。采用5 Gy X射线对细胞进行辐照(剂量率3.0 Gy/min)。照射后将细胞移入含5% CO2、37 ℃的恒温细胞孵箱中继续培养48 h。根据实验要求进行下一步操作。
1.2.2 细胞总RNA提取使用TRIzol Universal裂解细胞,1 mL TRIzol Universal试剂加0.2 mL氯仿,剧烈振荡15 s,室温放置3 min。4 ℃、12 000 r/min离心15 min。使用异丙醇沉淀后,用75%乙醇洗涤沉淀2次,离心去乙醇后晾干,用50 μL RNase-Free ddH2O溶解沉淀并测量浓度。
1.2.3 RT-qPCR及RNA测序分析将提取的总RNA按照说明书步骤先反转录成cDNA。采用荧光定量PCR仪(Bio-Rad)检测基因表达丰度。RT-qPCR反应的总体积为20 μL,包括9.2 μL cDNA模板,10 μL 2×SYBR绿色RT-qPCR混合物,0.4 μL 10 μmol/L正向引物和0.4 μL反向引物。所有操作均在冰上完成,PCR引物序列见表 1,α-Tubulin作为内参照基因,每组设2个复孔。采用2-ΔΔCt方法计算相对基因表达值。
| 引物名称 | 引物序列(5′→3′) |
| RRM2B | 上游:GCAGGACCGCTGTAGGAAAT |
| 下游:ATGACAAACCGGCGAGAACT | |
| α-Tubulin | 上游:CGGGCAGTGTTTGTAGACTTGG |
| 下游:CTCCTTGCCAATGGTGTAGTGC |
按照1.2.1的方法对细胞进行辐照处理后,提取RNA并使用NanoDrop进行质检,稀释20倍后用Qubit Flex(美国Invitrogen公司)进行RNA定量。mRNA富集后,取500 ng的mRNA使用VAHTS® Universal V8 RNA-seq Library Prep Kit for Illumina进行样品建库,使用VAHTS® RNA Multiplex Oligos Set 1 for Illumina进行测序接头连接。测序文库构建完成后,使用Agilent 2100 Bioanalyzer进行文库片段质检,7500 Real Time PCR System进行定量,将文库稀释到1.5 pmol/L,使用NextSeq 500/550 Mid Output Kit v2.5上机试剂盒,在Illumina NexSeq 550测序仪进行RNA seq测序。基因表达谱数据的获取步骤如下:首先,使用trim galore(v0.6.10)对原始数据进行接头去除和低质量reads的过滤;随后,使用HISAT2(v2.2.1)将清理后的fastq数据参考基因组进行比对;然后使用featureCount(v1.6)计算测序数据匹配到参考基因组上每个基因的reads读数,从而获取基因表达谱数据,使用“Transcripts Per Million” (TPM)方法对reads读数进行标准化处理。
差异基因通过R包DESeq2(v1.38.1)获取,筛选差异基因使用的阈值参数为:校正后的差异显著性Padj<0.05,且对数差异倍数|log2 Fold Change|>0.5。使用R包clusterProfiler(v4.6.2)进行富集分析,其中GO富集分析和GSEA分析分别使用的是函数enrichGO和gseKEGG。
1.2.4 TCGA数据库下载和分析TCGA-BRCA数据来自在线数据库(https://portal.gdc.cancer.gov/),数据分为正常组织组和肿瘤组织组,采用R语言和Perl语言进行数据处理和分析。
1.2.5 Western blot检测蛋白表达水平将处理好的细胞经消化离心后加入2倍体积的RIPA裂解提取蛋白,加入与RIPA等量的2×SDS后沸水煮15 min,使蛋白充分变性,随后进行蛋白电泳。蛋白转印至硝酸纤维素膜后,用新鲜配制的5%脱脂牛奶室温封闭1 h,室温下孵育一抗1.5 h,TBST在室温条件下洗涤3次后孵育二抗1 h,再次使用TBST洗涤3次后,使用Bio-Rad显影仪显影。
1.2.6 稳定敲低RRM2B细胞系的构建将4.4 μg shNC或shRRM2B-1/2、2.2 μg PSP、3.3 μg PMD的质粒加入500 μL无血清无双抗的DMEM并混匀,加入转染试剂PEI(20 μL),混匀后室温静置15 min。将混合液均匀加入HEK-293T细胞中,放入培养箱孵育7 h后换液。分别于48、72 h后用0.45 μm的滤器过滤HEK-293T上清病毒液。将收集的病毒液感染ZR-75-1细胞和MCF-7细胞,并于72 h后加入2 μg/mL嘌呤霉素,筛选2周后进行细胞鉴定。细胞实验中,shNC细胞为对照组,shRRM2B-1/2细胞为实验组,其中实验组中2种shRNA敲低效果均可,后续实验组均采用shRRM2B-1细胞。
1.2.7 免疫荧光染色将细胞用PBS清洗后,使用4%多聚甲醛固定30 min。用Triton X-100通透细胞,随后用配好的山羊血清封闭。加入一抗孵育2 h,用配好的山羊血清封闭液清洗3遍,每次5 min,避光条件下加入二抗孵育1 h。PBS清洗3遍后,加入DAPI避光孵育5 min。PBS清洗2遍后用中性树脂封片,荧光显微镜下观察及拍照。
1.2.8 CCK-8实验将处理好的细胞消化悬浮并计数(1×105/mL),置于96孔板中(100 μL/孔,设置3个复孔),恒温孵育4 h使细胞贴壁。培养板中加入10 μL的CCK-8溶液并孵育2 h,用酶标仪测定在450 nm处的光密度值[D(450)]。分别于24、48、72、96 h采用同样的方法测定D(450)值。用GraphPad绘制生长曲线。
1.2.9 克隆形成实验将处理好的细胞消化并计数,取约500个细胞接种于3.5 cm培养皿中,继续培养10 d,每3天换液。使用普通光学显微镜观察细胞克隆形成情况。弃去培养基后,用PBS洗涤2次,在室温条件下用4%多聚甲醛固定30 min。再用PBS洗涤2次,1% 结晶紫溶液染色10 min,用流动的清水清洗结晶紫溶液,晾干后显微镜下观察和拍照。
1.2.10 Hoechst/PI染色按照Hoechst 33342/PI双染试剂盒说明书操作。吸尽细胞内的培养基,用4%多聚甲醛固定30 min。用PBS洗涤3次,弃去PBS后,加入Hoechst33342染色液避光置于摇床5 min,PBS洗涤2次后加入PI染色液避光置于摇床10 min。PBS洗涤2次后用荧光防淬灭剂进行封片,然后在荧光显微镜下进行观察并拍照。
1.2.11 流式细胞术检测细胞凋亡按试剂盒说明书进行操作,细胞胰酶消化后用PBS洗涤,去除多余的胰酶。使用FITC结合液重悬细胞,分别加入5 μL Annexin V-FITC和10 μL PI染色液,轻轻混匀。室温避光孵育10 min,然后上机检测细胞的凋亡情况。
1.3 统计学分析采用SPSS 23.0软件进行数据分析,Prism8.0(GraphPad)软件绘制图形。计量资料以x±s表示,两组间比较采用t检验,实验重复2次。生存分析采用Kaplan-Meier法。P<0.05表示差异具有统计学意义。
2 结果 2.1 辐射处理后ZR-75-1细胞DNA修复基因RRM2B和DDB2表达增加将ZR-75-1细胞分为未辐射的对照组和辐射处理的实验组,辐射处理48 h后提取总RNA,对mRNA富集后进行转录组测序(图 1A)。结果显示,与对照组(0 Gy)比较,5 Gy辐射处理的细胞中有117个基因上调,197个基因下调(图 1B)。两组细胞差异基因的GO富集分析显示,辐射后ZR-75-1细胞中细胞质翻译、p53相关的信号转导、核糖体小亚基组装、p53介导的凋亡信号等通路显著上调,而DNA复制、核染色体分离、有丝分裂核分裂、细胞周期的负调控等通路显著下调(图 1C)。表明辐射刺激后细胞不仅有辐射相关损伤的表现,还有细胞对抗辐射产生的保护性通路的激活。对差异基因与DNA损伤修复相关基因集取交集(图 1D)发现,有16个相关基因表达下调,如PCNA、BRCA、FANC等;2个相关基因表达上调,分别是RRM2B和DDB2,其中RRM2B上调更显著(图 1E)。结果表明,DNA损伤修复相关基因RRM2B在辐射后乳腺癌细胞中的表达显著上调。

|
| A:乳腺癌细胞辐射处理及RNA测序示意图;B:两组细胞差异基因火山图;C:GO富集分析2组细胞的差异基因;D:韦恩图分析两组细胞差异基因和DNA损伤相关基因交集;E:两组细胞差异基因和DNA损伤相关基因交集的热图 图 1 RNA测序分析辐射诱导乳腺癌基因改变 |
2.2 RRM2B在乳腺癌组织中高表达且与患者预后负相关
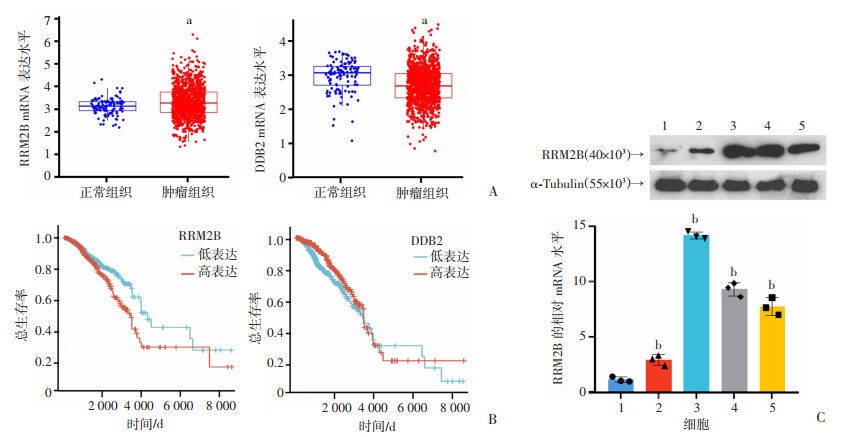
为了验证上调的RRM2B和DDB2基因在乳腺癌组织中的表达及与患者预后的关系,收集TCGA-BRCA数据库中1 231个乳腺浸润癌或正常乳腺组织资料。通过对比分析113个正常组织和1 118个肿瘤组织发现,RRM2B在肿瘤组织中的表达显著增高,而DDB2在正常组织中的表达显著增高(图 2A)。Kaplan-Meier分析显示,高表达RRM2B患者的OS显著低于低表达RRM2B患者(HR=1.45,P=0.025),而DDB2表达高低对OS的影响没有统计学意义(HR=0.75,P=0.077,图 2B)。Western blot、RT-qPCR检测结果显示,与正常乳腺细胞MCF-10A比较,4种乳腺癌细胞系(MDA-MB-231、MCF-7、ZR-75-1、BT-474)中RRM2B的蛋白水平和mRNA水平均显著升高(P<0.05,图 2C)。结果表明,RRM2B在乳腺癌组织、细胞中高表达,且RRM2B表达与乳腺癌患者的总生存期呈负相关。
|
| A:TCGA-BRCA数据库分析RRM2B和DDB2在肿瘤组织和正常组织中的表达 a: P<0.05,与正常组织比较;B:TCGA-BRCA数据库中RRM2B和DDB2对患者总生存率的影响;C:正常乳腺上皮细胞和乳腺癌细胞中RRM2B的蛋白表达和mRNA水平(n=3) 1: MCF-10A; 2: BT-474; 3: ZR-75-1; 4: MCF-7; 5: MDA-MB-231; b: P<0.05,与MCF-10A细胞比较 图 2 RRM2B和DDB2表达的乳腺癌数据库分析和细胞检测 |
2.3 敲低RRM2B增强辐射诱导的乳腺癌细胞DNA损伤
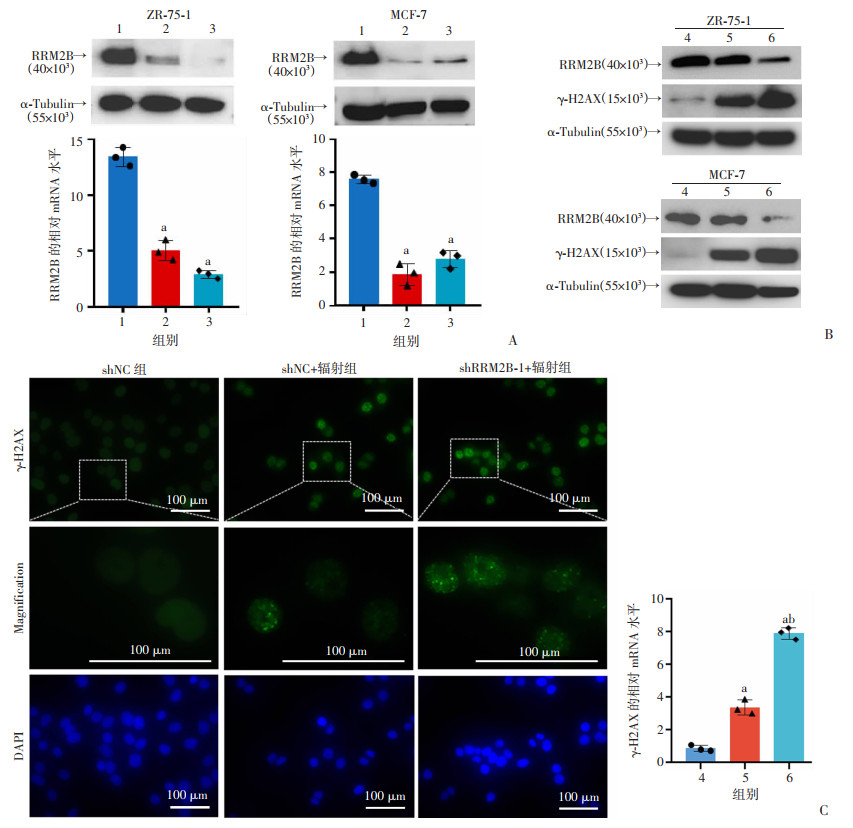
利用慢病毒构建稳定敲低RRM2B的ZR-75-1细胞和MCF-7细胞,分别提取对照组shNC和实验组shRRM2B-1/2中的蛋白和RNA鉴定敲低效果,结果显示RRM2B在两实验组细胞中显著敲低(P<0.05,图 3A),选择shRRM2B-1组细胞作为后续细胞实验组。当细胞发生DNA损伤时,H2AX的氨基酸保守序列发生磷酸化变成γ-H2AX,而γ-H2AX是细胞中感应DNA损伤最敏感的分子,与DNA损伤呈正相关。Western blot实验显示,与shNC组细胞比较,shRRM2B-1组细胞用5 Gy剂量照射后γ-H2AX明显增加(P<0.05,图 3B)。免疫荧光显示shRRM2B-1组的ZR-75-1细胞中,γ-H2AX含量(绿色荧光)也显著增加(P<0.05,图 3C)。结果表明,敲低RRM2B可促进辐射诱导的乳腺癌细胞发生DNA损伤。
|
|
1、4: shNC组; 2: shRRM2B-1组; 3: shRRM2B-2组;5:shNC+辐射组; 6:shRRM2B-1+辐射组; a:P<0.05,与shNC组比较,b:P<0.05,与shNC+辐射组比较 A:2种稳定敲低RRM2B乳腺癌细胞系中RRM2B的蛋白表达和mRNA水平比较;B:Western blot验证RRM2B下调后辐射对γ-H2AX的影响;C:荧光染色验证RRM2B下调后辐射对γ-H2AX的影响 图 3 敲低RRM2B增强辐射诱导的乳腺癌细胞DNA损伤 |
2.4 敲低RRM2B抑制辐射后乳腺癌细胞增殖
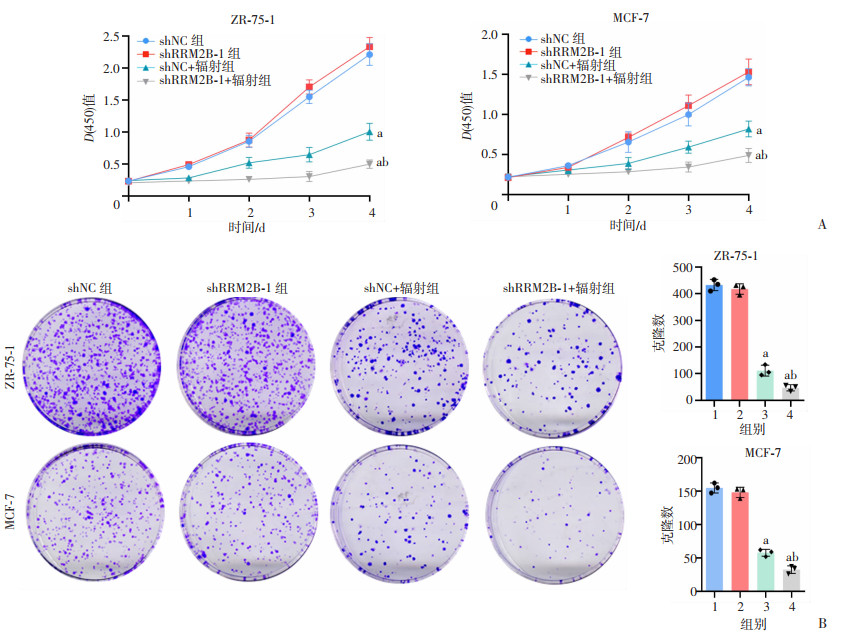
通过CCK-8和克隆形成实验验证敲低RRM2B对辐射后乳腺癌细胞增殖的影响。为了排除RRM2B敲低对正常乳腺癌细胞增殖的影响,对比shNC组和shRRM2B-1组的基础增殖情况,发现RRM2B敲低对细胞生长增殖影响较小,差异无统计学意义;而在辐射条件下,shRRM2B-1组增殖能力较shNC组明显减弱(P<0.05,图 4A)。克隆形成实验结果也显示,照射后shRRM2B-1组细胞克隆数显著减少(P<0.05,图 4B)。结果表明,敲低RRM2B可显著抑制辐射后乳腺癌细胞增殖。
|
|
a: P<0.05,与shNC组比较;b:P<0.05,与shNC+辐射组比较;1: shNC组; 2: shRRM2B-1组; 3: shNC+辐射组; 4: shRRM2B-1+辐射组 A:CCK-8实验验证敲低RRM2B后辐射对乳腺癌细胞增殖能力的影响;B:克隆形成实验验证敲低RRM2B后辐射对乳腺癌细胞增殖能力的影响 图 4 敲低RRM2B抑制辐射后乳腺癌细胞增殖 |
2.5 敲低RRM2B促进辐射诱导的乳腺癌细胞凋亡
Hoechst染色结果表明,在辐射条件下,shRRM2B-1组较shNC组细胞死亡更多(P<0.05,图 5A)。与Hoechst结果类似,凋亡结果显示,辐射后shRRM2B-1组的细胞凋亡比例更多(P<0.05,图 5B)。结果表明,敲低RRM2B后,乳腺癌细胞在辐射后细胞凋亡显著增加,即乳腺癌细胞的辐射敏感性增加。

|
|
1:shNC+辐射组;2:shRRM2B-1+辐射组;a: P<0.05,与shNC+辐射组比较 A:Hoechst/PI染色验证RRM2B下调后对辐射诱导乳腺癌细胞死亡的影响;B:流式细胞术验证RRM2B下调后对辐射诱导乳腺癌细胞凋亡的影响 图 5 敲低RRM2B促进辐射诱导的乳腺癌细胞凋亡 |
3 讨论
乳腺癌是女性高发的肿瘤之一,威胁着全球女性的健康。放射治疗是目前临床乳腺癌的重要治疗手段,较大程度地缓解了乳腺癌的进展,遏制其复发和转移的趋势[14]。然而,放疗抵抗在临床上较为常见,肿瘤细胞通过激活各种保护信号通路,减少或抵制射线造成的细胞损伤和凋亡。如何有效地避免放疗抵抗,仍是目前亟待解决的问题。
乳腺癌放疗抵抗与多种因素相关。有研究报道,通过靶向TGF-β信号传导可用于消除转移并减少放射抗性,从而提高乳腺癌患者的生存率[15-16]。ZHANG等[17]研究发现一种潜在预测乳腺癌放疗敏感的标志物FATS,其过表达可以使乳腺癌细胞中辐射诱导的细胞凋亡变得更敏感,高表达FATS的肿瘤患者更能从放射治疗中受益。ALSUBHI等[18]研究表明,CHK1和p53是乳腺癌放疗抵抗和早期局部复发的预测生物标志物,而CHK1抑制剂AZD7762对p53突变的乳腺癌细胞具有放射增敏作用。此外,乳腺癌干细胞也是放疗抵抗的重要原因[19-21]。放疗后乳腺癌干细胞中的HER2基因表达明显上调,并反过来促进放疗耐受、细胞生长和侵袭[22]。近年来,测序技术的发展使以高通量的方法来发现新的、重要的乳腺癌放疗抵抗因子成为可能,但目前国内外此类研究还较少。
RRM2B是核糖核苷酸还原酶的亚基之一,在DNA修复、细胞周期调节、线粒体DNA合成以及氧化应激抵抗等多种通路中发挥重要作用[23]。本研究中,通过RNA seq技术发现,RRM2B在辐射后的乳腺癌细胞中表达显著升高。研究表明,辐射能诱导细胞DNA单(双)链断裂或共价交联,而细胞通过启动不同的DNA修复机制应对损伤,并尽可能维持细胞周期和基因组稳定性[24]。作为DNA修复因子,RRM2B曾被报道通过多种途径激活DNA修复,促进DNA复制,提高细胞存活率[13]。这可能是RRM2B在辐射早期升高的重要原因。此外,RRM2B缺失能使辐射诱导的DNA损伤增加,细胞增殖减弱、凋亡增多,提示RRM2B在辐射增敏中扮演重要角色,阐明了RRM2B在乳腺癌放疗中的生物学特性,但具体的机制以及临床相关性有待进一步的验证。BROWN等[25]研究发现化疗能诱导肝母细胞瘤中的RRM2B显著上调,促进细胞存活和后续的复发。MOBARRA等[26]发现RRM2B的血清水平和组织表达水平可以作为结直肠癌进展和转移的可靠生物标志物。TIAN等[27]则发现RRM2B通过调节Egr-1/PTEN/Akt1来抑制肝癌细胞迁移和扩散。最近的一项研究表明,细胞缺氧时RRM2B表达增加可以维持复制并防止DNA损伤,为RRM2B在癌症中频繁地扩增提供了合理的解释。此外,缺氧模型中RRM2B的缺失显示肿瘤生长延迟、放射敏感性增加和细胞凋亡增加,凸显了RRM2B缺氧诱导的生物学重要性[28]。尽管RRM2B在不同的肿瘤中发挥着非常重要的作用,但其在乳腺癌中的作用和与辐射的关系并不明确,值得进一步深入研究和探讨。
综上所述,RRM2B是乳腺癌细胞为抵抗辐射损伤,实现自我保护机制而上调的重要因子。当RRM2B缺失后,乳腺癌细胞对辐射更敏感,能更好地发挥辐射对抑制肿瘤细胞增殖、促进细胞凋亡的作用。RRM2B是乳腺癌放射增敏的关键分子之一,有望成为解决乳腺癌放疗抵抗的潜在靶点,其具体的通路、机制及临床应用仍需进一步探索。
| [1] |
BARZAMAN K, KARAMI J, ZAREI Z, et al. Breast cancer: biology, biomarkers, and treatments[J]. Int Immunopharmacol, 2020, 84: 106535. |
| [2] |
KASHYAP D, PAL D, SHARMA R, et al. Global increase in breast cancer incidence: risk factors and preventive measures[J]. Biomed Res Int, 2022, 2022: 9605439. |
| [3] |
CRISCITIELLO C, CORTI C. Breast cancer genetics: diagnostics and treatment[J]. Genes (Basel), 2022, 13(9): 1593. |
| [4] |
HAUSSMANN J, CORRADINI S, NESTLE-KRAEMLING C, et al. Recent advances in radiotherapy of breast cancer[J]. Radiat Oncol, 2020, 15(1): 71. |
| [5] |
SHAH C, BAUER-NILSEN K, MCNULTY R H, et al. Novel radiation therapy approaches for breast cancer treatment[J]. Semin Oncol, 2020, 47(4): 209-216. |
| [6] |
VALENTE S A, SHAH C. The landmark series: adjuvant radiation therapy for breast cancer[J]. Ann Surg Oncol, 2020, 27(7): 2203-2211. |
| [7] |
ZHANG Y, HUANG Y, LI Z, et al. Exploring natural products as radioprotective agents for cancer therapy: mechanisms, challenges, and opportunities[J]. Cancers (Basel), 2023, 15(14): 3585. |
| [8] |
PETRONI G, CANTLEY L C, SANTAMBROGIO L, et al. Radiotherapy as a tool to elicit clinically actionable signalling pathways in cancer[J]. Nat Rev Clin Oncol, 2022, 19(2): 114-131. |
| [9] |
WU Y, SONG Y Q, WANG R Z, et al. Molecular mechanisms of tumor resistance to radiotherapy[J]. Mol Cancer, 2023, 22(1): 96. |
| [10] |
GILREATH C, BOERMA M, QIN Z Q, et al. The hypoxic microenvironment of breast cancer cells promotes resistance in radiation therapy[J]. Front Oncol, 2020, 10: 629422. |
| [11] |
CHO E C, KUO M L, CHENG J H, et al. RRM2B-mediated regulation of mitochondrial activity and inflammation under oxidative stress[J]. Mediators Inflamm, 2015, 2015: 287345. |
| [12] |
FUMAGALLI M, RONCHI D, BEDESCHI M F, et al. A novel RRM2B mutation associated with mitochondrial DNA depletion syndrome[J]. Mol Genet Metab Rep, 2022, 32: 100887. |
| [13] |
IQBAL W, DEMIDOVA E V, SERRAO S, et al. RRM2B is frequently amplified across multiple tumor types: implications for DNA repair, cellular survival, and cancer therapy[J]. Front Genet, 2021, 12: 628758. |
| [14] |
KAWIAK A. Molecular research and treatment of breast cancer[J]. Int J Mol Sci, 2022, 23(17): 9617. |
| [15] |
YADAV P, SHANKAR B S. Radio resistance in breast cancer cells is mediated through TGF-β signalling, hybrid epithelial-mesenchymal phenotype and cancer stem cells[J]. Biomed Pharmacother, 2019, 111: 119-130. |
| [16] |
SUN X Q, HE Z Y, GUO L, et al. ALG3 contributes to stemness and radioresistance through regulating glycosylation of TGF-β receptor Ⅱ in breast cancer[J]. J Exp Clin Cancer Res, 2021, 40(1): 149. |
| [17] |
ZHANG J, WU N, ZHANG T M, et al. The value of FATS expression in predicting sensitivity to radiotherapy in breast cancer[J]. Oncotarget, 2017, 8(24): 38491-38500. |
| [18] |
ALSUBHI N, MIDDLETON F, ABDEL-FATAH T M A, et al. Chk1 phosphorylated at serine345 is a predictor of early local recurrence and radio-resistance in breast cancer[J]. Mol Oncol, 2016, 10(2): 213-223. |
| [19] |
MAO X D, WEI X, XU T, et al. Research progress in breast cancer stem cells: characterization and future perspectives[J]. Am J Cancer Res, 2022, 12(7): 3208-3222. |
| [20] |
SHI H J, YANG Y S. Identification of inhibitory immune checkpoints and relevant regulatory pathways in breast cancer stem cells[J]. Cancer Med, 2021, 10(11): 3794-3807. |
| [21] |
HE L, WICK N, GERMANS S K, et al. The role of breast cancer stem cells in chemoresistance and metastasis in triple-negative breast cancer[J]. Cancers (Basel), 2021, 13(24): 6209. |
| [22] |
MONZEN S, TATARA Y, MARIYA Y, et al. HER2-positive breast cancer that resists therapeutic drugs and ionizing radiation releases sphingomyelin-based molecules to circulating blood serum[J]. Mol Clin Oncol, 2020, 13(6): 70. |
| [23] |
CHO E C, KUO M L, LIU X Y, et al. Tumor suppressor FOXO3 regulates ribonucleotide reductase subunit RRM2B and impacts on survival of cancer patients[J]. Oncotarget, 2014, 5(13): 4834-4844. |
| [24] |
CHATTERJEE N, WALKER G C. Mechanisms of DNA damage, repair, and mutagenesis[J]. Environ Mol Mutagen, 2017, 58(5): 235-263. |
| [25] |
BROWN A, PAN Q F, FAN L, et al. Ribonucleotide reductase subunit switching in hepatoblastoma drug response and relapse[J]. Commun Biol, 2023, 6(1): 249. |
| [26] |
MOBARRA N, GHOLAMALIZADEH H, ABDULHUSSEIN K A, et al. Serum level and tumor tissue expression of ribonucleotide-diphosphate reductase subunit M2 B: a potential biomarker for colorectal cancer[J]. Mol Biol Rep, 2022, 49(5): 3657-3663. |
| [27] |
TIAN H, GE C, LI H, et al. Ribonucleotide reductase M2B inhibits cell migration and spreading by early growth response protein 1-mediated phosphatase and tensin homolog/Akt1 pathway in hepatocellular carcinoma[J]. Hepatology, 2014, 59(4): 1459-1470. |
| [28] |
FOSKOLOU I P, JORGENSEN C, LESZCZYNSKA K B, et al. Ribonucleotide reductase requires subunit switching in hypoxia to maintain DNA replication[J]. Mol Cell, 2017, 66(2): 206-220.e9. |




