2. 401329 重庆,重庆知见生命科技有限公司;
3. 404000 重庆,重庆大学附属三峡医院放射科
2. Chongqing Zhijian Life Technology Co., Ltd, Chongqing, 401329;
3. Department of Radiology, Chongqing University Three Gorges Hospital, Chongqing, 404000, China
肺炎具有高发病率和死亡率,是全球所有年龄段死亡的主要感染性原因[1]。尽管自1990年以来肺炎死亡率大幅下降,但其对老年人的危害居高不下,据统计,2007-2017年间世界范围内70岁以上成年人肺炎感染死亡人数增加了33.6%[2]。2019年新型冠状病毒肺炎(新冠肺炎,COVID-19)给全球带来巨大的医疗负担,疫情的出现让人们再次重视肺炎这一全球性问题。细菌、病毒、真菌是肺部感染主要的病原体,不同病原体感染所致肺炎需要的治疗措施不同,因此肺部感染病原体的早期识别对指导治疗至关重要。
肺炎的诊断通常是通过临床病史及体征、病原学检查和影像学检查来确定。几乎所有病原体感染所致肺炎均可表现为发热、咳嗽、胸痛等,不具备特异性临床症状及体征。病原学检查耗时长,且受标本采集时间、方式等多因素的影响,病原体总体检出率较低且时效性差[3]。影像学检查,尤其是胸部CT,扫描速度快,可以提供与病理学相关的信息,并且可定量测量病变大小、肺部受累范围及严重程度,已成为诊断包括肺炎在内的肺部疾病的重要工具[4-5]。在肺炎CT图像征象中,病毒性肺炎典型征象为多发弥漫磨玻璃灶、间质性改变等,细菌性肺炎常表现为片状实变、充气支气管征、伴发空洞、磨玻璃影等,真菌性肺炎往往具有空洞、空气新月征、晕征等特点。然而在实际临床工作中大多数病例往往不具备上述典型征象且各种病原体感染所致肺炎影像征象多有重叠。肺炎的临床及影像表现缺乏特异性给病变的早期诊断带来挑战。
人工智能(artificial Intelligence,AI)技术的发展为肺炎的影像诊断提供了新思路和方法,其中深度学习(deep learning,DL)能自动从大规模医学图像数据集中提取隐含的疾病诊断特征,迅速成为医学图像分析的研究热点。近年来一些研究将人工智能用于胸部影像,如基于CT图像的新冠肺炎和普通肺炎的分类[6-7]、基于胸片的小儿细菌性肺炎和病毒性肺炎的分类[8-9]、基于胸部CT的肺炎型肺癌和肺炎的鉴别[10],均表现优异。然而基于胸部CT图像鉴别不同类型病原体感染所致肺炎的研究报道相对较少。因此,本研究旨在建立基于CT图像的AI预测模型,实现对不同类型病原体感染所致肺炎的早期鉴别诊断。
1 资料与方法 1.1 研究对象本研究回顾性分析了来自陆军军医大学附属第一医院(西南医院)2013-2020年的559例细菌、真菌及普通病毒(即非COVID-19病毒)肺炎患者,来自重庆大学附属三峡医院2020年的53例COVID-19患者,共纳入612例患者。本研究经陆军军医大学第一附属医院伦理委员会审批(KY2020036)。
纳入标准:①根据《中国成人社区获得性肺炎诊断和治疗指南(2016年版)》诊断的细菌性肺炎、真菌性肺炎、普通病毒性肺炎(即非COVID-19病毒性肺炎)患者以及经实验室检查确诊的COVID-19患者;②年龄≥18岁;③临床及胸部CT影像资料完整且图像有感染征象。排除标准:①图像质量差,伪影重,影响观察结果;②有严重的肺部并发症患者;③有胸部手术病史。
1.2 CT成像方案细菌、真菌及普通病毒肺炎患者来自陆军军医大学第一附属医院,COVID-19患者来自重庆大学附属三峡医院。图像数据从PACS系统中导出。胸部CT扫描范围从胸廓入口到肺底,采用德国Siemens公司的Sensation 16、Somatom Definition AS 64排螺旋CT机或Somatom Definition Flash双源螺旋CT机扫描。扫描参数:管电压100~120 kV,管电流为自动设置,螺距为1.2~1.5。重建参数:卷积核I70f,肺窗窗位-450~-600 HU,窗宽1 200~1 500 HU,层间距2 mm,层厚2 mm。本研究使用的图像均为肺窗平扫图像。
1.3 图像预处理与模型构建 1.3.1 图像预处理2名具有5年以上胸部CT影像诊断工作经验的放射科医师筛选出患者胸部CT图像中有感染征象的层面,在2名医师意见有分歧时,由第3名具有10年以上胸部CT影像诊断工作经验的放射科医师做决断。以患者为单位随机将图像按7 ∶1 ∶2的比例划分为训练集、验证集和测试集,将图像输入模型进行模型训练及验证。同时考虑到放射科医师进行阅片诊断时需要查看患者的完整CT图像层面,因此本研究将尝试图像融合方法,即将3张连续的CT图像融合制作数据集后输入模型进行模型训练及验证。
1.3.2 模型构建使用2种不同架构的模型,即卷积神经网络(convolutional neural networks,CNN)和基于自注意力机制的神经网络,分别测试这2种模型在胸部CT图像鉴别中的效果。卷积神经网络具有平移不变性和局部连接性,主要通过卷积操作和池化操作分层提取图像中的局部特征。基于自注意力机制的神经网络使用其自注意力机制和多层感知结构,通过复杂空间变换和长距离特征依赖获取全局特征表示。本研究从这2种模型架构中分别各选取具有代表性的2种网络进行建模以挑选出能完成本研究任务的最优模型,即2种卷积神经网络:Resnet_18、Efficientnet-b5,2种基于自注意力机制的神经网络:ViT、Swin-Transformer。将模型首先在ImageNet公开数据集中进行预训练得到预训练模型,采用预训练模型参数作为网络的初始化参数,以提高模型的特征提取效果并加速模型训练。使用本研究的训练集及验证集数据对预训练模型进行微调,对患者图像进行三分类(细菌、真菌、包括COVID-19在内的病毒性肺炎)及四分类(细菌、真菌、普通病毒、COVID-19肺炎)处理,经过多轮的迭代训练后从得到的模型文件中选择验证集准确率最高者作为实验用的最终模型。
在筛选出图像级的最优模型后,鉴于医师在进行阅片诊断时需要查看分析患者完整的CT图像,本研究提出通过图像融合的方法制作数据集提高模型的性能。
另一方面,与图像级的预测相比,在临床实践中患者级的预测结果更具有实际应用价值,因此本研究还进行了患者级的预测模型构建。与图像级的分类结果通过网络模型直接获得不同,患者级分类结果采用随机森林的方式获取。首先将患者的每张图像输入训练好的分类模型中,得到其输入分类器前的特征,然后将患者每张图像的特征融合输入到随机森林分类器中,得到每个患者的分类结果,如图 1所示。与简单的通过病例中所有图像类别占比最高的方式确定患者类别相比,随机森林的方法融合了患者所有的图像信息,同时能够计算图像特征重要性,提高模型分类准确率。
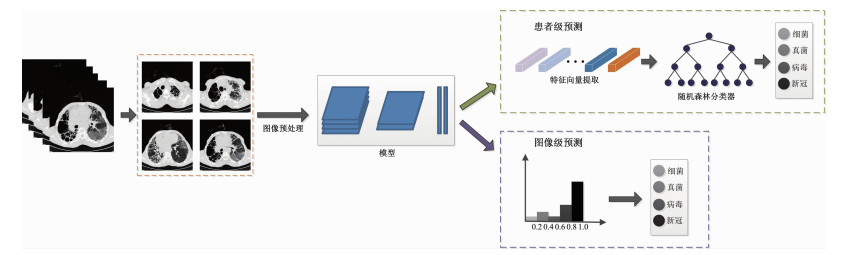
|
| 图 1 深度学习模型流程图 |
1.4 模型效能评估
模型效能评估在测试集中进行,测试集图像与训练集及验证集图像无交叉,为独立数据集,模型对测试集图像做出分类诊断,将模型在测试集中的预测结果与放射科医师阅片结果进行比较。医师均在不了解患者任何其他信息的情况下阅片做出诊断,在这个过程中,分别将测试集图像交给低年资医师(胸部CT影像诊断工作经验<10年)和高年资医师(胸部CT影像诊断工作经验≥10年)进行诊断,然后将医师的诊断结果与临床诊断结果进行比对,同时和模型预测的结果进行比较。
1.5 统计学分析采用SPSS 26.0统计软件进行分析,计数资料用例数(%)表示,组间比较采用χ2检验,P<0.05表示差异有统计学意义。采用接收者操作特征曲线下面积(area under curve,AUC)、准确率(accuracy,ACC)、精确率、召回率、特异性等评估模型效能。
2 结果 2.1 一般资料本研究共纳入612例患者,其中女性184例,男性428例,年龄(51.2±16.7)岁。在训练集、验证集和测试集中,患者性别差异无统计学意义(P=0.823>0.05)。细菌性肺炎患者225例(6 530张图像),真菌性肺炎患者187例(7 392张图像),普通病毒性肺炎患者147例(8 465张图像),COVID-19肺炎患者53例(7 936张图像)。数据集的纳入流程及在训练集、验证集和测试集中的具体划分如图 2及表 1所示。

|
| 图 2 数据的纳入流程图 |
| 数据集 | 三分类 | 四分类 | ||||||
| 病毒(含COVID-19) | 细菌 | 真菌 | 普通病毒 | COVID-19 | 细菌 | 真菌 | ||
| 训练集 | 139(12 031) | 157(4 652) | 130(5 141) | 102(6 259) | 37(5 772) | 157(4 652) | 130(5 141) | |
| 验证集 | 21(1 908) | 23(762) | 19(813) | 15(877) | 6(1 031) | 23(762) | 19(813) | |
| 测试集 | 40(2 462) | 45(1 116) | 38(1 438) | 30(1 329) | 10(1 133) | 45(1 116) | 38(1 438) | |
| 合计 | 200(16 401) | 225(6 530) | 187(7 392) | 147(8 465) | 53(7 936) | 225(6 530) | 187(7 392) | |
| 括号外数据为患者数量,括号内数据为图像数量 | ||||||||
2.2 深度学习模型效能
4种网络模型在图像水平的预测结果见表 2、图 3,表现最优的是Swin-Transformer模型。在此模型基础上使用融合图像制作数据集进行进一步测试,模型简称为Swin-Transformer_C模型,预测效能得到进一步提升,其与单张图像数据集的预测性能对比见表 3。因患者级的预测值更具有临床适用性及临床价值,使用Swin-Transformer_C模型进行患者级的预测,结果见表 4、图 4,可见模型在使用随机森林分类的患者级上的预测效能高于使用投票分类的表现。这是因为随机森林的方法融合图像信息,并且能够对图像信息进行重要性的排序,提高分类准确率。
| 模型名称 | 指标 | 四分类 | 三分类 | ||||||
| 普通病毒 | COVID-19 | 细菌 | 真菌 | 病毒 | 细菌 | 真菌 | |||
| Resnet_18 | 精确率 | 0.782 | 0.970 | 0.870 | 0.900 | 0.927 | 0.858 | 0.928 | |
| 召回率 | 0.867 | 0.912 | 0.962 | 0.778 | 0.944 | 0.953 | 0.820 | ||
| 特异性 | 0.913 | 0.992 | 0.959 | 0.965 | 0.938 | 0.955 | 0.975 | ||
| F1 | 0.822 | 0.940 | 0.913 | 0.835 | 0.936 | 0.903 | 0.871 | ||
| 准确率 | 0.873 | 0.910 | |||||||
| Efficientnet_b5 | 精确率 | 0.888 | 1.000 | 0.941 | 0.897 | 0.920 | 0.946 | 0.917 | |
| 召回率 | 0.947 | 0.914 | 0.946 | 0.898 | 0.956 | 0.948 | 0.856 | ||
| 特异性 | 0.957 | 1.000 | 0.983 | 0.958 | 0.921 | 0.984 | 0.967 | ||
| F1 | 0.917 | 0.955 | 0.943 | 0.896 | 0.938 | 0.947 | 0.885 | ||
| 准确率 | 0.925 | 0.925 | |||||||
| Vit | 精确率 | 0.859 | 0.960 | 0.772 | 0.653 | 0.901 | 0.941 | 0.731 | |
| 召回率 | 0.577 | 0.883 | 0.962 | 0.797 | 0.899 | 0.938 | 0.737 | ||
| 特异性 | 0.968 | 0.983 | 0.920 | 0.871 | 0.881 | 0.984 | 0.917 | ||
| F1 | 0.690 | 0.918 | 0.856 | 0.718 | 0.900 | 0.940 | 0.734 | ||
| 准确率 | 0.803 | 0.870 | |||||||
| Swin-Transformer | 精确率 | 0.963 | 0.999 | 0.973 | 0.856 | 0.956 | 0.905 | 0.912 | |
| 召回率 | 0.860 | 0.986 | 0.992 | 0.953 | 0.952 | 0.966 | 0.871 | ||
| 特异性 | 0.989 | 1.000 | 0.992 | 0.951 | 0.957 | 0.971 | 0.966 | ||
| F1 | 0.908 | 0.993 | 0.983 | 0.902 | 0.954 | 0.935 | 0.891 | ||
| 准确率 | 0.948 | 0.932 | |||||||
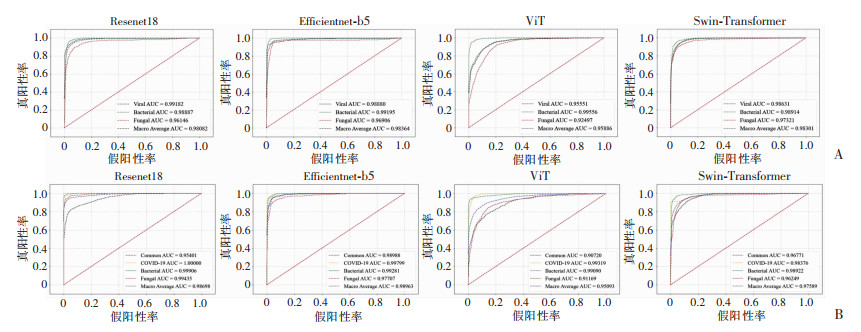
|
| A:三分类ROC曲线;B:四分类ROC曲线 图 3 不同网络模型图像级分类ROC曲线 |
| 模型 | 三分类 | 四分类 | |||
| ACC | AUC | ACC | AUC | ||
| Swin-Transformer | 0.925 | 0.983 | 0.948 | 0.976 | |
| Swin-Transformer_C | 0.931 | 0.989 | 0.952 | 0.990 | |
| Swin-Transformer_C为使用图像融合预处理方式所得模型 | |||||
| 模型 | 三分类 | 四分类 | |||
| ACC | AUC | ACC | AUC | ||
| Swin-Transformer_C+Vote | 0.963 | 0.974 | 0.951 | 0.969 | |
| Swin-Transformer_C+RF | 0.984 | 0.987 | 0.967 | 0.971 | |
| Swin-Transformer_C+Vote为使用投票方式获得的图像级结果,Swin-Transformer_C+RF为使用随机森林方式获得的患者级结果 | |||||
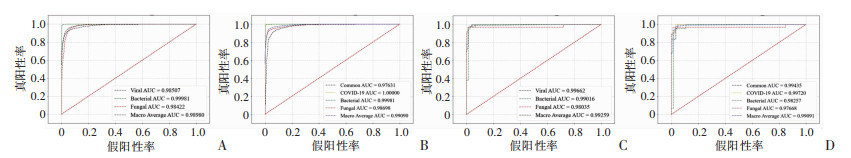
|
| A:图像级三分类ROC曲线;B:图像级四分类ROC曲线;C:患者级三分类ROC曲线;D:患者级四分类ROC曲线 图 4 Swin-Transformer_C模型图像级及患者级ROC曲线 |
2.3 深度学习模型和医师效能比较
低年资及高年资放射科医师分别对测试集图像在患者水平进行阅片,低年资医师诊断结果三分类准确率为0.504,四分类准确率为0.439;高年资医师诊断结果三分类准确率为0.545,四分类准确率为0.496,均低于深度学习模型。阅片结果与Swin-Transformer_C模型的患者级结果分类别效能对比见图 5。

|
| A:三分类结果;B:四分类结果 图 5 Swin-Transformer_C模型患者级ROC曲线及医师诊断分类结果 |
3 讨论 3.1 深度学习模型分析
卷积神经网络是深度学习的经典模型,在新冠肺炎期间,大量的研究将卷积神经网络模型用于将新冠肺炎与非新冠肺炎区分开来,并且实现了高准确率预测[11-13];Transformer网络是近年来另一个在图像中应用越来越广泛的模型,因此选取2个卷积神经网络模型(Resnet_18、Efficientnet_b5)和2个Transformer网络模型(ViT、Swin_Transformer)做对比分析。Resnet网络模型提出残差模块,大大提升了网络性能并在2015年的ImageNet视觉识别竞赛(ImageNet large scale visual recognition challenge,ILSVRC)中获得了图像分类的冠军[14],由于其高效性,在医学图像处理领域也得到了广泛应用,WU等[15]将Resnet模型用于乳腺癌筛查,KARTHIK等[16]将Resnet网络用于基于胸部X射线图像的新冠肺炎检测,均取得良好效果。Efficientnet网络利用NAS搜索技术分别从网络输入分辨率、网络深度、网络宽度三者考虑,搭建网络结构。该系列网络模型在效率和准确性上超过了之前所有的卷积神经网络模型[17]。MARQUES等[18]将Efficientnet用于新冠肺炎的自动检测;WANG等[19]将Efficientnet用于乳腺癌淋巴结转移的筛查。Transformer网络模型近年来在图像领域应用也越来越广泛,其中ViT模型结构凭借简单、可扩展性强成为了Transformer在计算机视觉领域应用中的里程碑著作[20]。CHEN等[21]将ViT模型与分割模型结合提高了医学图像分割效果,并在多个医学图像数据集中进行了试验,包括皮肤病变分割,X射线肺部分割,CT图像肺部分割,组织病理图像细胞分割等。Swin-Transformer模型提出时在多个公开数据集中获得了最佳效果,如COCO、ADE20K[22]。LEE等[23]将Swin-Transformer应用于脑部区域分割,腹部器官分割等。
Swin-Transformer模型框架流程如下。步骤一:将图片输入到Patch Partition模块中进行分块,每4×4个相邻的像素作为1个Patch,并在通道方向展平。若输入的是RGB三通道图片,每个patch包含16个像素,在3个颜色通道(R、G、B)展平,通过Patch Partition后图像由[H, W, 3]的尺寸变成了[H/4, W/4, 48]。然后再通过Linear Embeding层对每个像素的通道数据做线性变换,由48变成C,即图像由[H/4, W/4, 48]变成了[H/4, W/4, C]。步骤二:通过4个特征模块构建不同大小的特征图,除了模块1中先通过1个Linear Embeding层外,剩下3个模块均先通过1个Patch Merging层进行下采样,然后重复堆叠Swin Transformer Blocks结构。步骤三:将步骤二中提取的特征输入分类模块,得到最终输出。
从结果看,与Resnet、Efficientnet等CNN模型以及VIT模型相比,Swin-Transformer网络在针对本研究任务时特征提取能力更优,效果更好。这是因为Swin-Transformer采用多头自注意力机制使模型在浅层特征层便具有较大的感受野,而CNN模型则需要通过层层卷积逐步扩大视野。与ViT模型相比,Swin-Transformer采用了层级特征结构,梯度式降低特征尺度,增加模型多尺度特征信息,并且Swin-Transformer模型提出的Window Self-Attention模块较ViT模型中的Self-Attention模块计算复杂度大大降低,同时通过Shifted Window based Self-Attention避免了不同窗口之间无法进行信息交互的问题。最终Swin-Transformer模型在处理本研究任务中表现最为出色。
鉴于在临床工作中医师阅片时往往需要查看患者连续层面的图像,因此本研究采用的融合数据集和随机森林融合图像特征等方式使得模型的预测效果得到进一步的提升。
3.2 临床应用价值本研究建立了5种基于胸部CT图像的人工智能深度学习模型,用于对细菌性、真菌性、普通病毒性以及COVID-19肺炎的分类,均成功对肺炎进行了分类预测,其中Swin_Transformer_C模型在独立测试集上表现最优,在图像水平四分类(准确率0.953,AUC:0.991),三分类(准确率0.924,AUC:0.990);在患者水平四分类(准确率0.967,AUC:0.991),三分类(准确率0.984,AUC:0.993)。与人工阅片诊断结果比较,该模型的预测效能明显优于放射科医师。此模型是对肺炎病原体类型早期识别的一种新的尝试,通过快速、无创的方式实现肺炎类型的早期鉴别诊断,促进肺炎患者的早期目标性诊疗。
最常见的肺炎按感染病原体可分为细菌、真菌、病毒性肺炎,不同病原体感染所致肺炎所需治疗方式不同,COVID-19可归类于病毒性肺炎,尽管COVID-19已不作为需要采取强制隔离措施的传染性疾病,但其带来的影响并未消失,近期反复感染人群仍存在,远期COVID-19带来的影响并未得到确切验证。因此目前部分患者在就诊时仍会进行COVID-19检测,为满足这一需求,本研究依然进行并保留四分类建模及验证,以供在有COVID-19检测需求时进行快速预测。
在COVID-19持续大流行期间,CT检查被用作RT-PCR检测的重要且可靠的补充[24-25],体现了CT在肺部感染诊断中的重要作用。疫情迅速在全球蔓延,快速且可靠的诊断方法成为亟待解决的问题,研究者们开始探索使用人工智能来辅助影像诊断,随后使用卷积神经网络来区分COVID-19和一般肺炎[26-27]、使用深度学习及机器学习来特征性识别COVID-19[28-29]等相关研究逐渐开展。基于CT图像的AI模型结合了影像学检查和人工智能的优势,在上述特征性鉴别新冠肺炎的研究中均表现优异,基于胸部CT将多种不同类型肺炎进行分类鉴别相关的研究较少。在病例纳入方面,以往的研究纳入的病例多为实验室确诊的患者[7, 30],然而对于社区获得性肺炎,根据指南可采用临床确诊,尤其在门诊患者中,临床经验性确诊为推荐使用的方式。本研究纳入的是根据指南诊断的患者(包括临床确诊及实验室确诊患者),更接近真实世界的医疗环境,有助于模型的后续临床应用。在结果上,本研究建立的模型较之前基于CT的AI诊断识别肺炎模型准确性更高[6-7],与放射科医师相比能够更快做出诊断,且诊断的准确率高于放射科医师。在后续临床应用上,该模型可通过输入图像实现自动预测,这对于医疗资源匮乏的偏远地区影像学诊断将是重要的补充方式,另一方面,模型可以用于辅助训练低年资医师进行影像诊断。
然而,本研究有一定的局限性。其一,本研究未进行多中心验证,这是后续工作的重要部分;其二,疾病的诊断是多维度的,患者的症状、体征、实验室检查等临床信息在诊断过程中的作用不容忽视,本研究未整合临床信息建立临床-影像融合模型,在之后的研究中将完善这个部分。
综上所述,本研究建立的基于胸部CT图像的人工智能模型可有效鉴别不同病原体感染所致肺炎,能够提供准确的预测,辅助医师进行早期诊疗。
| [1] |
MURDOCH D R, HOWIE S R C. The global burden of lower respiratory infections: making progress, but we need to do better[J]. Lancet Infect Dis, 2018, 18(11): 1162-1163. |
| [2] |
ROTH G A, ABATE D, ABATE K H, et al. Global, regional, and national age-sex-specific mortality for 282 causes of death in 195 countries and territories, 1980-2017: a systematic analysis for the Global Burden of Disease Study 2017[J]. Lancet, 2018, 392(10159): 1736-1788. |
| [3] |
PERUMAL V, NARAYANAN V, RAJASEKAR S J S. Detection of COVID-19 using CXR and CT images using Transfer Learning and Haralick features[J]. Appl Intell, 2021, 51(1): 341-358. |
| [4] |
LONG C Q, XU H X, SHEN Q L, et al. Diagnosis of the coronavirus disease (COVID-19): rRT-PCR or CT?[J]. Eur J Radiol, 2020, 126: 108961. |
| [5] |
LEE E Y P, NG M Y, KHONG P L. COVID-19 pneumonia: what has CT taught us?[J]. Lancet Infect Dis, 2020, 20(4): 384-385. |
| [6] |
LI L, QIN L X, XU Z G, et al. Using artificial intelligence to detect COVID-19 and community-acquired pneumonia based on pulmonary CT: evaluation of the diagnostic accuracy[J]. Radiology, 2020, 296(2): E65-E71. |
| [7] |
ZHANG K, LIU X H, SHEN J, et al. Clinically applicable AI system for accurate diagnosis, quantitative measurements, and prognosis of COVID-19 pneumonia using computed tomography[J]. Cell, 2020, 181(6): 1423-1433. |
| [8] |
KERMANY D S, GOLDBAUM M, CAI W J, et al. Identifying medical diagnoses and treatable diseases by image-based deep learning[J]. Cell, 2018, 172(5): 1122-1131. |
| [9] |
RAJARAMAN S, CANDEMIR S, KIM I, et al. Visualization and interpretation of convolutional neural network predictions in detecting pneumonia in pediatric chest radiographs[J]. Appl Sci(Basel), 2018, 8(10): 1715. |
| [10] |
DU W, LUO X J, CHEN M. A practical deep learning model in differentiating pneumonia-type lung carcinoma from pneumonia on CT images: ResNet added with attention mechanism[J]. J Oncol, 2022, 2022: 1-9. |
| [11] |
IBRAHIM A U, OZSOZ M, SERTE S, et al. Pneumonia classification using deep learning from chest X-ray images during COVID-19[J]. Cogn Comput, 2021, 1-13. |
| [12] |
ABBAS A, ABDELSAMEA M M, GABER M M. Classification of COVID-19 in chest X-ray images using DeTraC deep convolutional neural network[J]. Appl Intell(Dordr), 2021, 51(2): 854-864. |
| [13] |
BARSHOOI A H, AMIRKHANI A. A novel data augmentation based on Gabor filter and convolutional deep learning for improving the classification of COVID-19 chest X-Ray images[J]. Biomed Signal Process Control, 2022, 72: 103326. |
| [14] |
HE K M, ZHANG X Y, REN S Q, et al. Deep residual learning for image recognition[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). June 27-30, 2016. Las Vegas: IEEE, 2016: 770-778. DOI: 10.1109/CVPR.2016.90.
|
| [15] |
WU N, PHANG J, PARK J, et al. Deep neural networks improve radiologists' performance in breast cancer screening[J]. IEEE Trans Med Imaging, 2020, 39(4): 1184-1194. |
| [16] |
KARTHIK R, MENAKA R, HARIHARAN M. Learning distinctive filters for COVID-19 detection from chest X-ray using shuffled residual CNN[J]. Appl Soft Comput, 2021, 99: 106744. |
| [17] |
TAN M, LE Q. Efficientnet: Rethinking model scaling for convolutional neural networks[EB/OL]. [2020-09-11]. http://doi.org/10.48550/arXiv.1905.11946.
|
| [18] |
MARQUES G, AGARWAL D, DE LA TORRE DÍEZ I. Automated medical diagnosis of COVID-19 through EfficientNet convolutional neural network[J]. Appl Soft Comput, 2020, 96: 106691. |
| [19] |
WANG J, LIU Q Y, XIE H T, et al. Boosted EfficientNet: detection of lymph node metastases in breast cancer using convolutional neural networks[J]. Cancers, 2021, 13(4): 661. |
| [20] |
DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16x16 words: transformers for image recognition at scale[EB/OL]. [2021-06-03]. https://doi.org/10.485501/arXiv.2010.11929.
|
| [21] |
CHEN B, LIU Y, ZHANG Z, et al. Transattunet: Multi-level attention-guided u-net with transformer for medical image segmentation[EB/OL]. [2022-07-09]. https://doi.org/10.48550/arXiv.2017.05274.
|
| [22] |
LIU Z, LIN Y T, CAO Y, et al. Swin transformer: hierarchical vision transformer using shifted windows[C]//2021 IEEE/CVF International Conference on Computer Vision (ICCV). [2021-10-17]. Montreal: IEEE, 2022: 9992-10002. DOI: 10.1109/ICCV48922.2021.00986.
|
| [23] |
LEE H H, BAO S X, HUO Y K, et al. 3D UX-net: a large kernel volumetric ConvNet modernizing hierarchical transformer for medical image segmentation[EB/OL]. [2023-05-02]. https://doi.org/10.48550./arXiv.2209.15076.
|
| [24] |
DANGIS A, GIERAERTS C, DE BRUECKER Y, et al. Accuracy and reproducibility of low-dose submillisievert chest CT for the diagnosis of COVID-19[J]. Radiol Cardiothorac Imaging, 2020, 2(2). |
| [25] |
MAJIDI H, NIKSOLAT F. Chest CT in patients suspected of COVID-19 infection: a reliable alternative for RT-PCR[J]. Am J Emerg Med, 2020, 38(12): 2730-2732. |
| [26] |
LIU C L, WANG X Y, LIU C B, et al. Differentiating novel coronavirus pneumonia from general pneumonia based on machine learning[J]. Biomed Eng Online, 2020, 19: 66. |
| [27] |
周奇浩, 张俊华, 普钟, 等. 结合深层密集聚合的新冠肺炎CT图像分类方法[J]. 计算机应用研究, 2023, 40(6): 1857-1863. ZHOU Q H, ZHANG J H, PU Z, et al. COVID-19 CT image classification method combined with deep layer dense aggregation[J]. Appl Res Comput, 2023, 40(6): 1857-1863. |
| [28] |
KASSANIA S H, KASSANIB P H, WESOLOWSKIC M J, et al. Automatic detection of coronavirus disease (COVID-19) in X-ray and CT images: a machine learning based approach[J]. Biocybern Biomed Eng, 2021, 41(3): 867-879. |
| [29] |
张淙越, 杨晓玲. 基于卷积神经网络的新冠肺炎CT图像识别系统[J]. 电脑与信息技术, 2022, 30(3): 12-14, 40. ZHANG C Y, YANG X L. COVID-19 CT image recognition system based on convolutional neural network[J]. Comput Inf Technol, 2022, 30(3): 12-14, 40. |
| [30] |
WANG G Y, LIU X H, SHEN J, et al. A deep-learning pipeline for the diagnosis and discrimination of viral, non-viral and COVID-19 pneumonia from chest X-ray images[J]. Nat Biomed Eng, 2021, 5(6): 509-521. |




