乙型肝炎病毒(hepatitis B virus, HBV)感染引起的慢性肝炎、肝硬化和肝癌是一个严重的公共卫生问题。目前我国慢性乙型肝炎患者大约有2 000万[1]。大量研究表明HBV感染中病毒基因组DNA整合到宿主基因组,通过多种机制发挥致癌作用[2-4]。病毒DNA与宿主基因组DNA进行基因整合,发生DNA序列缺失、重排、转位或扩增,改变宿主基因的功能或染色体的不稳定,促进细胞的癌变[5]。但是HBV DNA整合到宿主基因组的特征尚不清楚。
目前检测HBV DNA整合到宿主基因组的方法主要包括Alu-PCR[6-7]、反向巢式PCR(inv-PCR)[8]、高通量测序技术 [9-10]等。既往对整合肝癌细胞系中HBV DNA整合的研究主要采用传统的PCR技术,其灵敏度和特异性不能全面分析肝癌整合细胞系中HBV DNA的整合特征[11-12]。最近,基于病毒特异性靶序列捕获的二代测序技术已被用于检测低病毒载量[13]。因此,本研究采用HBV探针富集带有HBV序列的基因组片段,联合高通量测序后,建立一种新的生信分析方法策略,实现对人类肝癌整合细胞系HepG2.2.15与HepAD38进行HBV-宿主整合位点的分析,研究结果有助于深入研究HBV DNA整合与肝细胞癌(hepatocellular carcinoma, HCC)的关系,同时为该方法应用到临床HCC的诊断分析奠定基础。
1 材料与方法 1.1 主要试剂耗材人肝癌细胞株HepG2.2.15由重庆医科大学林永教授转赠,HepAD38细胞由四川大学华西医学院唐红教授转赠,并由重庆医科大学感染性疾病分子生物学教育部重点实验室长期保存;DMEM培养基及胰酶均购自Gibco公司;胎牛血清购自上海双洳生物科技有限公司;DNA提取试剂盒(QIAamp DNA mini kat)购自德国QIAGEN公司;pMDTM 19-T Vector和PCR扩增酶rTaq均购自TaKaRa公司;PCR产物纯化试剂盒购自美基生物公司;琼脂糖粉末购自北京擎科生物科技有限公司;引物合成与一代测序由上海生工生物工程股份有限公司完成。
1.2 方法 1.2.1 细胞培养与胞内基因组DNA提取HepG2.2.15与HepAD38细胞用含有1%青-链霉素、10%胎牛血清的DMEM培养基,在37 ℃、含二氧化碳浓度为5%的培养箱中培养,细胞基因组DNA提取采用QIAamp DNA mini kat试剂盒(详细操作见说明书),DNA浓度使用Nanodrop-500测定。
1.2.2 HBV DNA整合检测 1.2.2.1 Alu-PCR技术检测HBV DNA整合Alu-PCR扩增方法参考文献[14],并做一定优化,共进行3轮扩增(引物设计见表 1[6]):①第1轮扩增引物为Alu sense A5(或Alu antisense A3)、HBV引物HB1x(或HB1s、HB1c),采用单管扩增形式,每个样本做6套扩增。50 μL体系含25 μL rTaq、1 μL Alu引物、1 μL HBV引物、DNA 100 ng,加入ddH2O补足到50 μL,Alu引物与HBV引物浓度比为1 ∶10。扩增条件为94 ℃预变性2 min,每个循环包括94 ℃ 30 s、59 ℃ 30 s、70 ℃ 3 min,共10次循环,70 ℃补平延伸10 min。②使用尿嘧啶DNA糖苷酶(Uracil DNA Glycosylase, UNG)对第1轮扩增产物进行消化,每个扩增管加入1 μL UNG酶,37 ℃金属浴30 min,95 ℃ 10 min灭活酶活性。③第2轮扩增引物为Alu sense Tag5(或Alu antisense Tag3)、HBV引物HB2x(或HB2s、HB2c),Alu引物与HBV引物浓度比例为1 ∶1,各1 μL直接加入到UNG酶消化后的第1轮产物中,进行第2轮扩增,扩增条件为94 ℃预变性2 min,每个循环包括94 ℃ 30 s、65 ℃ 30 s、70 ℃ 3 min,共20次循环,采用降落PCR,每次循环退火温度降低0.5 ℃,直到55 ℃,在此退火温度上继续扩增20个循环,70 ℃补平延伸10 min。④第3轮扩增引物为Alu sense Tag5(或Alu antisense Tag3)、HBV引物HB3x(或HB3s、HB3c)。25 μL体系含rTaq 12.5 μL、引物各1 μL、第2轮产物1 μL、ddH2O 9.5 μL,扩增条件同第2轮扩增。第3轮PCR产物经过1%琼脂糖凝胶电泳确认目的条带产物并进行胶回收纯化。
| 引物名称 | 序列(5′→3′) | 注释 |
| UP5 | CAGUGCCAAGUGUUUGCUGACGCCAAAGUGCUGGGAUUA | Alu sense A5 |
| T3-515 | AUUAACCCUCACUAAAGCCUCGAUAGAUYRYRCCAYUGCAC | Alu antisense A3 |
| UP6 | CAAGTGTTTGCTGACGCCAAAG | Alu sense Tag5 |
| midT3 | TTAACCCTCACTAAAGCCTCG | Alu antisens Tag3 |
| pUTP | ACAUGAACCUUUACCCCGUUGC | HB1(HBx) |
| MM37 | TGCCAAGTGTTTGCTGACGC | HB2(HBx) |
| MM60 | CTGCCGATCCATACTGCGGAAC | HB3(HBx) |
| uPre 31 | GAGUUCUUCUUCUAGGGGACCUG | HB1(HBc) |
| MM31 | AGTGCGAATCCACACTC | HB2(HBc) |
| MM25 | GGAAGGAAAGAAGTCAGAAGG | HB3(HBc) |
| uPreS2 | ACACGGCGGUAUUUUGGGGTGGAG | HB1(HBs) |
| MM2R | CAGGCTCAGGGCATATTGACAA | HB2(HBs) |
| MD71 | YCCTGCTGGTGGCTCCAGTTC | HB3(HBs) |
| Y=(C, T); R=(C, G); U: 脱氧核苷三磷酸;带下划线的序列为Tag标签序列 | ||
1.2.2.2 TA克隆、一代测序及结果比对
纯化的胶回收产物克隆到pMDTM 19-T载体,每个样本挑选单克隆测序。测序结果采用美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)的BLAST检索以及加州大学圣克鲁兹分校数据库(University of California, Santa Cruz Database,UCSC)进行比对。
1.2.3 探针捕获和高通量测序技术检测HBV DNA整合位点提取的细胞基因组DNA使用Qubit核酸定量仪对DNA浓度进行精准定量质检,由北京艾吉泰康生物科技公司根据Illumina标准说明构建测序文库。采用针对HBV不同基因型(A-H)保守序列设计特异性捕获探针[15-16](由北京艾吉泰康生物科技公司设计合成),通过液相捕获高效、特异富集,富集后在NovaSeq 6000二代测序平台上进行PE150高通量、高深度测序。主要流程如图 1。
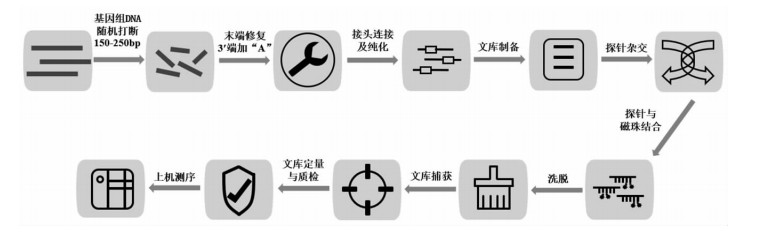
|
| 图 1 高通量捕获测序检测HBV DNA整合大致流程 |
1.2.4 数据处理与分析
二代测序(next generation sequencing, NGS)原始数据首先采用Trim momatic软件(v0.39)过滤低质量片段,按以下条件处理后得到高质量序列:①切除序列头尾碱基质量值小于15的碱基;②从序列5’至3’方向,以5 bp为窗口滑动,当窗口平均质量值小于15时,切除该窗口至序列3’末尾的序列;③去掉序列平均质量值低于15的序列;④去掉trim之后序列长度小于30 bp的序列。高质量序列采用BBAP(http://homepage.ntu.edu.tw/~youylin/)进行拼接,得到高质量unique reads,然后利用Burrows-Wheeler Aligner(BWA, v0.7.17)软件将其与Human人类(NCBI build 37,HG19)和HBV参考基因组(HE815465.1)进行比对。同时具有人类基因组序列和HBV基因组序列的unique reads即为包含HBV整合事件断点的序列,对这些断点进行分析,计算每种整合事件支持的unique reads数,并将其进行标准化,得到normalized number of support unique sequences (nnsus),进一步根据hg19基因组基因注释gtf信息,对整合事件进行注释。判断HBV整合事件的发生条件为nnsus≥1且支持该整合事件的unique reads数要大于等于3条。
1.2.5 Sanger测序验证整合事件的发生测序样本来自捕获测序后构建的文库样本,根据结果分析得到的病毒-宿主嵌合区段设计相应的PCR扩增引物,该引物能够扩增病毒整合到宿主基因组上的嵌合区段,PCR扩增产物进行Sanger测序,对HBV捕获测序结果进行验证。
2 结果 2.1 Alu-PCR技术检测HepG2.2.15与HepAD38细胞中HBV DNA整合为了探索HepG2.2.15与HepAD38细胞中HBV整合到基因组的特征,首先采用Alu-PCR技术对2个细胞系的基因组进行扩增检测,扩增产物经琼脂糖凝胶电泳后显示存在多个扩增产物(图 2)。进一步通过胶回收纯化并克隆到pMDTM19-T载体上测序,结果显示大部分目的条带为含Tag标签的非特异性序列,表明Alu-PCR难以高效地扩增出人类基因组与HBV基因组嵌合的整合序列。
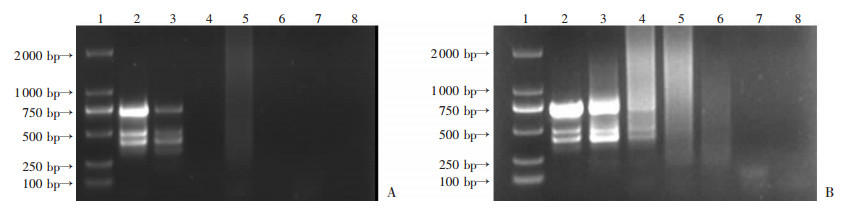
|
| A:Alu-PCR扩增HepG2.2.15细胞HBV整合 1: Marker 2 000; 2: HepG2.2.15 5x; 3: HepG2.2.15 5c; 4: HepG2.2.15 5s; 5: HepG2.2.15 3x; 6: HepG2.2.15 3c; 7: HepG2.2.15 3s; 8: 阴性对照;B:Alu-PCR扩增HepAD38细胞HBV整合 1: Marker 2 000; 2: HepAD38.5x; 3: HepAD38.5c; 4: HepAD38.5s; 5: HepAD38.3x; 6: HepAD38.3c; 7: HepAD38.3s; 8: 阴性对照 图 2 Alu-PCR技术扩增2个整合细胞系胞内基因组DNA中HBV DNA整合 |
2.2 高通量捕获测序技术检测HBV DNA整合特征
进一步利用高通量捕获测序技术,即通过针对HBV不同基因型的序列设计覆盖HBV全基因组的特异性探针,提取2个HBV整合的肝癌细胞系基因组DNA,利用探针杂交技术,捕获与富集目的序列,最后对Illumina平台测序结果进行生信分析,鉴定HBV-人类基因组的整合位点并进行特征分析。
测序结果显示,对照组HepG2细胞测得17.08 MB数据,HepG2.2.15和HepAD38细胞分别测得10.44 MB和6.63 MB的高质量序列,目标区域平均测序深度约为55倍,目标区域平均序列长度为145 bp,测序数据结果达到了本分析方法对HBV整合位点分析的最低测序深度要求(表 2)。通过捕获测序,在HepG2.2.15与HepAD38细胞基因组DNA中分别检测到12个和7个HBV整合位点。作为对照,HepG2细胞中未检测出HBV整合事件。HepG2.2.15细胞中的12个整合事件分别位于7条染色体上,包括chr1、chr2、chr7、chr9、chr17、chr19、chrX。HepAD38细胞中的7个整合事件分别位于4条染色体上,包括chr1、chr2、chr16、chrX。分析8条染色体上分布的所有整合事件所在的或其上下游基因位点(表 3),HepG2.2.15细胞中整合频率较高的基因有DPP7、TRIM56、FAM9B等,HepAD38细胞中整合频率较高基因有FNDC4、MIGA1、GPC3。
| 样本 | 原始测序数据/MB | 过滤后数据/MB | 目标序列平均读取长度/bp | 目标比对序列/MB | 目标序列捕获效率/% | 覆盖率/% | 平均深度 | 目标序列10×覆盖率/% |
| HepG2 | 17.08 | 16.73 | 140 | 0.06 | 100 | 3.44 | 0.16 | 0.29 |
| HepG2.2.15 | 10.66 | 10.44 | 145 | 9.43 | 100 | 18.89 | 55.61 | 8.90 |
| HepAD38 | 6.68 | 6.63 | 145 | 9.01 | 100 | 16.72 | 54.95 | 7.35 |
| 样本 | 断点所在染色体 | 人类染色体断点 | HBV断点 | HBV基因区域 | 支持序列数 | 整合模式 | 宿主基因注释 |
| HepG2.2.15 | Chr1 | 247911749 | 3 153 | S | 826 | Cis | AL390860.1 |
| Chr2 | 77967501 | 2 137 | C | 498 | Rev | AC012494.1 | |
| Chr2 | 77923838 | 1 027 | P | 474 | Rev | AC012494.1 | |
| Chr2 | 27775261 | 2 777 | P | 174 | Cis | C2orf16 | |
| Chr2 | 27915041 | 2 568 | P | 56 | Cis | SLC4A1AP | |
| Chr7 | 100755058 | 1 227 | P | 532 | Cis | TRIM56(Upstream) | |
| Chr7 | 51464631 | 1 116 | P | 145 | Rev | AC012441.1(Upstream) | |
| Chr9 | 140008778 | 1 638 | X | 1 006 | Rev | DPP7 | |
| Chr9 | 140011480 | 1 860 | C | 698 | Cis | DPP7 | |
| Chr17 | 76317491 | 1 346 | X | 14 | Rev | SFXN1 | |
| Chr19 | 10762420 | 1 227 | P | 16 | Rev | ILF3-DT(Downstream) | |
| ChrX | 9148657 | 2 178 | C | 522 | Rev | FAM9B | |
| HepAD38 | Chr1 | 78245320 | 1 440 | X | 45 | Rev | MIGA1 |
| Chr1 | 91834551 | 1 513 | X | 3 | Rev | HFM1 | |
| Chr2 | 27718953 | 1 495 | X | 74 | Rev | FNDC4(Upstream) | |
| Chr2 | 229723772 | 1 180 | P | 3 | Rev | PID1 | |
| Chr2 | 102365519 | 1 578 | X | 3 | Rev | AC079160.1(Upstream) | |
| Chr16 | 70423863 | 1 462 | X | 3 | Cis | TMEM233 | |
| ChrX | 132992184 | 687 | S | 22 | Rev | GPC3 |
研究结果显示, HBV整合到宿主基因组有正向插入和反向插入整合两种模式(图 3)。对19个整合事件中HBV整合断点在HBV基因组上的分布显示,大部分断点在HBV基因组nt1 000~nt2 000。部分断点分布在HBV基因组其他区域。在HepG2.2.15和HepAD38 2个整合细胞系中,整合事件在HBV基因组上的分布又有所不同。在HepG2.2.15细胞中的分布大部分在P基因区域,而在HepAD38细胞中则大部分在X基因区域,这可能和2个细胞系本身的特点有关。此外,研究结果表明,2个整合细胞株中的HBV DNA整合事件是随机发生的,未发现其对某条染色体或者某个基因有整合偏向性。
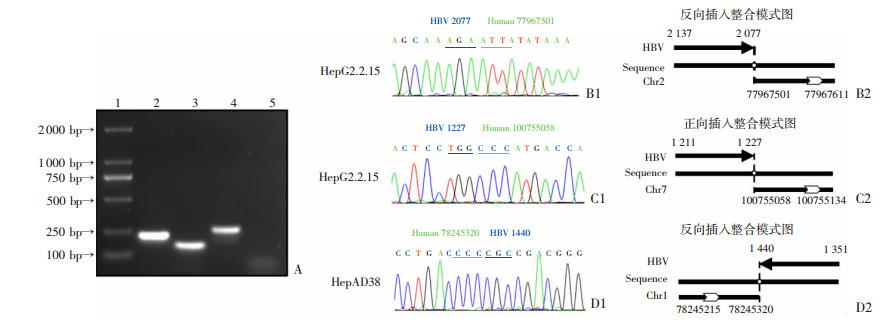
|
| A: PCR扩增产物凝胶电泳图 1: Marker 2 000;2: HepG2.2.15 chromosome 2; 3: HepG2.2.15 chromosome 7; 4: HepAD38 chromosome 1; 5: 阴性对照;B1、B2:HepG2.2.15细胞2号染色体上整合事件序列验证与整合事件模式图;C1、C2:HepG2.2.15细胞7号染色体上整合事件序列验证与整合事件模式图;D1、D2:HepAD38细胞1号染色体上整合事件序列验证与整合事件模式图 图 3 高通量捕获测序HBV整合事件反向验证 |
2.3 HepG2.2.15与HepAD38细胞系HBV DNA整合事件验证
为验证高通量捕获测序技术检测HBV整合事件的准确性,从2个细胞系中检测出的19个整合断点中,随机选取了3个整合断点(HepG2.2.15细胞系中的2号和7号染色体上的整合事件以及HepAD38细胞系中的1号染色体上的整合事件),通过设计引物(表 4) 对其进行反向验证,PCR扩增后得到目的条带(图 3A),目的条带测序后比对,结果与捕获测序技术检测的HBV整合特征相符(图 3,表 3),表明高通量捕获测序技术对HBV DNA整合事件检测的准确度较高。
| 样本 | 染色体位置 | 上游引物FP(5′→3′) | 下游引物RP(5′→3′) | 整合模式 |
| HepG2.2.15 | Chr2:100755058 | TACCTGAACCTTTACCCCGT | TGTAGAAATGGGGTCTTGCT | HBV-Human (Rev) |
| Chr7:77967501 | AAGTGTTGGGATTACAGGCG | AAACCACAAGAGTTGCCTGA | HBV-Human (Cis) | |
| HepAD38 | Chr1:78245321 | AAGTGTTGGGATTACAGGCG | AAGTGTTGGGATTACAGGCG | Human-HBV (Rev) |
3 讨论
HBV DNA整合可以发生在HBV感染的早期阶段[17],并可以持续存在于慢性感染的不同阶段。病毒DNA整合可能有两种生物学意义:①HBV在复制过程中形成的双链线性DNA(Double-stranded linear DNA, dslDNA)通过非同源重组的方式整合到宿主基因组中。由于HBsAg启动子区域位于HBV基因组中部,病毒DNA整合产生的S基因转录产物可能编码截断的HBsAg。已有研究报道,即使在肝脏内共价闭合环状DNA(Covalently closed circular DNA, cccDNA)转录沉默的情况下,HBV DNA整合也能产生HBsAg[18]。因此,在长期核苷酸类似物药物治疗的部分慢性乙型肝炎患者中,尽管cccDNA的转录活性降低或沉默,但是由于来自整合DNA编码的HBsAg持续性表达,血清中的HBsAg水平难以显著下降,这部分患者应该停用核苷酸类似物药物,而采用其他的抗病毒策略,因此,对慢性乙型肝炎患者整合DNA的检测有助于抗病毒疗效的优化。②病毒基因组的整合可能导致人类基因组功能的破坏,HBV整合导致细胞重要的基因表达异常,可能对肝细胞具有选择性生长优势并导致肝癌的发生。因此,研究HBV DNA整合到宿主基因组的特征有助于深入阐明HBV DNA整合与HCC发生、发展的关系。
HepG2.2.15和HepAD38是人肝癌细胞HepG2的HBV稳定细胞株[19-20],这2个细胞系已大量用于研究HBV的复制调控和HCC的调控机制。赵杨静等[11]曾采用Alu-PCR检测了HepAD38和HepG2.2.15中HBV DNA整合特征,在HepG2.2.15检测到来自HBx和HBc部分基因组的5个整合片段,而HepAD38细胞检测到HBx的2个整合片段,未检测出HBs的整合。本研究也曾用Alu-PCR方法进行了探索,发现Alu-PCR对HBV DNA整合到人类染色体上序列的扩增效率低,非特异性高,并且难以对HBV DNA整合事件进行定量分析。同时,由于人类基因组上的Alu位点具有偏向性,很难真实地反映样本中HBV DNA的整合。正是由于Alu-PCR检测方法的不足,本研究则利用探针捕获联合高通量测序技术和本课题组构建的生信分析流程,对HepG2.2.15和HepAD38 2个肝癌细胞系的HBV DNA整合特征进行深入研究。研究结果发现:①在2个细胞系上检测到的HBV整合事件高于既往Alu-PCR检测的结果,相对于Alu-PCR,本研究检测到HBs的整合,表明高通量捕获测序技术在检测病毒DNA的整合具有更高的灵敏度;②在HepAD38细胞中,HBx基因片段插入到宿主染色体上的序列最多,提示HBx在调控HCC的发生中具有重要的调控作用;③本研究在HepG2.2.15和HepAD38分别检测到12个和7个整合事件,分布在8个染色体上,这些整合位点在染色体上随机分布,没有偏向性整合。既往文献报道的整合基因位点与本研究发现的整合基因位点有所不同,可能是方法的不同导致。本研究分别在HepG2.2.15和HepAD38细胞中发现了以下整合频率较高的基因,HepG2.2.15细胞中:DPP7、TRIM56、FAM9B;HepAD38细胞中:FNDC4、MIGA1、GPC3。其中DPP7能够调控HepG2.2.15细胞的衰老和凋亡[21],TRIM56能够通过抑制HBV核心蛋白启动子来影响HBV病毒的复制[22],GPC3与HCC预后相关可作为HCC诊断和预后的生物标志物[23]。此外,分析结果显示HBV基因整合插入到宿主基因组上有正向插入整合和反向插入整合两种模式,两种不同的整合模式随机发生,这种不同模式的整合插入,可能会影响嵌合转录本的表达, 表明病毒通过整合到宿主基因组上,调控这些基因表达异常,促进病毒复制或癌症的发生、发展。
本研究的不足之处在于HepG2.2.15和HepAD38是人肝癌细胞HepG2的HBV稳定细胞株,检测到的HBV DNA整合事件可能与肝癌自然病程中HBV的作用存在差异,本研究将进一步利用建立的探针捕获和高通量测序技术来分析临床HBV感染的肝癌样本中病毒DNA的整合事件特征,以期找到潜在导致肝癌发生的靶点整合基因。
综上所述,本研究建立了一种探针捕获和高通量测序技术检测HBV肝癌细胞系中病毒DNA整合事件的方法,该方法能高效富集带有HBV基因组序列的片段,通过深度测序和构建的生信分析策略能反映样本整体背景下的病毒-宿主嵌合DNA片段特征。该技术有助于对临床慢性乙型肝炎患者HBV DNA整合特征的研究和阐明病毒DNA整合促进HCC发生、发展的机制研究,具有重要的应用价值。
| [1] |
COLLABORATORS P O. Global prevalence, treatment, and prevention of hepatitis B virus infection in 2016: a modelling study[J]. Lancet Gastroenterol Hepatol, 2018, 3(6): 383-403. |
| [2] |
HUANG H C, REN Z G, GAO X X, et al. Integrated analysis of microbiome and host transcriptome reveals correlations between gut microbiota and clinical outcomes in HBV-related hepatocellular carcinoma[J]. Genome Med, 2020, 12(1): 102. |
| [3] |
POLLICINO T, CAMINITI G. HBV-integration studies in the clinic: role in the natural history of infection[J]. Viruses, 2021, 13(3): 368. |
| [4] |
MASON W S, JILBERT A R, LITWIN S. Hepatitis B virus DNA integration and clonal expansion of hepatocytes in the chronically infected liver[J]. Viruses, 2021, 13(2): 210. |
| [5] |
RAMIREZ R, VAN BUUREN N, GAMELIN L, et al. Target- ed long-read sequencing reveals comprehensive architecture, burden, and transcriptional signatures from hepatitis B virus-associated integrations and translocations in hepatocellular carcinoma cell lines[J]. J Virol, 2021, 95(19): e0029921. |
| [6] |
MURAKAMI Y, SAIGO K, TAKASHIMA H, et al. Large scaled analysis of hepatitis B virus (HBV) DNA integration in HBV related hepatocellular carcinomas[J]. Gut, 2005, 54(8): 1162-1168. |
| [7] |
SAITTA C, TRIPODI G, BARBERA A, et al. Hepatitis B virus (HBV) DNA integration in patients with occult HBV infection and hepatocellular carcinoma[J]. Liver Int, 2015, 35(10): 2311-2317. |
| [8] |
MASON W S, GILL U S, LITWIN S, et al. HBV DNA integration and clonal hepatocyte expansion in chronic hepatitis B patients considered immune tolerant[J]. Gastroenterology, 2016, 151(5): 986-998.e4. |
| [9] |
FUJIMOTO A, FURUTA M, TOTOKI Y, et al. Whole-genome mutational landscape and characterization of noncoding and structural mutations in liver cancer[J]. Nat Genet, 2016, 48(5): 500-509. |
| [10] |
WANG A Q, WU L C, LIN J Z, et al. Whole-exome sequencing reveals the origin and evolution of hepato-cholangio -carcinoma[J]. Nat Commun, 2018, 9(1): 894. |
| [11] |
赵杨静, 李国力, 张恒辉, 等. 应用HBV-Alu-PCR研究肝癌细胞株中乙型肝炎病毒整合[J]. 中华实验和临床感染病杂志(电子版), 2015, 9(5): 134-138. ZHAO Y J, LI G L, ZHANG H H, et al. Identification of hepatitis B virus integration in human hepatocellular carcinoma cell lines by HBV-Alu-PCR[J]. Chin J Exp Clin Infect Dis Electron Ed, 2015, 9(5): 134-138. |
| [12] |
阮鹏, 何春萍, 黄超, 等. 改良Alu-PCR技术检测HepG2.2.15细胞HBV整合的研究[J]. 肝脏, 2022, 27(7): 785-788. RUAN P, HE C P, HUANG C, et al. Detection of hepatitis B viral DNA integration in HepG2.2. 15 cells by a modified Alu-PCR method[J]. Chin Hepatol, 2022, 27(7): 785-788. |
| [13] |
NO J S, KIM W K, CHO S, et al. Comparison of targeted next-generation sequencing for whole-genome sequencing of Hantaan orthohantavirus in Apodemus agrarius lung tissues[J]. Sci Rep, 2019, 9(1): 1-9. |
| [14] |
MINAMI M, POUSSIN K, BRÉCHOT C, et al. A novel PCR technique using Alu-specific primers to identify unknown flanking sequences from the human genome[J]. Genomics, 1995, 29(2): 403-408. |
| [15] |
ZHENG B, LIU X L, FAN R, et al. The landscape of cell-free HBV integrations and mutations in cirrhosis and hepato cellular carcinoma patients[J]. Clin Cancer Res, 2021, 27(13): 3772-3783. |
| [16] |
JANG J W, KIM J S, KIM H S, et al. Persistence of intrahepatic hepatitis B virus DNA integration in patients developing hepatocellular carcinoma after hepatitis B surface antigen seroclearance[J]. Clin Mol Hepatol, 2021, 27(1): 207-218. |
| [17] |
TU T, BUDZINSKA M, VONDRAN F, et al. Hepatitis B virus DNA integration occurs early in the viral life cycle in an in vitro infection model via sodium taurocholate cotransporting polypeptide-dependent uptake of enveloped virus particles[J]. J Virol, 2018, 92(11): e02007-17. |
| [18] |
MEIER M A, CALABRESE D, SUSLOV A, et al. Ubiquitous expression of HBsAg from integrated HBV DNA in patients with low viral load[J]. J Hepatol, 2021, 75(4): 840-847. |
| [19] |
SELLS M A, CHEN M L, ACS G. Production of hepatitis B virus particles in Hep G2 cells transfected with cloned hepatitis B virus DNA[J]. Proc Natl Acad Sci USA, 1987, 84(4): 1005-1009. |
| [20] |
LADNER S K, OTTO M J, BARKER C S, et al. Inducible expression of human hepatitis B virus (HBV) in stably transfected hepatoblastoma cells: a novel system for screening potential inhibitors of HBV replication[J]. Antimicrob Agents Chemother, 1997, 41(8): 1715-1720. |
| [21] |
HU X G, JIANG J H, NI C, et al. HBV integration-mediated cell apoptosis in HepG2.2.15[J]. J Cancer, 2019, 10(17): 4142-4150. |
| [22] |
TIAN X, DONG H J, LAI X Y, et al. TRIM56 impairs HBV infection and replication by inhibiting HBV core promoter activity[J]. Antivir Res, 2022, 207: 105406. |
| [23] |
JIANG D L, ZHANG Y S, WANG Y N, et al. Diagnostic accuracy and prognostic significance of Glypican-3 in hepatocellular carcinoma: a systematic review and meta-analysis[J]. Front Oncol, 2022, 12: 1012418. |




