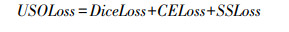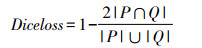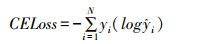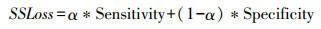2. 400044 重庆,重庆大学生物工程学院;
3. 611731 成都,电子科技大学生命科学与技术学院;
4. 400037 重庆,陆军军医大学(第三军医大学)第二附属医院生物医学信息研究与应用中心,临床医学研究中心
2. College of Bioengineering, Chongqing University, Chongqing, 400044;
3. School of Life Science and Technology, University of Electronic Science and Technology of China, Chengdu, Sichuan Province, 611731;
4. Bio-Med Informatics Research Center & Clinical Research Center, Second Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400037, China
精准的分割心脏磁共振成像(magnetic resonance imaging,MRI)可以有效提高心血管疾病评估与诊断准确性[1]。近年来,随着深度学习技术的发展,自动分割算法已经逐步取代手动分割。因此,研究人员也开始利用深度学习,实现医学图像的自动分割。例如,2017年的心脏自动诊断挑战(automated cardiac diagnosis challenge,ACDC)数据集,已被广大研究者用于构建和测试新的心脏分割方法[2],并在左心室、右心室和心肌分割中取得了较好效果。然而,ACDC数据集来源于同一临床中心,且使用相同成像设备采集了150名受试者影像。基于该数据集开发的影像分割算法,难以泛化于其他临床中心,尤其是在当患者有不同基础病,或在不同的影像设备中完成影像检查时,模型分割效果差异大,难以真正应用于临床。针对这一问题,MICCAI 2020心脏分割挑战赛M&Ms Challenge从4种不同影像设备中,采集了健康受试者以及有不同基础病患者的心脏影像,用于参赛者验证模型的泛化能力。因此,本文基于该数据集,研究多病种情况下跨影像设备的分割算法,从而验证模型分割结果的有效性与实用性。
目前,针对模型的泛化性差等问题,国内外已有相关研究致力于提升模型的泛化能力。例如,LIU等[3]使用一种具有无监督的多域自适应(unsupervised multi-domain adaptation, UMDA)和空间神经注意(spatial neural attention, SNA)结构的对称全卷积神经网络(symmetric full convolutional neural network, SFCNN),具有不需要对测试域进行任何标注的优点。ABRAHAM等[4]提出了一种混合损失Focal Tversky Loss(FTL),结合了Tversky指数和FocalLoss的优点,在维持分割精度的同时降低数据类别不平衡问题所带来的分割影响,提高模型在图像中的适用性。TAO等[5]基于短轴电影MRI图像实现了完全自动量化左心室(left ventricular,LV)功能,可实现对多供应商、多中心电影MRI数据的全自动、准确的分析。SUDRE等[6]提出了损失函数广义戴斯损失(Generalized Dice Loss, GDL),能有效解决图像类别数量不平衡的问题,提升不同物体图像的分割精度,使模型具有良好的鲁棒性。CHEN等[7]提出了一种新的无监督域自适应框架,称为协同图像和特征对齐(synergistic image and feature alignment,SIFA),以有效地将分割网络适应于未标记的目标域。尽管解决多中心数据问题的改善工作正在持续进行中,但是并未充分考虑数据不均衡问题。因此,在现有分割模型基础上设计适合医学影像分割的损失函数,能够更好地实现误差最小化,从而提升模型的预测能力和泛化能力。
因此,本研究针对来自不同疾病患者与厂商设备的心脏磁共振成像,基于敏感性、特异性以及几何相似度,构建了一种不平衡相似度优化损失函数,以增强基于深度模型在不同设备供应商或不同病种患者MRI影像分割中的通用性。
1 材料与方法 1.1 数据采集本文采用M&Ms Challenge公开的CMR心脏短轴位数据集(患者、健康受试者共320例)[8]。受试者包括:健康志愿者(normal,NOR);肥厚型心肌病(hypertrophic cardiomyopathy,HCM);扩张型心肌病(dilated cardiomyopathy,DCM)以及其他疾病患者。其他疾病患者包括:致心律失常性右室心肌病(arrhythmogenic right ventricular cardiomyopathy,AHS)、室性心动过速(ventricular tachycardia,ARV)、高血压性心脏病(hypertensive heart disease,HHD)、缺血性心脏病(ischemic heart disease,IHD)、左室心肌致密化不全(left ventricular noncompaction,LVNC)和一些罕见心脏病等。图 1为M&Ms Challenge提供的疾病类型、扫描设备等官方详细数据信息。
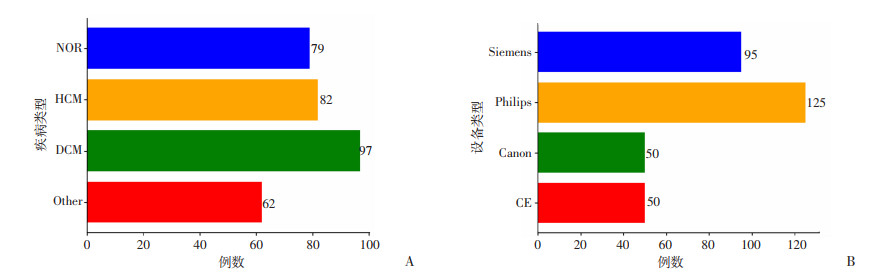
|
| A:多病种条件下影像数据分布;B:不同影像设备数据分布 图 1 多病种与不同影像设备MRI数据分组情况 |
根据临床诊断需求,CMR主要分割区域为右心室、左心室和左心肌3个区域,均为舒张末期与收缩末期。标注的主要规则有:①完全覆盖住左心室和右心室腔以及乳头肌;②不在基底部分进行心肌边界值插值;③避开肺动脉;④右心室的舒张末期必须比收缩末期表面积更大。数据分别使用西门子、飞利浦、佳能和通用电气(GE)磁共振设备进行成像(图 2)。
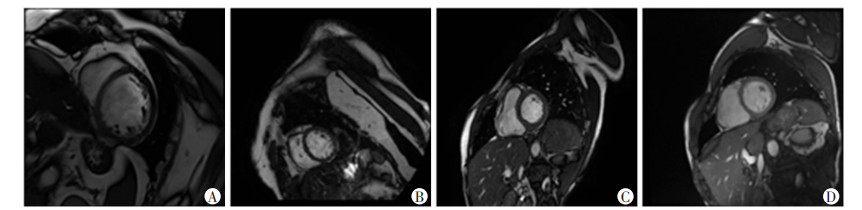
|
| A:西门子(Siemens);B:飞利浦(Philips);C:佳能(Canon);D:通用电气(GE) 图 2 4家不同厂商的心脏磁共振成像 |
1.2 分割算法 1.2.1 总体框架
为了解决数据不平衡问题并兼顾分割几何相似度,本实验利用敏感性、特异性作为损失函数的扩展基础,构建了不平衡相似度优化损失函数。该损失函数能够更好地平衡模型的分割精度和泛化能力,并对经典的UNet[9]、google提出的DeepLabV3+[10]和基于注意力机制的TransUNet[11]3种主流模型进行改进。
1.2.2 模型结构UNet:UNet网络结构,由用于捕获上下文的收缩路径和实现精确本地化的对称扩展路径组成。其主要结构分为左边压缩过程的下采样(提取特征)、右边解码过程的上采样(获取深层特征)以及跳跃连接(结合深浅层次特征)3个部分,是医学影像分割算法中最经典的模型结构之一。
DeepLabV3+:DeepLabV3+是一种语义分割架构, 在DeepLabV2和DeepLabV3基础之上进行修改,该网络结构在级联或并行中采用多孔卷积的模块,通过多个多孔速率来捕获多尺度上下文,实现在多个尺度上分割对象的效果。此外,在DeepLabV2的Atrous Spatial Pyramid Pooling模块增加了编码全局上下文的图像级特征,将底层特征与高层特征进一步融合,提升了分割边界的准确度。
TransUNet:近年来基于注意力机制的transformer深度模型,在各个任务上均取得了优异的成绩。TransUNet作为一款应用于医学的分割模型,结合了transformer与UNet的优点,重点改善了transformer过于关注全局上下文信息而导致特征分辨率低的缺点,利用CNN-Transformer的混合结构作为编码器,提取全局上下文信息,解码器对编码特征进行上采样,然后将其与高分辨率CNN特征图相结合,实现医学图像精准分割,在此过程中自注意力机制能够有效捕捉全局信息,多头注意力机制可以将其映射到多个空间,通过恢复局部空间信息来增强更精细的分割细节。
1.2.3 损失函数本文算法提出的不平衡相似度优化损失函数(USOLoss),由关注形态学相似性的戴斯损失函数(DiceLoss)、擅长于多分类的交叉熵损失函数(CELoss)以及更关注处理不平衡问题的特异性与灵敏度损失函数(SSLoss)三部分组成,如图 3所示。
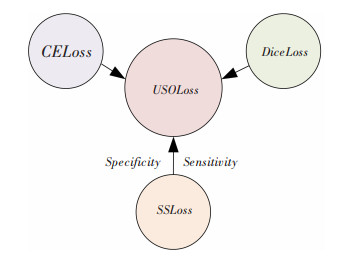
|
| 图 3 USOLoss函数结构图 |
在上述3种损失函数基础上,本文构建了一种不平衡相似度优化损失函数(USOLoss),其表达式为:
其中戴斯损失函数(DiceLoss)[12]是医学图像分割任务使用频率较高的损失函数之一,其表达式为:
其中P∩Q是2个图像的交集,P与Q分别是分割图像的真实标签与预测标签。
交叉熵损失函数(CELoss)[13]在分类任务中使用较多,由于分割也可以看作是像素级的分类任务,因此,交叉熵损失函数也可以应用到分割任务。多类别交叉熵损失函数的表达式如下:
其中yi是真实的类别标签,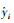
特异性与灵敏度损失函数(SSLoss)[14]该损失将灵敏度(Sensitivity)与特异性(Specificity)指标相结合,可用来处理高度不平衡数据集,侧重于关注真实阳性的事件,使用α用于平衡两者之间的权重,其表达式如下:
本研究中α=0.1。
1.3 统计学分析使用Python语言和PyCharm编程软件进行统计学分析。收集涉及的网络模型(DeepLabV3+、UNet和TransUNet)在使用USOLoss和未使用USOLoss算法前后的实验数据,采用2个常见的图像分割评价指标:戴斯相似系数(Dice similarity cofficient, DSC)和豪斯多夫距离(Hausdorff distance, HD) [15]作为衡量标准,针对连续性变量(即DSC和HD),使用配对t检验比较使用USOLoss算法前后的差异。在PyCharm软件中,利用Python scipy.stats模块中的ttest_rel函数进行计算,根据检验结果,得到每种网络模型的DSC和HD的2个指标的P值。在分析来自不同病种或设备的数据时,也进行了类似的统计分析。
2 结果为了验证模型的有效性,本文将M&Ms Challenge多中心数据集根据不同疾病和不同设备分别进行实验,并利用提出的新混合损失函数USOLoss分别改进Unet、DeepLabV3+与TransUNet,最后采用DSC与HD对模型的性能进行评价。
2.1 验证模型在不同疾病中的泛化能力将320例含标签的心脏序列,按疾病类型分为内部验证与外部验证,其中训练集含扩张型心肌病(DCM)77例,肥厚型心肌病(HCM)62例,健康受试者(NOR)59例,共198例,内部测试集分别为DCM 20例,HCM 20例,NOR 20例,外部测试集为其他疾病类型(Other)62例,疾病组数据分组设计如表 1所示。
| 疾病类型 | DCM | HCM | NOR | Other |
| 总数量 | 97 | 82 | 79 | 62 |
| 训练集 | 77 | 62 | 59 | 0 |
| 测试集 | 20 | 20 | 20 | 62 |
| 内外部验证 | 内部 | 内部 | 内部 | 外部 |
按照以上疾病分组对数据重新划分后,使用结合了USOLoss的UNet、DeepLabV3+与TransUNet进行模型训练,再使用戴斯相似系数与豪斯多夫距离进行评价,并与未使用USOLoss的模型性能进行对比,具体结果如表 2所示。
| 模型 | 戴斯相似系数DSC/%↑ | 豪斯多夫距离HD/mm↓ | |||||||
| DCM | HCM | NOR | Other | DCM | HCM | NOR | Other | ||
| UNet | 79.4±1.4 (79.2~79.5) |
77.4±1.0 (77.3~77.5) |
78.2±1.3 (78.1~78.4) |
75.9±1.3 (75.9~76.0) |
3.78±2.27 (3.55~4.02) |
4.13±3.32 (3.77~4.49) |
4.79±5.05 (4.22~5.37) |
1.70±2.41 (1.55~1.85) |
|
| USO-UNet | 80.0±1.1 (79.9~80.1) |
77.8±1.1 (77.6~77.9) |
77.9±1.5 (78.4~78.7) |
76.8±0.8 (76.8~76.9) |
3.64±1.61 (3.47~3.80) |
3.82±2.24 (3.57~4.06) |
4.48±3.69 (4.06~4.89) |
1.58±2.15 (1.45~1.71) |
|
| DeepLabV3+ | 79.1±1.7 (78.9~79.3) |
76.5±1.0 (76.4~76.6) |
77.6±1.5 (77.4~77.8) |
75.9±0.9 (75.8~75.9) |
3.88±1.89 (3.68~4.07) |
4.68±5.57 (4.08~5.29) |
4.55±5.13 (3.97~5.13) |
1.49±1.63 (1.39~1.59) |
|
| USO-DeepLabV3+ | 79.1±1.5 (78.9~79.2) |
77.0±1.0 (76.8~77.1) |
78.3±1.5 (78.1~78.5) |
76.5±0.8 (76.4~76.5) |
3.96±3.06 (3.64~4.27) |
3.83±2.23 (3.59~4.07) |
4.30±4.85 (3.75~4.85) |
1.47±1.81 (1.36~1.59) |
|
| TransUNet | 84.2±1.4 (82.7~85.7) |
78.0±1.8 (76.0~79.9) |
82.0±1.5 (80.3~83.7) |
80.1±1.8 (78.9~81.2) |
3.02±2.52 (2.75~3.28) |
3.04±3.17 (2.70~3.38) |
2.68±3.13 (2.32~3.03) |
2.61±2.24 (2.47~2.75) |
|
| USO-TransUNet | 84.5±1.5 (82.9~86.0) |
81.1±1.6 (79.3~82.8) |
83.3±1.4 (81.7~84.8) |
81.6±1.7 (80.6~82.7) |
3.05±1.04 (2.64~3.47) |
2.53±1.85 (2.33~2.73) |
2.15±1.82 (1.95~2.36) |
2.36±2.18 (2.23~ 2.49) |
|
| 内部验证采用扩张型心肌病(DCM)、肥厚型心肌病(HCM)、健康受试者(NOR)采集的数据;外部验证采用其他疾病类型(Other)采集的数据;算法在不同疾病类型的数据集上测试,使用USOLoss前后的结果经配对t检验计算,均存在统计学差异(P<0.05)。↑:DSC测试结果越接近1,相似度越高;↓:HD结果越小,边缘误差越小。 | |||||||||
从表 2结果可以看出,所提出的损失函数能够显著提升模型的分割性能,尤其对于TransUNet提升较为明显。各种模型对不同疾病类型MRI的预测效果如图 4所示。

|
| 蓝色ROI为左心室,绿色ROI为右心室,黄色ROI为左心肌 图 4 各种算法在疾病组中的预测效果图 |
2.2 验证模型在不同厂商设备的泛化能力
将320例含标签的心脏序列,按照设备类型分为内部验证与外部验证,其中训练集含飞利浦(Philips)105例、西门子(Siemens)75例和佳能(Canon)30例,共210例,内部测试集分别为飞利浦、西门子、佳能各20例,外部测试集为通用电气(GE)50例,设备组数据分组设计,如表 3所示。
| 设备类型 | Philips | Siemens | Canon | GE |
| 总数量 | 125 | 95 | 50 | 50 |
| 训练集 | 105 | 75 | 30 | 0 |
| 测试集 | 20 | 20 | 20 | 50 |
| 内外部验证 | 内部 | 内部 | 内部 | 外部 |
按照以上设备分组设计对数据重新划分后,分别进行与疾病组相同的实验步骤,在UNet、DeepLabV3+、TransUNet算法以及各自联合使用USOLoss,共6个实验模型来进行特征训练,内外部测试结果,如表 4所示。
| 模型 | 戴斯相似系数DSC/%↑ | 豪斯多夫距离HD/mm↓ | |||||||
| Philips | Siemens | Canon | GE | Philips | Siemens | Canon | GE | ||
| UNet | 76.3±2.0 (76.1~76.5) |
80.2±0.6 (80.1~80.2) |
77.5±1.8 (77.3~77.7) |
75.5±1.6 (75.4~75.7) |
4.87±6.74 (4.10~5.65) |
3.70±2.63 (3.43~3.97) |
4.87±6.03 (4.00~5.56) |
1.99±2.86 (1.79~2.19) |
|
| USO-UNet | 76.3±2.2 (76.0~76.5) |
80.7±0.5 (80.6~80.7) |
77.7±1.6 (77.7~78.0) |
75.4±1.6 (75.3~75.5) |
4.41±3.69 (3.99~4.84) |
3.66±1.79 (3.48~3.85) |
4.37±4.22 (3.91~4.82) |
1.94 ±2.75 (1.75~2.13) |
|
| DeepLabV3+ | 74.7±2.2 (74.5~75.0) |
79.7±0.6 (79.6~79.8) |
75.9±1.3 (75.8~76.0) |
74.1±1.0 (74.0~74.2) |
4.41±4.56 (3.88~4.93) |
3.99±2.13 (3.77~4.22) |
4.09±3.91 (3.66~4.51) |
1.82±2.11 (1.68~1.97) |
|
| USO-DeepLabV3+ | 75.8±6.8 (75.5~76.0) |
80.3±0.6 (80.3~80.4) |
77.4±1.5 (77.3~77.6) |
75.2±1.4 (75.1~75.3) |
4.42±2.28 (3.63~5.20) |
3.64±1.33 (3.50~3.78) |
4.28±5.39 (3.70~4.87) |
1.81±2.35 (1.64~1.97) |
|
| TransUNet | 82.2±1.7 (80.3~84.1) |
83.9±1.5 (82.5~85.5) |
82.2±1.9 (80.2~84.3) |
80.5±1.7 (79.4~81.6) |
2.02±1.76 (1.82~2.22) |
2.93±1.94 (2.73~3.13) |
1.90±1.69 (1.72~2.08) |
2.31±2.15 (2.16~2.46) |
|
| USO-TransUNet | 83.0±1.6 (81.1~84.8) |
84.4±1.4 (83.0~85.8) |
84.3±1.7 (82.4~86.1) |
81.5±1.5 (80.4~82.6) |
1.96±2.04 (1.73~2.19) |
2.92±2.28 (2.68~3.15) |
1.67±1.03 (1.56~1.79) |
2.08±1.41 (1.98~2.18) |
|
| 内部验证采用飞利浦(Philips)、西门子(Siemens)、佳能(Canon)采集的数据;外部验证采用通用电气(GE)采集的数据;算法在不同设备采集的数据集上测试,使用USOLoss前后的结果经配对t检验计算,均存在统计学差异(P<0.05)。↑:DSC测试结果越接近1,相似度越高;↓:HD结果越小,边缘误差越小。 | |||||||||
根据表 4评价指标的结果显示,本文算法在设备组的分割效果均有一定程度的提升。不同MRI扫描设备的预测效果如图 5所示。
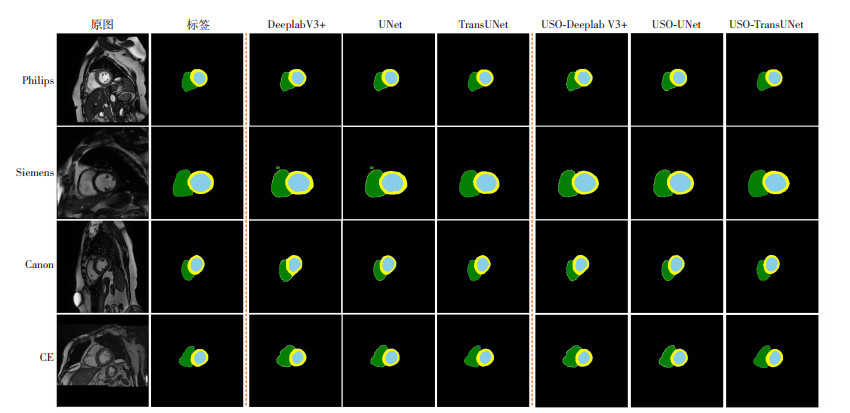
|
| 蓝色ROI为左心室,绿色ROI为右心室,黄色ROI为左心肌 图 5 本文算法在设备组中的预测效果图 |
3 讨论
心脏自动分割可用于帮助医师评估心脏病变的严重程度,了解心脏结构和功能,并帮助医师评估心肌损伤、诊断心肌病变等疾病,更好地规划治疗方案。虽然现有的交叉熵损失函数在分割任务中表现出色,但容易受到数据偏差和噪声干扰等问题的影响。针对这些问题,本研究提出了USOLoss损失函数,引入SSLoss以提高损失函数的可扩展性,其通过动态加权调整应对数据不平衡问题,有效克服了交叉熵损失函数的缺陷,提高了对模型的泛化力。
实验结果表明TransUNet分割效果整体要好于传统的CNN网络,这是因为TransUNet充分发挥了UNet与Transformer两者的优势,能通过恢复局部空间信息来增强更多的细节。采用本研究提出USOLoss后,3种算法整体获得了一定程度的提升,这是由于常见的图像分割损失函数,如DiceLoss和CELoss,没有考虑不均衡样本分布对分割结果的准确性影响。因此,受临床诊断中常用的特异性与灵敏度指标启发,本文引入SSLoss优化损失函数,进而提出了USOLoss来提升模型对图像纹理特征的辨别度,在测试未知疾病、不同设备等不同模态MRI数据时,降低错误预测的概率。
根据以上2个不同组别的实验结果展示:①本实验中TransUNet网络模型的分割效果要优于传统CNN网络模型;②本实验提出的USOLoss函数的改进对比原有的深度网络,无论是设备组还是疾病组,分割效果有着明显的提升,从而证明新混合损失函数的改进对不同模态的心脏MRI能够做出更加准确的分割;③外部验证中,其分割效果的提升同样明显,从而论证出本实验的方法可用于提升主流分割模型的泛化能力。
虽然USOLoss损失函数在面向多病种和多设备的数据集中,使模型性能和泛化能力有了一定的提升,但在后续研究需要考虑不同中心的影像差异,如分辨率、信噪比、对比度等因素,这些因素可能会影响分割模型的泛化性能。同时,不同病灶大小和形状等差异,也会对模型的泛化性造成影响,后续实验在USOLoss的基础上,可综合运用多种优化方案,联合使用跨域适应和迁移学习等新的深度强化技术。例如,LONG等[16]提出了一种新的深度自适应网络(DAN)结构,将深度卷积神经网络推广到领域自适应场景,成功地减少数据集在新的领域偏差并增强了任务特定层中的可转移性。AN等[17]基于分层分类和迁移学习的方法提出了一个新的训练框架,用于构建小数据集的疾病检测和分类的深度学习模型,他们使用多输入数据集,并采用叠加集成方法进行最终分类,进而提高精度。CUI等[18]提出了一种基于生成对抗网络(generative adversarial networks, GAN)双向跨模的无监督域自适应框架,该框架能够有效解决无标签心脏图像的分割。以上方法都将有助于构建通用性强的医学图像分割模型,进一步提高心脏分割的效果和应用价值来满足临床需要,提高医学诊断和治疗水平。
综上,本研究提出了不平衡相似度优化损失函数USOLoss,从而提升心脏自动分割模型的泛化能力。主流分割算法在使用了USOLoss后,心脏分割模型的鲁棒性均得到了有效提升,证明新损失函数的普适性。但由于数据集样本量少,为保证足够的训练数据,本研究只采用了一组外部测试集,后续工作将纳入更多外部测试数据。实验的评测指标相比于传统单中心大数据测试的结果还有所差距,其中分割结果会出现心室分割不全、标签混淆、识别不出等问题。未来将进一步基于不平衡相似度优化损失函数的深度学习网络结构进行优化,从而提高模型的泛化能力。
| [1] |
FRIEDRICH M G, SECHTEM U, SCHULZ-MENGER J, et al. Cardiovascular magnetic resonance in myocarditis: a JACC White Paper[J]. J Am Coll Cardiol, 2009, 53(17): 1475-1487. |
| [2] |
BAUMGARTNER C F, KOCH L M, POLLEFEYS M, et al. An exploration of 2D and 3D deep learning techniques for cardiac MR image segmentation[C]//International Workshop on Statistical Atlases and Computational Models of the Heart. Cham: Springer, 2018: 111-119. 10.1007/978-3-319-75541-0_12.
|
| [3] |
LIU J P, LIU H, GONG S B, et al. Automated cardiac segmentation of cross-modal medical images using unsupervised multi-domain adaptation and spatial neural attention structure[J]. Med Image Anal, 2021, 72: 102135. |
| [4] |
ABRAHAM N, KHAN N M. A novel focal tversky loss function with improved Attention U-Net for lesion segmentation[J]. IEEE International Symposium on Biomedical Imaging (ISBI), 2019, 683-687. |
| [5] |
TAO Q, YAN W J, WANG Y Y, et al. Deep learning-based method for fully automatic quantification of left ventricle function from cine MR images: a multivendor, multicenter study[J]. Radiology, 2019, 290(1): 81-88. |
| [6] |
SUDRE C H, LI W Q, VERCAUTEREN T, et al. Generalised dice overlap as a deep learning loss function for highly unbalanced segmentations[J]. Deep Learn Med Image Anal Multimodal Learn Clin Decis Support, 2017, 2017: 240-248. |
| [7] |
CHEN C, DOU Q, CHEN H, et al. Unsupervised bidirectional cross-modality adaptation via deeply synergistic image and feature alignment for medical image segmentation[J]. IEEE Trans Med Imaging, 2020, 39(7): 2494-2505. |
| [8] |
CAMPELLO V M, GKONTRA P, IZQUIERDO C, et al. Multi-centre, multi-vendor and multi-disease cardiac segmentation: the M&Ms challenge[J]. IEEE Trans Med Imaging, 2021, 40(12): 3543-3554. |
| [9] |
RONNEBERGER O, FISCHER P, BROXT. U-net: convolutional networks for biomedical image segmentation[EB/OL]. 2015: arXiv: 1505.04597. https://arxiv.org/abs/1505.04597.
|
| [10] |
CHEN L C, ZHU Y K, PAPANDREOU G, et al. Encoder-decoder with atrous separable convolution for semantic image segmentation[C]//European Conference on Computer Vision. Cham: Springer, 2018: 833-851. DOI: 10.1007/978-3-030-01234-2_49.
|
| [11] |
CHEN J N, LU Y Y, YU Q H, et al. TransUNet: transformers make strong encoders for medical image segmentation[EB/OL]. 2021: arXiv: 2102.04306. https://arxiv.org/abs/2102.04306.
|
| [12] |
MILLETARI F, NAVAB N, AHMADI S A. V-net: fully convolutional neural networks for volumetric medical image segmentation[C]//2016 Fourth International Conference on 3D Vision (3DV). IEEE, 2016: 565-571. DOI: 10.1109/3DV.2016.79.
|
| [13] |
WANG Y S, MA X J, CHEN Z Y, et al. Symmetric cross entropy for robust learning with noisy labels[C]//2019 IEEE/CVF International Conference on Computer Vision (ICCV). IEEE, 2020: 322-330. DOI: 10.1109/ICCV.2019.00041.
|
| [14] |
BROSCH T, TANG L Y, YOUNGJIN YOO, et al. Deep 3D convolutional encoder networks with shortcuts for multiscale feature integration applied to multiple sclerosis lesion segmentation[J]. IEEE Trans Med Imaging, 2016, 35(5): 1229-1239. |
| [15] |
HUTTENLOCHER D P, KLANDERMAN G A, RUCKLIDGE W J. Comparing images using the Hausdorff distance[J]. IEEE Trans Pattern Anal Mach Intell, 1993, 15(9): 850-863. |
| [16] |
LONG M S, CAO Y, WANG J M, et al. Learning transferable features with deep adaptation networks[C]//Proceedings of the 32nd International Conference on International Conference on Machine Learning. ACM, 2015: 97-105. DOI: 10.5555/3045118.3045130.
|
| [17] |
AN G Z, AKIBA M, OMODAKA K, et al. Hierarchical deep learning models using transfer learning for disease detection and classification based on small number of medical images[J]. Sci Rep, 2021, 11(1): 1-9. |
| [18] |
CUI H F, CHANG Y W, JIANG L, et al. Bidirectional cross-modality unsupervised domain adaptation using generative adversarial networks for cardiac image segmentation[J]. Comput Biol Med, 2021, 136: 104726. |