2. 400042 重庆,陆军特色医学中心神经内科
2. Department of Neurology, Army Medical Center of PLA, Chongqing, 400042, China
阿尔茨海默病(Alzheimer’s disease, AD)是一种神经退行性疾病,其特征在于含有β-淀粉样蛋白(Amyloid β, Aβ)的细胞外斑块和tau蛋白的细胞内神经原纤维缠结(neurofibrillary tangles, NFT)[1]。在65岁及以上的美国人中,AD位居第五大死因,且死亡人数呈逐年增加趋势[2]。胰淀素(islet amyloid polypeptide, IAPP)是由胰岛β细胞分泌的一种含有37-氨基酸残基的长肽[3]。IAPP沉积是2型糖尿病(diabetes mellitus type 2,T2DM)的典型病理特征,且错误折叠的IAPP可以在AD和T2D患者的大脑中聚集和沉积[4-5]。目前,IAPP与Aβ相互作用对AD的影响,仍没有达成共识。有研究表明,IAPP作为一种调节葡萄糖稳态的脑肠神经肽调节剂, 广泛存在于脑中[6],IAPP与Aβ可通过交叉播种相互作用,形成神经元外淀粉样斑块沉积,并促进Aβ聚集,造成认知功能损伤,导致AD的发生发展[7-9]。另一些研究发现,可溶性IAPP与AD的Aβ具有高亲和力,并抑制Aβ的纤维生成和细胞毒性,减少Aβ斑块生成[10-11]。动物实验研究表明,在小鼠外周注射IAPP或其非淀粉样蛋白生成类似物普兰林肽后,Aβ从大脑转移到血液增加,改善AD小鼠认知功能[12-15]。IAPP成为AD治疗和预防的潜在分子靶点。对IAPP分子功能及其与Aβ之间的相互作用对研究AD的发病机制和治疗具有重要意义。
circRNA是一类缺乏典型的5’帽和3’多聚A尾,并共价闭合的新的非编码RNA,circRNA对核酸外切酶的消化具有高度抗性并且相对稳定,通过海绵化特定的microRNA来调节基因表达,虽然它们被认为是非编码RNA,但一些circRNA已被证明具有产生肽的潜力[16]。有研究发现,circRNA在哺乳动物的大脑中具有高度代表性,在衰老、神经发育和突触活动中发挥调节作用,可能导致AD的发展[17-18]。
目前,IAPP对AD的作用尚不清楚。与AD相关的circRNA的功能了解甚少,但circRNA的稳定性和特异性表达谱表明circRNA是治疗AD的候选者,并受到广泛关注。本研究利用基因芯片技术检测IAPP腹腔注射后AD小鼠脑组织中circRNA和mRNA表达谱变化,并对筛选的差异转录本进行分析,探究IAPP腹腔注射对AD小鼠脑组织中circRNA表达谱的影响,识别和验证关键circRNA,为开发治疗AD的circRNA相关核苷酸药物提供新的分子靶点。
1 材料与方法 1.1 主要试剂和仪器IAPP购自重庆市思恩特医疗器械有限公司,Arraystar Mouse circRNA Array V2及配套试剂盒购自美国Arraystar公司,TRIzol试剂和SuperScriptTM Ⅲ RT master mix试剂盒购自美国Invitrogen公司,RNA酶试剂盒购自美国Qiagen公司,2×PCR master mix试剂盒购自美国Arraystar公司。
NanoDrop Ⓡ ND-1000分光光度仪为美国NanoDrop公司产品,Agilent Scanner G2505C、Agilent Feature Extraction (v11.0.1.1)软件、GeneSpring GX (v12.1)软件为美国Agilent公司产品,QuantStudio5 Real-time PCR System为美国Applied Biosystems公司产品。
1.2 小鼠脑组织样本脑组织样本取自腹腔注射IAPP溶液和相同剂量磷酸盐缓冲液的10月龄雄性APP/PS1转基因AD模型小鼠和C57野生型老年小鼠[C57WT,购自南京君科生物工程有限公司,动物许可证号:SCXK(苏)2020-0009],体质量20~30 g,AD小鼠和C57 WT小鼠各6只,使用随机数字表法分为4组:AD+IAPP、WT+IAPP、AD+PBS与WT+PBS,每组各3只小鼠。IAPP溶液浓度0.5 μmol/L,溶剂为PBS,剂量200 μg/kg,每日1次。腹腔注射IAPP溶液干预时间均为10周,结束后立即颈椎脱位处死,剥离完整脑组织,保存在-80 ℃冰箱中备用。实验过程中遵循实验动物伦理规定。
1.3 基因芯片测序和数据分析总RNA提取按照TRIzol试剂盒说明操作。使用Arraystar Mouse circRNA Array V2及配套试剂盒完成基因芯片杂交;NanoDrop Ⓡ ND-1000测定RNA浓度和纯度;Agilent Scanner G2505C对基因芯片进行扫描。根据扫描结果,使用Agilent Feature Extraction软件读取芯片信号强度和获得原始数据;GeneSpring GX(v12.1)软件将原始数据标准化处理后进行差异表达分析。差异表达的circRNA和mRNA筛选条件为:差异倍数的绝对值≥1.5,P值<0.05且FDR<0.5。
1.4 qRT-PCR验证circRNA的表达选取具有潜在生物学功能的6个差异表达的circRNA进行qRT-PCR检测。使用TRI Reagent抽提AD+IAPP与WT+IAPP脑组织总RNA,NanoDrop Ⓡ ND-1000测定RNA浓度和纯度。按照SuperScriptTM Ⅲ Reverse Transcriptase kit说明书,将提取的RNA逆转录为cDNA。逆转录条件为:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,40个循环。使用Primer 5.0设计引物,引物序列见表 1。将所得cDNA按照2×PCR master mix试剂盒说明进行扩增,反应条件为:95 ℃ 10 min;95 ℃ 10 s,60 ℃ 60 s,40个循环。以甘油醛-3-磷酸脱氢酶(GAPDH)为内参,由反应曲线得出阈值循环参数后,应用QuantStudioTM 5 real-time PCR仪分析荧光阈值(cyclethreshold,Ct)。使用2-ΔΔCt法计算目的基因的相对表达量。重复3次实验,结果取平均值。
| 基因名(CircRNA) | 引物序列 | 产物长度/bp |
| circRNA_35138 | 正向: 5′-TAGCGTCTGCCCTAAGAGGTA-3′ 反向: 5′-CCACTTGTGCCAATTTGTTGT-3′ |
181 |
| circRNA_38989 正向引物 |
正向: 5′-TCCTGAAGCGGACTCACCAA-3′ 反向: 5′-GGATTTGCCAATACCTGTCTCACT-3′ |
192 |
| circRNA_22593 正向引物 |
正向: 5′-ATGTGGGTGGCTTATCTGG-3′ 反向: 5′-ACTGCGGATGTTGGTTGTT-3′ |
61 |
| circRNA_36807 | 正向: 5′-TTCGGGATGAGCACAAGGT-3′ 反向: 5′-GGCTGATTGTCATGGTGGAA-3′ |
71 |
| circRNA_38835 | 正向: 5′-GCTTATCGTGCCAACTTCTGT-3′ 反向: 5′-GAGTGATGTCAAAAAGTGACCTGT-3′ |
105 |
| circRNA_45921 | 正向: 5′-ATGTGGGAGCGTGGAGATAA-3′ 反向: 5′-CATCACGGTCAGCATTCTTG-3′ |
298 |
| GAPDH | 正向: 5′-CACTGAGCAAGAGAGGCCCTAT-3′ 反向: 5′-GCAGCGAACTTTATTGATGGTATT-3′ |
144 |
1.5 GO富集分析与KEGG通路富集分析
将差异表达的mRNA导入GO数据库(http://www.geneontology.org)进行富集分析,对分子功能、生物过程和细胞成分3个领域的GO条目上差异表达基因的数目进行分析,从而获得显著富集的GO条目和预测差异表达基因的生物学功能。将差异表达的mRNA导入KEGG数据库(http://www.genome.jp/kegg)进行通路富集分析,获得差异表达的mRNA显著富集的信号通路。
1.6 CeRNA网络构建CeRNA假说表明mRNA和circRNA转录本可以通过microRNA反应元件与microRNA靶向结合来相互调控基因表达。为了寻找mRNA和circRNA与microRNA的潜在靶点,采用基于TargetScan和miRanda的miRNA靶点预测数据库进行预测。根据预测结果,通过合并mRNA和circRNA共同的靶向miRNA,利用Cytoscape构建ceRNA网络图。
1.7 统计学分析差异表达基因筛选使用R-4.1,应用Graphpad Prism 8.0统计学软件进行数据分析和统计图绘制,组间比较采用独立样本t检验,以P<0.05为差异有统计学意义。
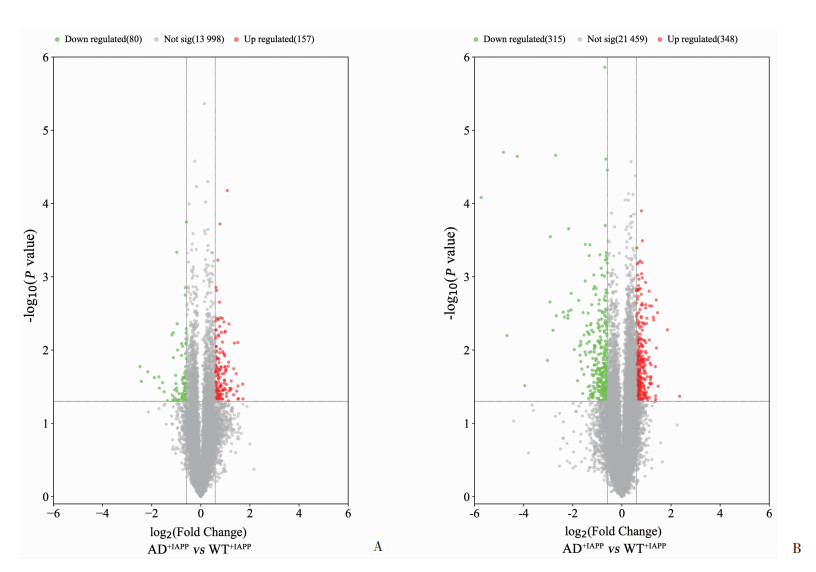
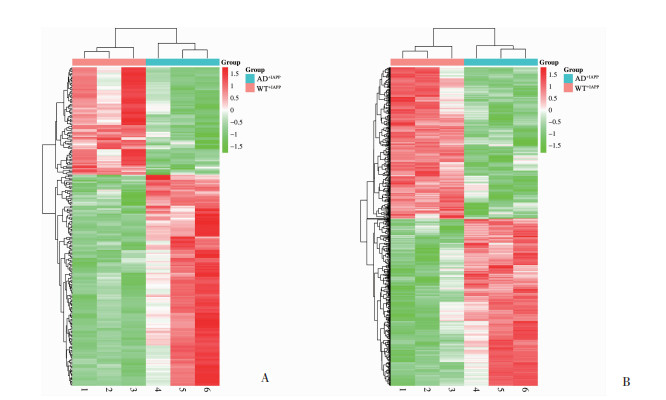
2 结果 2.1 circRNA和mRNA的差异表达分析AD+IAPP与WT+IAPP芯片测序结果显示,显著差异表达的circRNA共有237个,其中157个上调,80个下调;显著差异表达mRNA共有663个,其中348个上调,315个下调,见图 1。热图显示,AD+IAPP与WT+IAPP小鼠脑组织的circRNA和mRNA基因表达谱存在显著差异,见图 2。差异表达前10位的circRNA和mRNA见表 2。这些差异表达的circRNA和mRNA在AD+PBS与WT+PBS小鼠之间并不存在差异表达,见表 3,说明这些差异表达的circRNA和mRNA均受IAPP调节。
|
| 红色为显著上调基因; 绿色为显著下调基因; 灰色为无差异变化的基因 图 1 AD+IAPP与WT+IAPP小鼠比较差异表达circRNA(A)和mRNA(B)的火山图 |
|
| 1~3:WT+IAPP组;4~6:AD+IAPP组;纵轴为基因聚类,1行代表 1个基因;红色表示上调,绿色表示下调,颜色越亮基因相对表达量越高 图 2 AD+IAPP与WT+IAPP小鼠比较差异表达circRNA(A)和mRNA(B)的热图 |
| CircRNA | mRNA | |||||||
| 基因名称 | 差异倍数 | P值 | 上/下调 | 基因名称 | 差异倍数 | P值 | 上/下调 | |
| circRNA_39081 | 3.288 | 0.046 | 上调 | Gm42417 | 2.736 | 0.003 | 上调 | |
| circRNA_26540 | 3.262 | 0.029 | 上调 | Paip2 | 2.652 | 0.009 | 上调 | |
| circRNA_22892 | 2.881 | 0.045 | 上调 | Cdon | 2.637 | 0.002 | 上调 | |
| circRNA_39877 | 2.872 | 0.046 | 上调 | Cmtm3 | 2.457 | 0.003 | 上调 | |
| circRNA_43335 | 2.856 | 0.031 | 上调 | Erbb3 | 2.429 | 0.003 | 上调 | |
| circRNA_24245 | 5.562 | 0.016 | 下调 | Ttr | 28.286 | 0.000 | 下调 | |
| circRNA_45921 | 5.346 | 0.026 | 下调 | 1500015O10Rik | 19.173 | 0.000 | 下调 | |
| circRNA_29625 | 4.465 | 0.019 | 下调 | F5 | 6.497 | 0.000 | 下调 | |
| circRNA_007951 | 3.705 | 0.023 | 下调 | Aldh1a2 | 5.236 | 0.003 | 下调 | |
| circRNA_19469 | 3.269 | 0.023 | 下调 | Tmem212 | 5.066 | 0.004 | 下调 | |
| CircRNA | mRNA | |||||||
| 基因名称 | 差异倍数 | P值 | 上/下调 | 基因名称 | 差异倍数 | P值 | 上/下调 | |
| circRNA_39081 | 1.558 | 0.262 | 无差异 | Gm42417 | 1.054 | 0.755 | 无差异 | |
| circRNA_26540 | 1.651 | 0.080 | 无差异 | Paip2 | 2.017 | 0.120 | 无差异 | |
| circRNA_22892 | 1.348 | 0.166 | 无差异 | Cdon | 1.055 | 0.445 | 无差异 | |
| circRNA_39877 | 1.352 | 0.086 | 无差异 | Cmtm3 | 1.073 | 0.745 | 无差异 | |
| circRNA_43335 | 1.439 | 0.083 | 无差异 | Erbb3 | 1.167 | 0.444 | 无差异 | |
| circRNA_24245 | 3.270 | 0.179 | 无差异 | Ttr | 5.045 | 0.169 | 无差异 | |
| circRNA_45921 | 3.634 | 0.194 | 无差异 | 1500015O10Rik | 4.355 | 0.147 | 无差异 | |
| circRNA_29625 | 2.566 | 0.235 | 无差异 | F5 | 3.456 | 0.146 | 无差异 | |
| circRNA_007951 | 2.241 | 0.141 | 无差异 | Aldh1a2 | 2.526 | 0.143 | 无差异 | |
| circRNA_19469 | 2.273 | 0.125 | 无差异 | Tmem212 | 1.734 | 0.240 | 无差异 | |
2.2 qRT-PCR验证circRNA的表达
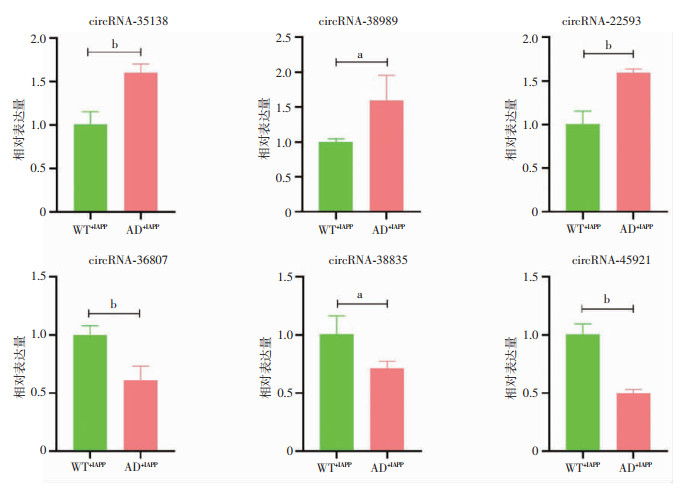
为了验证芯片结果的可靠性,挑选具有潜在生物学功能的6个差异表达的circRNA进行qRT-PCR验证,结果显示,AD+IAPP与WT+IAPP相比,circRNA-35138、circRNA-38989、circRNA-22593表达显著上调(P<0.05),circRNA-36807、circRNA-38835、circRNA-45921表达显著下调(P<0.05),见图 3。qRT-PCR检测结果与基因芯片结果一致,提示circRNA基因芯片测序结果可靠。
|
|
a: P<0.05, b: P<0.01 AD为阿尔茨海默病鼠;WT为野生型老年鼠;IAPP为胰淀素 图 3 AD+IAPP与WT+IAPP小鼠脑组织目的基因相对表达量 |
2.3 差异表达mRNA的GO和KEGG通路富集分析
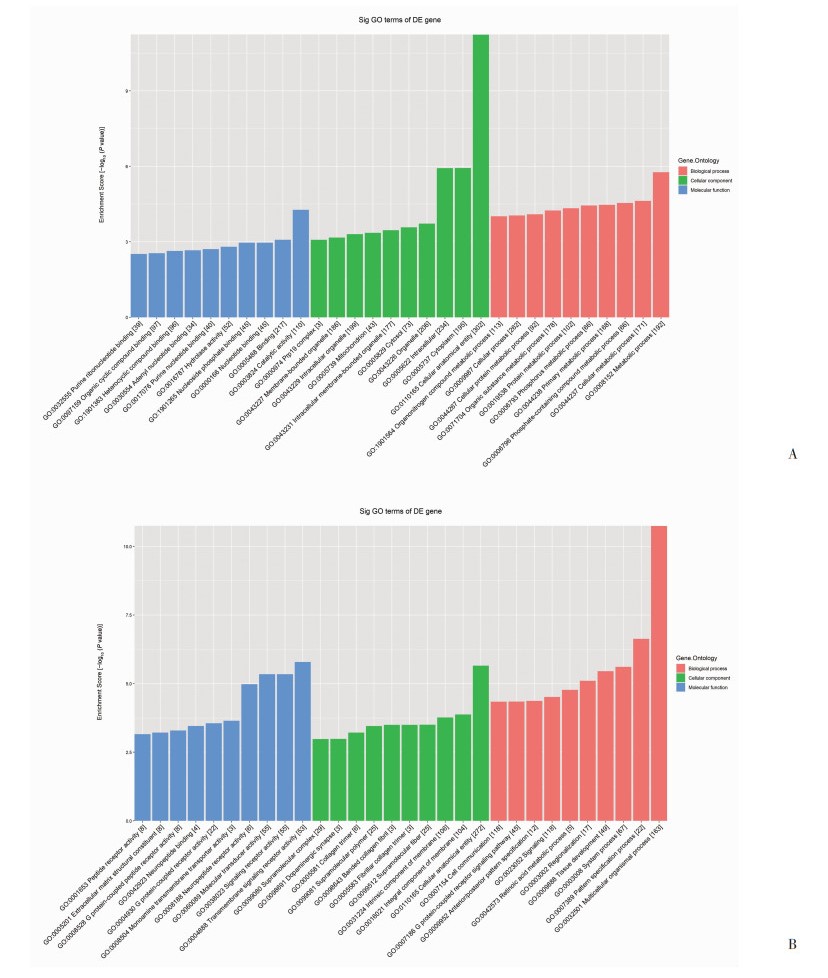
GO富集分析显示:663个差异表达的mRNA被富集到1 019个GO条目,其中348个上调mRNA被富集到525个GO条目。315个下调的mRNA被富集到494个GO条目,差异均有统计学意义(P<0.05)。差异表达上调的mRNA在分子功能领域主要富集于核苷磷酸结合、催化剂活性等;在细胞组分领域主要富集于细胞器、细胞质等;在生物过程领域主要富集于含磷酸盐化合物代谢过程、初级代谢过程等。差异表达下调的mRNA在分子功能领域主要富集于跨膜信号受体活性、分子传导活性等;在细胞组分领域主要富集于膜固有成分、超分子纤维等;在生物过程领域主要富集于组织发育、系统进程等。差异表达mRNA在分子功能、细胞成分、生物过程3个领域富集评分前10的GO条目见图 4。
|
| A:显著上调的差异表达mRNA的GO条目;B:显著下调的差异表达mRNA的GO条目 图 4 AD+IAPP与WT+IAPP小鼠相比差异表达的mRNA在分子功能、细胞成分和生物学过程3个领域的GO分析 |
KEGG富集分析显示: 663个差异表达的mRNA被富集到16个通路,其中348个上调的mRNA被富集到8个通路,315个下调的mRNA被富集到8个通路,差异均有统计学意义(P<0.05)。差异表达上调的mRNA主要富集于胰岛素分泌、氧化磷酸化、帕金森病等信号通路;差异表达下调的mRNA主要富集于神经活性配体-受体相互作用、钙信号通路、酪氨酸代谢、苯丙氨酸代谢等信号通路。KEGG富集分析评分前10位的通路见图 5。
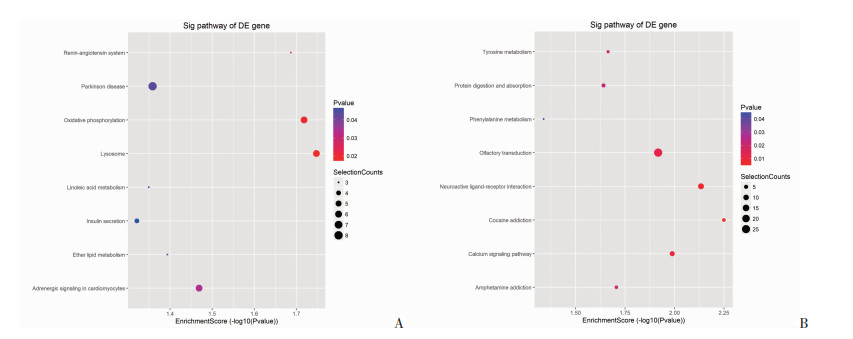
|
| A:显著上调的差异表达mRNA的富集通路; B: 显著下调的差异表达mRNA的富集通路 图 5 AD+IAPP与WT+IAPP小鼠相比差异表达的mRNA的KEGG分析 |
2.4 CeRNA分析
AD+IAPP与WT+IAPP小鼠相比,circRNA_45921显著下调,qRT-PCR验证结果与芯片结果相同。因此,选择circRNA_45921作为miRNA靶点构建ceRNA网络, 见图 6。CeRNA网络分析表明,circRNA_45921与miR-34c-5p靶向结合具有较高的预测评分。miR-34c-5p与真核细胞延伸因子2激酶(eukaryotic elongation factor 2 kinase,Eef2k)靶向结合具有较高的预测评分。合并circRNA_45921和Eef2k的共同靶向miRNA,获得了circRNA_45921的ceRNA网络circRNA_45921/miR-34c-5p/Eef2k调控网络。
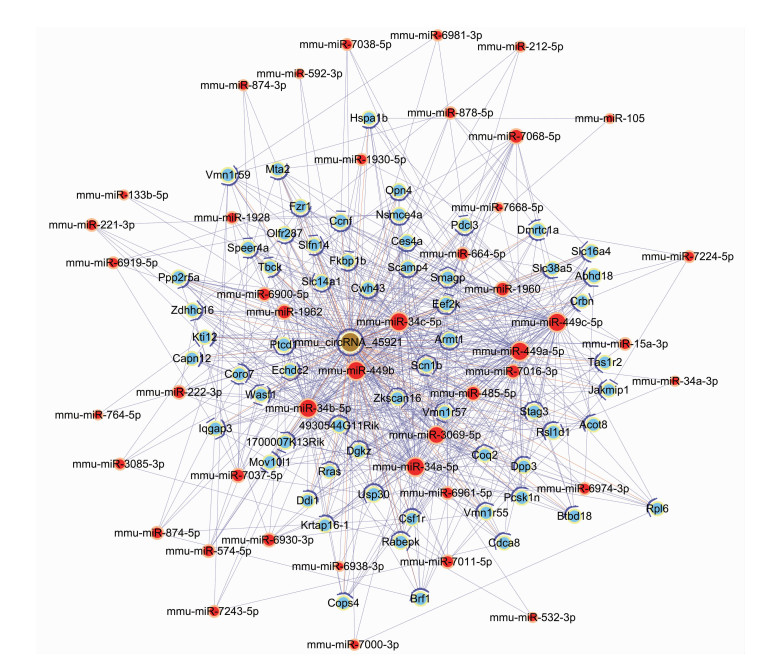
|
| 棕色表示 circRNA;红色表示 microRNA;蓝色表示 mRNA 图 6 circRNA_45921的ceRNA分析 |
3 讨论
在生理条件下,IAPP与胰岛素协同从胰岛β细胞释放,共同调节糖代谢[19]。在AD或T2DM小鼠,IAPP可因聚集和沉积而失去功能,形成有毒的低聚物和淀粉样纤维[20]。研究发现,IAPP可与Aβ相互作用共同沉积在脑内,加重AD病理[7-8]。本研究采用基因芯片技术,AD+IAPP与WT+IAPP测序结果比较,共有663个差异mRNA,包括上调的Paip2、Cdon和下调的Ttr。多聚(A)结合蛋白相互作用蛋白2(Paip2)是一种翻译抑制蛋白,有研究发现抗Paip2自身抗体的产生与ApoE4有关,APOE是迟发型阿尔兹海默症(AD)的主要风险因子,Paip2可能与脑内炎性病理相关。抗Paip2自身抗体升高与MCI和AD的风险显著相关,认为其可能是AD的潜在诊断生物标志物[21-22]。癌基因下调的细胞黏附分子相关蛋白(Cdon)是一种凋亡诱导蛋白,其下调对于促进SH-SY5Y神经细胞存活是必需的[23]。在体试验表明,Cdon可能与TNF-α、Il-1β相互作用调节神经炎症。Cdon敲除小鼠的血脑屏障紧密性导致白细胞浸润减少,小胶质细胞和星形胶质细胞活化减少以及神经元存活增加[24]。转甲状腺素(Ttr)是血浆和脑脊液中甲状腺素和视黄醇的载体蛋白,已被证明可以与Aβ结合。不仅如此,Ttr还被认为可以防止Aβ沉积[25]。ZISKIN等[26]通过磷酸化tau免疫染色发现,在皮层区域,Ttr蛋白沉积的下方观察到磷酸化tau聚集体,这表明Ttr和tau病理之间存在潜在联系。腹腔注射IAPP使得Paip2和Cdon的表达上调,Ttr的表达下调,可能导致Aβ的沉积增加、Tau蛋白磷酸化和神经炎症相关分子表达增加。提示腹腔注射IAPP可能通过Paip2、Cdon和Ttr调节AD的发生和发展[27]。一项与本研究类似的实验结果显示,腹腔注射IAPP降低了大脑皮质和海马Aβ的沉积,增加脑脊液Aβ水平。同时,腹腔注射IAPP改善了转基因AD鼠的认知功能,而对野生型老年鼠没有影响[13]。本研究为IAPP的分子机制提供了新的见解,IAPP通过Paip2、Cdon或Ttr调节Aβ、tau病理和脑内炎性病理,有待进一步研究。
AD+IAPP与WT+IAPP比较,显著差异表达的circRNA共有237个。包括上调的circRNA_39081和下调的circRNA_45921等。相关研究表明,circRNA_39081的亲本基因Sparcl1在人和小鼠海马中均存在circRNA转录本(如hsa_circ_0127250, mmu_circ_0011950)。circRNA_45921的亲本基因Phka2在人前额叶、颞叶、间脑存在circRNA转录本(如hsa_circ_0140034)[28]。本研究通过circRNA芯片测序小鼠脑组织存在Sparcl1和Phka2的circRNA转录本。研究表明,circPhka2在人脑内是一种环状且稳定的转录物,主要位于细胞质中。circPhka2可以靶向结合miR-574-5p,上调SOD2和降低Bax表达,促进细胞增殖,抑制凋亡和氧化应激[29]。并且miR-574-5p在3′UTR具有与BACE1(β-site amyloid precursor protein cleaving enzyme 1,β-secretase)结合位点。BACE1不仅促进Aβ的沉积,而且增加的BACE1活性可能影响正常的突触功能,导致认知障碍[30]。AD小鼠腹腔注射IAPP使circRNA_45921表达下调,IAPP可能通过circRNA_45921/miR-574-5p/BACE1轴调控Aβ沉积和AD的进展。CircPAIP2是一种保留内含子的circRNA,它上调记忆相关的亲代基因Paip2[31]。IAPP腹腔注射可能通过相关circRNA上调Paip2、Cdon和下调Ttr的表达,调节AD的发展。同时,circRNA_39081和circRNA_45921也可能是潜在的AD诊断分子标志物和治疗靶点。
本研究GO和KEGG富集分析结果表明,IAPP可能通过细胞器、胰岛素分泌、氧化磷酸化、帕金森病等途径参与调节AD的发生发展。相关研究发现,IAPP可使细胞膜过度弯曲而破坏膜结构,导致AD和帕金森病的发生[32]。IAPP和胰岛素是由胰岛β细胞共包装和协同分泌,胰岛素抵抗可能导致AD病理,IAPP破坏囊泡功能,与阿尔茨海默病和帕金森病的发病机制有关[33-34]。IAPP诱导tau蛋白磷酸化,在体外和体内增强tau蛋白的神经元毒性,导致小鼠突触功能障碍和记忆缺陷[35]。本研究为IAPP参与AD调节的作用机制研究提供了新的方向,IAPP可能通过具有调节胰岛素分泌、帕金森病或氧化磷酸化作用的基因(如COX6c)改变AD的病理表现。
CeRNA结果表明,circRNA_45921/miR-34c-5p/Eef2k调控网络显示较高预测评分。已有研究表明,miR-34c-5p的过度表达显著降低了微管相关tau蛋白(microtubule-associated protein tau,MAPT)的表达[36]。Eef2k诱导Eef2过度磷酸化,导致蛋白质合成受到抑制。Eef2k减少改善了记忆缺陷和海马长时程增强损伤,而不改变Aβ病理[37]。这提示circRNA_45921/miR-34c-5p/Eef2k调控网络可能在IAPP调节AD发生发展过程中发挥作用,也为IAPP作用的分子机制提供新的思路。
本研究取材自小鼠脑组织进行芯片测序,没有进行细胞水平更加深入的研究,以验证所确定的circRNA功能及其与IAPP之间的作用关系;结果显示Ttr下调,相关研究表明Ttr可能是治疗AD的靶点[38],本研究没有对IAPP功能进行进一步验证。
综上所述,本研究结果表明AD+IAPP与WT+IAPP相比,两组小鼠脑组织中circRNA和mRNA的表达谱均发生改变。差异表达的circRNA如circRNA_45921,可能通过circRNA_45921/miR-574-5p/BACE1轴调节Aβ沉积和AD的进展。差异表达的mRNA如Paip2、Cdon和Ttr等可能导致Aβ的沉积增加、Tau蛋白磷酸化和神经炎症相关分子表达增加。差异表达的mRNA主要通过胰岛素分泌、氧化磷酸化、神经活性配体-受体相互作用等通路参与调节AD的发生发展。circRNA_45921/miR-34c-5p/Eef2k调控网络可能在IAPP调节AD的发生发展中起作用。
利益冲突声明 所有作者声明不存在利益冲突
作者贡献声明 侯明亮:实验操作、论文撰写;马琳秋、李金平、黄洁、廖旗荣、杨红岩:数据整理;李小雄、马琳秋:统计学分析;周华东:研究指导、论文修改、经费支持
| [1] |
KNOPMAN D S, AMIEVA H, PETERSEN R C, et al. Alzheimer disease[J]. Nat Rev Dis Primers, 2021, 7: 33. DOI:10.1038/s41572-021-00269-y |
| [2] |
2022 Alzheimer's disease facts and figures[J]. Alzheimers Dement, 2022, 18(4): 700-789. DOI: 10.1002/alz.12638.
|
| [3] |
WESTERMARK P, ANDERSSON A, WESTERMARK G T. Islet amyloid polypeptide, islet amyloid, and diabetes mellitus[J]. Physiol Rev, 2011, 91(3): 795-826. DOI:10.1152/physrev.00042.2009 |
| [4] |
ZHANG Y, SONG W H. Islet amyloid polypeptide: another key molecule in Alzheimer's pathogenesis?[J]. Prog Neurobiol, 2017, 153: 100-120. DOI:10.1016/j.pneurobio.2017.03.001 |
| [5] |
MORENO-GONZALEZ I, EDWARDS Ⅲ G, SALVADORES N, et al. Molecular interaction between type 2 diabetes and Alzheimer's disease through cross-seeding of protein misfolding[J]. Mol Psychiatry, 2017, 22(9): 1327-1334. DOI:10.1038/mp.2016.230 |
| [6] |
KAPURNIOTU A. Enlightening amyloid fibrils linked to type 2 diabetes and cross-interactions with Aβ[J]. Nat Struct Mol Biol, 2020, 27(11): 1006-1008. DOI:10.1038/s41594-020-00523-z |
| [7] |
HU R D, ZHANG M Z, CHEN H, et al. Cross-seeding interaction between β-amyloid and human islet amyloid polypeptide[J]. ACS Chem Neurosci, 2015, 6(10): 1759-1768. DOI:10.1021/acschemneuro.5b00192 |
| [8] |
BHARADWAJ P, SOLOMON T, SAHOO B R, et al. Amylin and beta amyloid proteins interact to form amorphous hetero complexes with enhanced toxicity in neuronal cells[J]. Sci Rep, 2020, 10: 10356. DOI:10.1038/s41598-020-66602-9 |
| [9] |
JACKSON K, BARISONE G A, DIAZ E, et al. Amylin deposition in the brain: a second amyloid in Alzheimer disease?[J]. Ann Neurol, 2013, 74(4): 517-526. DOI:10.1002/ana.23956 |
| [10] |
YAN L M, VELKOVA A, TATAREK-NOSSOL M, et al. IAPP mimic blocks Abeta cytotoxic self-assembly: cross-suppression of amyloid toxicity of Abeta and IAPP suggests a molecular link between Alzheimer's disease and type Ⅱ diabetes[J]. Angew Chem Int Ed Engl, 2007, 46(8): 1246-1252. DOI:10.1002/anie.200604056.[PubMed |
| [11] |
YAN L M, VELKOVA A, TATAREK-NOSSOL M, et al. Selectively N-methylated soluble IAPP mimics as potent IAPP receptor agonists and nanomolar inhibitors of cytotoxic self-assembly of both IAPP and Aβ40[J]. Angewandte Chemie Int Ed, 2013, 52(39): 10378-10383. DOI:10.1002/anie.201302840 |
| [12] |
AFTABIZADEH M, TATAREK-NOSSOL M, ANDREETTO E, et al. Blocking inflammasome activation caused by β-amyloid peptide (aβ) and islet amyloid polypeptide (IAPP) through an IAPP mimic[J]. ACS Chem Neurosci, 2019, 10(8): 3703-3717. DOI:10.1021/acschemneuro.9b00260 |
| [13] |
ZHU H, WANG X, WALLACK M, et al. Intraperitoneal injection of the pancreatic peptide amylin potently reduces behavioral impairment and brain amyloid pathology in murine models of Alzheimer's disease[J]. Mol Psychiatry, 2015, 20(2): 252-262. DOI:10.1038/mp.2014.17 |
| [14] |
ADLER B L, YARCHOAN M, HWANG H M, et al. Neuroprotective effects of the amylin analogue pramlintide on Alzheimer's disease pathogenesis and cognition[J]. Neurobiol Aging, 2014, 35(4): 793-801. DOI:10.1016/j.neurobiolaging.2013.10.076 |
| [15] |
SCHULTZ N, JANELIDZE S, BYMAN E, et al. Levels of islet amyloid polypeptide in cerebrospinal fluid and plasma from patients with Alzheimer's disease[J]. PLoS One, 2019, 14(6): e0218561. DOI:10.1371/journal.pone.0218561 |
| [16] |
IDDA M L, MUNK R, ABDELMOHSEN K, et al. Noncoding RNAs in Alzheimer's disease[J]. Wiley Interdiscip Rev RNA, 2018, 9(2): e1463. DOI:10.1002/wrna.1463 |
| [17] |
SONG C H, ZHANG Y F, HUANG W Y, et al. Circular RNA Cwc27 contributes to Alzheimer's disease pathogenesis by repressing Pur-α activity[J]. Cell Death Differ, 2022, 29(2): 393-406. DOI:10.1038/s41418-021-00865-1 |
| [18] |
MO D D, LI X P, RAABE C A, et al. Circular RNA encoded amyloid beta peptides-a novel putative player in alzheimer's disease[J]. Cells, 2020, 9(10): 2196. DOI:10.3390/cells9102196 |
| [19] |
BHAGAT V, VERCHERE C B. A small molecule improves diabetes in mice expressing human islet amyloid polypeptide[J]. Islets, 2023, 15(1): 12-15. DOI:10.1080/19382014.2022.2163829 |
| [20] |
BOCCARDI V, MURASECCO I, MECOCCI P. Diabetes drugs in the fight against Alzheimer's disease[J]. Ageing Res Rev, 2019, 54: 100936. DOI:10.1016/j.arr.2019.100936 |
| [21] |
YOSHIKAWA T, WU J F, OTSUKA M, et al. ROCK inhibition enhances microRNA function by promoting deadenylation of targeted mRNAs via increasing PAIP2 expression[J]. Nucleic Acids Res, 2015, 43(15): 7577-7589. DOI:10.1093/nar/gkv728 |
| [22] |
SHIM S M, KOH Y H, KIM J H, et al. Author Correction: a combination of multiple autoantibodies is associated with the risk of Alzheimer's disease and cognitive impairment[J]. Sci Rep, 2022, 12: 2328. DOI:10.1038/s41598-021-04556-2 |
| [23] |
ULUCA B, LEKTEMUR ESEN C, SARITAS ERDOGAN S, et al. NFI transcriptionally represses CDON and is required for SH-SY5Y cell survival[J]. Biochim Biophys Acta Gene Regul Mech, 2022, 1865(2): 194798. DOI:10.1016/j.bbagrm.2022.194798 |
| [24] |
CHAPOULY C, HOLLIER PL, GUIMBAL S, et al. Desert hedgehog-driven endothelium integrity is enhanced by Gas1 (growth arrest-specific 1) but negatively regulated by Cdon (cell adhesion molecule-related/downregulated by oncogenes)[J]. Arterioscler Thromb Vasc Biol, 2020, 40(12): e336-e349. DOI:10.1161/ATVBAHA.120.314441 |
| [25] |
SOUSA J C, CARDOSO I, MARQUES F, et al. Transthyretin and Alzheimer's disease: where in the brain?[J]. Neurobiol Aging, 2007, 28(5): 713-718. DOI:10.1016/j.neurobiolaging.2006.03.015 |
| [26] |
ZISKIN J L, GREICIUS M D, ZHU W, et al. Neuropathologic analysis of Tyr69His TTR variant meningovascular amyloidosis with dementia[J]. Acta Neuropathol Commun, 2015, 3: 43. DOI:10.1186/s40478-015-0216-0 |
| [27] |
张国新, 彭琴玉, 郭笑迪, 等. 胰岛淀粉样多肽在阿尔茨海默病发病机制中的作用研究进展[J]. 卒中与神经疾病, 2022, 29(3): 291-295, 299. ZHANG G X, PENG Q Y, GUO X D, et al. Research progress on the role of islet amyloid polypeptide in the pathogenesis of Alzheimer’s disease[J]. Stroke Nerv Dis, 2022, 29(3): 291-295, 299. DOI:10.3969/j.issn.1007-0478.2022.03.019 |
| [28] |
RYBAK-WOLF A, STOTTMEISTER C, GLAŽAR P, et al. Circular RNAs in the mammalian brain are highly abundant, conserved, and dynamically expressed[J]. Mol Cell, 2015, 58(5): 870-885. DOI:10.1016/j.molcel.2015.03.027 |
| [29] |
YANG X B, LI X Y, ZHONG C H, et al. Circular RNA circPHKA2 relieves OGD-induced human brain microvascular endothelial cell injuries through competitively binding miR-574-5p to modulate SOD2[J]. Oxid Med Cell Longev, 2021, 2021: 3823122. DOI:10.1155/2021/3823122 |
| [30] |
KU T, LI B, GAO R, et al. NF-κB-regulated microRNA-574-5p underlies synaptic and cognitive impairment in response to atmospheric PM2.5 aspiration[J]. Part Fibre Toxicol, 2017, 14(1): 34. DOI:10.1186/s12989-017-0215-3 |
| [31] |
LU D, XU A D. Mini review: circular RNAs as potential clinical biomarkers for disorders in the central nervous system[J]. Front Genet, 2016, 7: 53. DOI:10.3389/fgene.2016.00053 |
| [32] |
BRENDER J R, SALAMEKH S, RAMAMOORTHY A. Membrane disruption and early events in the aggregation of the diabetes related peptide IAPP from a molecular perspective[J]. Acc Chem Res, 2012, 45(3): 454-462. DOI:10.1021/ar200189b |
| [33] |
PRUZIN J J, NELSON P T, ABNER E L, et al. Review: Relationship of type 2 diabetes to human brain pathology[J]. Neuropathol Appl Neurobiol, 2018, 44(4): 347-362. DOI:10.1111/nan.12476 |
| [34] |
ANGUIANO M, NOWAK R J, LANSBURY P T Jr. Protofibrillar islet amyloid polypeptide permeabilizes synthetic vesicles by a pore-like mechanism that may be relevant to type Ⅱ diabetes[J]. Biochemistry, 2002, 41(38): 11338-11343. DOI:10.1021/bi020314u |
| [35] |
ZHANG G, MENG L, WANG Z, et al. Islet amyloid polypeptide cross-seeds tau and drives the neurofibrillary pathology in Alzheimer's disease[J]. Mol Neurodegener, 2022, 17(1): 12. DOI:10.1186/s13024-022-00518-y |
| [36] |
WU H, HUANG M, LU M J, et al. Regulation of microtubule-associated protein tau (MAPT) by miR-34c-5p determines the chemosensitivity of gastric cancer to paclitaxel[J]. Cancer Chemother Pharmacol, 2013, 71(5): 1159-1171. DOI:10.1007/s00280-013-2108-y |
| [37] |
BECKELMAN B C, YANG W Z, KASICA N P, et al. Genetic reduction of eEF2 kinase alleviates pathophysiology in Alzheimer's disease model mice[J]. J Clin Investig, 2019, 129(2): 820-833. DOI:10.1172/jci122954 |
| [38] |
ANKARCRONA M, WINBLAD B, MONTEIRO C, et al. Current and future treatment of amyloid diseases[J]. J Intern Med, 2016, 280(2): 177-202. DOI:10.1111/joim.12506 |




