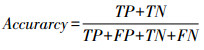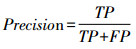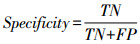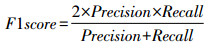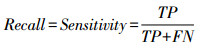2. 400038 重庆,陆军军医大学(第三军医大学)生物医学工程与影像医学系数字医学教研室;
3. 400037 重庆,陆军军医大学(第三军医大学)第二附属医院泌尿外科;
4. 030013 太原,山西省肿瘤医院普外科
2. Department of Digital Medicine, Faculty of Biomedical Engineering and Imaging Medicine, Army Medical University (Third Military Medical University), Chongqing, 400038;
3. Department of Urology, Second Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400037;
4. Department of General Surgery, Shanxi Cancer Hospital, Taiyuan, Shanxi Province, 030013, China
前列腺癌(prostate cancer, PCA)是全球男性癌症死亡率第二的疾病,成年人比例增加和人口老龄化为主要原因[1]。预计到2023年,其发病率还将翻一番[2]。前列腺癌的临床治疗选择取决于TNM分期。根治性前列腺切除术是治愈局限性前列腺癌(T1、T2期)最有效的方法之一,以内分泌治疗为主的治疗方式主要应用于T3、T4期及转移性前列腺癌[3]。术前明确区分T1和T2期病变与T3和T4期病变对PCA的治疗和预后具有重要意义[4]。多参数前列腺MR成像(multiple parameter magnetic resonance imaging, mpMRI)是目前检测、定位和诊断最准确的成像方式[5]。前列腺T2WI成像可以显示准确的前列腺分带解剖结构,为PCA的TNM分期诊断提供比CT更重要的信息[6]。由于低年资医师阅片经验不足常导致前列腺癌的漏诊率、误诊率较高,导致前列腺癌患者的预后效果相差较大[7]。与临床医师的人工阅片相比,深度学习可以从MRI影像中自动提取更多可分离的特征,大大提高诊断准确率,同时,也可大大减轻临床医师的工作量[8]。
近年来,随着人工智能在医学领域的发展,很多学者尝试将各种神经网络应用于前列腺癌的医学图像研究[9-12]。CAI等[13]提出一种基于Swin-Transformer (SwinT)的弱监督学习方法(multiple instance learning network based on SwinT,MIST)应用于H&E病理组织切片,实现了精准度为0.784,AUC为0.933的最佳分类性能;WU等[14]发现在预测胶质瘤IDH状态变化方面,SwinT网络可以得到平均AUC最高为0.965(内部测试)和0.842(外部测试)的分类结果;KHOSRAVI等[15]开发了一种计算方法以区分前列腺癌和前列腺良性病变,最高AUC可达0.89。LIU等[16]通过开发一种Textured-深度学习模型来区分临床有意义的前列腺癌和临床无意义的前列腺癌(AUC为0.85)。虽然大多模型在前列腺分带、癌灶、周围结构的分割和分类任务上已达到了高年资放射科医师的水平,但研究多采用单一模型,缺乏模型间的比较,前列腺癌智能T分期诊断的准确性和分类效能仍有待提高[17]。
本研究将T≤T2c期的前列腺癌患者定义为低中危组,T≥T3a期患者定义为高危组。我们拟将Dense-Net、Res-Net、SwinT及Vision-Transformer (ViT)多种深度学习网络模型用于前列腺癌智能T分期诊断,以筛选出最优分类模型,为下一步前列腺癌T分期的智能评估提供依据,有望为临床决策的选择提供支撑,减轻医师工作量。
1 资料与方法 1.1 临床资料回顾性收集陆军军医大学第二附属医院2020年1月至2022年1月和山西省肿瘤医院2018年4月至2020年6月共200例前列腺患者MRI图像。依据纳入标准:①经过病理证实的前列腺癌患者;②扫描规范的T2WI/fT2WI序列;③完整的临床T分期报告。排除标准:①接受过内分泌治疗(n=10);②存在明显伪影或图像质量差(n=22);③接受过尿管插管术或前列腺根治术(n=16)。最终152例前列腺癌患者的T2WI或fT2WI图像纳入研究。研究经医院伦理委员会审批(2022-研第321-01和2021051)。
1.2 数据集的构建将152位患者按7 ∶1 ∶2比例简单随机划分为训练集(n=107)、验证集(n=15)、测试集(n=30)[18]。不同医院的数据集分布如表 1所示。根据临床T分期诊断报告及患者治疗方式的选择,本研究将T≤T2c期者定义为低中危组,T≥T3a期者定义为高危患者。
| 组别 | 训练集 | 验证集 | 测试集 | 总计 |
| 低中危组(T≤T2c) | ||||
| A医院 | 12 | 2 | 4 | 18 |
| B医院 | 50 | 6 | 12 | 68 |
| 高危组(T≥T3a) | ||||
| A医院 | 6 | 2 | 4 | 12 |
| B医院 | 39 | 5 | 10 | 54 |
| 总计 | 107 | 15 | 30 | 152 |
| A院:山西省肿瘤医院;B院:陆军军医大学第二附属医院 | ||||
1.3 深度模型的训练和验证 1.3.1 实验设备
本研究操作系统为Windows系统,硬件部分:CPU:AMD A8-5550M APU with Radeon(tm) HD Graphics、GPU:NVIDIA TITAN X(Pascal) 12G;软件部分:ITK-Snap、MicroDicom Viewer、Python3.7.1、Pytorch+ Cuda:1.8.0+cu111。集成开发环境:Pytroch 1.7,Torchvision 0.8.0。超参数设置为:epoch:150、freeze epoch:50、batch size:16、learning rate:0.0001.优化器采用Adam优化器,损失函数为:CrossEntropy函数。
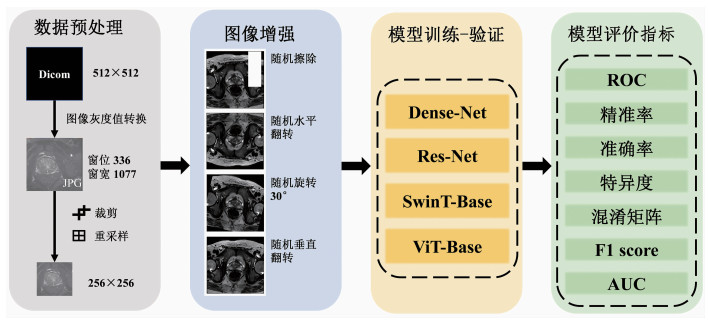
1.3.2 数据预处理首先对MRI图像进行格式转换,通过使用Python3.7.1将Dicom格式的医学图像灰度值转化为CT值(Hu),本研究中采用的窗位、窗宽分别设置为336、1 077,然后再转换为JPG格式。采用Dense-Net、Res-Net、ViT及SwinT网络训练模型。提取MRI图像ROI区域,保留前列腺及毗邻结构,大小为256×256。再经过归一化处理,传入网络进行训练(图 1)。
|
| 图 1 深度模型训练与验证过程 |
1.3.3 深度学习模型的构建
因训练集数据量有限,可能会产生严重的过拟合现象,从而使模型的分类性能下降。本实验在不改变数据集主要特征的前提下,通过随机擦除、随机水平翻转、旋转30°、垂直翻转等方法进行数据增强(图 1),有效增加数据集的数量,避免过拟合现象的发生[19]。
当前研究使用较多的深度学习分类模型,如HUANG等[20]提出的Dense-Net模型,通过引入从任意层到后续层的直接连接,缓解了梯度消失问题,加强了特征传递,并大大减小了参数和计算量;HE等[21]提出的Res-Net模型通过特征映射和残差映射两种方式优化了网络加深造成的梯度爆炸和梯度消失问题;不同于传统的卷积神经网络(convolutional neural networks, CNN)和循环神经网络(recurrent neural network,RNN),Transformer是由Google团队提出的一种自然语言处理领域(natural language processing,NLP)的经典模型,整个网络结构完全由Attention机制组成,不采用RNN的顺序结构,使得模型可以并行训练,很好地解决了长距离依赖的问题,并且符合当前的硬件(主要指GPU)环境[21-22];ViT基于Transformer模型来处理视觉领域的任务[23];SwinT网络以ViT作为起点,借鉴CNN的分层结构,选取Res-Net网络的精华,拥有了滑动窗口和层级式Transformer的两大特点[24]。
本研究共选择4种具有代表性的深度学习分类算法进行研究,Dense-Net、Res-Net、ViT及SwinT都是近年较为热门的分类网络。首先在4个深度学习网络上进行迁移学习,网络模型在数据集上训练之后,获得预训练模型,并得到预训练模型的初始权重,后将参数迁移到新的模型微调即可进行数据集的分类。本研究搭载Dense-Net、Res-Net、ViT和SwinT网络的预训练权重,在其基础上进行微调,首先输入图像经过预处理过后,传入网络模型中进行训练。优化器均选择Adam优化器,损失函数为:CrossEntropy函数,为了保证公平性,实验过程中每个网络均采用相同的训练集,测试集以及超参数,保存验证集上损失最小的Epoch所对应的模型权重文件。
训练阶段完成后,加载保存好的损失最小的权重在测试集上进行推理。使用相同测试集对训练好的四个网络模型进行测试,输出高危组和低中危组的结果,比较不同模型的分类性能。计算各网络模型的空间复杂度(floating point operations, FLOPs)、时间复杂度(参数量)和模型大小[25]。利用Grad-CAM工具分别对Dense-Net、Res-Net、ViT和SwinT网络保存的模型进行热力图可视化。
1.3.4 深度学习模型的性能评价指标采用混淆矩阵展示各模型的分类结果[26]。使用精准率(accuracy)、准确率(precision)、特异度(specificity)、混淆矩阵、召回率的调和均值(F1 score)、召回率(recall)、ROC曲线以及AUC对4种网络模型区分前列腺癌T分期低中危组、高危组的分类性能进行评估[27]。其相关公式表达如式(1)~(5)所示:
其中TP,TN,FP,FN分别表示真阳性、真阴性、假阳性、假阴性的数量。混淆矩阵是一种可视化工具,以预测值作为横轴、真实值作为纵轴形成矩阵图。其中,TP、TN数量越大,FP、FN的数量越小,说明分类效果越好[28]。ROC曲线是根据二分类,以真阳性率(灵敏度)为纵坐标,假阳性率(1-特异度)为横坐标绘制的曲线,AUC值越接近1,说明分类效果越好。
1.4 统计学分析采用SPSS 25.0软件进行统计学处理,组间差异采用方差分析,以P<0.05表示存在统计学差异。
2 结果 2.1 患者一般资料本研究共纳入152例患者,均为男性。研究样本被随机分为训练集、验证集和测试集。患者的临床资料见表 2。各子集患者的年龄、BMI和游离前列腺特异性抗原与总前列腺特异性抗原的比值(free/total prostate specific antigen, f/t-PSA)均无统计学差异。
| 变量 | 训练集 | 验证集 | 测试集 | P |
| 年龄 | 72.3±6.4 | 71.2±4.7 | 70.2±6.8 | 0.943 |
| BMI | 24.31±2.18 | 24.01±2.98 | 23.98±2.48 | 0.871 |
| f/t-PSA | 0.15±0.09 | 0.16±0.11 | 0.17±0.10 | 0.856 |
2.2 分类结果
对于前列腺癌医院数据集的分类任务,各网络的效果对比如表 3所示。从表中可以看出,Dense-Net、Res-Net,ViT和SwinT网络的精确度分别为0.587、0.410、0.600、0.680,SwinT网络分类效果准确性最高。
| 网络模型 | 准确率 | 特异性 | F1 score | 精确度 | AUC |
| Dense-Net | 0.587 | 0.630 | |||
| T1-T2 | 0.609 | 0.250 | 0.781 | ||
| T3-T4 | 0.426 | 0.778 | 0.527 | ||
| Res-Net | 0.410 | 0.477 | |||
| T1-T2 | 0.585 | 0.032 | 0.750 | ||
| T3-T4 | 0.667 | 0.989 | 0.538 | ||
| ViT | 0.600 | 0.648 | |||
| T1-T2 | 0.692 | 1.000 | 0.739 | ||
| T3-T4 | 0.100 | 0.333 | 0.412 | ||
| SwinT | 0.680 | 0.708 | |||
| T1-T2 | 0.652 | 0.333 | 0.731 | ||
| T3-T4 | 0.571 | 0.833 | 0.421 |
2.3 混淆矩阵
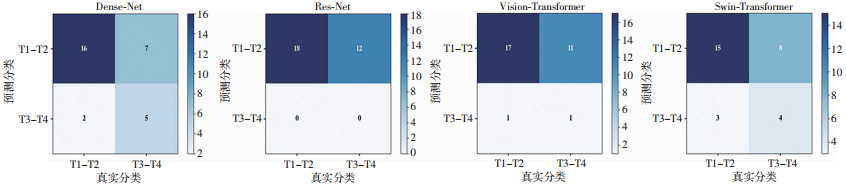
混淆矩阵如图 2所示。从混淆矩阵中可以看出,以患者为单位,Dense-Net、Res-Net、ViT和SwinT网络的假阳性例数分别是7、12、11、8,假阴性例数分别是2、0、1、3。Swin-Transformer真阳性和真阴性为15和4,假阳性和假阴性为8和3,病例分布仅次于Dense-Net。
|
| A:Dense-Net网络混淆矩阵;B:Res-Net网络混淆矩阵;C:ViT网络模型混淆矩阵;D:SwinT网络模型混淆矩阵 图 2 不同网络的混淆矩阵 |
2.4 ROC曲线对比情况
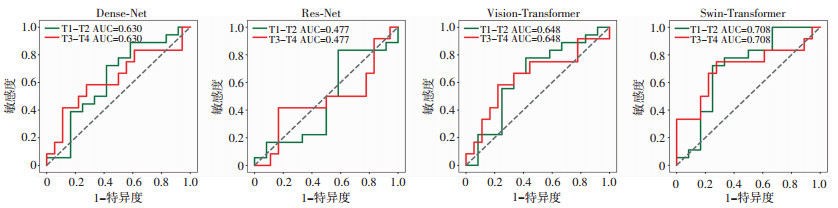
以每位患者的MRI图像为单位,FRP为横轴、TPR为纵轴绘制各网络的ROC曲线,结果如图 3所示。除了Res-Net网络外(AUC为0.477),Dense-Net、ViT和SwinT网络AUC值均大于0.5,分别为0.630、0.648、0.708,其中SwinT网络效果最优。
|
| 图 3 不同网络的ROC曲线 |
2.5 热力图可视化
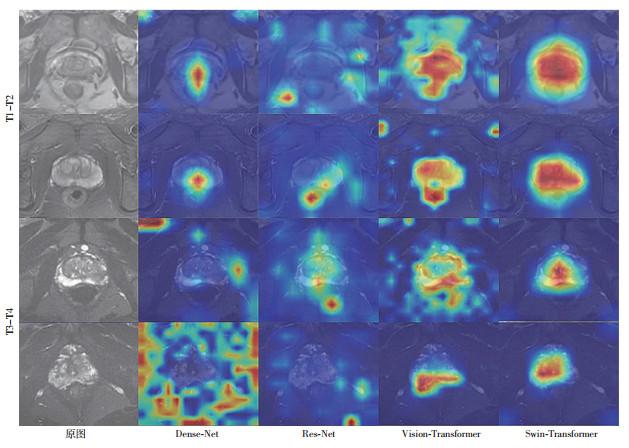
为了进一步了解深度学习网络的可解释性,我们借助梯度加权类激活映射(Grad-CAM)算法使模型结果可视化,对模型的注意力机制进行探究。Grad-CAM图的高度激活区是模型预测的主要贡献者。选取测试集低中危组和高危组各2例患者,分别对每个模型生成热力图(图 4)。Swin-Transformer的注意力最准确,基本可以准确定位于前列腺。
|
| 红色区域显示相对更好的结果 图 4 各种网络模型在低中危组和高危组不同层面的网络热力图可视化 |
3 讨论
近年来,人工智能深度学习在医学图像处理领域有重要应用,主要体现人工智能辅助医学图像分割、疾病智能诊断,疾病预后评估等方面[29]。虽然目前关于前列腺癌的研究已取得很多成果,但研究模型较单一,缺乏模型间的比较[10-12]。本研究收集多中心数据进行多种深度学习模型的训练和测试,挑选性能最优的模型,为下一步智能化评估前列腺癌T分期提供支撑。
Dense-Net模型采用直接连接的方式加强了特征传递,大大减小了参数和计算量,使得其尺寸最小;Res-Net模型在编码器和解码器的卷积运算中增加了残差结构,以提高模型性能[30];ViT是首个用于计算机视觉领域的纯Transformer模型;SwinT以ViT作为起点,借鉴CNN的分层结构,选取Res-Net网络的亮点,实现了滑动窗口和分层表示的两大特点。滑动窗口实现局部自注意力的计算,以及相邻窗口的交互,减少计算复杂度;分层结构实现不同尺度的图像适配,提供各个尺度的维度信息。在小窗内计算自注意力,利用图像的局部性先验知识,减少全局计算造成的计算浪费;从而可以实现任何尺寸图像的输入[31]。相反,ViT网络通过全局自注意力机制达到全局计算,只能使用低分辨率的图像输入。总之,SwinT网络不仅可用于图像分类,还可用于图像分割、目标检测等一系列视觉下游任务,能够适应更广泛的视觉任务。
从网络的性能参数以及各种评价指标可以得出,ViT和SwinT网络整体性能优于Dense-Net、Res-Net网络。从混淆矩阵结果可以得出SwinT网络假阳性、假阴性分别为8、3,分类错误率分别仅高于Dense-Net假阳性和假阴性各1例,相差较小。从ROC曲线图可以得出,Res-Net网络的AUC值为0.477,无法用来分类;Dense-Net、ViT和SwinT这3个网络AUC值均大于0.5,可实现前列腺癌的低中危组和高危组的分类。分类效果最好的是SwinT,其AUC值为0.708,略高于张洪涛等[27]通过手动分割217例患者后实现的最高诊断效能——AUC值为0.686[DWI(b值为1 000 s/m2)+DWI(b值为2 000 s/m2)],但略低于张洪涛等[27]实现的73.33%的精确度(SwinT网络分类的精确度为68.04%)。造成本研究准确率较低的原因可能是:本研究未对感兴趣区域进行分割和重点标记,使得网络模型未能进行注意力集中的学习。
热力图将模型的预测性能可视化,颜色越深的区域对结果预测贡献越大[32]。从Grad-CAM的角度来看,Res-Net和Dense-Net网络主要集中在前列腺的周边区域,注意力分散,冗余图像特征较多,结果可信度较低,类似于SwinT模型的不准确结果。然而,ViT和SwinT网络注意力则主要集中在前列腺整个区域和周围入侵的结构,表明这两个网络模型能够注意到与分类任务相关的重要区域,从而得到较为可靠的分类结果。与传统二进制预测的Norgram图相比,热力图实现了前列腺病灶的可视化,有助于放射科医师和泌尿外科医师快速发现和定位病灶,减少诊断时间,提高诊疗效率。
本研究仍存在一些局限性,需要在下一步工作中改进:①本研究仅收集两个医院的样本,样本量和样本来源仍需扩增,下一步拟构建大样本,多中心数据集;②本研究为基于MRI图像直接进行的分类,未对重要结构进行分割标注,下一步我们拟在模型训练之前对图像中前列腺、癌灶、膀胱、精囊腺体、直肠、尿道括约肌等重要结构进行分割标注,以减少无关特征对于模型训练的影响,提高分类性能和T分期诊断效率;③本研究模型仍为2D网络模型,准确性有限,可能会忽略病灶和结构的三维信息,下一步我们拟充分利用MRI图像的层间信息,创建和优化3D网络模型,提高T分期诊断效率。
本研究利用深度学习网络来构建前列腺癌的智能T分期诊断模型Swin-T,分类性能优于Dense-Net、Res-Net及ViT网络,智能诊断的效率最高,有助于节省医生诊断时间,节约医疗成本,提高诊断效率。
| [1] |
XIA C F, DONG X S, LI H, et al. Cancer statistics in China and United States, 2022: profiles, trends, and determinants[J]. Chin Med J (Engl), 2022, 135(5): 584-590. |
| [2] |
MADDAMS J, UTLEY M, MØLLER H. Projections of cancer prevalence in the United Kingdom, 2010-2040[J]. Br J Cancer, 2012, 107(7): 1195-1202. |
| [3] |
陈江华, 王子明. 泌尿系统疾病[M]. 北京: 人民卫生出版社, 2015. CHEN J H, WANG Z M. Urinary disease[M]. Beijing: People's Medical Publishing House, 2015. |
| [4] |
王颖, 刘金丰. 肿瘤CT与MRI诊断[M]. 广州: 广东科技出版社, 2018. WANG Y, LIU J F. CT and MRI diagnosis in oncology[M]. Guangzhou: Guangdong Technology Publish House, 2018. |
| [5] |
HOEKS C M A, BARENTSZ J O, HAMBROCK T, et al. Prostate cancer: multiparametric MR imaging for detection, localization, and staging[J]. Radiology, 2011, 261(1): 46-66. |
| [6] |
王良, LI Q B, ALBERTO V H. 前列腺MRI解剖分区[J]. 中华放射学杂志, 2020, 54(10): 1038-1040. WANG L, LI Q B, ALBERTO V H. MR imaging of prostate zonal anatomy[J]. Chin J Radiol, 2020, 54(10): 1038-1040. |
| [7] |
SCHRÖDER F H, HUGOSSON J, ROOBOL M J, et al. Screening and prostate cancer mortality: results of the European Randomised Study of Screening for Prostate Cancer (ERSPC) at 13 years of follow-up[J]. Lancet, 2014, 384(9959): 2027-2035. |
| [8] |
TRAN K A, KONDRASHOVA O, BRADLEY A, et al. Deep learning in cancer diagnosis, prognosis and treatment selection[J]. Genome Med, 2021, 13(1): 152. |
| [9] |
周飞燕, 金林鹏, 董军. 卷积神经网络研究综述[J]. 计算机学报, 2017, 40(6): 1229-1251. ZHOU F Y, JIN L P, DONG J. Review of convolutional neural network[J]. Chin J Comput, 2017, 40(6): 1229-1251. |
| [10] |
ISHIOKA J, MATSUOKA Y, UEHARA S, et al. Computer-aided diagnosis of prostate cancer on magnetic resonance imaging using a convolutional neural network algorithm[J]. BJU Int, 2018, 122(3): 411-417. |
| [11] |
ZHONG X R, CAO R M, SHAKERI S, et al. Deep transfer learning-based prostate cancer classification using 3 Tesla multi-parametric MRI[J]. Abdom Radiol, 2019, 44(6): 2030-2039. |
| [12] |
刘想, 高歌, 韩超, 等. 基于3D U-Net模型实现T2WI图像中前列腺的区域分割及临床验证[J]. 中国临床医学影像杂志, 2022, 33(1): 33-38, 42. LIU X, GAO G, HAN C, et al. Regional segmentation of prostate and clinical evaluation in T2WI image based on 3D U-Net model[J]. J China Clin Med Imaging, 2022, 33(1): 33-38, 42. |
| [13] |
CAI H B, FENG X B, YIN R M, et al. MIST: multiple instance learning network based on Swin Transformer for whole slide image classification of colorectal adenomas[J]. J Pathol, 2023, 259(2): 125-135. |
| [14] |
WU J F, XU Q, SHEN Y Q, et al. Swin transformer improves the IDH mutation status prediction of gliomas free of MRI-based tumor segmentation[J]. J Clin Med, 2022, 11(15): 4625. |
| [15] |
KHOSRAVI P, LYSANDROU M, ELJALBY M, et al. A deep learning approach to diagnostic classification of prostate cancer using pathology-radiology fusion[J]. J Magn Reson Imaging, 2021, 54(2): 462-471. |
| [16] |
LIU Y K, ZHENG H X, LIANG Z R, et al. Textured-based deep learning in prostate cancer classification with 3T multiparametric MRI: comparison with PI-RADS-based classification[J]. Diagnostics (Basel), 2021, 11(10): 1785. |
| [17] |
吴哲, 庞亚, 明智, 等. 人工智能技术在鼻咽癌放疗危及器官自动勾画中的应用研究[J]. 实用肿瘤学杂志, 2021, 35(2): 137-141. WU Z, PANG Y, MING Z, et al. Application of artificial intelligence technology in automatic delineation of organs at risk in nasopharyngeal carcinoma radiotherapy[J]. Pract Oncol J, 2021, 35(2): 137-141. |
| [18] |
GRIMES D A, SCHULZ K F. An overview of clinical research: the lay of the land[J]. Lancet, 2002, 359(9300): 57-61. |
| [19] |
余奕盈. 基于深度学习的图像分类[J]. 数字技术与应用, 2022, 40(1): 108-110. YU Y Y. Image classification based on deep learning[J]. Digit Technol Appl, 2022, 40(1): 108-110. |
| [20] |
HUANG G, LIU Z, VAN DER MAATEN L, et al. Densely connected convolutional networks[C]//2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). IEEE, 2017: 2261-2269. DOI: 10.1109/CVPR.2017.243.
|
| [21] |
HE K M, ZHANG X Y, REN S Q, et al. Deep residual learning for image recognition[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). IEEE, 2016: 770-778. DOI: 10.1109/CVPR.2016.90.
|
| [22] |
LIU Z, LIN Y, CAO Y, et al. Swin transformer: Hierarchical vision transformer using shifted window[C]//2021 Proceedings of the IEEE/CVF International Conference on Computer Vision (ICCV). 2021: 10012-10022.
|
| [23] |
DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16x16 words: transformers for image recognition at scale[EB/OL]. arXiv, 2020. https://arxiv.org/abs/2010.11929.
|
| [24] |
LIU Z, LIN Y T, CAO Y, et al. Swin transformer: hierarchical vision transformer using shifted windows[C]//2021 IEEE/CVF International Conference on Computer Vision (ICCV). IEEE, 2022: 9992-10002. DOI: 10.1109/ICCV48922.2021.00986.
|
| [25] |
刘学思, 聂瑞, 张和华, 等. 基于Swin Transformer网络的肺结核影像自动分类效果评价[J]. 中国医疗设备, 2022, 37(8): 25-31, 65. LIU X S, NIE R, ZHANG H H, et al. Effect evaluation of automatic classification of pulmonary tuberculosis images based on swin transformer network[J]. China Med Devices, 2022, 37(8): 25-31, 65. |
| [26] |
于营, 杨婷婷, 杨博雄. 混淆矩阵分类性能评价及Python实现[J]. 现代计算机, 2021(20): 70-73, 79. YU Y, YANG T T, YANG B X. Confusion matrix classification performance evaluation and python implementation[J]. Mod Comput, 2021(20): 70-73, 79. |
| [27] |
张洪涛, 胡泽宇, 盛复庚, 等. 双参数MRI影像组学机器学习预测前列腺癌T分期价值研究[J]. 人民军医, 2021, 64(6): 538-541, 558. ZHANG H T, HU Z Y, SHENG F G, et al. Study on the value of machine learning in predicting T staging of prostate cancer with two parameters MRI imageology[J]. People Mil Surg, 2021, 64(6): 538-541, 558. |
| [28] |
孙兆男, 崔应谱, 刘想, 等. 基于深度学习的前列腺mpMRI序列分类的可行性研究[J]. 临床放射学杂志, 2022, 41(8): 1559-1564. SUN Z N, CUI Y P, LIU X, et al. Feasibility of prostate mpMRI sequence classification based on deep learning[J]. J Clin Radiol, 2022, 41(8): 1559-1564. |
| [29] |
吴毅, 张小勤. 人工智能在医学图像处理中的研究进展与展望[J]. 第三军医大学学报, 2021, 43(18): 1707-1712. WU Y, ZHANG X Q. Artificial intelligence in medical image processing: progress and prospect[J]. J Third Mil Med Univ, 2021, 43(18): 1707-1712. |
| [30] |
JHA D, SMEDSRUD P H, RIEGLER M A, et al. ResUNet: an advanced architecture for medical image segmentation[C]//IEEE International Symposium on Multimedia (ISM). IEEE, 2020: 225-2255. DOI: 10.1109/ISM46123.2019.00049.
|
| [31] |
DAN Y P, ZHU Z N, JIN W S, et al. S-Swin Transformer: simplified Swin Transformer model for offline handwritten Chinese character recognition[J]. Peerj Comput Sci, 2022, 8: e1093. |
| [32] |
HOU Y, ZHANG Y H, BAO J, et al. Artificial intelligence is a promising prospect for the detection of prostate cancer extracapsular extension with mpMRI: a two-center comparative study[J]. Eur J Nucl Med Mol Imaging, 2021, 48(12): 3805-3816. |