氮芥(nitrogen mustard,NM)属于军用糜烂性毒剂[1-2],其渗透性强,靶器官众多,可迅速造成皮肤、呼吸道、眼等的急性损伤[3]。有研究显示,与NM结构类似的糜烂性毒剂硫芥造成呼吸系统损伤的远后效应在所有损伤中排首位,提示应重视糜烂性毒剂对呼吸系统的影响[4-5]。NM引起机体损伤的机制复杂,其中备受关注的机制之一是:NM分子中含有2个活跃的亲电氯乙基,可作为2个烷化位点,与细胞中含有亲核基团的大分子(如DNA、蛋白质等)进行烷化反应[6]。DNA是NM最为敏感的靶分子;正是由于对DNA的烷化损伤,使NM及其衍生物也作为一类化疗药物在临床上广泛应用[7]。然而,NM对正常细胞的DNA损伤依然值得关注,这关系到研发特效抗毒药物以及减轻临床应用的副作用。目前,关于NM引起的DNA损伤、修复作用及其机制尚未完全阐明。
研究表明,NM除了可以引起DNA的链内和链间交联,还可引起DNA-蛋白交联(DNA-protein crosslink,DPC),即NM通过其2个氯乙基分别烷化DNA和蛋白质,并在二者之间形成稳定的共价交联[8]。例如,本实验室前期发现O6-甲基鸟嘌呤-DNA甲基转移酶(O6-methylguanine-DNA methyltransferase,MGMT)可在NM的作用下与DNA形成DPC(MGMT-DPC,mDPC)[9],该蛋白可作为研究NM致DPC损伤的靶蛋白。因此,修复DPC是对抗NM损伤的重要方式之一。DVC1是近来发现的蛋白水解酶,可能参与DPC的蛋白水解修复[10-11]。本实验室前期研究发现DVC1参与了NM的损伤修复[9],然而,其具体作用尚未深入阐明。本研究通过体外实验,利用人支气管上皮细胞16HBE建立NM染毒模型,结合DVC1 siRNA干扰,以MGMT为DPC的代表性靶蛋白,初步探讨DVC1在NM致DPC损伤修复中的作用。
1 材料与方法 1.1 实验试剂人支气管上皮细胞16HBE为本室保存。胎牛血清(无支原体)和RPMI1640培养基购自美国HyClone公司;lipofectamine 2000脂质体转染试剂盒购自美国Sigma公司;DVC1过表达质粒购自中国义翘神州公司、si-DVC1购自中国吉玛公司。小鼠抗人MGMT、Ub抗体购自美国Santa Cruz公司;兔抗人DVC1抗体购自美国Abcam公司;兔抗人p-H2AX、ERCC1、Rad51、Ku70、β-actin、GAPDH、H3抗体购自美国CST公司。细胞总蛋白、细胞核浆蛋白提取试剂盒购自中国碧云天公司。BCA蛋白定量试剂盒购自美国Fisher Scientific公司。DNAiso购自日本TaKaRa公司。SYBR Green荧光染料购自中国瑞楚生物。NM盐酸盐、FITC购自美国Sigma公司。NSC697923购自美国Selleck公司。
1.2 细胞处理16HBE细胞用含10%胎牛血清的RPMI1640培养基传代培养。细胞长至融合度为70%~80%时进行实验。NM盐酸盐用PBS溶解后即进行细胞染毒1 h,维持染毒终浓度为50 μmol/L。根据不同实验进行分组及处理,(1)总DPC(total DPC, tDPC)随时间变化趋势检测:对照组为正常培养的细胞;1 h组:50 μmol/L NM染毒1 h后结束培养;6 h组:染毒后更换正常培养基并持续至6 h;24 h组:染毒后更换正常培养基并持续至24 h;si-DVC1组:用si-DVC1进行细胞转染2 d(si-DVC1序列为:5′-GCACUGUUUGUUCAGUUUAAC-3′);si-DVC1+NM组:si-DVC1进行细胞转染2 d后进行NM染毒,根据实验需要培养至相应时间;NSC697923+NM组:1 μmol/L NSC697923预处理1 h后进行NM染毒,根据实验需要培养至相应时间。(2)MGMT、mDPC变化及影响因素、DNA损伤修复通路检测:对照组、NM组、si-DVC1组、si-DVC1+NM组同前述;si-NC组:转染随机对照siRNA 2 d(si-NC寡核苷酸序列为:5′-UUCUCCGAACGUGUCACGUTT-3′);si-NC+NM组:转染随机对照siRNA 2 d后进行NM染毒;pcDNA组:转染随机对照pcDNA 2 d;pcDNA+NM组:转染随机对照pcDNA 2 d后进行NM染毒;DVC1组:转染DVC1过表达质粒2 d;DVC1+NM组:转染DVC1过表达质粒2 d后进行NM染毒;MG132组:10 μmol/L MG132处理细胞2 h;MG132+NM组:10 μmol/L MG132处理细胞2 h后进行NM染毒。
1.3 Western blot检测蛋白表达细胞培养结束后,加入细胞裂解液收集细胞总蛋白,或利用细胞质/核蛋白提取试剂盒分别收集胞质/胞核蛋白。测定蛋白浓度后,取40 μg蛋白样品加入上样缓冲液后煮沸变性上样。用质量分数为10%的SDS-PAGE凝胶电泳,将蛋白应用湿性转膜法转印至PVDF膜上,用含1% BSA封闭液封闭2 h后,用MGMT、DVC1、p-H2AX、ERCC1、Rad51、Ku70、β-actin、GAPDH、H3抗体4 ℃过夜孵育。洗膜后加入二抗稀释液室温孵育1 h。再次洗膜后加入ECL发光液,用VILBER FUSION FX7成像系统曝光。
1.4 总DPC(tDPC)含量测定细胞裂解及DPC提取参照KIIANITSA等[12]报道的方法并加以改进。DNAiso裂解细胞后,用无水乙醇将DNA沉淀并清洗DNA。TE缓冲液溶解DNA后,利用SYBR Green标记DNA并通过测定荧光值对DNA进行定量。取含10 μg DNA的样品,加入1 μL FITC母液避光孵育1 h以结合交联蛋白,利用无水乙醇将DNA沉淀并清洗。TE缓冲液重新溶解DNA后,利用酶标仪在495 nm激发光波长处测量各样本FITC的荧光值。
1.5 Slot blot检测取30 μg前述利用SYBR Green定量的DNA样品,用TBS补充体积至200~500 μL并过滤。向Slot blot装置各上样孔中加入样品,利用真空泵将样品吸附至装置中的NC膜上。用1% BSA封闭液封闭NC膜1 h,MGMT一抗4 ℃孵育过夜。洗膜后加入二抗稀释液室温孵育1 h。再次洗膜后加入ECL发光液,用VILBER FUSION FX7成像系统曝光。
1.6 免疫沉淀实验在吸附柱中加入树脂并用PBS清洗1次。加入1 mg蛋白样品,4 ℃混匀孵育1 h。离心收集蛋白滤过液。向蛋白滤过液中加入10 μg Ub一抗,用IP裂解/清洗缓冲液补充混合液体积至300 μL,4 ℃下孵育过夜。将样品加入新的含蛋白A/G琼脂糖的吸附柱,4 ℃下缓慢旋转混匀孵育1 h,离心去除缓冲液。用IP裂解/清洗缓冲液清洗复合物后,向吸附柱中加入2×SDS上样缓冲液煮沸变性,离心收集滤过蛋白。采用Western blot检测方法,经MGMT一抗孵育、洗膜、二抗孵育,ECL化学发光检测泛素化的MGMT蛋白表达。
1.7 GST下拉实验将DVC1 cDNA序列构建到pet-GST载体上并测序验证。将质粒转入大肠杆菌系统,经细菌培养小量表达GST-DVC1蛋白,超声法提取细菌总蛋白。经SDS-PAGE电泳鉴定GST-DVC1融合蛋白的表达后,将总蛋白加入50~70 μL GST-Beads,4 ℃摇床上反应30~60 min,离心弃上清,重复上述步骤以使凝胶珠上结合更多GST-DVC1融合蛋白。用200 μL PBS洗涤GST-Beads,离心弃上清。用500 μL NETN缓冲液悬浮GST-Beads,加入20 μL 16HBE细胞裂解提取的总蛋白,4 ℃摇床上反应4~8 h,经离心、洗涤,Western blot检测MGMT和Ub表达。
1.8 统计学分析各组样本均独立重复3次。计量资料以x±s表示,采用SPSS 20. 0统计软件对数据进行单因素方差分析。检验水准:α=0.05。
2 结果 2.1 DVC1对tDPC含量的影响用si-DVC1构建DVC1表达缺陷细胞模型,结果显示DVC1蛋白的表达被显著抑制(图 1A)。为观察DVC1在清除DPC过程中是否发挥泛素酶作用,以E2泛素结合酶抑制剂NSC697923作为对照,发现si-DVC1+NM组1、6、24 h的tDPC变化趋势与NSC697923+NM组类似,24 h与1、6 h相比有更多tDPC累积(图 1B),提示DVC1在泛素被激活之后发挥作用,可能与E2泛素结合酶类似或晚于E2酶作用。
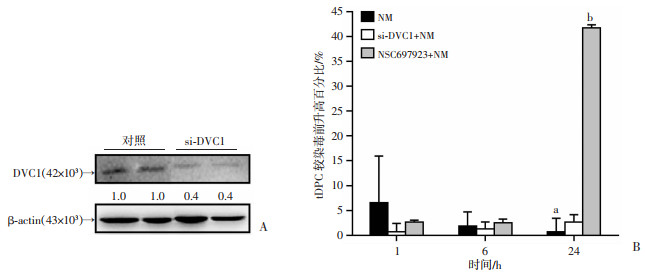
|
| A:si-DVC1的干扰效果;B:细胞在染毒后1、6、24 h tDPC含量相对染毒前增长的百分比 NM:仅NM染毒;si-DVC1+NM:NM合并si-DVC1处理;NSC697923+NM:NM合并NSC697923处理;a:P < 0.05, 与NM处理1 h比较;b:P < 0.01, 与NSC697923+NM处理1、6 h比较 图 1 si-DVC1及泛素结合酶抑制剂处理下tDPC的时间变化趋势 |
2.2 DVC1对DNA-MGMT交联(mDPC)含量的影响
为进一步研究DVC1对DPC的影响,选取MGMT作为形成DPC的代表性交联蛋白进行后续实验。NM染毒后1 h,Western blot检测26 kD原型MGMT蛋白(26 kD-MGMT)表达水平降低,且MGMT主要表达于细胞核(图 2A)。然而,MGMT在染毒后与DNA形成交联(mDPC)(图 2B)。DVC1过表达可降低26 kD-MGMT水平,相反,si-DVC1干预后,可提高26 kD-MGMT水平;在NM暴露后仍然可以看到各自的类似表现(图 2C)。si-DVC1干预可导致mDPC含量升高(图 2D),且(55~72)×103处多泛素化MGMT水平升高(图 2E)。上述结果提示,NM染毒造成泛素化MGMT水平升高及非泛素化MGMT水平降低,同时引起MGMT与DNA的交联。DVC1缺失加重交联和泛素化,提示抑制DVC1妨碍泛素化MGMT从交联复合体上清除。
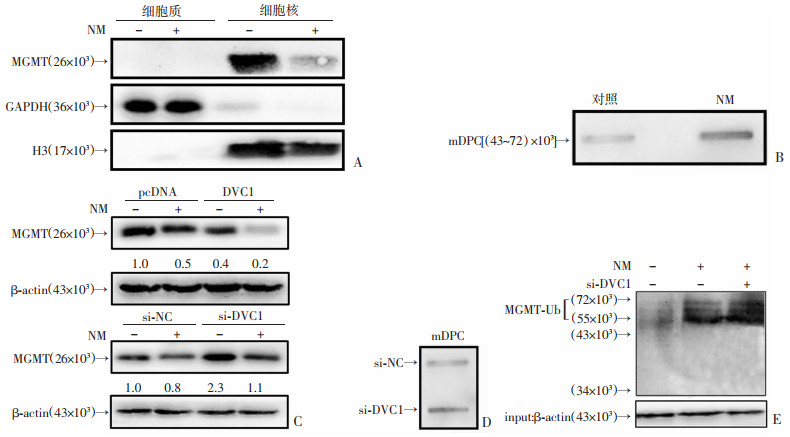
|
| A:Western blot检测26 kD原型MGMT蛋白在16HBE细胞质及细胞核内的表达; B:Slot blot检测NM染毒后MGMT与DNA形成mDPC; C:pcDNA-DVC1过表达质粒及si-DVC1处理对26 kD原型MGMT蛋白表达水平的影响; D:Slot blot检测si-DVC1处理后mDPC的含量 si-NC组为转染siRNA随机对照寡核苷酸的细胞; E:免疫共沉淀检测si-DVC1处理后MGMT的泛素化水平 图 2 MGMT在染毒后的变化及DVC1对其的影响 |
2.3 DVC1通过泛素途径影响DPC含量
采用GST下拉实验明确DVC1是否对MGMT有特异性识别作用。结果证实,DVC1和MGMT没有直接结合的相互作用,然而DVC1可以结合泛素分子(图 3A),提示DVC1对mDPC的影响与结合MGMT上的泛素有关。蛋白酶体抑制剂MG132处理细胞可降低核内26 kD-MGMT的水平(图 3B),与si-DVC1作用类似的是,MG132也可引起mDPC的显著升高(图 3C、D),提示通过抑制蛋白酶体阻止泛素化MGMT水解将导致交联于DNA的MGMT无法移除。
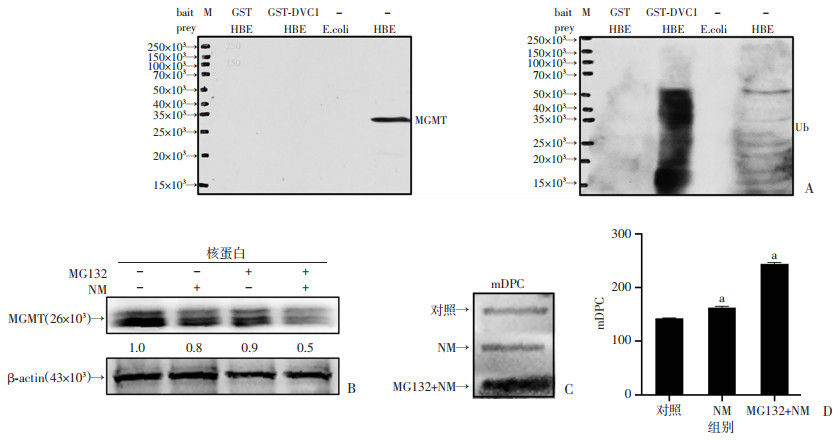
|
| A:GST下拉实验检测DVC1与MGMT蛋白(左)和泛素(Ub,右)的直接结合能力;B:蛋白酶体抑制剂MG132处理后26 kD原型MGMT在细胞核中的表达变化;C:MG132处理后mDPC的含量变化;D:C图的灰度定量分析结果 a:P < 0.05, 与对照组比较 图 3 DVC1参与MGMT泛素化过程 |
2.4 DVC1对DNA损伤修复的影响
考虑到NM本身和DPC修复过程可能引起多种形式的DNA损伤,本研究选择ERCC1、Ku70、Rad51作为核苷酸切除修复、非同源末端连接和同源重组修复方式的代表蛋白,检测了DVC1对其他DNA损伤修复的影响。结果显示,NM染毒可造成ERCC1蛋白表达轻微升高,si-DVC1提高了ERCC1蛋白在非染毒状态下的本底表达。合并NM染毒后,ERCC1表达较染毒前轻微升高,与si-NC组升高趋势类似。NM染毒造成Rad51表达升高,si-DVC1也造成Rad51本底表达升高(P < 0.05),但与si-NC相比,si-DVC1处理降低了Rad51的相对表达水平(P < 0.05)。与此同时,si-DVC1处理较si-NC组升高了Ku70的相对表达水平(P < 0.05,图 4),提示DVC1缺失对细胞DNA损伤修复的方式有影响。
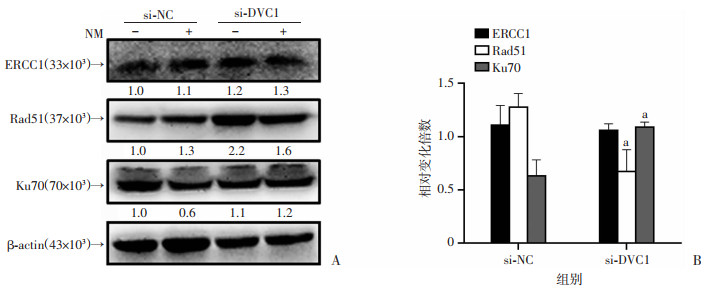
|
| A:si-DVC1处理后ERCC1、Rad51及Ku70在NM染毒后的表达水平;B:蛋白相对染毒前的变化倍数 a:P < 0.05, 与si-NC组比较 图 4 DVC1对DNA损伤修复通路的影响 |
3 讨论
作为一种军用糜烂性毒剂和肿瘤化疗药物,NM可对细胞DNA造成严重损伤。这在抗肿瘤方面是有益作用,可干扰肿瘤细胞的增殖[13-14],但在正常细胞中,就成为触发正常细胞损伤的因素。因此,其致糜烂作用强,也能在化疗时引起严重的副作用。目前有多种学说尝试阐明NM的损伤机制,其中,NM对DNA的损伤是重要的学说之一[15]。DNA受损增加了细胞增殖、蛋白质合成的风险,这也是增殖旺盛的细胞对NM更为敏感的原因。
烷化剂对靶分子造成的烷化反应与烷化剂分子内的烷化基团个数有关。仅有1个烷化基团的烷化剂,即单功能烷化剂,仅能与DNA上1个碱基位点反应[16]。NM分子中含有2个亲电的氯乙基基团,属于双功能烷化剂[17],因而对DNA造成的损伤形式更为多样且严重,包括仅利用一个氯乙基造成DNA上单碱基/核苷酸位点的烷化,而另一个氯乙基仍游离,以及利用2个氯乙基同时对2个靶位点交联。DPC即DNA和蛋白质之间发生的交联,是NM作为双功能烷化剂引起的交联种类之一,只是2个烷化靶点并非全部位于DNA,而是分别位于DNA和蛋白质[15]。因为蛋白质共价、稳定地结合于DNA,不易移除[8],从而使交联蛋白对DNA正常的解旋、复制和转录产生阻碍作用,也可能引起继发的链断裂[18]。
在酵母中发现的一种DPC加工蛋白酶Wss1,可降解酶(TOP1交联)及非酶(甲醛诱导)交联DPC[19-20]。这使得DPC的蛋白水解修复途径被关注。通过生物信息学比对,发现Wss1在高等真核生物中的同源蛋白为DVC1[21]。因为交联蛋白能阻止DNA复制酶的移动,其水解也势必需要通过DNA复制受阻引发复制压力而触发[22]。巧合的是,有研究发现DVC1的表达及功能与DNA复制及细胞周期密切相关[23],这些都提示DVC1可能参与修复DPC的作用。本研究结果显示,正常细胞在NM损伤之后,随时间进展有清除DPC的趋势;但DVC1缺失后,DPC含量却随时间上升,说明在修复阶段,DVC1的缺失影响了清除DPC的过程。NSC697923抑制E2泛素结合酶后有类似的DPC持续上升的结果。E2泛素结合酶在泛素链的延长和识别特异的E3泛素连接酶并将泛素链转移到靶蛋白上有重要作用[24],E2泛素结合酶的抑制可能在一定程度上影响了交联蛋白泛素化的最后步骤,从而使交联蛋白无法被E3酶识别并被转移。研究发现DVC1可通过泛素化信号被招募[20]。si-DVC1的作用与NSC697923类似,提示DVC1参与修复NM诱导的DPC的作用,可能发生在泛素链形成之后。
为进一步阐明DVC1的作用及其与泛素化过程的关系,本研究以MGMT作为交联蛋白的代表,观察DVC1对MGMT蛋白本身及mDPC的影响。MGMT是此前已被证实的能被NM交联到DNA链上的蛋白,本课题组此前证实MGMT被移除是依赖于泛素化的[25],但尚不清楚DVC1对MGMT的具体影响。MGMT主要存在于细胞核,与形成DPC的位置一致,且mDPC在NM染毒后升高。但如仅通过Western blot检测MGMT蛋白在26×103的位置,却发现在NM损伤之后有显著降低。考虑到这可能由于MGMT在被交联后,需被泛素化,而泛素化将导致MGMT分子量变化,从而使其在SDS-PAGE上的位置升高,本研究检测si-DVC1干预后MGMT的泛素化表达,结果证实DVC1的抑制使MGMT泛素化增强,说明MGMT的泛素化并不受DVC1缺失的影响,但mDPC清除过程却发生阻滞。这可能是因为DVC1的抑制干扰了MGMT泛素化后的水解,从而使泛素化MGMT累积。
本实验证实DVC1无法与MGMT结合,却能够结合泛素分子,说明DVC1对DPC的清除并非依赖交联蛋白本身的特异性,而是广泛的作用,其机制可能与结合泛素分子有关。这也佐证了干预DVC1的效果与干预E2泛素结合酶类似,当泛素分子无法通过E2泛素结合酶形成泛素链并聚集于DPC时,也无法募集DVC1到DPC处,从而限制了DPC的清除。
被泛素链标记的蛋白最后将被转移到蛋白酶体,蛋白酶体是蛋白质泛素化降解的最后环节[26-27]。利用蛋白酶体抑制剂MG132处理细胞显著降低了Western blot检测到的MGMT原型的水平,但mDPC含量却升高,提示MGMT原型降低的同时,可能存在更多的MGMT交联于DNA并且被泛素化,但却因蛋白酶体被抑制,即使被泛素化也无法完成降解。MG132对mDPC的影响与si-DVC1引起MGMT泛素化水平升高相吻合,提示DVC1发挥作用的时段主要在交联蛋白已被泛素化标记到被转移至蛋白酶体之间。
DPC的长时间存在势必作为DNA复制和翻译的阻遏物,解旋和复制的压力可能会继发更严重的DNA损伤,如单链甚至双链断裂[20]。DNA损伤有多种修复方式[28],通过检测DVC1缺失条件下各修复蛋白的改变情况,发现未染毒条件下,DVC1缺失可造成细胞生理状态下的DNA损伤,并导致ERCC1和Rad51升高,提示此时主要通过核苷酸切除、同源重组进行修复。但DVC1缺失对染毒引起的ERCC1变化影响并不显著,而是促使染毒条件下Ku70相对上调,以及Rad51相对下调。ERCC1、Ku70、Rad51分别是核苷酸切除修复、非同源末端连接和同源重组的关键蛋白[29-32],三者的表达变化提示DVC1的缺失可能促进DNA修复方式的改变,同源重组弱化,非同源末端连接增强。二者都是修复双链断裂的重要方式[33],不同之处在于,同源重组以姐妹染色单体的另一条未受损DNA链为模板,其修复过程保守,属无错修复;而非同源末端连接通过修复蛋白直接将断裂的DNA双链连接,属易错修复[34]。DVC1缺失造成二者在修复DNA上的平衡发生改变,提示细胞此时以一种更易错的方式进行DNA修复,虽然不利于保持基因组的稳定,但可以维持细胞的基本生物活动。有研究报道DVC1与PCNA共定位,其缺失将造成细胞周期阻滞[23],我们推测此时细胞无法正常进入S期,因而不满足同源重组的条件,故只能以非同源末端连接进行修复。
综上所述,本研究发现DVC1可与泛素分子结合,并参与NM诱导的DPC损伤的修复。DVC1缺失将导致交联蛋白泛素化和DPC含量升高,与抑制E2泛素结合酶和蛋白酶体的作用类似。该结果进一步揭示了DVC1在NM致DPC损伤修复中的作用,为研究NM防治措施提供了潜在靶点。
| [1] |
YUE L J, WEI Y X, CHEN J, et al. Abundance of four sulfur mustard-DNA adducts ex vivo and in vivo revealed by simultaneous quantification in stable isotope dilution-ultrahigh performance liquid chromatography-tandem mass spectrometry[J]. Chem Res Toxicol, 2014, 27(4): 490-500. |
| [2] |
ZHANG Y J, YUE L J, NIE Z Y, et al. Simultaneous determination of four sulfur mustard-DNA adducts in rabbit urine after dermal exposure by isotope-dilution liquid chromatography-tandem mass spectrometry[J]. J Chromatogr B Analyt Technol Biomed Life Sci, 2014, 961: 29-35. |
| [3] |
LIN Y, DONG Y, CHEN J, et al. Gas chromatographic-tandem mass spectrometric analysis of β-lyase metabolites of sulfur mustard adducts with glutathione in urine and its use in a rabbit cutaneous exposure model[J]. J Chromatogr B Analyt Technol Biomed Life Sci, 2014, 945/946: 233-239. |
| [4] |
NIE Z Y, ZHANG Y J, CHEN J, et al. Monitoring urinary metabolites resulting from sulfur mustard exposure in rabbits, using highly sensitive isotope-dilution gas chromatography-mass spectrometry[J]. Anal Bioanal Chem, 2014, 406(21): 5203-5212. |
| [5] |
DARCHINI-MARAGHEH E, BALALI-MOOD M. Delayed complications and long-term management of sulfur mustard poisoning: recent advances by Iranian researchers (part Ⅰ of Ⅱ)[J]. Iran J Med Sci, 2018, 43(2): 103-124. |
| [6] |
STEINRITZ D, LÜLING R, SIEGERT M, et al. Alkylated epidermal creatine kinase as a biomarker for sulfur mustard exposure: comparison to adducts of albumin and DNA in an in vivo rat study[J]. Arch Toxicol, 2021, 95(4): 1323-1333. |
| [7] |
IQUBAL A, IQUBAL M K, SHARMA S, et al. Molecular mechanism involved in cyclophosphamide-induced cardiotoxicity: old drug with a new vision[J]. Life Sci, 2019, 218: 112-131. |
| [8] |
GROEHLER A 4th, VILLALTA P W, CAMPBELL C, et al. Covalent DNA-protein cross-linking by phosphoramide mustard and nornitrogen mustard in human cells[J]. Chem Res Toxicol, 2016, 29(2): 190-202. |
| [9] |
CHENG J, YE F, DAN G R, et al. Bifunctional alkylating agent-mediated MGMT-DNA cross-linking and its proteolytic cleavage in 16HBE cells[J]. Toxicol Appl Pharmacol, 2016, 305: 267-273. |
| [10] |
STINGELE J, BELLELLI R, ALTE F, et al. Mechanism and regulation of DNA-protein crosslink repair by the DNA-dependent metalloprotease SPRTN[J]. Mol Cell, 2016, 64(4): 688-703. |
| [11] |
REINKING H K, KANG H S, GOTZ M J, et al. DNA structure-specific cleavage of DNA-protein crosslinks by the SPRTN protease[J]. Mol Cell, 2020, 80(1): 102-113.e6. |
| [12] |
KⅡANITSA K, MAIZELS N. A rapid and sensitive assay for DNA-protein covalent complexes in living cells[J]. Nucleic Acids Res, 2013, 41(9): e104. |
| [13] |
BRAYBOY L M, CLARK H, KNAPIK L O, et al. Nitrogen mustard exposure perturbs oocyte mitochondrial physiology and alters reproductive outcomes[J]. Reproductive Toxicol, 2018, 82: 80-87. |
| [14] |
PUYO S, MONTAUDON D, POURQUIER P. From old alkylating agents to new minor groove binders[J]. Crit Rev Oncol, 2014, 89(1): 43-61. |
| [15] |
JAN Y H, HECK D E, LASKIN D L, et al. DNA damage signaling in the cellular responses to mustard vesicants[J]. Toxicol Lett, 2020, 326: 78-82. |
| [16] |
SOSIC A, ZURAVKA I, SCHMITT N K, et al. Direct and topoisomerase Ⅱ mediated DNA damage by bis-3-chloropiperidines: the importance of being an earnest G[J]. ChemMedChem, 2017, 12(17): 1471-1479. |
| [17] |
GOSWAMI D G, KANT R, AMMAR D A, et al. Acute corneal injury in rabbits following nitrogen mustard ocular exposure[J]. Exp Mol Pathol, 2019, 110: 104275. |
| [18] |
NAKAMURA J, NAKAMURA M. DNA-protein crosslink formation by endogenous aldehydes and AP sites[J]. DNA Repair, 2020, 88: 102806. |
| [19] |
BALAKIREV M Y, MULLALLY J E, FAVIER A, et al. Wss1 metalloprotease partners with Cdc48/Doa1 in processing genotoxic SUMO conjugates[J]. eLife, 2015, 4: e06763. |
| [20] |
VAZ B, POPOVIC M, RAMADAN K. DNA-protein crosslink proteolysis repair[J]. Trends Biochem Sci, 2017, 42(6): 483-495. |
| [21] |
STINGELE J, BELLELLI R, BOULTON S J. Mechanisms of DNA-protein crosslink repair[J]. Nat Rev Mol Cell Biol, 2017, 18(9): 563-573. |
| [22] |
STINGELE J, HABERMANN B, JENTSCH S. DNA-protein crosslink repair: proteases as DNA repair enzymes[J]. Trends Biochem Sci, 2015, 40(2): 67-71. |
| [23] |
MÓROCZ M, ZSIGMOND E, TÓTH R, et al. DNA-dependent protease activity of human Spartan facilitates replication of DNA-protein crosslink-containing DNA[J]. Nucleic Acids Res, 2017, 45(6): 3172-3188. |
| [24] |
CARLA B, MARCO T. Identification and characterization of physiological pairing of E2 ubiquitin-conjugating enzymes and E3 ubiquitin ligases[J]. Methods Mol Biol Clifton N J, 2023, 2581: 13-29. |
| [25] |
CHENG J, YE F, DAN G R, et al. Formation and degradation of nitrogen mustard-induced MGMT-DNA crosslinking in 16HBE cells[J]. Toxicology, 2017, 389: 67-73. |
| [26] |
BARD J A M, GOODALL E A, GREENE E R, et al. Structure and function of the 26S proteasome[J]. Annu Rev Biochem, 2018, 87: 697-724. |
| [27] |
YASUDA S, TSUCHIYA H, KAIHO A, et al. Stress- and ubiquitylation-dependent phase separation of the proteasome[J]. Nature, 2020, 578(7794): 296-300. |
| [28] |
LEE N S, KIM S, JUNG Y W, et al. Eukaryotic DNA damage responses: Homologous recombination factors and ubiquitin modification[J]. Mutat Res Mol Mech Mutagen, 2018, 809: 88-98. |
| [29] |
FARIDOUNNIA M, FOLKERS G, BOELENS R. Function and interactions of ERCC1-XPF in DNA damage response[J]. Molecules, 2018, 23(12): 3205. |
| [30] |
SABATELLA M, PINES A, SLYSKOVA J, et al. ERCC1-XPF targeting to psoralen-DNA crosslinks depends on XPA and FANCD2[J]. Cell Mol Life Sci, 2020, 77(10): 2005-2016. |
| [31] |
GAO C, JIN G X, FORBES E, et al. Inactivating mutations of the IK gene weaken Ku80/Ku70-mediated DNA repair and sensitize endometrial cancer to chemotherapy[J]. Cancers, 2021, 13(10): 2487. |
| [32] |
BONILLA B, HENGEL S R, GRUNDY M K, et al. RAD51 gene family structure and function[J]. Annu Rev Genet, 2020, 54: 25-46. |
| [33] |
MURMANN-KONDA T, SONI A, STUSCHKE M, et al. Analysis of chromatid-break-repair detects a homologous recombination to non-homologous end-joining switch with increasing load of DNA double-strand breaks[J]. Mutat Res Toxicol Environ Mutagen, 2021, 867: 503372. |
| [34] |
SCULLY R, PANDAY A, ELANGO R, et al. DNA double-strand break repair-pathway choice in somatic mammalian cells[J]. Nat Rev Mol Cell Biol, 2019, 20(11): 698-714. |




