目前,肺癌仍然是全球癌症死亡的主要原因[1]。肺癌患者中约85%为非小细胞癌(non-small cell lung carcinoma,NSCLC),其中最常见的组织型是肺腺癌(lung adenocarcinoma,LUAD)[2-4]。LUAD患者的五年总生存率仅为15%,多数患者首诊即为晚期。此外,既往多数分子亚型的研究中并未考虑生存状况,而生存率的特异性往往对分子亚型有较大影响,在疾病进展、治疗反应等方面尚存在一定差异[5]。因此,LUAD的预后预测和亚型分类研究对生存率、生活质量的提升较为关键。
在既往研究中,对癌症的预后预测分析多使用临床数据[6-8],存在样本不均衡、样本量小等问题,进而影响癌症预后预测的精确度。伴随新一代高通量测序技术的发展,海量组学信息为癌症患者的预后情况提供了精确预测的基础,有利于为患者提供个性化治疗[9-10]。但组学数据存在高维度、高噪音和稀疏性的特点,特征提取和降维对于组学数据的分析至关重要。针对日益增长的高通量转录组学数据,机器学习、深度学习等人工智能方法的作用则逐步被应用和推广。目前,随机生存森林(random survival forest,RSF)[11]、CoxBoost[12]、Cox-nnet[13]、DeepSurv[14]等模型已被用于癌症的生存预后预测领域。但当前多数模型仅限于生存分析、无法执行亚型分型,且对于高维组学信息的处理能力有限。
自编码器(autoencoder,AE)是一种新型深度学习方法,可重构数据以生成新的特征,进而达到对高维特征进行降维的效果[15]。因此,本文拟在AE的基础上,构建一种包含特征降维、亚型分型和生存分析等功能的深度学习模型,旨在高效、准确地完成预后预测。
本文收集来源于癌症基因组图谱(the Cancer Genome Atlas, TCGA)数据库的转录组学数据,使用基于堆叠、监督的深度学习方法执行LUAD患者的预后预测分析,旨在从高通量、高维度的组学数据中获得和生存相关的关键特征基因,进而准确、全面地预测患者预后情况。
1 材料与方法 1.1 数据收集和预处理数据来源于TCGA。从肺癌数据库(TCGA-LUAD database)中下载转录组学数据和临床数据,包含生存时间和生存状态;共收集555例样本,60 660个基因。本文数据和方法的技术路线如图 1所示。
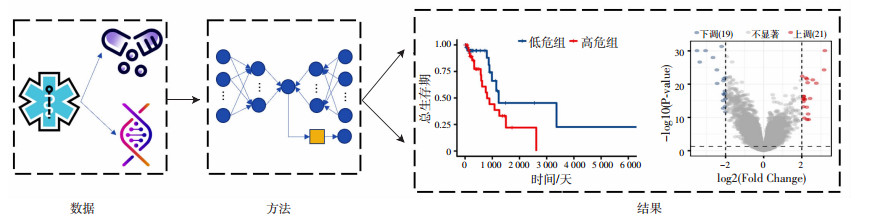
|
| 图 1 全文技术路线图 |
纳入和排除标准:纳入非阴性生存价值和组织学诊断为LUAD的样本;剔除数据中缺失值超过20%的特征、生存状态为存活但生存时间为零的样本。最终纳入临床数据475例样本(25 481个基因),其中存活353例,死亡122例。
使用归一化规范数据,将所有组学数据均按比例被缩放入[0, 1]区间,旨在消除因不同特征数据之间的测量数值大小差异产生的噪音。
1.2 预测模型构建 1.2.1 堆叠监督自编码器模型在AE中,逐层无监督的训练可达到良好的特征提取结果,但其提取的特征往往缺乏针对性,进而引发精确度降低和稳定性不足等一系列问题[15]。作为深度学习领域一种有效的降维方式,栈式自编码器(stacked autoencoder,SAE)是一类由多个自编码器堆叠起来的有分层结构的深层神经网络。相较于AE,SAE通过多次降维寻优训练,在其隐藏层拥有更好的特征压缩和提取效果。
基于Cox回归网络的自编码器(auto encoder with cox regression network,AECOX)和监督自编码器可为改良SAE提供思路[16-17]。因此,本研究将SAE和Cox-PH模型联合用于生存预测领域,提出一种利用Cox-PH回归网络以执行有监督训练的自编码器,即堆叠监督自编码器(stacked supervised autoencoder,SSAE)。在解码的同时,自编码器的隐藏层h通过Cox-PH回归网络进行预测并回传参与权重更新。在以逐层堆叠的方式提取包含目标变量信息特征的同时,SSAE还可进行生存分析。SSAE模型的结构如图 2所示。
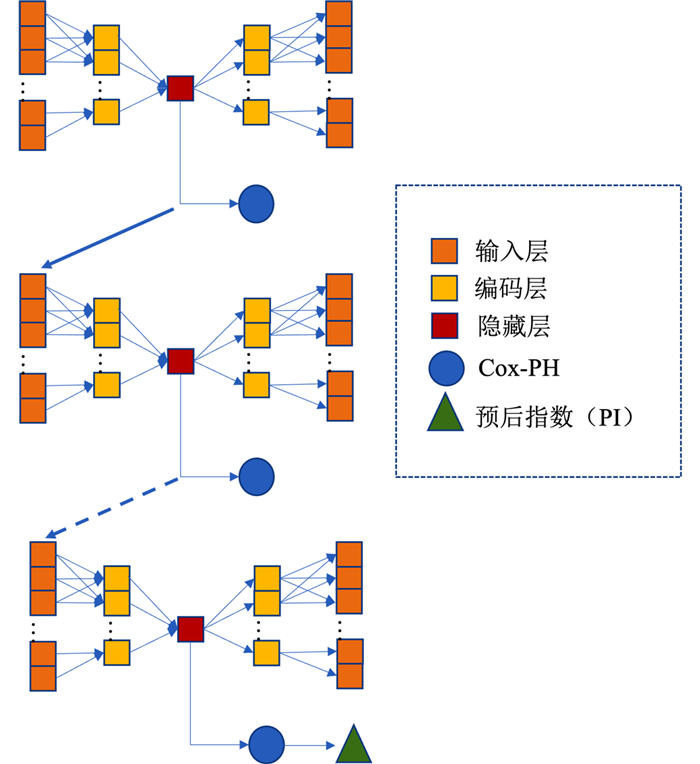
|
| 图 2 SSAE的结构 |
网络的超参数包括网络的深度、大小、学习率、超参数λ1、超参数λ2。使用Python包Optunity运行随机超参数优化搜索。从预定义的范围内,使用Sobol求解器对每个超参数进行采样。利用训练集和验证集寻找最优超参数,以验证集的一致性指数(concordance index,CI)最大的设置为超参数确定标准。
1.2.2 对比模型对比模型包含RSF和DeepSurv,均采用与SSAE相同的超参数寻优策略。
RSF模型由多个独立的决策树(decision tree, DT)组成,各决策树随机接受一个样本的子集,并在树中的各个分支点随机选择一个变量子集进行预测。
在DeepSurv模型中,将Cox回归模型连接在由多个全连接层和舍弃层组成的神经网络的末端,进而执行生存预后预测。
1.3 评价指标为评估方法的性能,我们对数据进行了10次重采样。在各次重采样的迭代中,我们在随机选择的80%训练集上训练模型,并在20%的测试集上评估性能。
利用CI和Log-rank检验P值评估模型性能[18-19]。CI被广泛用于评估生存预后模型,其取值范围为0~1。CI值越高表明模型的预测能力越强。
依据模型输出的预后指数(prognostic index,PI)中值对风险比进行二分类,并在高危组和低危组之间进行Log-rank检验,进而获得P值,P值越低代表对高危组和低危组的区分能力越强。
1.4 模型训练和统计学分析基于Python 3.7平台和PyTorch 1.7.1框架构建和训练模型,采用R 4.2.0和SPSS 25.0完成生信分析和生存分析。
在生信分析中,使用R包“DESeq2”对转录组学数据集进行差异分析,其中差异基因的筛选阈值为|log2FC|>2且P<0.05。在生信分析中,使用火山图和热图进行可视化展示,旨在直观了解差异基因的整体分布。
2 结果 2.1 预后预测性能比较为评估模型在LUAD转录组学数据集上的预测能力,将SSAE模型与RSF和DeepSurv两种模型进行对比研究。在测试集上,SSAE模型的CI(中位值0.58)显著高于RSF模型(中位值0.54)和DeepSurv模型(中位值0.55),如图 3所示。
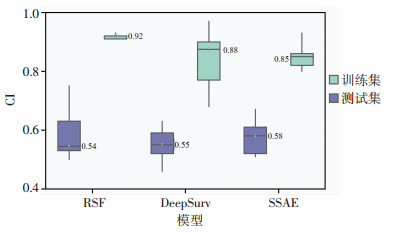
|
| 图 3 各模型预测性能的比较-CI |
利用各模型预测PI值的中位数对生存风险进行二分类,并利用Log-rank检验P值评估高风险组、低风险组Kaplan-Meier生存曲线间的生存差异。在测试数据集中,SSAE模型的P值(0.05)明显低于Deep Surv(P=0.10)和RSF(P=0.15),如图 4所示。
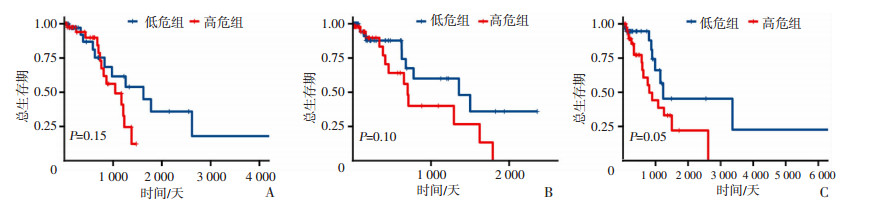
|
| A:RSF;B:DeepSurv:C:SSAE 图 4 各模型预测性能的比较-P值 |
2.2 生存分析
为进一步验证分子亚型对生存结局的影响,利用本文纳入的全部数据集进行生存分析,单因素分析结果表明:相较于低风险亚型(n=238),高风险亚型(n=237)的预后更差,差异有统计学意义(HR:2.833,95%CI:1.82~4.41,Log-rank检验P<0.001)。
为排除混杂因素造成的影响,将年龄、性别、病理分期及亚型分组纳入COX回归模型,多因素分析结果表明:病理分期(HR、95%CI和P值见表 1)、风险亚型(HR: 2.841,95%CI:1.907~4.232,Log-rank检验P<0.001)对生存结局的影响有统计学意义。
| 变量 | 样本(n=475) | HR | 95% CI | P |
| 年龄 | 475 | 1.018 | 0.999~1.037 | 0.063 |
| 性别 | ||||
| 男 | 256 | |||
| 女 | 219 | 0.822 | 0.571~1.187 | 0.293 |
| 病理分期 | ||||
| Ⅰ | 254 | |||
| Ⅱ | 118 | 2.160 | 1.356~3.441 | 0.001 |
| Ⅲ | 78 | 3.932 | 2.464~6.275 | <0.001 |
| Ⅳ | 25 | 3.809 | 1.926~7.535 | <0.001 |
| 风险亚型 | ||||
| 低风险 | 238 | |||
| 高风险 | 237 | 2.841 | 1.907~4.232 | <0.001 |
单因素和多因素生存分析一致表明,风险亚型对生存结局影响差异有统计学意义,详见图 5、表 1。
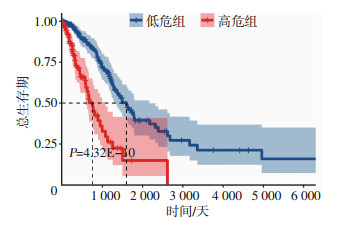
|
| 图 5 Kaplan-Meier单因素生存分析 |
2.3 生信分析
在生信分析中,使用本文纳入的全部数据集(P<0.001)进行基因差异表达分析,选出差异基因40个(包含上调21个、下调19个)。在上调基因中进行排序和筛选,结果提示IGFBP1、ANXA13、MUC2、CIDEC、NTSR1、DSG3为代表性上调基因,这些基因和相应P值如表 2所示。这些代表性上调基因是影响LUAD患者预后的关键靶点,并在既往研究中得到了证实[20-25],可为后续诊治决策提供依据。
| 基因 | P |
| IGFBP1 | 4.01E-25 |
| ANXA13 | 1.19E-19 |
| MUC2 | 7.72E-19 |
| CIDEC | 9.51E-19 |
| NTSR1 | 1.09E-18 |
| DSG3 | 9.15E-18 |
为从整体上了解差异基因的分布,对SSAE方法获得的基因构建火山图和热图,对差异基因分布进行了整体展示,如图 6所示。
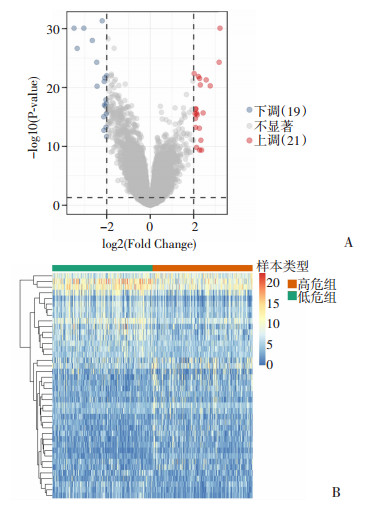
|
| A:火山图 横坐标:示差异倍数(高危组/低危组,取2为底的对数) 纵坐标:示-log10(P.adjust) 图中每个点:示一个转录组学基因 蓝色和红色的点:示显著差异表达基因, 红色的点表示其基因表达量是上调的(高危组/低危组),蓝色的点表示其基因表达量是下调的(高危组/低危组),灰色的点代表这些基因在两组间无显著差异;B:热图 小方格:示基因,其颜色表示该基因表达量大小,表达量越大颜色越深(红色为高表达,蓝色为低表达) 第一行:示样本分组 行:示每个基因不同样本中的表达量情况列表示每个样品中所有差异基因的表达量情况 图 6 生信分析 |
3 讨论
相较于以临床指标数据为基础的探索研究[6-8],基于大数据的基因组、转录组、蛋白组、代谢组等组学研究有利于全面揭示疾病特异性细胞群体的表型和功能,识别癌症诊断的生物标志物和新的可治疗靶点。基于组学数据的预后预测对癌症临床诊治的规划意义较大,已成为当下研究的热点问题。既往关于LUAD预后预测研究较多,但亚型分类的研究较少、且方法较为单一。联合深度学习方法和组学数据以预测癌症预后有广阔的研究前景。
目前,深度学习方法在LUAD的辅助诊治中的应用较少,且多集中于癌症标志物的筛选和亚型分类领域,预后预测的相关研究较为缺乏。GU等[26]利用堆叠去噪自编码器(stacked denoise autoencoder,SDA)对LUAD进行亚型分类,并区分为两种亚型(P=1.00E-02)。LI等[27]选择随机森林、单变量Cox回归等方法评估各个生存相关基因的预后相关性,进而将LUAD区分为高风险组和低风险组。ZHAO等[28]利用机器学习方法计算出LUAD的mRNA干性指数(mRNAsi),并通过加权相关网络分析确定与预后密切相关的关键基因。相较于既往研究,本文模型不仅可检测潜在生物标志物、执行亚型分类,而且在预后预测中有较大的应用潜力。
在现有研究基础上,本文提出了使用高通量、高维度数据预测癌症患者预后生存情况的SSAE,此模型是自编码器[29]和Cox-PH模型[30]的结合和延伸。AE的优势在于重构原始数据以消除噪声,高效提取有用特征,更适用于基因表达数据的降维。在传统的无监督AE中添加Cox回归预测可做到有监督地压缩高维度特征,使特征包含更丰富的生存相关的信息和更低的噪音信息。此外,SSAE模型的栈式结构有利于让前一个自编码器的隐藏层进入下一个自编码器,进而展开更深度的特征压缩和提取。这一方面有利于充分地运用自编码器的特征压缩能力,另一方面可提升模型的预后预测能力。相较于RSF、DeepSurv等既往经典方法,SSAE模型展示出了更好的预后预测能力,在深度神经网络应用于生存分析方面进行了新的拓展。随着更多大规模的研究,SSAE的提出可补充和丰富生存分析的方法学研究,在高通量组学预后预测领域发挥更多优势。
生信分析不仅有利于揭示影响LUAD预后结局的潜在生物标志物,而且可验证SSAE模型在生存预测领域的优异性能。IGFBP1在高风险地区人群中过度表达,提示其可能与LUAD患者的肿瘤学过程有关[20]。在LUAD细胞系(Calu-3、LTEP-a-2和NCI-H1395)中,ANXA13的表达高于正常肺细胞系BEAS2B。ANXA13的缺乏可抑制Calu-3细胞的增殖、侵袭和迁移,ANXA13的过表达可增强NCI-H1395细胞的增殖、侵袭和迁移,这提示ANXA13作为LUAD治疗靶点的可能性[21]。MUC2主要富集于免疫反应和癌症相关信号通路中,提示MUC2与LUAD患者预后相关[22]。ARF-like GTPase 14通过CIDEC/ERK/p38信号通路在LUAD的发病机制中发挥重要作用[23]。NTSR1在TCGA-LUAD肿瘤组织中被鉴定为上调,过表达的NTSR1与LUAD患者生存率低有关[24]。DEG3和KRT14可干扰细胞凋亡途径,被视为区分LUAD和LUSC的关键基因。研究表明,DSG3和KRT14可作为NSCLC患者的诊断标志物[25]。本文方法可精准发现关键基因,为后续癌症机制的研究、治疗靶点的选择奠定基础,具有重要的临床意义。
当然,本研究仍存在一定不足,需在后续研究中进行改进。多组学数据的纳入可为预测提供更多信息,进而有效提升CI等预测指标。此外,本研究的样本量相对有限。在未来研究中,有待于纳入更多来源、更多疾病的数据集,开展多组学、大样本量验证研究,继续提升并优化模型的有效性和泛化性。
综上所述,本文基于堆叠、监督的理念构建了SSAE模型,在LUAD预后预测分析中的性能优于传统方法,具有较强的竞争力。LUAD潜在亚型的区分有助于为患者提供个性化的治疗,进而为临床诊治提供决策支持,具有极大的应用价值和推广前景。
| [1] |
SUNG H, FERLAY J, SIEGEL R L, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. |
| [2] |
SPELLA M, STATHOPOULOS G T. Immune resistance in lung adenocarcinoma[J]. Cancers (Basel), 2021, 13(3): 384. |
| [3] |
ZAPPA C, MOUSA S A. Non-small cell lung cancer: current treatment and future advances[J]. Transl Lung Cancer Res, 2016, 5(3): 288-300. |
| [4] |
COURAUD S, ZALCMAN G, MILLERON B, et al. Lung cancer in never smokers-A review[J]. Eur J Cancer, 2012, 48(9): 1299-1311. |
| [5] |
LIU L P, LU L, ZHAO Q Q, et al. Identification and validation of the pyroptosis-related molecular subtypes of lung adenocarcinoma by bioinformatics and machine learning[J]. Front Cell Dev Biol, 2021, 9: 756340. |
| [6] |
QIU M, YANG S, WANG M, et al. Prognostic evaluation of esophageal cancer patients with stages Ⅰ-Ⅲ[J]. Aging (Albany NY), 2020, 12(14): 14736-14753. |
| [7] |
HOESSEINI A, OFFERMAN M P J, VAN DE WALL-NEECKE B J, et al. Physicians' clinical prediction of survival in head and neck cancer patients in the palliative phase[J]. BMC Palliat Care, 2020, 19(1): 176. |
| [8] |
李娟, 石锐, 余萍, 等. 癌症相关性乏力预测非小细胞肺癌患者手术预后的临床研究[J]. 华西医学, 2013, 28(09): 1411-1414. LI J, SHI R, YU P, et al. A clinical study of predicting role of cancer-related fatigue for the post-surgery prognosis in patients with non-small cell Lung cancer[J]. West China Med J, 2013, 28(9): 1411-1414. |
| [9] |
CHEN X, LAN H, HE D, et al. Analysis of autophagy-related signatures identified two distinct subtypes for evaluating the tumor immune microenvironment and predicting prognosis in ovarian cancer[J]. Front Oncol, 2021, 11: 616133. |
| [10] |
TIAN R, LI Y, LIU Q, et al. Identification and validation of an immune-associated RNA-binding proteins signature to predict clinical outcomes and therapeutic responses in glioma patients[J]. Cancers (Basel), 2021, 13(7): 1730. |
| [11] |
TAYLOR J M G. Random survival forests[J]. J Thorac Oncol, 2011, 6(12): 1974-1975. |
| [12] |
WEYER V, BINDER H. A weighting approach for judging the effect of patient strata on high-dimensional risk prediction signatures[J]. BMC Bioinformatics, 2015, 16(1): 294. |
| [13] |
CHING T, ZHU X, GARMIRE L X. Cox-nnet: An artificial neural network method for prognosis prediction of high-throughput omics data[J]. PLoS Comput Biol, 2018, 14(4): e1006076. |
| [14] |
KATZMAN J L, SHAHAM U, CLONINGER A, et al. DeepSurv: personalized treatment recommender system using a Cox proportional hazards deep neural network[J]. BMC Med Res Methodol, 2018, 18(1): 24. |
| [15] |
李炜, 宋威, 王晨妮, 张雨轩. 标签约束的半监督栈式自编码器分类算法[J]. 小型微型计算机系统, 2019, 40(03): 488-492. LI W, SONG W, WANG C N, et al. Label regularization semi-supervised stacked autoencoder classification algorithm[J]. J Chin Comput Syst, 2019, 40(3): 488-492. |
| [16] |
HUANG Z, JOHNSON T S, HAN Z, et al. Deep learning-based cancer survival prognosis from RNA-seq data: approaches and evaluations[J]. BMC Med Genomics, 2020, 13(S5): 41. |
| [17] |
闫静, 张雪英, 李凤莲, 等. 基于栈式监督AE和可变加权ELM的回归预测算法及应用[J]. 计算机工程: 1-10[2022-12-02]. DOI: 10.19678/j.issn.1000-3428.0062416. YAN J, ZHANG X Y, LI F L, et al. Regression prediction model combining stack supervised AE and variable weighted ELM[J]. Comput Eng, 2022, 48(8)p: 62-69, 76. DOI: 10.19678/j.issn.1000-3428.0062416. |
| [18] |
THOMPSON J A, CHRISTENSEN B C, MARSIT C J. Methylation-to-expression feature models of breast cancer accurately predict overall survival, distant-recurrence free survival, and pathologic complete response in multiple cohorts[J]. Sci Rep, 2018, 8(1): 5190. |
| [19] |
WANG W, ZHANG X, DAI D Q. DeFusion: a denoised network regularization framework for multi-omics integration[J]. Brief Bioinform, 2021, 22(5): bbab057. |
| [20] |
WU Z X, HUANG X, CAI M J, et al. Development and validation of a prognostic index based on genes participating in autophagy in patients with lung adenocarcinoma[J]. Front Oncol, 2022, 11: 799759. |
| [21] |
XUE G, ZHANG C, ZHENG G, et al. Annexin A13 predicts poor prognosis for lung adenocarcinoma patients and accelerates the proliferation and migration of lung adenocarcinoma cells by modulating epithelial-mesenchymal transition[J]. Fundam Clin Pharmacol, 2020, 34(6): 687-696. |
| [22] |
TAO Y, LI Y, LIANG B. Comprehensive analysis of microenvironment-related genes in lung adenocarcinoma[J]. Future Oncol, 2020, 16(24): 1825-1837. |
| [23] |
GUO F, YUAN D, ZHANG J, et al. Silencing of ARL14 gene induces lung adenocarcinoma cells to a dormant state[J]. Front Cell Dev Biol, 2019, 7: 238. |
| [24] |
MAO S, CHEN Z, WU Y, et al. Crosstalk of eight types of rna modification regulators defines tumor microenvironments, cancer hallmarks, and prognosis of lung adenocarcinoma[J]. J Oncol, 2022, 2022: 1-19. |
| [25] |
DONG Y, LI S, SUN X, et al. Desmoglein 3 and keratin 14 for distinguishing between lung adenocarcinoma and lung squamous cell carcinoma[J]. Onco Targets Ther, 2020, Volume 13: 11111-11124. |
| [26] |
GU T, ZHAO X. Integrating multi-platform genomic datasets for kidney renal clear cell carcinoma subtyping using stacked denoising autoencoders[J]. Sci Rep, 2019, 9(1): 16668. |
| [27] |
LI Y, GE D, GU J, et al. A large cohort study identifying a novel prognosis prediction model for lung adenocarcinoma through machine learning strategies[J]. BMC Cancer, 2019, 19(1): 886. |
| [28] |
ZHAO M, CHEN Z, ZHENG Y, et al. Identification of cancer stem cell-related biomarkers in lung adenocarcinoma by stemness index and weighted correlation network analysis[J]. J Cancer Res Clin Oncol, 2020, 146(6): 1463-1472. |
| [29] |
HINTON G E, SALAKHUTDINOV R R. Reducing the dimensionality of data with neural networks[J]. Science, 2006, 313(5786): 504-507. |
| [30] |
LIU Q, HU P. Association analysis of deep genomic features extracted by denoising autoencoders in breast cancer[J]. Cancers, 2019, 11(4): 494. |




