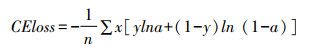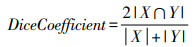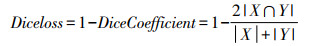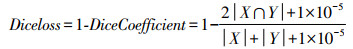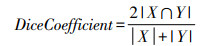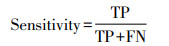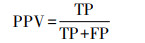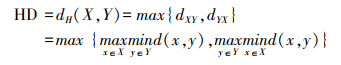2. 400044 重庆,重庆大学:生物工程学院;
3. 400038 重庆,陆军军医大学(第三军医大学)生物医学工程与影像医学系数字医学教研室;
4. 030001 太原,山西医科大学组织胚胎学教研室;
5. 030013 太原,山西省肿瘤医院普外科
2. College of Bioengineering, Chongqing University, Chongqing, 400044;
3. Department of Digital Medicine, Faculty of Biomedical Engineering and Imaging Medicine, Army Medical University (Third Military Medical University), Chongqing, 400038;
4. Department of Histology and Embryology, School of Basic Medicine, Shanxi Medical University, Taiyuan, Shanxi Province, 030001;
5. Department of General Surgery, Shanxi Cancer Hospital, Taiyuan, Shanxi Province, 030013, China
前列腺是男性泌尿生殖系统重要的器官,前列腺病变(如增生、癌变等)为临床常见的疾病且严重影响男性健康,前列腺癌发病率居全球男性恶性肿瘤第2位,仅次于肺癌。在我国前列腺癌发病率位居第六位,并呈现逐年上升趋势[1-2]。磁共振(MRI)检查对软组织分辨能力强,是目前前列腺疾病的主要诊断方法。前列腺的解剖结构较为复杂,包括前列腺纤维基质带(anterior fibrous muscular stroma,AFMS)、中央带(central zone,CZ)、外周带(peripheral zone,PZ)、移行带(transition zone,TZ)4个分带。病变好发于这4个区域,其中癌症好发于PZ,增生好发于TZ。若能准确分割这些区域,则能快速量化这些区域的体积,能够快速辅助检测诊断前列腺增生和前列腺癌等疾病,可大大提高临床医师的诊断工作效率和准确率。但是,传统的手动分割方法耗时耗力,效率低下,经验不足的年轻医师往往存在误诊和漏诊。近年来,国内外基于深度学习用于医学图像分割的方法被广泛报道,如胡学刚等[3]提出了一种基于全卷积DenseNet的分割方法,在公开数据集上Dice相似性系数达到93.25%。KURATA等[4]基于卷积神经网络对122例患者的子宫进行了分割,精度(Dice)达到了82%;LEE等[5]报道了MRI智能分割脑部区域的方法,精度(DSC)达到93%。AFZALI等[6]利用深度学习技术实现了肺部病灶区域的智能精确分割,其左右肺部平均DSC值分别达到97.1%和96.1%。然而,目前人工智能辅助前列腺的分割研究较少。
基于此,本研究利用人工智能深度学习技术提出了以Unet为框架,Vgg16网络作为主干的Vgg16-Unet模型,对前列腺4个分带进行智能分割研究,并可以快速计算四分带体积等参数,有助于辅助检测诊断前列腺疾病。
1 资料与方法 1.1 临床资料收集山西省肿瘤医院放射科2018年1月至2020年10月的50例前列腺癌患者MRI图像。纳入标准:①因前列腺特异性抗原(PSA)增加或直肠指检怀疑PCA而进行MRI扫描;②规范的T2WI序列。排除标准:①内分泌治疗史;②图像质量较差,结构不清晰;③接受前列腺癌根治术或经尿道电切术[7]。最终33例前列腺癌患者MRI影像被纳入研究。此外,还选取了荷兰奈梅亨拉德堡德大学医学中心前列腺MRI公开数据集的346例患者T2WI图像[8]。最终379例前列腺患者被纳入研究。根据临床研究分类法则的随机分配原则[9],保证训练集所有样本不出现在验证集与测试集的前提下,所有患者按7 ∶1 ∶2比例随机划分为训练集(n=265)、验证集(n=38)、测试集(n=76),此研究获山西省肿瘤医院伦理委员会批准(批件号:2021051)。
1.2 数据集构建山西省肿瘤医院患者(n=33)采集轴位T2WI,层厚4.5 mm,公开数据集患者(n=346)T2WI,层厚3.6 mm。设备型号为:GE Signa TwinSpeed (Excite) 1.5 T磁共振,共得到7 766张Dicom图像,其中5 444张训练图像;744张验证图像;1 578张测试图像,其中训练集手工分割图像由2名放射医师勾画核定,其中1名采用Amria软件人工手动逐层标注前列腺前纤维基质带、中央带、外周带、移形带,另外1名对勾勒的手工分割图像进行修改核对。
1.3 方法 1.3.1 实验设备本实验采用的硬件工具为:CPU:AMD A8-5550M APU with Radeon(tm) HD Graphics;GPU:NVIDIA TITAN X(Pascal) 12G。软件工具为:Windows系统;ITK-Snap;MicroDicomViewer;python3.7.1;pytorch+cuda:1.8.0+cu111。
1.3.2 数据预处理前列腺MRI影像中前列腺所占区域面积较小且与其相邻结构相比,其对比度较低,这对于精准分割前列腺区域带来了困难。本研究采用的预处理过程如图 1所示,首先对前列腺MRI图像进行格式转换,通过使用Python(3.7.1)中第三方库pydicom,simpleITK将医学Dicom格式[10]数据转化为JPG图像格式;接着将Dicom中像素归一化到0~1之间,调整Dicom图像的窗宽窗位,将其图像的灰度值转为CT值(单位Hu),本研究中采用的窗位,窗宽分别为336、1 077,最后对开窗过后的图像利用中值滤波器对其进行去噪处理。
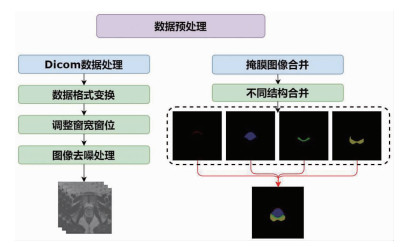
|
| 图 1 前列腺癌患者MRI及手工分割图像数据预处理过程 |
将前列腺AFMS、CZ、PZ、TZ所对应的手工分割合并至一张图像上,经过预处理后的Dicom图像分别与手工分割图像互相对应,随机抽取1张前列腺MRI影像以及其手工分割图像,见图 2。
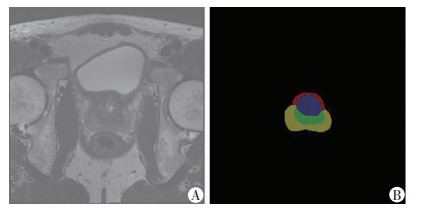
|
| A:经过预处理后的前列腺MRI图像;B:相应手工分割图像 图 2 前列腺癌患者MRI及手工分割图像 |
1.3.3 实验过程
本研究提出了Vgg16-Unet模型,在编码器部分使用了Vgg16网络[11]作为主干,采用3个3×3的卷积核,经过了ReLU(非线性激活函数)层以及BN(正则化层)层,既保证了感受野的相同,增加了模型的非线性表达能力,又可以防止训练过程中出现梯度爆炸以及过拟合现象。训练过程中使用Vgg16网络在ImageNet[12]数据集上将训练好的预训练权重进行迁移学习[13],解码器部分还原Unet网络[14]右半部分(图 3)。
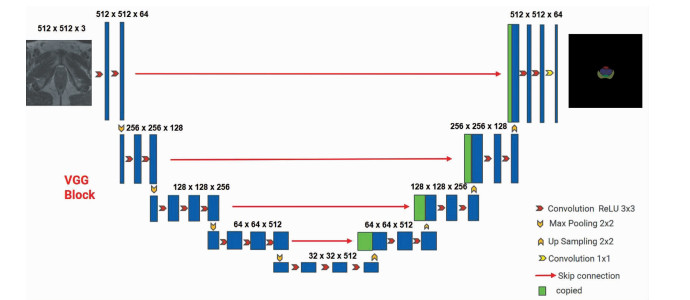
|
| 图 3 Vgg16-Unet模型框架图 |
1.3.3.1 训练过程
训练过程分为2个阶段:冻结阶段;解冻阶段。在冻结阶段,模型的主干被冻结,特征提取网络不变化,此时占用的显存较小仅对网络进行微调。在解冻阶段,模型的主干不被冻结,特征提取网络会发生改变,占用的显存较大,网络所有的参数都会发生改变。通过设计两阶段的训练模式,不仅加速了网络的收敛,同时也提升了模型收敛的效果,模型的具体参数见表 1。
| 层(类别) | 输出 | 填充 | 步长 | 每层参数 |
| 卷积(1) | [64, 512, 512] | 1 | 36 928 | |
| ReLU BN(1) | [64, 512, 512] | |||
| 最大池化(1) | [64, 256, 256] | 2 | ||
| 卷积(2) | [128, 256, 256] | 1 | 147 584 | |
| ReLU BN(2) | [128, 256, 256] | |||
| 最大池化(2) | [128, 128, 128] | 2 | ||
| 卷积(3) | [256, 128, 128] | 1 | 590 080 | |
| ReLU BN(3) | [256, 128, 128] | |||
| 最大池化(3) | [256, 64, 64] | 2 | ||
| 卷积(4) | [512, 64, 64] | 1 | 2 359 808 | |
| ReLU BN(4) | [512, 64, 64] | |||
| 最大池化(4) | [512, 32, 32] | 2 | ||
| 卷积(5) | [512, 32, 32] | 1 | 2 359 808 | |
| ReLU BN(5) | [512, 32, 32] | |||
| 上采样(1) | [512, 64, 64] | |||
| 拼接(1) | [1 024,64,64] | |||
| 卷积(6) | [512, 64, 64] | 1 | 2 359 808 | |
| ReLU BN(6) | [512, 64, 64] | |||
| 上采样(2) | [512, 128, 128] | |||
| 拼接(2) | [768, 128, 128] | |||
| 卷积(7) | [256, 128, 128] | 1 | 590 080 | |
| ReLU BN(7) | [256, 128, 128] | |||
| 上采样(3) | [256, 256, 256] | |||
| 拼接(3) | [384, 256, 256] | |||
| 卷积(8) | [128, 256, 256] | 1 | 147 584 | |
| ReLU BN(8) | [128, 256, 256] | |||
| 上采样(4) | [128, 512, 512] | |||
| 拼接(4) | [192, 512, 512] | |||
| 卷积(9) | [64, 512, 512] | 1 | 36 928 | |
| ReLU BN(9) | [64, 512, 512] | |||
| 卷积(10) | [5, 512, 512] | 1 | 325 |
1.3.3.2 Vgg16-Unet模型架构
Vgg16-Unet网络由编码器和解码器两部分组成,编码器模块采用Vgg16网络部分,由5层卷积层与4层最大池化层组成(图 4)。输入图像经过编码器编码后会得到4个由浅层到深层的不同尺度的特征信息,然后通过Unet中的“跳跃”连接与解码器模块中相同尺度的特征图像在通道方向上进行“融合”,弥补了图像在下采样过程中丢失的边缘信息。解码器部分主要由上采样模块和卷积层构成,其中上采样模块采用的是双线性插值[15](bilinear interpolation)方法,且上采样的次数与编码器中池化层数目相对同。输入图像经过编码器部分的特征提取过后,传入解码器中再通过多次上采样的过程,最终得到与输入图像等同大小的分割预测图像。
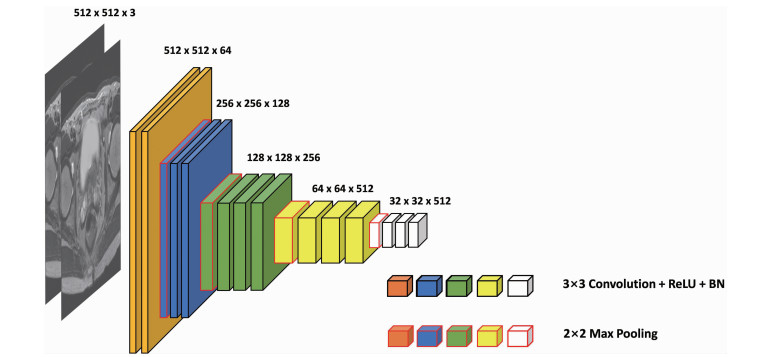
|
| 图 4 Vgg16-Unet主干网络框架 |
1.3.3.3 训练细节和参数设置
模型训练部分:初始学习率为0.01,学习率下降策略采用余弦退火方式(cosine annealing);优化器方面,选取带有动量的SGD优化器,动量(momentum)为0.9,权重衰减(weight decay)为1×10-4,训练过程中冻结阶段和解冻阶段的Batchsize分别为10、8,训练轮次为100轮,前50轮为冻结阶段,后50轮为解冻阶段,选择损失最小的模型保存其权重。
1.3.4 损失函数本实验采用以1 ∶3加权比例混合Dice损失函数、Cross Entropy交叉熵损失函数作为损失函数。
Cross Entropy交叉熵损失函数经常用于分类问题中,而且由于交叉熵涉及到计算每个类别的概率,本实验归根到底可以理解为4分类问题,因此搭配Softmax函数进行损失计算。Cross Entropy交叉熵损失函数公式如下。
其中CEloss:损失值;n:Batchsize;x:预测向量维度;y:经过独热编码后的真实值对应x维度上的标签,1或0;a:独热编码格式输出的预测标签,1或0(经过softmax函数)。
Dice系数是一种相似度度量函数,通常用来评判两个样本之间的相似度,值域为[0, 1],Dice系数越接近于1,表明分割精度越高,公式如下。
其中,|X∩Y|表示两个集合的交集,|X+Y|表示两个集合的并集。对于本实验任务而言,X,Y分别表示手工分割与网络预测图像。Dice损失函数的计算公式如下。
为了防止出现零除以及过拟合现象,在Diceloss的基础上添加了smooth指标,本实验中smooth=1×10-5,因此Dice损失函数计算公式如下。
1.3.5 评价指标本研究主要以医学图像分割常用的评价指标[16]对不同网络的预测结果与手工分割之间进行对比评价,公式如下。
① Dice系数:
② Sensitivity灵敏度:
③ PPV阳性预测率:
④ HD豪斯多夫距离:
其中TP、TN、FP、FN分别指真实正样本、真实负样本、假正样本、假负样本的数量。为了消除非常小的离群值子集的影响,本实验中使用的是HD95,即公式(7)的结果HD的值乘以95%。
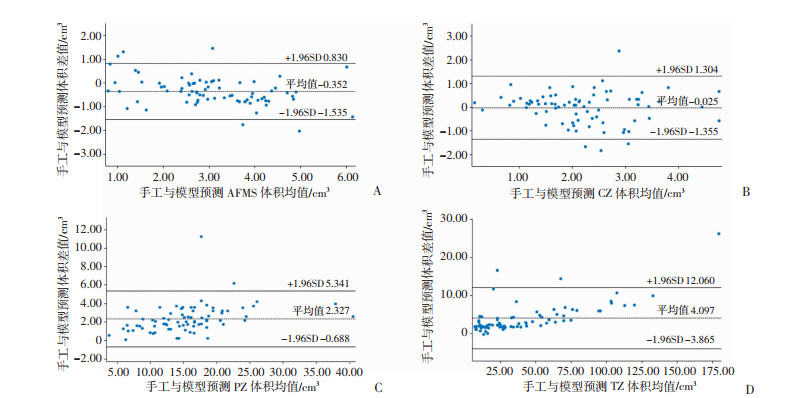
2 结果 2.1 各模型在测试集上的分割结果本研究构建的Vgg16-Unet模型,在测试集上进行推理,对比Unet、Unet++、ResUnet++ 3种经典Unet与其衍生模型,结果如表 2、3所示,模型对AFMS、CZ、PZ、TZ 4个区域实现了较为准确的分割,其平均DSC值、95%HD距离分别为:56.95%、47.28%、82.75%、89.28%和20.84、20.02、15.39、11.20 mm。图 5所示为测试集上Vgg16-Unet模型预测和医师手工标注的前列腺4个分带的体积的Bland-Altman一致性分析,二者一致性较好,大部分差值均位于95%CI内。
| 评价指标 模型 | AFMS | CZ | PZ | TZ |
| 相似性系数DSC(%) | ||||
| Vgg16-Unet | 56.95 | 47.28 | 82.75 | 90.63 |
| Unet++ | 56.75 | 43.07 | 80.62 | 89.09 |
| Unet | 56.92 | 43.24 | 80.58 | 88.85 |
| ResUnet++ | 53.28 | 43.38 | 79.94 | 87.87 |
| 灵敏度Sensitivity(%) | ||||
| Vgg16-Unet | 62.58 | 43.65 | 80.98 | 87.90 |
| Unet++ | 57.42 | 41.40 | 79.40 | 90.36 |
| Unet | 62.82 | 42.94 | 81.70 | 87.19 |
| ResUnet++ | 69.35 | 51.17 | 80.35 | 85.72 |
| 阳性预测率PPV(%) | ||||
| Vgg16-Unet | 52.25 | 43.28 | 83.35 | 90.17 |
| Unet++ | 56.09 | 44.88 | 80.26 | 87.85 |
| Unet | 52.03 | 43.54 | 79.49 | 90.58 |
| ResUnet++ | 43.26 | 37.65 | 79.55 | 90.12 |
| 95%豪斯多夫距离HD95/mm | ||||
| Vgg16-Unet | 20.84 | 20.02 | 15.39 | 11.20 |
| Unet++ | 20.83 | 21.13 | 15.76 | 12.78 |
| Unet | 20.78 | 21.20 | 17.10 | 12.64 |
| ResUnet++ | 19.99 | 20.38 | 15.79 | 13.26 |
| 模型 | AFMS | P | CZ | P | PZ | P | TZ | P |
| Vgg16-Unet | 2.50±1.26 | 0.001 | 1.92±1.18 | 0.764 | 15.31±7.28 | 0.001 | 37.01±30.75 | 0.001 |
| Unet++ | 2.93±1.40 | 0.029 | 1.76±1.32 | 0.053 | 15.12±7.13 | 0.001 | 34.64±29.83 | 0.001 |
| Unet | 2.97±1.30 | 0.001 | 1.92±1.13 | 0.956 | 15.46±7.83 | 0.001 | 34.47±29.90 | 0.001 |
| ResUnet++ | 3.94±1.66 | 0.001 | 2.60±1.37 | 0.001 | 15.24±6.87 | 0.001 | 33.99±29.41 | 0.001 |
| 手工计算体积 | 2.68±1.72 | 0.001 | 2.11±1.32 | 0.001 | 15.13±8.75 | 0.001 | 36.69±31.22 | 0.001 |
|
| A:模型预测与手工标注AFMS体积均值差值;B:模型预测与手工标注CZ体积均值差值;C:模型预测与手工标注PZ体积均值差值;D:模型预测与手工标注TZ体积均值差值 图 5 Vgg16-Unet模型预测与手工标注的前列腺分带体积差值比较 |
2.2 各模型训练过程收敛情况
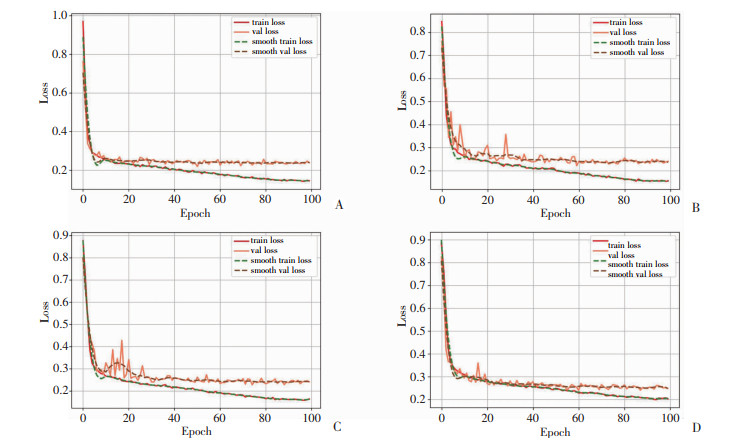
经过比对不同模型在相同超参数下训练过程的收敛情况,Vgg16-Unet模型在整个训练过程中保持了相对稳定的状态,收敛速度远远快于Unet、Unet++、Res-Unet++3个模型,其在约20个epoch处模型就已经完成拟合,大大缩短了训练周期,从而提高了前列腺分区的分割效率(图 6)。
|
| A:Vgg16-Unet模型训练过程收敛情况;B:Unet模型训练过程收敛情况;C:Unet++模型训练过程收敛情况;D:Res-Unet++模型训练过程收敛情况 图 6 各模型训练过程中收敛速率和稳定性对比 |
2.3 智能分割与手工分割对比
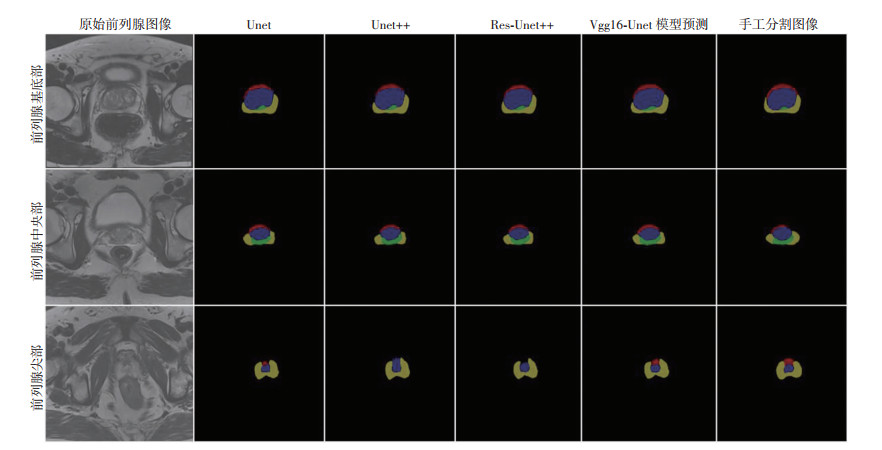
为了更加直观地比较Vgg16-Unet模型与其他模型分割效果,在测试集中以前列腺基底部、中央部、尖部为3个平面,选取3张MRI影像的分割结果进行可视化对比展示(图 7)。在前列腺尖部的分割中Vgg16-Unet模型,能更精准地捕获小结构的边缘信息。总体看来,模型对整个测试集的推理结果与手工分割图像保持了较高的一致性。
|
| 图 7 各模型前列腺分带分割可视化结果比较 |
3 讨论 3.1 智能分割结果分析
近年来,人工智能深度学习在医学图像处理领域应用广泛,主要体现人工智能辅助医学图像分割、疾病智能诊断、疾病预后评估等方面[17]。本研究基于深度学习方法,构建Vgg16-Unet模型,对前列腺MRI图像进行了更为精细的分割研究,深化其临床应用。本研究结果表明:前列腺4个分带的智能分割结果能够快速计算前列腺分区体积,基本满足了临床应用,有助于临床医师快速检测诊断前列腺癌和前列腺增生等疾病,能够节省医疗资源,减轻医师工作量,提高诊断效率。
从网络的分割效果来看,Unet模型虽然分割精度较稳定,但其分割结果的假阳性较高,细小特征的提取效果不好;ResUet++和Unet++模型加深了网络的层数,同时分别采用了残差结构、密集连接,填补了Unet“跳跃”连接中空白的部分,但是随着网络层数的加深,不仅增加了计算参数量,而且在训练过程中出现了过拟合现象,反而导致其分割结果较差。相比于经典的Unet模型,Vgg16-Unet模型引入了Vgg16的特征提取模块,多次采用3×3的卷积核,在保证相同感受野的同时,提升了模型在下采样过程中的多尺度特征提取能力,还充分利用了Imagenet数据集上训练好的预训练权重进行迁移学习,大大加快了模型的收敛速度,显著提升了模型的分割效率。模型在PZ、TZ区域分割的效果较好,其DSC值分别达到了82.75%、90.63%,95%Hausdorff距离分别控制在15.39、11.20 mm,而对AFMS、CZ的分割效果则不理想,其DSC值分别为56.95%、47.28%,95%Hausdorff距离达到了20.84、20.02 mm,估计为数据层厚较大,分区体积较小所导致,正如REN等[18-19]报道分割目标体积小,分割的难度越大,精度越低。本实验结果与刘想等[20]的智能分割结果基本一致。
3.2 创新与不足之处目前关于前列腺智能分割大部分研究都是针对易分割区域展开的,如KHAN等 [21]的研究中将前列腺CZ、TZ部分合并为CG(中央腺体),对前列腺两个大结构区域分割,PZ和CG的最高DSC值分别达到了78.9%、92.8%;ZAVALA-ROMERO等[22]在组合模型和单模型之间进行消融实验,最终在前列腺PZ上分割的DSC值最高达到了81.1%;BARDIS等[23]在前列腺PZ、TZ区域上实现了智能分割,其精度(DSC值)达到77.4%、91.0%;同样,CUOCOLO等[24]在PZ和TZ上的分割精度(DSC值)分别为71.8%和87.5%,而LI等[25]只对前列腺整个区域进行分割研究,在测试集上分割准确率(DSC值)达到了91.03%。然而,这些智能分割研究主要针对整个前列腺、外周带或移形带,但并未对中央带和膀胱下基质进行分割。本研究对前列腺进行了更细致的分割,包括AFMS、CZ、PZ、TZ 4个区域,更能够获取前列腺包括各个分区详细的解剖和病理信息,精细化的分割对前列腺疾病,包括前列腺癌和良性前列腺增生等诊断更有意义。
然而,本研究仍存在不足之处:①基于患者的DICOM图像较少,层厚较厚,以及GPU算力受限,实验未使用3D深度学习模型,忽略了图像的空间信息,分割精度仍有待提高;②单中心数据量较少,样本量仍需扩大。今后我们将从2D/3D维度,选取大样本、多中心数据,加强前列腺分区的智能分割研究。
本研究构建的Vgg16-Unet模型实现了对前列腺4个分区的精准分割,可以快速计算分区体积,有助于临床医师快速检测诊断前列腺癌、前列腺增生等疾病,提高诊断效率,节省医疗资源,减轻医师工作量。
| [1] |
XIA C F, DONG X S, LI H, et al. Cancer statistics in China and United States, 2022: profiles, trends, and determinants[J]. Chin Med J (Engl), 2022, 135(5): 584-590. |
| [2] |
SIEGELR L, MILLER K D, JEMAL A. Cancer statistics, 2015[J]. CA A Cancer J Clin, 2015, 65(1): 5-29. |
| [3] |
胡学刚, 杨洪光. 基于全卷积DenseNet的前列腺MRI分割新方法[J]. 计算机应用研究, 2020, 37(6): 1886-1889, 1894. HU X G, YANG H G. Novel prostate MRI segmentation method based on full convolutionDenseNet[J]. Appl Res Comput, 2020, 37(6): 1886-1889, 1894. |
| [4] |
KURATA Y, NISHIO M, KIDO A, et al. Automatic segmentation of the uterus on MRI using a convolutional neural network[J]. Comput Biol Med, 2019, 114: 103438. |
| [5] |
LEE B, YAMANAKKANAVAR N, CHOIJ Y. Automatic segmentation of brain MRI using a novel patch-wise U-net deep architecture[J]. PLoS One, 2020, 15(8): e0236493. |
| [6] |
AFZALI A, BABAPOUR MOFRAD F, POULADIAN M. 2D statistical lung shape analysis using chest radiographs: modelling and segmentation[J]. J Digit Imaging, 2021, 34(3): 523-540. |
| [7] |
ZABIHOLLAHY F, SCHIEDA N, KRISHNA JEYARAJ S, et al. Automated segmentation of prostate zonal anatomy on T2-weighted (T2W) and apparent diffusion coefficient (ADC) map MR images using U-Nets[J]. Med Phys, 2019, 46(7): 3078-3090. |
| [8] |
LITJENSG, DEBATS O, BARENTSZ J, et al. Computer-aided detection of prostate cancer in MRI[J]. IEEE Trans Med Imaging, 2014, 33(5): 1083-1092. |
| [9] |
GRIMES D A, SCHULZ K F. An overview of clinical research: the lay of the land[J]. Lancet, 2002, 359(9300): 57-61. |
| [10] |
刘燕茹, 罗利霞, 刘明芳. Windows系统上DICOM图像开窗显示方法实现[J]. 通讯世界, 2020, 27(1): 150. LIU Y R, LUO L X, LIU M F. Realization of window display method of DICOM image on Windows system[J]. Telecom World, 2020, 27(1): 150. |
| [11] |
ALSHAMMARI A. Construction of VGG16 convolution neural Network (VGG16_CNN) classifier with NestNet-based segmentation paradigm for brain metastasis classification[J]. Sensors (Basel), 2022, 22(20): 8076. |
| [12] |
DENG J, DONG W, SOCHER R, et al. ImageNet: a large-scale hierarchical image database[C]//2009 IEEE Conference on computer vision and pattern recognition, 2009: 248-255. DOI: 10.1109/CVPR.2009.5206848.
|
| [13] |
HEK M, GIRSHICK R, DOLLÁR P. Rethinking ImageNet pre-training[EB/OL]. [2018-11-21]. https://arxiv.org/abs/1811.08883.
|
| [14] |
YU J, CAI L, CHEN C, et al. Cascade Path Augmentation Unet for bladder cancer segmentation in MRI[J]. Med Phys, 2022, 49(7): 4622-4631. |
| [15] |
KIRKLAND E J. Bilinear interpolation[M]//Advanced computing in electron microscopy. Boston: Springer US, 2010: 261-263. DOI: 10.1007/978-1-4419-6533-2_12.
|
| [16] |
VUM H, GRIMBERGEN G, NYHOLM T, et al. Evaluation of multislice inputs to convolutional neural networks for medical image segmentation[J]. Med Phys, 2020, 47(12): 6216-6231. |
| [17] |
吴毅, 张小勤. 人工智能在医学图像处理中的研究进展与展望[J]. 第三军医大学学报, 2021, 43(18): 1707-1712. WU Y, ZHANG X Q. Artificial intelligence in medical image processing: progress andprospect[J]. J Third Mil Med Univ, 2021, 43(18): 1707-1712. |
| [18] |
REN X H, XIANG L, NIE D, et al. Interleaved 3D-CNNs for joint segmentation of small-volume structures in head and neck CT images[J]. Med Phys, 2018, 45(5): 2063-2075. |
| [19] |
吴哲, 庞亚, 明智, 等. 人工智能技术在鼻咽癌放疗危及器官自动勾画中的应用研究[J]. 实用肿瘤学杂志, 2021, 35(2): 137-141. WU Z, PANG Y, MING Z, et al. Application of artificial intelligence technology in automatic delineation of organs at risk in nasopharyngeal carcinoma radiotherapy[J]. Pract Oncol J, 2021, 35(2): 137-141. |
| [20] |
刘想, 高歌, 韩超, 等. 基于3D U-Net模型实现T2WI图像中前列腺的区域分割及临床验证[J]. 中国临床医学影像杂志, 2022, 33(1): 33-38, 42. LIU X, GAO G, HAN C, et al. Regional segmentation of prostate and clinical evaluation in T2WI image based on 3D U-Net model[J]. J China Clin Med Imaging, 2022, 33(1): 33-38, 42. |
| [21] |
KHAN Z, YAHYA N, ALSAIH K, et al. Evaluation of deep neural networks for semantic segmentation of prostate in T2W MRI[J]. Sensors (Basel), 2020, 20(11): 3183. |
| [22] |
ZAVALA-ROMERO O, BRETOA L, XU I R, et al. Segmentation of prostate and prostate zones using deep learning: a multi-MRI vendor analysis[J]. Al, 2020, 196(10): 932-942. |
| [23] |
BARDIS M, HOUSHYAR R, CHANTADULY C, et al. Segmentation of the prostate transition zone and peripheral zone on MR images with deep learning[J]. Radiol Imaging Cancer, 2021, 3(3): e200024. |
| [24] |
CUOCOLO R, COMELLI A, STEFANO A, et al. Deep learning whole-gland and zonal prostate segmentation on a public MRI dataset[J]. J Magn Reson Imaging, 2021, 54(2): 452-459. |
| [25] |
LI Y C, LIN C, ZHANG Y, et al. Automatic segmentation of prostate MRI based on 3D pyramid pooling Unet[J]. Med Phys, 2023, 50(2): 906-921. |