胃癌是我国癌症相关死亡的第三大癌种[1],其中肠型胃癌是最为常见的胃癌亚型,其发生规律遵循Correa模式,即浅表性胃炎→慢性萎缩性胃炎→肠上皮化生→异型增生→胃癌[2]。胃黏膜肠上皮化生(intestinal metaplasia, IM)(以下简称肠化)作为肠型胃癌的重要癌前状态,是胃癌发生的独立高危因素[3],然而肠化发生的调控机制尚未完全阐明。
肠化是指正常胃黏膜细胞转换为类似肠上皮细胞形态的疾病阶段,该过程胃黏膜细胞的基因表达模式重编程为肠上皮细胞表达谱,导致细胞表型重塑[4]。肠化组织仅有极少量的基因突变、拷贝数变异和染色体改变[5],提示非基因组序列变化的基因表达调控机制,比如表观遗传状态失衡,可能是肠化发生的重要原因。
组蛋白修饰是一种重要的表观调控机制,以组蛋白H3K27ac(组蛋白H3第27位赖氨酸乙酰化修饰)为标记的增强子(enhancer)是其调控的重要方式之一[6-7]。增强子是一类远端DNA顺式作用元件,增强子区域包含转录因子结合基序,通过招募谱系特异性转录因子、辅因子以及染色质重塑复合物,提高启动子活性,可以激活或增强基因转录水平[8],从而调控细胞命运、器官发育和疾病发生。大量研究表明,组蛋白H3K27ac修饰是活性增强子的典型标志,可用于识别和鉴定基因组中的活性增强子[9-13]。由于表观调控可能在肠化细胞转录重塑和表型转换中发挥关键作用,本研究采用新近报道的少量细胞染色质靶向捕获(Cleavage Under Targets and Tagmentation, CUT&Tag)测序技术,检测全基因组水平的组蛋白H3K27ac修饰状态,结合高通量测序,首次系统绘制肠化组织的增强子重编程图谱,并初步解析增强子调控肠化基因表达的转录因子网络,揭示肠化发生的表观遗传新机制。
1 材料与方法 1.1 临床生物样本本研究收集21例正常胃窦黏膜、41例胃窦肠化组织,均来自本院内镜检查和治疗患者。肠化组织判断标准为白光内镜下的绒毛样改变、黏膜发白、黏膜表面粗糙、颗粒状不平,以及高清放大内镜下的亮蓝嵴及白色不透明物质等特征[14-15]。液氮保存收集样本,组织病理类型均由两名病理医师确认,内镜下特征与病理结果相一致的样本用于后续检测。本研究已通过陆军特色医学中心伦理审批[医研伦审(2021)第260号],样本收集前已获患者知情同意。
1.2 CUT&Tag文库构建及测序将冻存胃黏膜组织解冻后研磨,通过碘沙醇梯度溶液提取细胞核后用台盼蓝染色计数,收集100K的细胞核使用Hyperactive© Universal CUT&Tag Assay Kit for Illumina(Vazyme #TD903)试剂盒,采用H3K27ac抗体(Abclonal,货号A7253),参考试剂盒标准实验流程,构建文库并上机测序。
1.3 转录组测序抽提RNA后并质控(RIN值≥7,RNA含量≥1 μg)后,用Ribo-Zero rRNA Removal Reagent (H/M/R) (Illumina, USA)去除rRNA,并对提取的RNA进行片段化。逆转录合成双链cDNA,连接接头并扩增,纯化产物后再次质检,并上机测序。
1.4 CUT&Tag seq及RNA-seq数据分析利用以下工具或软件包进行:fastp对测序数据进行质控,cutadapt去接头,hisat2进行基因组比对,sambamba进行去重。随后调用MACS寻找H3K27ac富集的基因组区域,然后用edgeR鉴定差异H3K27ac信号(差异峰:差异倍数≥2倍、FDR<0.05)。采用featureCount统计基因表达量,进一步用edgeR进行差异表达分析(差异基因:差异倍数≥2倍、FDR<0.05)。在评估高变异增强子基因座(variant enhancer loci, VEL)与基因表达关系时,参考既往研究策略[13],将预测的增强子分配给临近靶基因,即在启动子H3K27ac富集区域,转录起始位点(transcription start site, TSS)的500 bp内进行注释。
1.5 功能富集(Gene Ontology, GO)分析及转录因子调控网络绘制GO富集:调用clusterProfiler工具对存在VEL且表达水平同向变化的基因进行GO分析,P<0.05时认为差异具有统计学意义。使用HOMER软件富集目标区域的转录因子结合基序,预测增强子相关转录因子,使用Cytoscape软件绘制转录因子调控网络。
2 结果 2.1 高质量表观组和转录组测序数据获取用于绘制正常胃黏膜和肠化组织H3K27ac修饰的全基因组分布图谱为描述肠化组织的组蛋白H3K27ac修饰特征,对正常胃窦黏膜及胃窦肠化组织进行H3K27ac CUT&Tag及RNA-seq建库和测序(图 1A)。质控分析显示:CUT&Tag测序数据Reads数均大于5×106个、Peak数均大于2×104个、FRiP(fraction of reads in peaks)分数大于10%;RNA-seq数据Reads数均大于5×107个、核糖体RNA比例小于10%;表观组和转录组数据的基因组比对率均大于90%、重复率均小于30%(图 1B)。因此,两种组织的两个组学数据均达到质控标准,数据质量均一、差异无统计学意义,满足下游分析要求(图 1B)。分析H3K27ac修饰的基因组分布情况,发现两种组织的H3K27ac信号都主要分布在启动子、基因间及内含子区域(图 1C),即正常胃黏膜和肠化组织H3K27ac修饰的全基因组分布特征无明显差异,并且与其他组织类型的分布情况相似[9]。

|
| A: 研究流程图:胃镜收集21例正常胃窦黏膜及41例胃窦肠化黏膜,经病理证实后进行H3K27ac CUT&Tag-seq及RNA-seq;B: H3K27ac CUT&Tag测序数据和RNA-seq数据的总Reads数、比对率、重复率及FRiP值/rRNA比例 C: H3K27ac信号在各样本基因组分布的全景图谱 图 1 CUT&Tag测序绘制胃黏膜肠化组织的H3K27ac修饰全景图谱 |
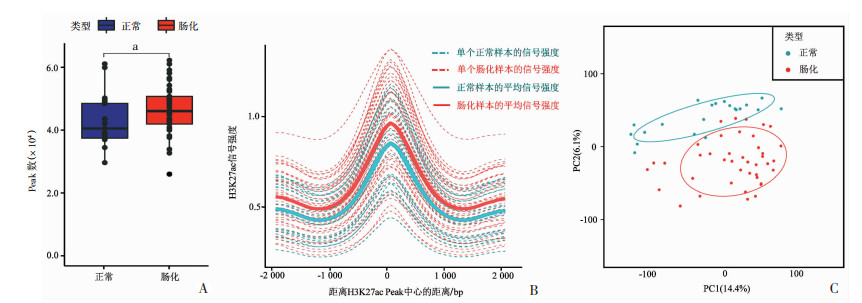
2.2 肠化组织H3K27ac信号峰的数量和强度均显著高于正常胃黏膜
分析全基因组范围内H3K27ac修饰的数量和信号强度发现,与正常胃黏膜相比,肠化组织H3K27ac信号峰不仅数量更多(肠化组织46 767个,正常胃黏膜42 853个,P=0.033, 图 2A),并且信号强度明显增加(图 2B)。对所有样本H3K27ac修饰数据进行主成分分析(Principal Component Analysis, PCA),降维结果显示,肠化组织H3K27ac修饰与正常胃黏膜的全局性特征明显不同(图 2C)。这些结果提示,肠化过程发生了广泛的H3K27ac修饰重塑,这种表观遗传改变可能是调控肠化基因表达的重要原因。
|
| A: 正常及肠化组织H3K27ac CUT&Tag-seq的Peak数a:P<0.05;B: 所有样本H3K27ac峰信号强度分布图;C: 依据H3K27ac修饰对各样本进行的主成分分析 图 2 肠化组织发生H3K27ac修饰重编程,导致增强子信号强度显著增加 |
2.3 肠化组织的增强子重编程可能调控肠化相关基因表达及其细胞生物学功能
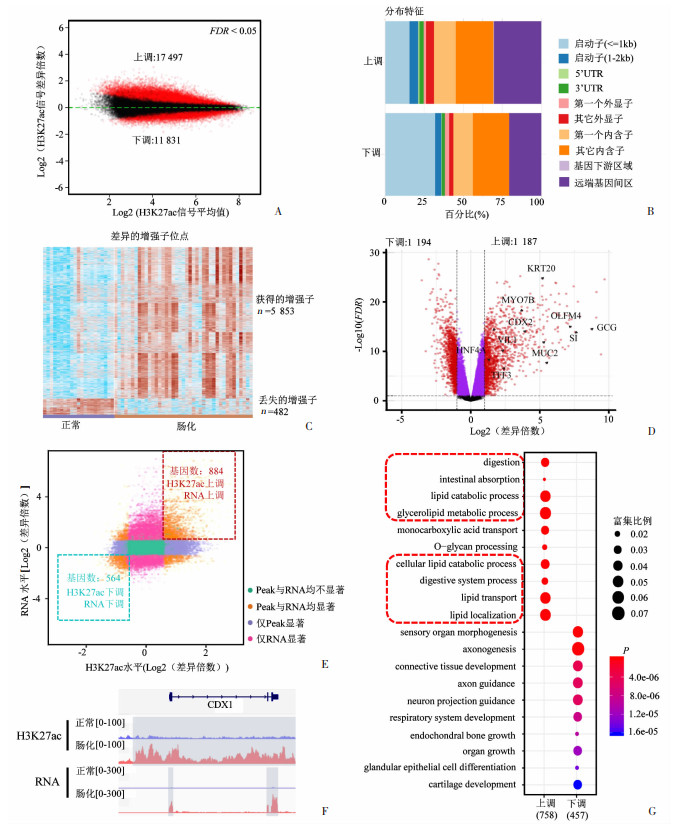
两种组织中存在差异的H3K27ac信号峰为29 328个,占全部信号峰数量的26.2%(图 3A)。值得注意的是,肠化组织显著增强的H3K27ac信号峰大部分远离启动子,位于基因间及内含子区,即增强子区域[16](图 3B)。因此,我们去除启动子区域(即距离转录起始位点2 kb内[12])的H3K27ac信号峰,只关注增强子区域。两种组织差异的高变异增强子基因座(VEL)为6 335个,其中肠化组织中5 853个增强子的强度显著升高,占所有VEL的92.4%(图 3C)。这表明增强子活性增加或重编程是肠化的典型表观遗传学特征,可能在上调肠化相关基因表达中发挥重要作用。
|
| A: 正常胃黏膜及肠化组织的H3K27ac差异信号FDR<0.05;B: 肠化组织上调及下调的H3K27ac信号的基因组分布;C: 热图显示肠化相比正常胃黏膜获得与丢失的高变异增强子基因座FC≥ 2,FDR<0.05;D: 火山图显示肠化组织的差异基因表达谱FC≥ 2,FDR<0.05;E: 肠化差异基因的H3K27ac信号与mRNA表达水平;F: IGV可视化展示肠化组织中CDX1基因的增强子H3K27ac修饰水平与RNA表达丰度;G: GO分析增强子可能调控的肠化基因集及其生物学功能 图 3 H3K27ac增强子重编程可能调控肠化相关转录谱征及信号通路活性 |
增强子调控的肠化相关基因至少满足两个特征:该基因增强子H3K27ac信号升高,且mRNA表达水平同步上调。转录组分析发现,肠化组织1 187个基因表达显著上调,包括MUC2、TFF3、VIL1、KRT20、GCC、SI、MYO7B、OLFM4等已知的肠道特异性基因,以及CDX1、CDX2、HNF4A等转录因子[17-20];肠化组织表达下调基因为1 194个(图 3D)。整合分析肠化组织的增强子谱征和差异表达基因,发现884个基因增强子信号与基因表达水平同步升高、564个基因增强子信号与基因表达水平均下调,分别占肠化差异表达基因总数的29.1%和17.4%(图 3E)。以公认的肠化标志物CDX1[3]为例,IGV可视化显示肠化组织中其增强子信号明显升高,伴随RNA表达上调,提示增强子可能激活CDX1基因表达(图 3F)。进一步GO分析提示,这些增强子上调基因主要富集于消化吸收、营养物质转运和代谢等肠上皮细胞生物学过程(图 3G)。这些数据提示增强子重编程可能调控肠化上皮的细胞生物学功能。
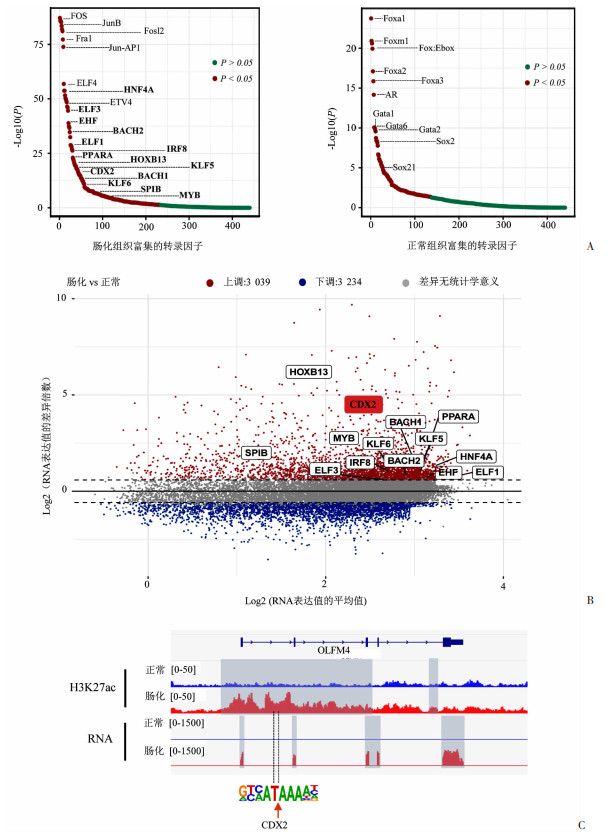
2.4 CDX2等增强子相关转录因子形成调控网络,可能在增强子重编程驱动胃黏膜肠化过程中发挥重要作用增强子通过其转录因子结合基序,招募转录因子激活基因表达。分析肠化相关增强子的转录因子结合基序发现,肠化组织获得(Gain)的增强子区域富集了CDX2、HNF4α、KLF5等14个肠上皮细胞功能相关的转录因子[17, 21];而肠化组织丢失(Loss)的增强子区域富含SOX2、SOX21等11个胃黏膜细胞功能相关的转录因子[22](图 4A)。这些肠上皮相关转录因子在肠化组织中表达上调(图 4C),可能发挥重要调控作用。比如,CDX2可能结合肠化标志基因OLFM4[19]的增强子,转录激活OLFM4基因表达(图 4B),但具体的调控作用仍需进一步的实验验证。上述结果不仅发现肠化组织中多个新的增强子相关转录因子,也揭示肠化过程存在胃-肠黏膜功能相关的转录因子转换现象。
|
| A: 获得与丢失的VEL区域上分别富集的转录因子,P<0.05的基因标记为红色;B: 肠化增强子基因座富集的转录因子中共有14个在肠化组织中高表达;C: IGV可视化展示增强子可能与转录因子CDX2协同调控肠化基因OLFM4表达 图 4 增强子区域可能募集CDX2等转录因子调控肠化相关基因表达 |
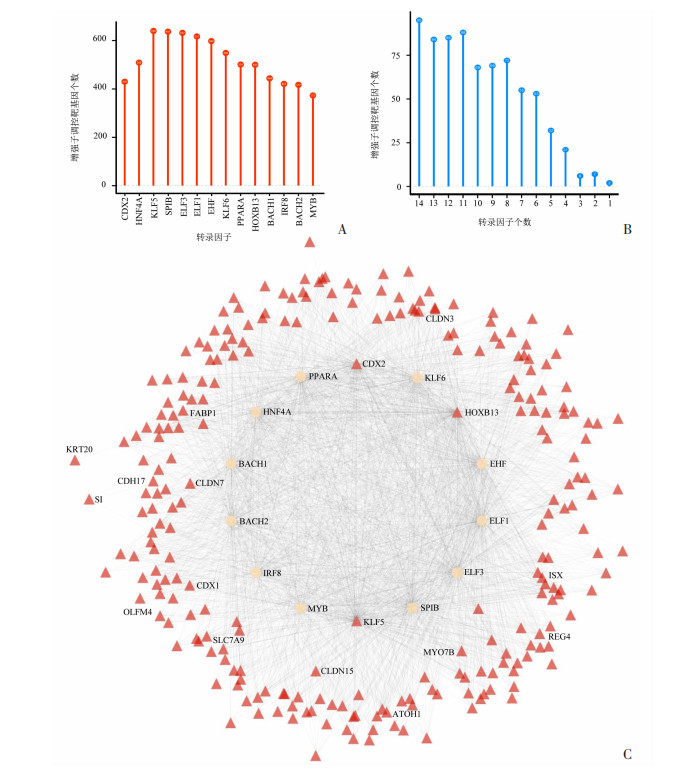
细胞表型转换过程通常由多个转录因子形成网络,协同调控基因表达谱[23]。分析发现每个肠化相关转录因子均有可能调控400~600个基因(图 5A),但肠化组织这类增强子激活的基因仅有884个(图 3E,右上象限红框所示),提示肠化过程中多个转录因子可能共同结合某个/某些增强子,协同调控肠化基因表达。因此,我们绘制转录因子调控网络,发现这些转录因子可能通过广泛互作,协同调控肠化基因表达(图 5C),转录因子之间及转录因子与目的基因之间的明确调控关系尚待进一步验证。有意思的是,在转录因子调控网络中,逐一随机去除某个转录因子,增强子调控的基因数量呈非线性降低趋势(图 5B)。因此,增强子重编程可能通过招募多个转录因子组成调控网络,共同决定肠化相关基因表达。
|
| A: 增强子相关转录因子调控的潜在肠化靶基因数量;B: 在14个转录因子组成的调控网络中,按一次一个的方式依次随机去除转录因子后,网络中受增强子调控的靶基因数目;C: 潜在肠化相关转录因子的调控网络内圈:示增强子相关的转录因子外圈:示可能受其调控的下游靶基因(FC ≥ 2) 图 5 增强子相关转录因子与其可能的靶基因共同形成肠化的转录调控网络 |
3 讨论
肠化胃黏膜的表型和功能发生显著变化,但肠化的表观遗传修饰状态,及其调控机理尚待深入研究。本研究利用少量细胞CUT&Tag测序技术,系统描绘肠化组织中组蛋白H3K27ac修饰的全基因组分布情况,发现肠化组织的增强子活性显著增加。既往研究显示,肠化组织的基因组DNA甲基化水平高于正常组织[5]。DNA甲基化修饰通常发生在CpG岛,导致染色质凝聚为异染色质,阻止转录起始复合物结合基因启动子区域,也影响增强子活性,进而抑制基因表达[7]。肠化关键转录因子CDX2则可以上调增强子活性,调控肠化表型[20]。这些结果提示肠化过程中存在增强子活性改变,但缺乏系统研究。本研究发现增强子重编程是肠化过程的一个重要表观修饰特征,因此DNA水平甲基化修饰、组蛋白水平增强子重塑等多种表观调控机制可能相互对话,协同调控肠化基因表达,共同决定肠化细胞的分化谱系。
增强子重编程可能通过招募14个转录因子形成互作网络,调控肠化相关基因表达。除CDX2和HNF4α为已有报道外,本研究发现KLF5、HOXB13和ELF3等12个转录因子可能调控肠化相关基因表达,进而参与胃黏膜肠化过程。这些新发现的转录因子在肠道上皮发育、表型维持中发挥关键作用[21, 24-25]。KLF5是胚胎内胚层祖细胞转录因子,KLF5基因敲除小鼠的肠黏膜杯状细胞和肠内分泌细胞分化障碍、顶部刷状缘缺失[26],HOXB13是远端结肠和直肠正常上皮细胞标志物,ELF3则调节小肠上皮细胞形态发生和终末分化。这些研究提示,胃黏膜肠化是一个异常的细胞分化过程,在增强子的作用下多个肠上皮细胞发育相关转录因子异常激活,调控下游基因表达,导致胃黏膜细胞部分获得肠上皮细胞表型和功能。本研究还发现CDX2可能是重要的调控枢纽,其在调控肠化相关靶基因的同时还受到多个转录因子的调控。另外KLF5可能通过增强子受到自身调节,符合核心转录调控环中关键转录因子的特征,说明其可能在肠化的过程中发挥更加重要的作用。
在这个转录因子网络中,CDX2和HNF4α持续活化不仅诱导肠化表型,也促进肠型胃癌恶性生物学行为,与胃癌患者临床预后密切相关[11, 13]。因此深入研究其它转录因子是否影响肠化向肠型胃癌的进展过程,有望揭示新的调控机制和干预靶标。
综上所述,本研究首次系统绘制胃黏膜肠化的H3K27ac增强子重编程图谱,发现增强子可能重塑以14个转录因子为核心的互作网络,进一步调控肠化相关基因表达。由于胃黏膜肠化是肠型胃癌发生的重要病理阶段[2],深入揭示其表观调控规律有助于解析肠型胃癌发生机制,对于胃癌早期预防具有一定理论意义。
| [1] |
ZHENG R S, ZHANG S W, ZENG H M, et al. Cancer incidence and mortality in China, 2016[J]. J Natl Cancer Cent, 2022, 2(1): 1-9. |
| [2] |
BUSINELLO G, ANGERILLI V, PARENTE P, et al. Molecular landscapes of gastric pre-neoplastic and pre-invasive lesions[J]. Int J Mol Sci, 2021, 22(18): 9950. |
| [3] |
CHEN H Y, HU Y, LU N H, et al. Caudal type homeoboxes as a driving force in helicobacter pylori infection-induced gastric intestinal metaplasia[J]. Gut Microbes, 2020, 12(1): 1-12. |
| [4] |
LARIO S, RAMÍREZ-LÁZARO M J, BRUNET-VEGA A, et al. Coding and non-coding co-expression network analysis identifies key modules and driver genes associated with precursor lesions of gastric cancer[J]. Genomics, 2022, 114(3): 110370. |
| [5] |
HUANG K K, RAMNARAYANAN K, ZHU F, et al. Genomic and epigenomic profiling of high-risk intestinal Metaplasia reveals molecular determinants of progression to gastric cancer[J]. Cancer Cell, 2018, 33(1): 137-150.e5. |
| [6] |
HÖRNBLAD A, REMESEIRO S. Epigenetics, enhancer function and 3D chromatin organization in reprogramming to pluripotency[J]. Cells, 2022, 11(9): 1404. |
| [7] |
CARTER B, ZHAO K J. The epigenetic basis of cellular heterogeneity[J]. Nat Rev Genet, 2021, 22(4): 235-250. |
| [8] |
SHLYUEVA D, STAMPFEL G, STARK A. Transcriptional enhancers: from properties to genome-wide predictions[J]. Nat Rev Genet, 2014, 15(4): 272-286. |
| [9] |
LI Q L, LIN X, YU Y L, et al. Genome-wide profiling in colorectal cancer identifies PHF19 and TBC1D16 as oncogenic super enhancers[J]. Nat Commun, 2021, 12(1): 6407. |
| [10] |
HUANG H, HU J Y, MARYAM A, et al. Defining super-enhancer landscape in triple-negative breast cancer by multi-omic profiling[J]. Nat Commun, 2021, 12(1): 2242. |
| [11] |
SHENG T T, HO S W T, OOI W F, et al. Integrative epigenomic and high-throughput functional enhancer profiling reveals determinants of enhancer heterogeneity in gastric cancer[J]. Genome Med, 2021, 13(1): 158. |
| [12] |
COHEN A J, SAIAKHOVA A, CORRADIN O, et al. Hotspots of aberrant enhancer activity punctuate the colorectal cancer epigenome[J]. Nat Commun, 2017, 8: 14400. |
| [13] |
OOI W F, XING M J, XU C, et al. Epigenomic profiling of primary gastric adenocarcinoma reveals super-enhancer heterogeneity[J]. Nat Commun, 2016, 7: 12983. |
| [14] |
FUKUTA N, IDA K, KATO T, et al. Endoscopic diagnosis of gastric intestinal metaplasia: a prospective multicenter study[J]. Dig Endosc, 2013, 25(5): 526-534. |
| [15] |
SHAH S C. Improving the endoscopic detection and management of gastric intestinal Metaplasia through training: a practical guide[J]. Gastroenterology, 2022, 163(4): S0016-S5085(22)00897-6[pii]. |
| [16] |
ANDERSSON R, SANDELIN A. Determinants of enhancer and promoter activities of regulatory elements[J]. Nat Rev Genet, 2020, 21(2): 71-87. |
| [17] |
HE Q J, LIU L M, WEI J G, et al. Roles and action mechanisms of bile acid-induced gastric intestinal metaplasia: a review[J]. Cell Death Discov, 2022, 8(1): 158. |
| [18] |
KOIDE T, KOYANAGI-AOI M, UEHARA K, et al. CDX2-induced intestinal metaplasia in human gastric organoids derived from induced pluripotent stem cells[J]. iScience, 2022, 25(5): 104314. |
| [19] |
ZHANG P, YANG M R, ZHANG Y D, et al. Dissecting the single-cell transcriptome network underlying gastric premalignant lesions and early gastric cancer[J]. Cell Rep, 2019, 27(6): S2211-S1247(19)30525-X[pii]. |
| [20] |
SINGH H, SERUGGIA D, MADHA S, et al. Transcription factor-mediated intestinal metaplasia and the role of a shadow enhancer[J]. Genes Dev, 2022, 36(1/2): 38-52. |
| [21] |
FUJII Y, YOSHIHASHI K, SUZUKI H, et al. CDX1 confers intestinal phenotype on gastric epithelial cells via induction of stemness-associated reprogramming factors SALL4 and KLF5[J]. Proc Natl Acad Sci USA, 2012, 109(50): 20584-20589. |
| [22] |
SINGH H, HA K, HORNICK J L, et al. Hybrid stomach-intestinal chromatin states underlie human Barrett's metaplasia[J]. Gastroenterology, 2021, 161(3): 924-939.e11. |
| [23] |
CHEN Y, XU L, LIN R Y, et al. Core transcriptional regulatory circuitries in cancer[J]. Oncogene, 2020, 39(43): 6633-6646. |
| [24] |
JEDLICKA P, GUTIERREZ-HARTMANN A. Ets transcription factors in intestinal morphogenesis, homeostasis and disease[J]. Histol Histopathol, 2008, 23(11): 1417-1424. |
| [25] |
MARRA G. An "expressionistic" look at serrated precancerous colorectal lesions[J]. Diagn Pathol, 2021, 16(1): 4. |
| [26] |
BELL S M, ZHANG L Q, XU Y, et al. Kruppel-like factor 5 controls villus formation and initiation of cytodifferentiation in the embryonic intestinal epithelium[J]. Dev Biol, 2013, 375(2): 128-139. |




