肠上皮化生(intestinal metaplasia, IM)(以下简称肠化)是正常胃黏膜细胞被类似肠上皮形态的细胞所取代的病理阶段[1-3],是一种重要的肠型胃癌癌前状态。流行病学研究显示,肠化的诱因包括慢性幽门螺杆菌感染、胆汁反流、烟酒摄入以及饮食结构不均衡等。新近研究发现,肠化上皮具有与正常胃黏膜上皮截然不同的转录组特征[4],但肠化发生的分子调控机制尚不甚清楚,目前也缺乏逆转肠化的有效治疗方法。由于肠化组织的基因组序列改变较少(染色体拷贝数变化和突变负荷极低),提示表观遗传状态紊乱可能是肠化发生的重要原因之一,但这个领域尚缺乏深入研究[5]。
表观遗传调控指在基因组碱基序列不变的情况下,决定基因在何时何处(时空特异性)表达的调控机制,组蛋白修饰是其中一种重要形式。以沉默子(silencer)为代表的远距调控元件是一种重要的抑制性表观调控方式[6],由多梳抑制复合体2 (polycomb repressive complex 2,PRC2)催化的组蛋白H3上第27位赖氨酸三甲基化修饰(H3K27me3)所标记[7]。沉默子区域的H3K27me3修饰可招募抑制性调节因子,改变染色质凝聚状态或阻碍激活因子结合,进而关闭临近基因转录程序,调控细胞分化/细胞命运决定、器官发育和肿瘤发生发展等生理和病理过程[6, 8-9]。
与正常胃黏膜细胞相比,肠化细胞的形态和功能发生明显转换,提示沉默子等表观修饰状态可能发生显著变化。因此,本研究利用针对H3K27me3修饰的染色质靶向捕获(CUT&Tag)测序技术,系统描绘胃黏膜肠化这一病理过程中的全基因组沉默子分布特征;整合转录组数据,探讨沉默子对肠化相关基因的调控机理。为深入认识肠化发生机制,开发阻断肠化进展的肠型胃癌预防策略提供新思路。
1 对象与方法 1.1 研究对象课题收集22例人正常胃窦样本和39例人胃窦肠化样本,均由陆军特色医学中心消化内科提供。纳入标准:①年龄、性别不限;②白光内镜下观察到红白相间(以白为主)的胃窦黏膜(肠化黏膜),ME-NBI观察可见特征性亮蓝嵴[10];③ HE染色显示肠化的经典病理改变,包括胃黏膜内散在分布的杯状细胞、刷状缘的形成等。排除标准:孕妇、凝血功能障碍者。样本由内镜下活检获取后液氮保存。临床样本采集方案已由陆军特色医学中心伦理委员会通过[医研伦审(2021)第260号]。
1.2 方法 1.2.1 冻存组织细胞核的提取取冰冻样本(胃窦正常黏膜或肠化黏膜),采用Omni-ATAC实验流程提取组织细胞核[11],台盼蓝染色检查细胞核完整性并计数,稀释至105个,用于后续CUT&Tag文库构建。
1.2.2 CUT&Tag文库构建和测序按诺唯赞TD903试剂盒说明书进行。将提取的细胞核用H3K27me3抗体及二抗识别目标DNA序列,并用pA-Tn5捕获目标DNA序列,同时片段化。随后,将纯化产物进行PCR扩增,并将纯化PCR产物进行浓度测定,完成CUT&Tag文库构建,采用Illumina HiSeq-X测序仪进行测序。
1.2.3 RNA文库构建和测序将正常胃窦样本和肠化样本进行总RNA的提取并检测RNA是否降解(RIN值≥ 7),获得足够量(1~4 μg)的合格RNA样品。利用oligo dT磁珠与mRNA的poly(A)尾特异性结合从而去除其他RNA,获得mRNA样品,同时对提取的mRNA进行片段化。随后,合成并纯化双链cDNA,连接接头并纯化产物进行PCR扩增,对PCR产物进行纯化后得到文库。文库质检合格后,上机测序。
1.2.4 CUT&Tag测序数据处理利用Fastp联合cutadpat去除接头(adaptor);通过使用Hisat2工具将去除接头的测序数据比对到基因组上,该步骤可查看比对率,并得到比对后的SAM文件;利用sambamba将SAM文件转换为BAM文件,进一步用Picard去除重复的reads,得到应用于后续分析的unique reads。因H3K27me3 CUT&Tag数据有部分存在于启动子,在对沉默子进行研究前,采用bedtools工具将H3K27me3 CUT&Tag数据进行启动子区(距离基因转录起始位点2 kb范围内的DNA区域)去除后,再进行后续操作[12]。
1.2.5 利用Ngsplot对沉默子信号进行可视化利用MACS2对过滤后的数据进行Peak calling,根据功能元件的基因组坐标处理BAM文件,计算TSS、peak和genebody区域的富集信号丰度,绘制峰线图。
1.2.6 利用ChIPseeker注释基因组上的沉默子分布情况利用Macs peak calling分析后得到的BED文件,同时准备好基因组注释库,加载ChIPseeker包,对H3K27me3信号峰所在位置进行注释,执行plotAnnoBar和plotAnnoPie两条命令对信号峰所在的位置信息进行可视化。
1.2.7 整合分析CUT&Tag和RNA-seq数据利用edgeR对RNA和H3K27me3 CUT&Tag数据进行差异分析,得到差异基因表达以及差异染色质区域的矩阵,再整合差异染色质状态所调控的靶基因,进行可视化分析。统计沉默子信号强度减弱且表达上调的靶基因,以及沉默子信号强度增强且表达下调的靶基因,分别进行GO以及KEGG分析。
1.2.8 转录因子结合基序富集分析以及转录因子调控网络的绘制利用edgeR工具分析肠化及正常组织的转录组差异,整合沉默子信号减弱区域,确定用于预测的基因组区间范围。利用Homer预测沉默子信号丢失区域的转录因子基序,除限定区间范围,其他均使用默认参数。根据Q-value富集程度对转录因子进行排名,展示前10个富集程度最高的转录因子基序;选取在肠化组织高表达的转录因子,绘制调控网络图。
2 结果 2.1 采用H3K27me3 CUT&Tag测序技术,获得全基因组沉默子分布图谱本研究收集肠化胃窦黏膜和正常胃窦黏膜,进行H3K27me3 CUT&Tag及RNA-seq测序(图 1A)。全面分析测序数据的质控指标显示(图 1B),样本DNA片段数(reads number)均大于5×106,提示获得足够多H3K27me3修饰信号,满足下游分析需求;基因组比对率(mapping rate)均大于90%,提示文库无显著的接头污染等情况;片段重复率(duplication rate)均小于30%,提示文库复杂性良好;信号峰数量(peak count)均大于1×104,表明捕获H3K27me3信号有较集中的富集趋势;位于信号峰区域的DNA片段比例(fraction of reads in peaks, FRiP)均大于25%,提示捕获到的沉默子信号富集在染色质上特定区域,可用于下游分析;正常与肠化组织的质控指标上无显著差异,可排除数据质量不均一导致假阳性结果。RNA数据质控分析显示,目标片段在基因编码区高度富集,且RNA-seq测序深度足够(图 1C)。
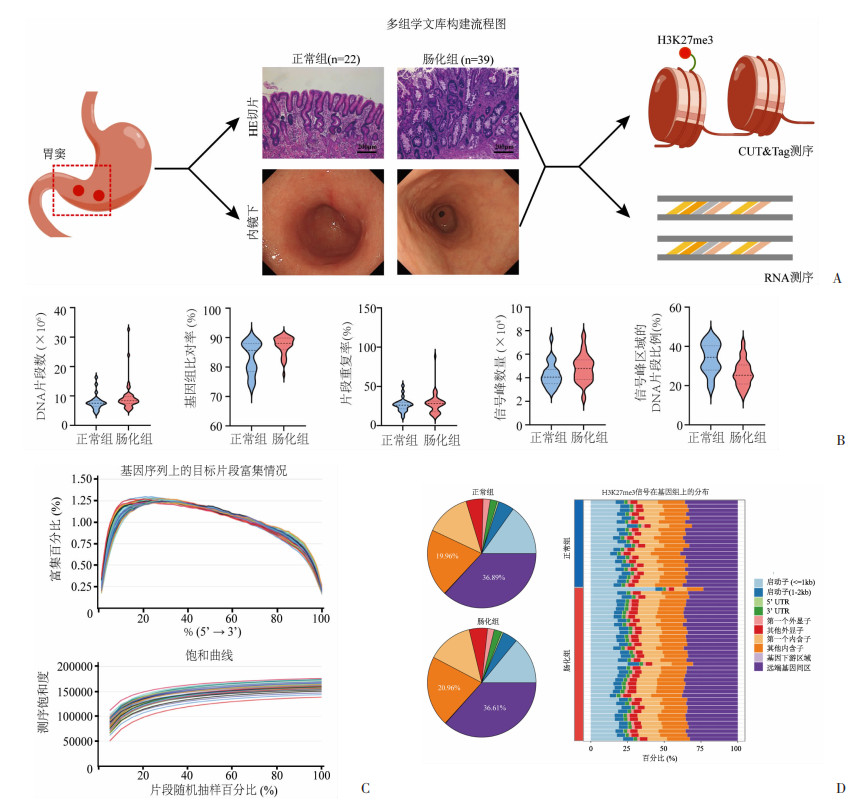
|
| A: 胃窦正常组织、肠化组织的H3K27me3 CUT&Tag、RNA测序流程图;B: H3K27me3 CUT&Tag文库质量的质控分析 从左至右:总体DNA片段数、比对率、重复序列比例、信号峰数量、信号峰区域的DNA片段比例;C: RNA测序数据的质控分析 上:Reads片段在基因编码区上的分布;下:转录组测序深度稀释图;D: H3K27me3沉默子信号在各(组)样本基因组上分布 左:各组整体沉默子信号的分布;右:各样本沉默子信号的分布 图 1 高质量H3K27me3 CUT&Tag及RNA-seq测序数据和H3K27me3修饰的基因组分布特征 |
分析H3K27me3修饰的基因组分布表明,两种组织的沉默子均主要分布在远端的基因间区以及非编码的内含子区域(图 1D),这与既往报道染色质上的沉默子定位特征一致。因此,两种组学数据质量较好,全面展示了两种组织中沉默子信号的全基因组分布图谱。
2.2 肠化组织沉默子信号强度显著降低,与正常组织相比发生了显著的沉默子重塑对比沉默子信号强度发现,肠化组织的平均信号强度显著低于正常组织(肠化组织为0.69,正常组织为0.80,P < 0.05),说明肠化组织存在沉默子丢失现象(图 2A)。为分析肠化组织中全基因组的沉默子信号丢失区域,我们利用MAnorm2工具对H3K27me3测序信号进行标准化处理,鉴定了肠化组织与正常组织之间信号强度显著差异的H3K27me3修饰位点。统计结果表明,肠化组织在15 881个基因组区域上获得了沉默子信号,在14 962个基因组区域上丢失了沉默子信号。这些数据说明,肠化组织不仅存在沉默子获得事件,同时存在大量的沉默子丢失现象(图 2B)。
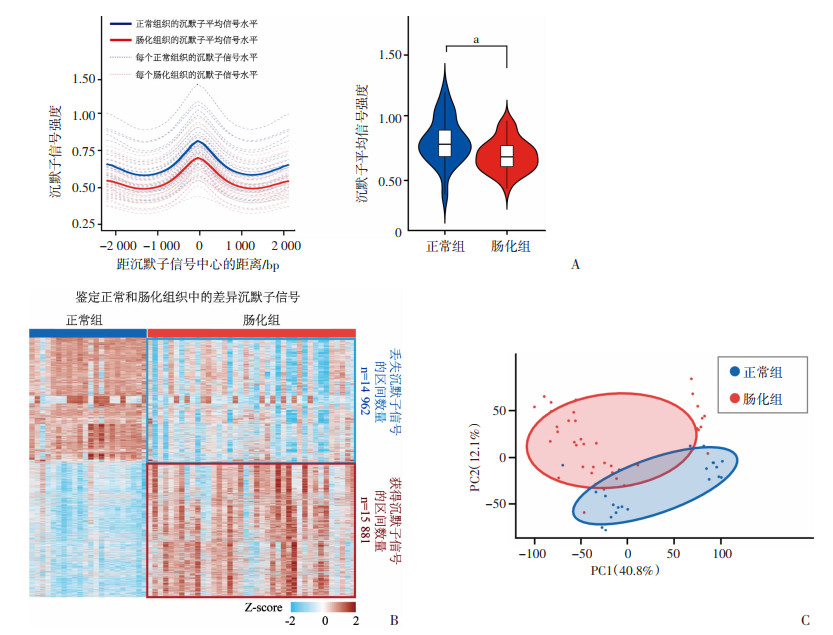
|
| A: 胃窦正常与肠化组织中沉默子信号强度的可视化(左)以及量化比较(右) a: P < 0.05;B: 热图展示两组样本间差异的沉默子信号 FC > 1.5, FDR < 0.05;C.依据沉默子特征对各样本进行的主成分分析 图 2 肠化组织中重塑的H3K27me3沉默子信号 |
对两组样本的H3K27me3沉默子信号进行主成分分析(PCA analysis)降维展示,发现肠化组织的沉默子特征与正常组织显著不同(图 2C)。上述结果表明,正常组织和肠化组织之间的沉默子信号存在显著差异,肠化组织的沉默子信号强度整体降低,发生沉默子重塑。
2.3 沉默子重塑可能调控肠化相关基因表达及肠化细胞的多种代谢通路由于沉默子是下调基因转录活性的重要表观调控机制,我们进一步筛选肠化组织中沉默子调控的下游靶基因。两种组织转录组存在显著差异:肠化组织中有1 449个基因表达上调,包括CDX1、CDX2、TFF3、MUC2、HNF4α、ATOH1[13]和ISX[14]等肠上皮黏膜表达的基因;肠化组织下调的基因有1 312个(图 3A)。整合分析沉默子数据与差异表达基因,发现872个基因沉默子信号上升且表达水平降低,占肠化低表达基因的66.5%;197个基因沉默子信号下调且表达水平升高,占肠化高表达基因的13.6%(图 3B),提示沉默子重塑可能在调控肠化组织基因表达中发挥重要作用。使用IGV工具可视化展示已知肠化标志物CDX1基因[15]的沉默子信号和RNA测序数据,发现肠化组织CDX1基因H3K27me3信号下调,伴随RNA表达水平上调,提示CDX1表达可能受沉默子调控(图 3C)。统计肠化组织可能受沉默子调控的所有基因,KEGG分析显示,沉默子重塑可能调控多条代谢通路和细胞生物学功能,比如上调脂肪酸降解和丙酮酸代谢通路;同时也可能下调胃酸分泌相关通路(图 3D)。这些代谢通路与肠化表型密切相关[16-20],提示沉默子重塑可能通过调控肠化相关基因表达,影响肠化细胞的物质代谢和生物学过程。
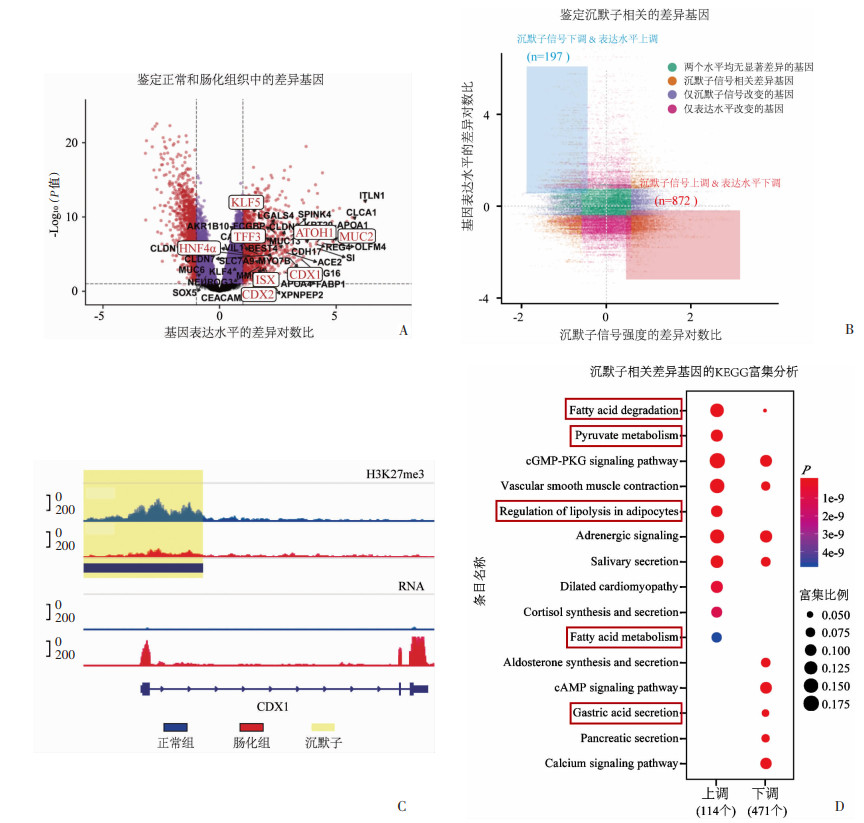
|
| A: 胃窦正常与肠化组织中的差异表达基因 FC>2,FDRP < 0.05;B: 各基因的沉默子信号与表达水平;C: 肠化特异性基因CDX1在正常和肠化组织中的沉默子信号及转录水平;D: 受沉默子调控的基因的KEGG富集分析 图 3 沉默子相关靶基因及生物学功能 |
2.4 转录因子ATOH1和ONECUT2可能被招募到沉默子丢失区域,形成转录因子网络,进而调控肠化相关基因表达
组蛋白H3K27me3修饰丢失后,沉默子区域染色质开放性增加,有利于招募转录因子,激活基因表达。分析肠化组织沉默子丢失区域的转录因子结合基序(TF motif),发现一系列转录因子可能被招募,图 4A中列举了排名前10位的预测转录因子。由于ATOH1和ONECUT2在肠化组织中表达水平显著上调(图 4B),提示它们可能调控肠化相关基因表达。我们聚焦这两个转录因子,比对全基因组沉默子信号减弱区域(图 4C),发现:①ATOH1和ONECUT2均可能调控自身表达,形成正反馈环路;②ATOH1和ONECUT2均可能调控其余8个转录因子;③ONECUT2可能调控ATOH1表达;④ATOH1和ONECUT2各自可能调控一系列重要的肠化相关基因。比如,沉默子丢失可能招募ATOH1至CDX1基因调控区域,直接上调肠化关键转录因子CDX1表达(图 4D)。
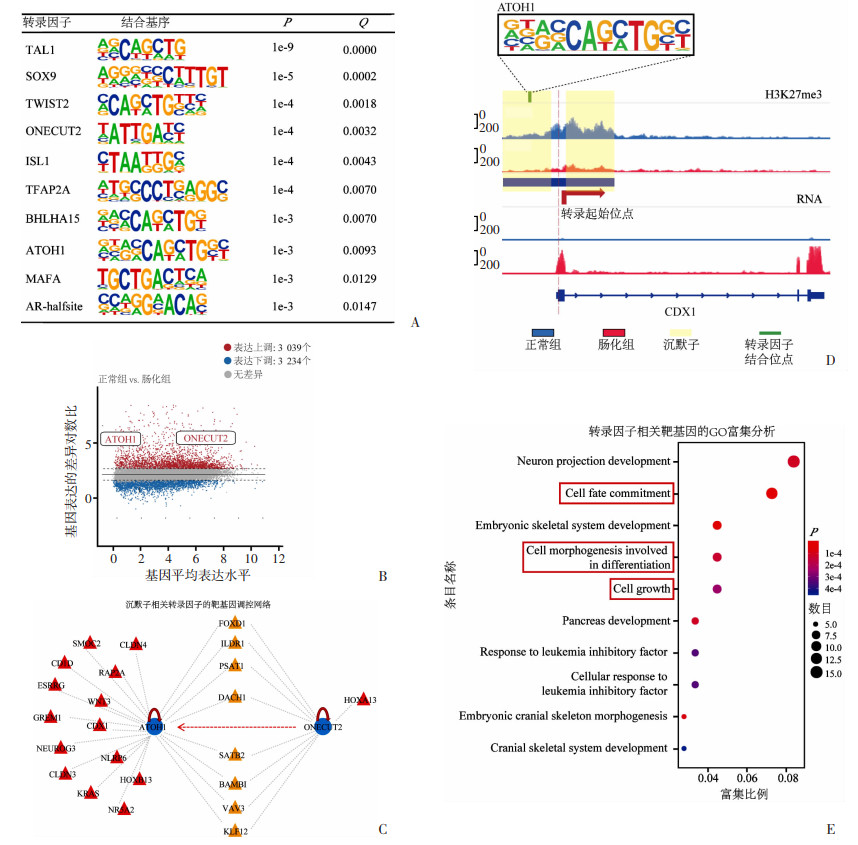
|
| A: 沉默子信号丢失的基因组区域上的转录因子富集情况 B: 沉默子相关转录因子ATOH1和ONECUT2在胃窦肠化样本中表达水平显著上调 FC>1.5, FDRP < 0.05;C: 转录因子ATOH1与ONECUT2可能调控的沉默子丢失基因,其中黄色标记同时受到二者调控的靶基因;D: ATOH1促进CDX1表达的沉默子调控机制;E: ATOH1与ONECUT2潜在调控的沉默子丢失基因的GO富集分析 图 4 沉默子相关转录因子ATOH1和ONECUT2调控肠化相关基因的机制 |
对ATOH1和ONECUT2可能调控的靶基因进行GO-BP分析,发现这些基因主要参与细胞命运决定、分化发育等重要生物学过程(图 4E)。以上数据提示,肠化过程中沉默子重编程,可能异常激活多个转录因子,导致胃黏膜细胞分化谱系异常,而其中ATOH1和ONECUT2这两个转录因子可能发挥关键调控作用。
3 讨论胃黏膜肠化是一种重要的肠型胃癌癌前状态,较难逆转,显著增加癌变风险,是胃癌发生的关键病理阶段[3]。表观修饰如DNA甲基化水平在肠化组织出现全局性改变,与肠化进展密切相关[5],但其它表观遗传学机制如组蛋白修饰如何调控肠化进程尚未全面探索。本研究系统描绘了肠化组织的全基因组沉默子分布图谱,首次报道沉默子重编程是肠化的重要表观调控特征。
组蛋白修饰可通过染色质免疫共沉淀技术(ChIP-seq)、CUT&Tag-seq以及CUT&RUN-seq等测序技术检测,其中ChIP-seq应用最为广泛,但所需样本量大。近年来新兴测序技术如CUT&Tag-seq和CUT&RUN-seq具有多重优势,比如步骤简便、实验周期短、建库样本的细胞量要求低、信号峰特异性好、成本低廉[21-22]。这两项新兴技术原理相似,CUT&Tag-seq是CUT&RUN-seq的升级版,采用Tn5转座酶复合物,切割染色质同时增加建库引物接头,简化建库步骤,可快速获得高质量信号峰[22-23]。由于内镜活检组织的细胞量较少,本研究采用CUT&Tag-seq技术鉴定沉默子。
本研究发现肠化组织的沉默子信号显著降低,发生广泛的重编程,可能调控大量肠化相关基因,影响胃黏膜上皮的营养物质代谢和胃酸分泌过程。沉默子与DNA甲基化,均为抑制性表观调控方式,两者在胚胎发育、肿瘤发生等过程中协同发挥作用[24-25]。既往研究发现肠化组织的启动子DNA高甲基化与肠化程度呈正相关[5],而本研究发现肠化组织远端调控元件的沉默子重塑特征,提示两者可能协同作用,在肠化过程中联合发挥抑制性表观调控效应。
肠化过程中多个转录因子可能被招募至组蛋白H3K27me3修饰信号丢失区域,其中ATOH1与ONECUT2可能形成互作网络,调控肠化相关基因表达。ATOH1表达于胚胎发育期的肠道黏膜上皮细胞,受NOTCH信号激活,进而诱导肠上皮干细胞分化,维持潘氏细胞、杯状细胞和内分泌细胞数量[26]。此外,Barrett食管组织中ATOH1被肠上皮转录因子CDX2诱导,参与肠化过程[27]。这些结果提示,除已知的CDX1/2等转录因子外,ATOH1与ONECUT2也可能参与调控胃黏膜肠化细胞的表型和功能转换。
综上,胃黏膜肠化组织存在全基因组沉默子重塑为特征的表观修饰紊乱。沉默子重塑可能招募ATOH1与ONECUT2等转录因子,形成调控网络,影响肠化相关基因表达。这些发现有助于深入理解胃黏膜肠化的发生规律,为开发表观靶向药物,逆转肠化和预防胃癌提供了新思路。
| [1] |
CORREA P. Human gastric carcinogenesis: a multistep and multifactorial process: first American cancer society award lecture on cancer epidemiology and prevention[J]. Cancer Res, 1992, 52(24): 6735-6740. |
| [2] |
GOLDENRING J R, MILLS J C. Cellular plasticity, reprogramming, and regeneration: metaplasia in the stomach and beyond[J]. Gastroenterology, 2022, 162(2): 415-430. |
| [3] |
GIROUX V, RUSTGI A K. Metaplasia: tissue injury adaptation and a precursor to the dysplasia-cancer sequence[J]. Nat Rev Cancer, 2017, 17(10): 594-604. |
| [4] |
LARIO S, RAMÍREZ-LÁZARO M J, BRUNET-VEGA A, et al. Coding and non-coding co-expression network analysis identifies key modules and driver genes associated with precursor lesions of gastric cancer[J]. Genomics, 2022, 114(3): 110370. |
| [5] |
HUANG KK, RAMNARAYANAN K, ZHU F, et al. Genomic and epigenomic profiling of high-risk intestinal metaplasia reveals molecular determinants of progression to gastric cancer[J]. Cancer Cell, 2018, 33(1): 137-150. e5. |
| [6] |
SEGERT J A, GISSELBRECHT SS, BULYK M L. Transcriptional silencers: driving gene expression with the brakes on[J]. Trends Genet, 2021, 37(6): 514-527. |
| [7] |
HUANG D, PETRYKOWSKA H M, MILLER B F, et al. Identification of human silencers by correlating cross-tissue epigenetic profiles and gene expression[J]. Genome Res, 2019, 29(4): 657-667. |
| [8] |
PANG B X, SNYDER M P. Systematic identification of silencers in human cells[J]. Nat Genet, 2020, 52(3): 254-263. |
| [9] |
NGAN C Y, WONG C H, TJONG H, et al. Chromatin interaction analyses elucidate the roles of PRC2-bound silencers in mouse development[J]. Nat Genet, 2020, 52(3): 264-272. |
| [10] |
WEI N, MULMI SHRESTHA S, SHI R H. Markers of gastric intestinal metaplasia under digital chromoendoscopy: syste matic review and meta-analysis[J]. Eur J Gastroenterol Hepatol, 2021, 33(4): 470-478. |
| [11] |
CORCES M R, TREVINO A E, HAMILTON E G, et al. An improved ATAC-seq protocol reduces background and enables interrogation of frozen tissues[J]. Nat Methods, 2017, 14(10): 959-962. |
| [12] |
NAKKA K, HACHMER S, MOKHTARI Z, et al. JMJD3 activated hyaluronan synthesis drives muscle regeneration in an inflammatory environment[J]. Science, 2022, 377(6606): 666-669. |
| [13] |
SINGH H, HA K, HORNICK J L, et al. Hybrid stomach-intestinal chromatin states underlie human Barrett's metaplasia[J]. Gastroenterology, 2021, 161(3): 924-939. e11. |
| [14] |
SUE S, SHIBATA W, KAMETA E, et al. Intestine-specific homeobox (ISX) induces intestinal metaplasia and cell proliferation to contribute to gastric carcinogenesis[J]. J Gastroenterol, 2016, 51(10): 949-960. |
| [15] |
SILBERG D, FURTH E, TAYLOR J, et al. CDX1 protein expression in normal, metaplastic, and neoplastic human alimentary tract epithelium[J]. Gastroenterology, 1997, 113(2): 478-486. |
| [16] |
KANEMITSU T, YAO K S, NAGAHAMA T, et al. Extending magnifying NBI diagnosis of intestinal metaplasia in the stomach: the white opaque substance marker[J]. Endoscopy, 2017, 49(6): 529-535. |
| [17] |
HANDA O, NAITO Y, YOSHIKAWA T. Redox biology and gastric carcinogenesis: the role of helicobacter pylori[J]. Redox Rep, 2011, 16(1): 1-7. |
| [18] |
KATSURAHARA M, KOBAYASHI Y, IWASA M, et al. Reactive nitrogen species mediate DNA damage in helicobacter pylori-infected gastric mucosa[J]. Helicobacter, 2009, 14(6): 552-558. |
| [19] |
HATA M, KINOSHITA H, HAYAKAWA Y, et al. GPR30-expressing gastric chief cells do not dedifferentiate but are eliminated via PDK-dependent cell competition during development of metaplasia[J]. Gastroenterology, 2020, 158(6): 1650-1666. e15. |
| [20] |
KON S, ISHIBASHI K, KATOH H, et al. Cell competition with normal epithelial cells promotes apical extrusion of transformed cells through metabolic changes[J]. Nat Cell Biol, 2017, 19(5): 530-541. |
| [21] |
PARK P J. ChIP-seq: advantages and challenges of a maturing technology[J]. Nat Rev Genet, 2009, 10(10): 669-680. |
| [22] |
KAYA-OKUR H S, WU S J, CODOMO C A, et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells[J]. Nat Commun, 2019, 10(1): 1930. |
| [23] |
SKENE P J, HENIKOFF S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites[J]. eLife, 2017, 6: e21856. |
| [24] |
BLACKLEDGE N P, KLOSE R J. The molecular principles of gene regulation by polycomb repressive complexes[J]. Nat Rev Mol Cell Biol, 2021, 22(12): 815-833. |
| [25] |
PIUNTI A, SHILATIFARD A. The roles of polycomb repressive complexes in mammalian development and cancer[J]. Nat Rev Mol Cell Biol, 2021, 22(5): 326-345. |
| [26] |
LIN X, GAUDINO S J, JANG KK, et al. IL-17RA-signaling in Lgr5+ intestinal stem cells induces expression of transcription factor ATOH1 to promote secretory cell lineage commitment[J]. Immunity, 2022, 55(2): 237-253. e8. |
| [27] |
TAMAGAWA Y, ISHIMURA N, UNO G, et al. Notch signaling pathway and Cdx2 expression in the development of barrett's esophagus[J]. Lab Investig, 2012, 92(6): 896-909. |




