2. 100071 北京, 解放军总医院第五医学中心呼吸与危重症医学科;
3. 100071 北京,军事科学院军事医学研究院微生物流行病研究所,病原微生物生物安全国家重点实验室;
4. 518000 广东 深圳,深圳大学总医院呼吸与危重症医学科
2. Department of Respiratory and Critical Care Diseases, Fifth Medical Centre, Chinese PLA General Hospital, Beijing, 100071;
3. State Key Laboratory of Pathogen and Biosecurity, Institute of Microbiology and Epidemiology, Academy of Military Medical Sciences, Beijing, 100071;
4. Department of Respiratory and Critical Care Diseases, General Hospital of Shenzhen University, Shenzhen, Guangdong Province, 518000, China
铜绿假单胞菌(Pseudomonas aeruginosa,P.aeruginosa)是一种机会致病性人类病原体,广泛分布于各种环境中。铜绿假单胞菌可在住院患者中引起慢性感染或急性危及生命的感染;患有囊性纤维化、中耳炎、角膜炎和严重烧伤以及免疫功能低下的人群[1-2]较易引发铜绿假单胞菌的感染。铜绿假单胞菌是一种适应性强的细菌,在长期抗生素治疗的情况下,铜绿假单胞菌是具耐药性的微生物种之一。随着全世界铜绿假单胞菌菌株的耐药性持续增加及新药研发的短缺,迫切需要探索抗菌药物替代疗法,噬菌体疗法被证明是控制铜绿假单胞菌感染的理想方法[3-5]。
有研究显示噬菌体具有治疗铜绿假单胞菌感染的潜力,尤其是多重耐药菌引起的感染[6-7]。噬菌体疗法的有效性在小鼠感染多重耐药性铜绿假单胞菌模型和体外肺囊性纤维化模型中得到证实[8-9]。铜绿假单胞菌噬菌体对感染性角膜炎、烧伤伤口感染、肠源性脓毒症和慢性中耳炎均具有治疗作用[10-16]。一般而言使用针对不同宿主的噬菌体混合物(鸡尾酒)会降低噬菌体疗法的疗效;因为在相对狭窄的裂解谱范围内细菌会出现耐受性[17]。然而也有研究人员发现使用6种裂解性噬菌体混合物对治疗铜绿假单胞菌引起的临床中耳感染具有很好的疗效。
据估计,在全球范围内,噬菌体(1031个)远比细菌数量要多[18]。截至2022年9月,来自NCBI的数据库中有2 111个噬菌体基因组序列已被公布。然而,仅有其中一部分噬菌体可以在体外或动物感染模型中具有抗菌活性。在2 111个噬菌体中,有152个是铜绿假单胞菌噬菌体,在过去的几年里有20多种针对铜绿假单胞菌的噬菌体通过动物模型测试具有很好的抗菌活性[19-20]。与地球上大量的噬菌体相比,现有分离的噬菌体数量仍然相对有限。因此,迫切需要从环境中分离裂解性较强的噬菌体治疗由多重耐药菌引起的感染。在本研究中,以实验室保存的铜绿假单胞菌Pa175为宿主菌从医院污水中分离裂解性噬菌体,对其生物学特性、全基因组序列及进化树进行研究,为噬菌体作为抗生素替代治疗耐药菌感染奠定基础。分离新型铜绿假单胞菌噬菌体,分析其基因组和裂解谱,将有助于更全面地了解铜绿假单胞菌噬菌体的抗菌潜力。
1 材料与方法 1.1 主要材料 1.1.1 样品来源本研究所用铜绿假单胞菌、鲍曼不动杆菌、肺炎克雷伯菌和金黄色葡萄球菌均由解放军总医院第五医学中心各临床科室通过痰液、肺泡灌洗液、血液、脑脊液及分泌物等分离得到,并利用本院检验科微生物室VITEK 2.0全自动微生物分析系统进行菌种鉴定和药敏试验。研究所用细菌保存至军事科学院军事医学研究院微生物流行病研究所未知病原分析室-80 ℃冰箱。
1.1.2 主要试剂和仪器0.22 μm微孔过滤器购自美国Pall Corporation公司;DNaseⅠ、蛋白酶K、RNaseⅠ购自赛默飞世尔科技(中国)有限公司;PEG8000购自赛默飞世尔科技(中国)有限公司;酚∶氯仿∶异戊醇液(25 ∶24 ∶1)、DNA平衡酚购自北京索莱宝科技有限公司;电热恒温培养箱、恒温摇床购自上海一恒科学仪器有限公司;低温高速离心机购自美国贝克曼库尔特有限公司;透射电子显微镜购自飞利浦电子公司。
2×Trans Taq® High Fidelity(Hi Fi) PCR Super Mix(北京全式金生物技术有限公司);二代测序建库用试剂盒E7645 NEBNext® UltraTMⅡ DNA Library Prep Kit for Illumina®(美国,纽英伦生物技术有限公司);二代测序仪(美国,亿明达高通量测序仪);本实验室提供的SGI4700高性能计算机服务器;CLC Genomics Workbench 12.0高通量测序分析软件(德国,QIAGEN)。
1.1.3 培养基及溶液配制LB液体培养基(1%胰化蛋白胨,0.5%酵母提取物,1% NaCl);LB固体培养基(在LB液体培养基中添加1.5%琼脂粉);PBS缓冲液[0.2 mol/L Na2HPO4,0.2 mol/L NaH2PO4,pH=7.4];10×TEN缓冲液[0.1 mol/L Tris-Cl(pH=8.0)],0.01 mol/L EDTA(pH=8.0),1 mol/L NaCl];SM Buffer [NaCl 5.8 g,MgSO4·7H2O 2.0 g,1 mol/L Tris·HCl(pH=7.5)50 mL,2%明胶5 mL,加蒸馏水至1 L,1×105Pa灭菌20 min]。
1.2 方法 1.2.1 细菌培养及鉴定使用接种环蘸取库存Pa175甘油菌液,通过分区划线法接种至LB固体培养基上,37 ℃恒温培养箱过夜培养;用无菌枪头挑取形态单一菌落接种至LB液体培养基中,37 ℃、180 r/min恒温摇床振荡培养至对数期[D(600)=0.6~0.8],PCR反应模板备用。使用细菌通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-GGY- TACCTTGTTACGACTT-3′)[21]对铜绿假单胞菌(Pa175、Pa13、Pa135、Pa140、Pa175、Pa1473和Pa2526)进行16S rRNA基因扩增。PCR反应体系(50 μL):2×Taq Master Mix 25 μL,Pa175菌液模板1 μL,上、下游引物(10 μmol/L)各2 μL,RNase-Free ddH2O 20.0 μL。PCR反应条件:PCR反应设置退火温度为60 ℃,其余条件参照产品使用说明书,共设置30个循环。取PCR产物10 μL,用含1.5%琼脂糖凝胶行电泳检测。取40 μL检测成功的PCR产物送至北京诺赛基因组研究中心有限公司行一代测序,测序结果在GenBank中进行BLAST比对分析确定菌种。
1.2.2 噬菌体的分离与纯化取1 L中国人民解放军总医院第五医学中心南院区污水站未经过任何处理的污水,4 ℃、12 000 r/min离心15 min,去沉淀留上清,用0.22 μm微孔过滤器过滤,收集滤液备用;取100 μL对数期铜绿假单胞菌Pa175[D(600)=0.6~0.8] 加至4 mL污水滤液和2 mL的3×LB液体培养基中,混匀后室温静置1 h,置于37 ℃恒温摇床继续培养6 h,待混合液体由浑浊变澄清后,4 ℃、12 000 r/min离心10 min,收集上清液,用0.22 μm微孔滤器过滤,所得滤过液即为噬菌体原液备用。
取100 μL上一步骤滤过液与500 μL对数期上述菌液[D(600)=0.6~0.8]混合均匀,倒入半固体琼脂平板中央,铺双层琼脂平板;用上述方法梯度稀释滤过液,在合适的稀释梯度下铺设双层平板并查看噬菌斑及记录数目;用带滤芯的移液器吸头吸取铺设LB固体平板上形态规则的单个噬菌斑液,反复吹吸至对数期Pa175[D(600)=0.8]菌液中,37 ℃、180 r/min恒温摇床震荡培养,直至澄清。取1.5 mL,置于新的Eppendorf管中,4 ℃、12 000 r/min离心3 min,用注射器吸取上清,用0.22 μm滤器过滤再次收集滤液;以此滤液为基础铺双层平板法观察噬菌斑形态,重复3次,直至噬菌斑形态均一为止,最终将滤过液储存于4 ℃保存。
1.2.3 噬菌体的超离纯化及电镜下形态观察取噬菌体原液35 mL加至50 mL超速离心管(Beckman公司)中,用10 mL带针头的灭菌注射器从超速离心管底层缓缓注入10 mL 30%(质量体积比)蔗糖溶液,配平后(注意要完全配平,精确到0.001 g),4 ℃、30 000 r/min离心3 h;倒出离心管中所有液体并将管口倒置于桌面上直至底层蔗糖完全流出为止,在用断枪头吸取200 μL 1×TEN缓冲液缓慢注入离心管底部,4 ℃过夜。
取50 μL超速离心纯化的噬菌体原液滴在含碳膜的铜网上吸附过夜,用2%的磷钨酸(PTA)负染5 min,使用Philips TECNAI-10型透射电子显微镜观察噬菌体形状,并正式命名为IME307。
1.2.4 噬菌体裂解谱测定随机从库存中选取31株铜绿假单胞菌、5株肺炎克雷伯菌、6株鲍曼不动杆菌及3株金黄色葡萄球菌,采用双层平板法和点板稀释法鉴定噬菌体裂解谱,观察有无噬菌斑,每次实验重复3次。
1.2.5 噬菌体最佳感染复数(MOI)的测定参照WANG等[22]的方法略有改动。为了确定最佳MOI,将IME307与Pa175宿主菌液分别按照MOI为10、1、0.1、0.01、0.001混合对数期宿主细菌和噬菌体悬液,加入新鲜的LB培养基补足至10 mL,37 ℃,220 r/min振荡培养90 min后,4 000 r/min离心10 min,除去底层细菌沉淀、收集上清液,使用0.22 μm的微孔过滤器过滤,通过双层平板法测定噬菌体的滴度。每次独立实验重复3次。
1.2.6 噬菌体一步生长曲线的测定参照WANG等[22]的方法略有改动,具体如下。取1 mL培养至对数期菌液Pa175[D(600)=0.8],4 ℃、8 000 r/min,离心1 min,取1 mL的PBS重悬沉淀,再用PBS倍比梯度稀释菌液后涂板,计算菌落数目,确定在对数期[D(600)=0.8]1 mL菌液所含细菌数目。
将Pa175接种至5 mL新鲜LB液体培养基中,培养至对数期[D(600)=0.8]后,4 ℃、5 000 r/min,离心2 min,去上清,沉淀重悬于5 mL的LB培养基,使Pa175绝对数达到上一步骤中对数期细菌数目。取100 μL噬菌体IME307(本实验所用MOI=0.01)加至上述混合液中;37 ℃恒温箱放置2 min,使噬菌体能够充分吸附到细菌表面,室温下5 000 r/min离心2 min,去除未能吸附的噬菌体上清,用5 mL的LB液体培养基洗2遍沉淀,去除清洗液再加入5 mL新鲜的LB培养基,用移液枪轻轻吹吸混匀沉淀,置于37 ℃恒温摇床220 r/min震荡培养;自加入噬菌体开始时间为0 min,分别在0、10、20、30、40、50、60、80和90 min等各个时间点取样品,并测定噬菌体在每个时间点下的滴度;以时间为横坐标,以取样下每个时间点噬菌体滴度的对数值为纵坐标绘制生长曲线。
1.2.7 噬菌体温度和pH敏感性的测定取1 mL噬菌体液IME307分别置于4、40、50、60、70、80、90和100 ℃恒温水浴中静置1 h。采用双层平板法测定IME307滴度,实验过程重复3次做统计分析。
将噬菌体原液IME307以1 ∶10的体积倍比稀释分别置于pH为2.0、3.0、4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0和12.0的SM Buffer中,37℃静置1 h。采用双层平板法测定各试验组噬菌体滴度,设置3个重复平行试验。
1.2.8 噬菌体核酸的提取采用蛋白酶K/SDS的方法并结合WANG等[22]的方法略有改动,提取纯化的噬菌体核酸,具体方法如下:取600 μL噬菌体原液,加入DNaseⅠ(0.2 mg/mL)和RNase A(0.2 mg/mL)各3 μL使得各自终浓度为1 μg/mL,37 ℃过夜消化以去除混合液中宿主菌核酸,80 ℃恒温灭活DNaseⅠ和RNase A 15 min;在向混合液中分别加入24 μL、0.5 mol/L的EDTA(使得终浓度20 mmol/L),2.5 μL、20 mg/mL的蛋白酶K(终浓度50 μg/mL),30 μL、10%SDS。终浓度0.5%(质量体积比),56 ℃水浴1 h;加入上一步骤中等体积平衡酚,温和震荡1 min,室温下12 000 r/min离心5 min,转移上层亲水相置于新Eppendorf管中;加入上一步骤等体积酚-氯仿-异戊醇(25 ∶24 ∶1),温和震荡1 min,室温下12 000 r/min离心5 min,转移上层亲水相于新的Eppendorf管中;加入等体积异丙醇,-20 ℃放置3 h以上;4 ℃、13 000 r/min离心20 min,缓慢倒掉上清;加入1 mL、75%冰乙醇(-20 ℃提前放置),室温静置1 min,4 ℃、12 000 r/min离心2 min,缓慢倒掉乙醇,室温开盖放置10 min,使乙醇完全挥发;在Eppendorf管的底层加入30 μL去离子水溶解DNA沉淀,储存于-20℃备用。
1.2.9 噬菌体全基因组测序及全基因组序列分析按照E7645 NEBNext®UltraTMⅡ DNA Library Prep Kit for Illumina®试剂盒使用说明书对已提取噬菌体基因组构建文库,通过本实验室Illumina-MiSeq测序平台上机测序。测序所得原始数据由本实验室工作人员通过高性能计算机服务器使用Newbler 2.90软件拼接序列,得到噬菌体全基因组序列。使用RAST在线数据库[22](http://www.rast.nmpdr.org)对开放阅读框(open reading frame,ORF)初步确定,所有的ORF使用NCBI中氨基酸序列比对工具BLASTp(http://www.ncbi.nlm.nih.gov/BLAST/)手动验证。将基因组注释序列提交到NCBI,获取序列号MN096267。
1.2.10 比较基因组学及噬菌体进化树的构建噬菌体末端酶大亚基是一种保守的噬菌体蛋白。因此,基于末端酶大亚基氨基酸序列构建噬菌体IME307系统发育树。从NCBI中下载与IME307末端酶大亚基氨基酸序列相似性最高的30余种不同噬菌体,通过CLC Genomics Workbench 12.0软件分别提取噬菌体末端酶大亚基氨基酸序列,使用MEGA 6.0软件制成基于末端酶大亚基氨基酸序列的进化树。
将IME307通过BlastN(https://blast.ncbi.nlm.nih.gov/Blast.cgi)全基因组序列比,找出最为相近的几种噬菌体序列,使用CLC Genomics Workbench 12.0和Mauve software 2.3.1软件进行基因多重比对分析,分析全基因组相似处和差异。
2 结果 2.1 铜绿假单胞菌Pa175的鉴定本研究所用宿主菌Pa175分离自解放军总医院第五医学中心呼吸与危重症医学科一诊断为支气管扩张伴感染的痰液样本,根据GenBank数据库中BLAST对Pa175的16S rRNA测序基因序列进行相似性比对,结合解放军总医院第五医学中心检验科利用VITEK 2.0全自动微生物分析,结果显示Pa175为多重耐药性铜绿假单胞菌,其药敏结果如表 1所示。
| 抗菌药物 | MIC/(μg/mL) | 敏感性 | 抗菌药物 | MIC/(μg/mL) | 敏感性 | |
| 环丙沙星Ciprofloxacin | ≥4 | R | 氨苄西林Ampicillin | ≥32 | S | |
| 庆大霉素Gentamicin | ≥16 | R | 氨苄西林舒巴坦钠Ampicillin sulbactam | ≥32 | S | |
| 亚胺培南Imipenem | ≥16 | S | 氨曲南Aztreonam | ≥64 | R | |
| 美罗培南Meropenem | ≥16 | S | 哌拉西林/他唑巴坦钠Piperacillin/tazobactam | ≥128 | S | |
| 哌拉西林Piperacillin | ≥128 | S | 呋喃妥因Nitrofurantoin | ≥512 | S | |
| 复方新诺明Cotrimoxazole | ≥320 | S | 头孢哌酮舒巴坦钠Cefoperazone/sulbactam | ≥64 | S | |
| 左氧氟沙星Levofloxacin | ≥8 | S | 头孢吡肟Cefepime | ≥64 | R | |
| 头孢他啶Ceftazidime | ≥64 | S | 头孢曲松Ceftriaxone | ≥64 | R | |
| 头孢呋辛酯Cefuroxime axetil | ≥64 | R | 妥布霉素Tobramycin | ≥16 | R | |
| 头孢呋辛钠Cefuroxime sodium | ≥64 | R | 头孢替坦Cefotetan | ≥64 | R |
2.2 IME307噬菌体的分离
本实验以Pa175为研究对象从解放军总医院第五医学中心医院污水中成功分离得到1株裂解性噬菌体,命名为vB_PaS_IME307(以下简称IME307)。IME307经过多次单斑纯化后能够在铺有双层LB的平板上形成透明的噬菌斑,在噬菌斑的周围可以形成半透明的晕环(图 1A),这种征象的形成可能是噬菌体多糖解聚酶存在的宏观表现。将图 1A噬菌斑中央白点用灭菌抢头挑选出并重新培养得到新的耐受菌,再次用IME307对其进行侵染,通过铺双层平板法在图 1B中并未发现噬菌斑,这种现象可能是因为Pa175能够迅速对IME307进行耐受。
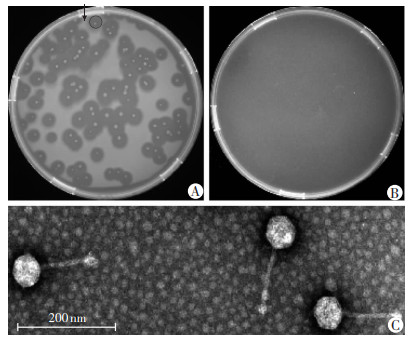
|
| A:噬菌体噬菌斑箭头示晕环,黑色圆圈内为噬菌斑;B: 噬菌体耐受性图;C:噬菌体透射电镜结果 图 1 噬菌体vB_PaS_IME307的形态特征 |
2.3 噬菌体形态特征
经透射电子显微镜对IME307进行形态观察,发现IME307拥有一个正二十面体的头部,其直径为(66± 2)nm;为长尾噬菌体,长度为(116±2)nm(图 1C)。根据ICTV制定的新国际病毒分类方法,该噬菌体被正式命名为vB_PaS_IME307,IME307为有尾噬菌体目、长尾噬菌体科(Siphoviridae)噬菌体。
2.4 噬菌体的裂解谱IME307能够对31株中的2株铜绿假单胞菌进行裂解,无法裂解金黄色葡萄球菌、鲍曼不动杆菌及肺炎克雷伯菌等(表 2),这表明IME307裂解谱相对较窄,具有严格的宿主特异性,仅对个别菌株(Pa175和Pa135)具有裂解性。图 2所示IME307对Pa175具有较强裂解性,对Pa135裂解性较弱,对Pa1473和Pa2526没能形成透明噬菌斑。
| 细菌 | 细菌编号 | 噬菌体敏感性 |
| Pseudomonas aeruginosa | 135/175/ | + |
| Pseudomonas aeruginosa | 13/140/21/28/80/83/180/769/784/808/825/962/1104/1106/1133/1198/1200/1306/1397/1768/1783/1850/2120/2122/2128/2130/1473/2526 | - |
| Klebsiella pneumoniae | 1183/226/2060/2061/409 | - |
| Acinetobacter baumannii | 387/333/2040/2/6/406 | - |
| Streptococcus agalactiae | 253/331/1323/ | - |
| +:裂解;-:不裂解 | ||
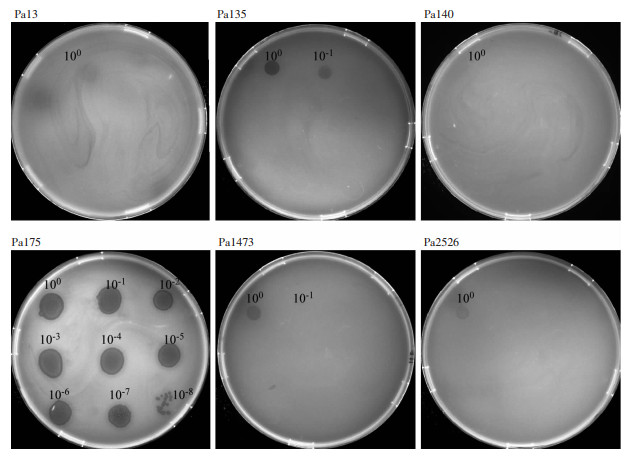
|
| 图 2 噬菌体vB_PaS_IME307的裂解图谱 |
2.5 噬菌体的最佳感染复数及一步生长曲线
据图 3A所示IME307与Pa175按一定比例混合,当以MOI=0.1时,噬菌体IME307可产生最大滴度(图 3A),这表明0.1是IME307的最佳MOI。
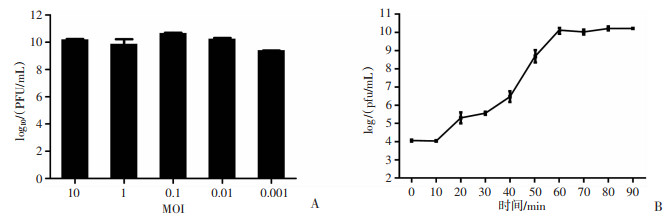
|
| A:噬菌体最佳MOI柱状图;B:IME307一步生长曲线图 图 3 噬菌体vB_PaS_IME307的MOI测定及一步生长曲线 |
据图 3B所示,噬菌体IME307的潜伏期约10 min,增殖时间在10~60 min。在60 min以后进入稳定期,爆发量=稳定期噬菌体滴度/感染初细菌数目。爆发量代表了单个宿主菌被裂解后平均释放的子代噬菌体数量,IME307噬菌体的爆发量为150 PFU/cell。
2.6 噬菌体对温度和pH的稳定性从图 4中可以看出,IME307处在pH=5~11时,噬菌体能够保持很好的滴度,当pH=12时噬菌体的滴度明显下降。IME307在60 ℃以下噬菌体的滴度相对稳定,当温度上升10 ℃以后噬菌体滴度明显下降,在100 ℃时噬菌体全部失活,不具有裂解性。
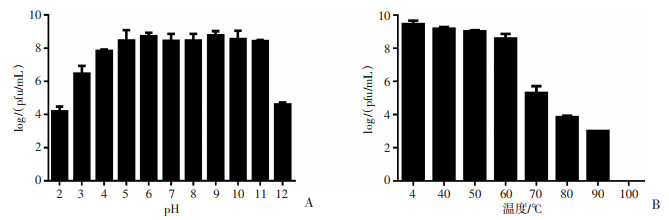
|
| A:pH=2~12时IME307的滴度曲线;B:温度4~100 ℃时IME307对温度敏感性滴度曲线 图 4 噬菌体IME307对pH和热稳定性 |
2.7 噬菌体的全基因组分析及进化树分析
对IME307噬菌体进行高通量测序并对测序数据进行拼接,分析发现该噬菌体为双链DNA噬菌体,全基因组大小为51 545 bp,GC含量占56.10%。噬菌体vB_PaS_IME307的功能基因呈现多样性,可编码多种噬菌体的结构蛋白基因,其基因组中存在97个开放阅读框(ORF)。运用NCBI的序列比对软件BLASTp进行噬菌体功能基因的ORF注释结果见表 3。其中45个ORF为假想蛋白(hypothetical protein),33个ORF功能已知。ORF45(穿孔蛋白)是噬菌体裂解相关基因,可导致细胞壁破裂而使细菌溶解。结构蛋白ORF48是噬菌体主要衣壳蛋白,与末端酶包装蛋白ORF46和ORF47邻近。ORF49编码噬菌体尾部相关蛋白,具有收缩和运动功能,该基因簇在噬菌体感染、吸附宿主菌时发挥重要作用。ORF13(假定噬菌体阻遏物)、ORF26(核酸内切酶)、ORF76(溶原性噬菌体整合酶)、ORF97(核酸外切酶)。经过对比分析并未发现该噬菌体中存在毒力基因,这表明该噬菌体具有较好的生物安全性。
| ORF序列 | 功能 | 启始 | 终止 | 正负链 | GenBank编号 | 一致性(%)(aa) | E值 |
| 1 | ERF family protein | 790 | 59 | - | WP_034080569.1 | 99.18% | 1e-179 |
| 2 | hypothetical protein | 1 110 | 853 | - | QDH93774.1 | 100% | 3e-54 |
| 3 | hypothetical protein | 1 305 | 1 141 | - | MBX5587495.1 | 100% | 2e-29 |
| 4 | hypothetical protein | 1 415 | 1 302 | - | QDH93776.1 | 100% | 5e-16 |
| 5 | hypothetical protein | 1 669 | 1 412 | - | WP_049264142.1 | 100% | 3e-53 |
| 6 | hypothetical protein D3p071 | 2 091 | 1 666 | - | NP_061562.1 | 100% | 2e-96 |
| 7 | hypothetical protein | 2 336 | 2 223 | - | QDH93779.1 | 100% | 5e-16 |
| 8 | hypothetical protein D3p072 | 2 822 | 2 595 | - | NP_061563.1 | 100% | 5e-47 |
| 9 | hypothetical protein | 3 102 | 2 839 | - | QDH93781.1 | 100% | 7e-57 |
| 10 | hypothetical protein | 3 232 | 3 345 | + | QDH93782.1 | 100% | 3e-17 |
| 11 | hypothetical protein | 3 634 | 3 452 | - | WP_132892723.1 | 100% | 3e-36 |
| 12 | hypothetical protein | 3 989 | 3 867 | - | QDH93784.1 | 100% | 2e-20 |
| 13 | Putative phage repressor | 4 927 | 4 212 | - | WP_053092377.1 | 100% | 1e-172 |
| 14 | Cro/CI-family transcriptional regulator | 5 028 | 5 207 | + | WP_023113651.1 | 100% | 9e-34 |
| 15 | hypothetical protein | 5 221 | 5 421 | + | WP_023125075.1 | 100% | 7e-40 |
| 16 | hypothetical protein | 5 441 | 5 644 | + | WP_023127506.1 | 100% | 9e-39 |
| 17 | phage replication protein | 5 720 | 6 520 | + | WP_124133257.1 | 100% | 0.0 |
| 18 | ATP-binding protein | 6 510 | 7 325 | + | WP_049264146.1 | 100% | 0.0 |
| 19 | hypothetical protein | 7 318 | 7 470 | + | QDH93791.1 | 100% | 2e-27 |
| 20 | hypothetical protein | 7 467 | 7 586 | + | QDH93792.1 | 100% | 3e-19 |
| 21 | hypothetical protein | 7 583 | 7 798 | + | QDH93793.1 | 100% | 2e-44 |
| 22 | recombination protein NinB | 7 791 | 8 207 | + | WP_014603756.1 | 100% | 7e-97 |
| 23 | Phage chromosome segregation protein | 8 204 | 8 548 | + | QDH93795.1 | 100% | 3e-76 |
| 24 | hypothetical protein | 8 545 | 8 874 | + | QDH93796.1 | 100% | 6e-74 |
| 25 | DUF1364 family protein | 8 871 | 9 173 | + | WP_023875435.1 | 100% | 5e-66 |
| 26 | Phage-associated homing endonuclease | 9 170 | 10 063 | + | QDH93798.1 | 100% | 0.0 |
| 27 | hypothetical protein | 10 147 | 10 341 | + | QDH93799.1 | 100% | 6e-39 |
| 28 | hypothetical protein | 10 338 | 10 565 | + | QDH93800.1 | 100% | 2e-48 |
| 29 | hypothetical protein | 10 562 | 10 939 | + | QDH93801.1 | 100% | 3e-85 |
| 30 | hypothetical protein | 10 936 | 11 151 | + | QDH93802.1 | 100% | 2e-43 |
| 31 | hypothetical protein | 11 148 | 11 378 | + | QDH93803.1 | 100% | 2e-48 |
| 32 | hypothetical protein | 11 375 | 11 758 | + | WP_017001698.1 | 100% | 7e-88 |
| 33 | hypothetical protein | 11 758 | 12 036 | + | WP_225001937.1 | 100% | 1e-61 |
| 34 | hypothetical protein | 12 036 | 13 049 | + | WP_049264154.1 | 100% | 0.0 |
| 35 | antitermination protein Q | 13 046 | 13 444 | + | WP_017001695.1 | 100% | 1e-92 |
| 36 | hypothetical protein | 13 444 | 13 608 | + | QDH93808.1 | 100% | 3e-31 |
| 37 | hypothetical protein | 13 643 | 13 846 | + | QDH93809.1 | 100% | 2e-42 |
| 38 | hypothetical protein | 14 076 | 14 423 | + | WP_170954280.1 | 100% | 6e-75 |
| 39 | hypothetical protein | 14 420 | 14 617 | + | QDH93811.1 | 100% | 1e-40 |
| 40 | Cell wall hydrolyses involved in spore germination | 14 617 | 15 033 | + | QDH93812.1 | 100% | 4e-97 |
| 41 | Phage protein | 15 033 | 15 206 | + | WP_162962015.1 | 100% | 3e-29 |
| 42 | hypothetical protein | 15 216 | 15 464 | + | WP_023875424.1 | 100% | 2e-54 |
| 43 | Endopeptidase | 15 457 | 15 912 | + | WP_023875424.1 | 100% | 2e-54 |
| 44 | hypothetical protein | 15 942 | 16 112 | + | QDH93816.1 | 100% | 1e-29 |
| 45 | Phage holin | 16 109 | 16 444 | + | QDH93817.1 | 100% | 5e-78 |
| 46 | Phage terminase, small subunit | 16 586 | 17 026 | + | QDH93818.1 | 100% | 5e-97 |
| 47 | Phage terminase large subunit | 17 028 | 18 689 | + | QDH93819.1 | 100% | 0.0 |
| 48 | Phage capsid and scaffold | 18 749 | 20 752 | + | QDH93820.1 | 100% | 0.0 |
| 49 | Phage tail fibers | 20 815 | 21 036 | + | WP_176600165.1 | 100% | 2e-44 |
| 50 | Phage protein | 21 033 | 21 257 | + | QDH93822.1 | 100% | 2e-41 |
| 51 | Phage portal protein | 21 250 | 22 635 | + | QDH93823.1 | 100% | 0.0 |
| 52 | Phage portal protein | 22 628 | 23 632 | + | QDH93824.1 | 100% | 0.0 |
| 53 | phage gp6-like head-tail connector protein | 23 629 | 24 003 | + | MCO3804694.1 | 100% | 6e-84 |
| 54 | hypothetical protein | 24 237 | 24 668 | + | QDH93826.1 | 100% | 5e-98 |
| 55 | phage head closure protein | 24 665 | 25 006 | + | WP_263822137.1 | 100% | 3e-77 |
| 56 | Phage protein | 25 006 | 25 575 | + | MCO3804691.1 | 100% | 1e-138 |
| 57 | hypothetical protein | 25 557 | 26 384 | + | QDH93829.1 | 100% | 0.0 |
| 58 | methyltransferase domain-containing protein | 26 395 | 26 949 | + | WP_034020418.1 | 100% | 2e-134 |
| 59 | hypothetical protein | 26 942 | 27 388 | + | WP_015994276.1 | 100% | 1e-102 |
| 60 | hypothetical protein | 27 388 | 27 759 | + | QDH93832.1 | 100% | 4e-86 |
| 61 | Phage tail fibers | 27 947 | 28 441 | + | WP_015994278.1 | 100% | 9e-117 |
| 62 | hypothetical protein | 28 451 | 28 813 | + | QDH93834.1 | 100% | 3e-81 |
| 63 | hypothetical protein | 28 972 | 29 127 | + | QDH93835.1 | 100% | 2e-29 |
| 64 | Phage tail length tape-measure protein1 | 29 158 | 31 911 | + | QDH93836.1 | 100% | 0.0 |
| 65 | hypothetical protein | 31 908 | 32 372 | + | WP_015994282.1 | 100% | 9e-108 |
| 66 | DUF1833 family protein | 32 369 | 32 857 | + | WP_126633578.1 | 100% | 1e-115 |
| 67 | hypothetical protein | 32 862 | 33 425 | + | QDH93839.1 | 100% | 1e-135 |
| 68 | hypothetical protein | 33 480 | 33 887 | + | WP_003158964.1 | 100% | 1e-94 |
| 69 | Phage tail fiber protein | 33 859 | 36 588 | + | QDH93841.1 | 100% | 0.0 |
| 70 | hypothetical protein | 36 649 | 38 733 | + | QDH93842.1 | 100% | 0.0 |
| 71 | exopolysaccharide production protein | 39 884 | 38 730 | - | QDH93843.1 | 100% | 0.0 |
| 72 | hypothetical protein | 40 849 | 40 163 | - | WP_124096903.1 | 100% | 2e-166 |
| 73 | hypothetical protein | 41 328 | 40 849 | - | QDH93845.1 | 100% | 7e-112 |
| 74 | hypothetical protein | 41 461 | 41 724 | + | QDH93846.1 | 100% | 3e-57 |
| 75 | hypothetical protein | 41 708 | 41 839 | + | QDH93847.1 | 100% | 5e-20 |
| 76 | integrase | 43106 | 41 889 | - | QDH93848.1 | 100% | 0.0 |
| 77 | hypothetical protein | 43 787 | 43 386 | - | QDH93849.1 | 100% | 5e-94 |
| 78 | hypothetical protein | 44 014 | 44 014 | - | QDH93850.1 | 100% | 3e-49 |
| 79 | hypothetical protein | 44 346 | 44 014 | - | QDH93851.1 | 100% | 5e-73 |
| 80 | hypothetical protein | 44 678 | 44 343 | - | WP_267736549.1 | 100% | 1e-74 |
| 81 | hypothetical protein | 45 046 | 44 675 | - | WP_267736548.1 | 100% | 2e-83 |
| 82 | hypothetical protein | 45 501 | 45 043 | - | QDH93854.1 | 100% | 1e-104 |
| 83 | hypothetical protein | 45 719 | 45 498 | - | WP_126895028.1 | 100% | 8e-46 |
| 84 | hypothetical protein | 46 036 | 45 716 | - | QDH93856.1 | 100% | 6e-72 |
| 85 | hypothetical protein | 46 472 | 46 029 | - | WP_236079895.1 | 100% | 7e-104 |
| 86 | dual specificity protein phosphatase family protein | 46 960 | 46 475 | - | WP_134260534.1 | 100% | 1e-117 |
| 87 | hypothetical protein | 47 091 | 46 945 | - | WP_208539801.1 | 97.92% | 1e-25 |
| 88 | hypothetical protein | 47 480 | 47 175 | - | WP_267736543.1 | 100% | 1e-67 |
| 89 | hypothetical protein | 47 473 | 48 060 | - | WP_267736542.1 | 100% | 1e-141 |
| 90 | hypothetical protein | 48 263 | 48 057 | - | WP_267736541.1 | 100% | 9e-41 |
| 91 | hypothetical protein | 48 435 | 48 256 | - | QDH93863.1 | 100% | 1e-35 |
| 92 | hypothetical protein | 49 490 | 48 435 | - | QDH93864.1 | 100% | 0.0 |
| 93 | hypothetical protein | 49 711 | 49 487 | - | WP_017001718.1 | 100% | 2e-47 |
| 94 | hypothetical protein | 49 893 | 49 708 | - | KYO84492.1 | 100% | 5e-37 |
| 95 | hypothetical protein | 50 473 | 49 874 | - | QDH93867.1 | 100% | 2e-147 |
| 96 | single-stranded DNA-binding protein | 50 983 | 50 537 | - | MCO3804737.1 | 100% | 4e-105 |
| 97 | exonuclease | 51 510 | 50 980 | - | MCO3804736.1 | 100% | 3e-126 |
全基因组序列经BLASTn比对(https://blast.ncbi.nlm.nih.gov/Blast.cgi),以此揭示与IME307序列最相近的噬菌体基因组。对比分析后结果显示:IME307与Pseudomonas phage PAJU2噬菌体(GenBank:AP009624.1)最为相似,全基因组序列为41%覆盖率及94.06%的序列一致性;与铜绿假单胞菌噬菌体Pseudomonas phage JBD44(GenBank:KU199710.1)10%全基因组覆盖率和93.85%的一致性,图 5显示IME307与以上2个铜绿假单胞菌噬菌体通过Easyfig_win_2.1行全基因组线性比对,结果表明IME307序列相对较新颖,与目前NCBI中其余噬菌体序列同源性较低。
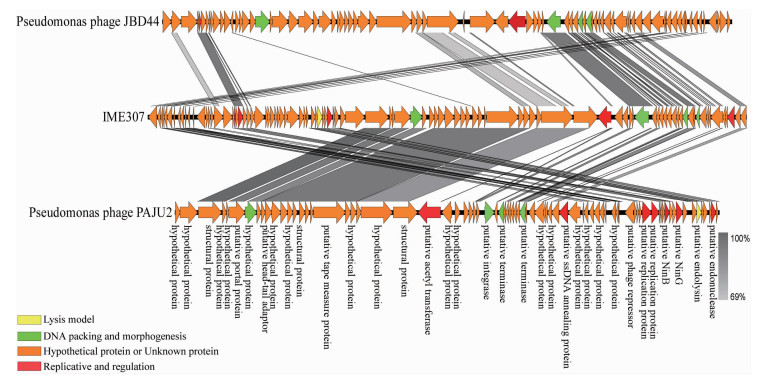
|
| IME175基因序列与JBD44和PAJU2噬菌体基因序列相似度不高;颜色灰度代表基因相似性高低噬菌体根据功能不同分为4类(裂解相关基因、DNA组装、复制调节相关基因、假想蛋白) 图 5 噬菌体全基因组比较与序列比对 |
为进一步分析IME307与其他噬菌体的亲缘关系,基于末端酶大亚基氨基酸序列构建进化树(图 6),从图 6中可看出,IME307与Pseudomonas phage VbPaeS PMG1、Lenoonostoo phage phiLN12、Lacttobacillus phage LL-Ku等噬菌体都位于Mccleskeyvirinae病毒属。因此,IME307属于有尾噬菌体目,长尾噬菌体(Siphoviridae),Mccleskeyvirinae病毒属。
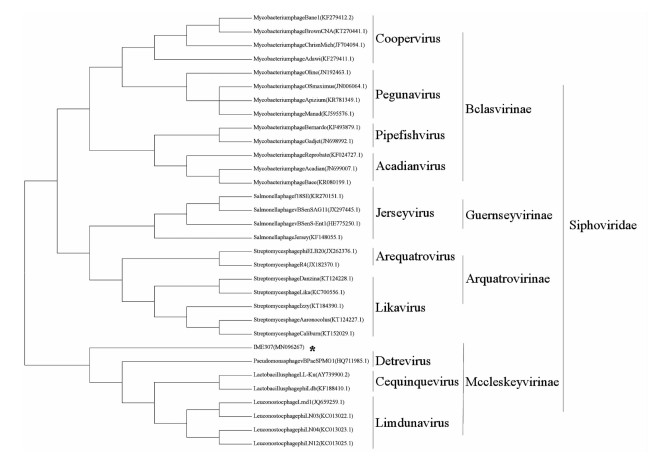
|
| 分支点上的数字代表可信度,数值越接近100,可信度越强;标尺越短代表亲缘关系越近;括号内代表GenBank序列登录号;IME307属于Mccleskeyvirinae病毒亚科 图 6 IME-307系统发育树分析 |
3 讨论
铜绿假单胞菌(Pseudomonas aeruginosa,PA)是一种机会性环境病原体,非发酵型革兰阴性杆菌,广泛分布于自然界,为土壤中最常见的细菌之一,通常在患有囊性纤维化的30%儿童和高达80%的25岁及以上成年人中定植[23],因此可能会导致慢性肺部感染、肺功能下降,甚至死亡。铜绿假单胞菌表面通常具有较厚的荚膜多糖(capsular polysaccharide,CPS),CPS是细菌感染宿主过程中的一个重要毒力因子,长期和反复的使用广谱抗菌药物后,铜绿假单胞菌对抗生素的耐药性会逐渐增强,并导致多重耐药性铜绿假单胞菌菌株的产生。作为细菌的天敌,噬菌体的研究得到了许多研究者的关注。噬菌体裂解细菌会形成噬菌斑,根据之前的一项研究表明,噬菌斑的大小是由噬菌体的一些特性所引起的,如噬菌体吸附速率、爆发量大小、裂解时间、噬菌体的大小和扩散率等[24];这些因素可能会导致噬菌斑形态及大小的差异。在噬菌体复制周期中,其晚期阶段主要是噬菌体裂解宿主菌释放子代噬菌体颗粒的过程,在裂解过程中会合成裂解相关蛋白,解聚酶是一种可以破坏细菌表面荚膜多糖的蛋白;表观上显示噬菌斑的周围被晕环包围,这种现象是因为在形成晕环的过程中,由于解聚酶的过度产生,细菌表面荚膜被破坏并降解为低聚糖重复单元,解聚酶通过琼脂层向外周扩散。有研究表明噬菌体解聚酶较噬菌体本身具有更宽泛的宿主谱,并对小鼠感染细菌具有很好的治疗作用[25],因此噬菌体解聚酶拥有较好的应用潜力。在本研究中IME307噬菌体所形成的噬菌斑周围可以看见透明晕环,晕环面积的大小随着时间的延长不断扩大,因此推断该噬菌体可能具有编码解聚酶相关的基因。在IME307噬菌斑的中央有白色的斑点,这种现象可能是本研究选择的宿主菌Pa175中含有溶原性噬菌体亦或者Pa175迅速对IME307耐受所致;挑选IME307噬菌斑的中央白色斑点通过铺双层板法再次进行噬菌体侵染,并未发现新的噬菌斑,在对IME307全序列基因进行详细注释分析发现,IE307基因中含有整合酶、切离酶、抑制子repressor等与溶原功能相关的基因,因此我们推论IME307为溶原性噬菌体。
本研究从解放军总医院第五医学中心未经处理的污水中成功分离到1株针对铜绿假单胞菌的噬菌体,命名为IME307。该噬菌体对环境具有较好的适应性,能够在较宽的温度和跨度较大的pH范围(5~11)内保持很好的滴度及活性,说明噬菌体IME307能耐受较强的酸碱性及高温环境,具有较强的适应性。不同的铜绿假单胞菌噬菌体其潜伏期、裂解期、裂解量存在较大差异。N4样噬菌体DSS3Φ2和EE36Φ具有更长的潜伏期,其潜伏期分别为3 h和2 h,其爆发裂解量分别为350和1 500[26];LP14具有785个PFU/感染细菌的大爆发量[27],而YH6约100个PFU/感染细菌[28]。在对IME307生物学特性进行研究发现;在MOI=0.1时,IME307具有潜伏期短(少于10 min),增殖到60 min以后达到稳定期,因此IME307具有潜伏期短、爆发量大的特点。进一步通过Illumina MiSeq高通量测序平台对IME307进行全基因组测序,并对测序数据拼接得到全基因组序列,IME307为双链DNA噬菌体,基因组大小为51 545 bp,G+C含量为56.1%。尽管我们通过2代测序对IME307进行全基因组测序,并得到全基因组序列,然而未能对该噬菌体进行末端鉴定,未能找到该噬菌体末端重复序列。结合李莎莎[29]及邢少贞[30]等构建查找末端序列的方法即高频序列就是噬菌体基因组的末端序列;我们将拼接的IME307噬菌体基因组序列与噬菌体下机原始数据通过CLC genomics workbench 12.0进行mapping并在高通量测序的原始数据中查找高频序列,结果未能发现IME307中存在高频序列,然而噬菌体末端仍旧是我们非常值得注意的一个问题,这在以后的噬菌体研究中会特别关注。
噬菌体进化树分析及全基因组序列比对分析,发现IME307噬菌体全基因组与其他铜绿假单胞菌噬菌体具有较大差异,其中与相似性最高的Pseudomonas phage PAJU2噬菌体进行比较,仅41%的全基因组覆盖率,在这41%序列里面,仅有94.06%的相似性。因此推断本研究所涉及的噬菌体具有一定程度的独特性及新颖性。
噬菌体疗法作为一种耐药菌感染的生物治疗方法,目前已引起科学界的重视,但要将其真正应用于临床仍有许多问题要解决,如噬菌体裂解谱较窄、噬菌体具有宿主特异性和噬菌体数量较少等、细菌耐药及进化后快速耐受噬菌体、机体启动免疫反应被快速清除噬菌体、噬菌体制剂安全等问题[19, 31]。因此,需要分离筛选更多的不同裂解谱的宽谱噬菌体、人工进化突变株、噬菌体改造等,构建容量巨大的噬菌体库,以此覆盖噬菌体裂菌谱。在对IME307裂解谱进行研究时发现,IME307对铜绿假单胞菌Pa175具有较强的裂解作用,但裂解谱较窄,仅能裂解31株中的2株铜绿假单胞菌,不能裂解其他种属细菌;这表明IME307具有高效特异性和高度专一性,这与大多数噬菌体具有共同特性。但这种特异性在高效、靶向性地杀灭致病菌的同时会造成噬菌体裂解谱狭窄及应用范围受限,然而噬菌体“鸡尾酒”疗法有望能有效解决这一问题[32]。在今后的研究中,我们将寻找铜绿假单胞菌分型方法,并针对不同型别的铜绿假单胞菌分离不同类别的噬菌体,利用噬菌体“鸡尾酒”制剂以此扩大铜绿假单胞菌的裂解谱。
| [1] |
CLARK S T, GUTTMAN D S, HWANG D M. Diversification of Pseudomonas aeruginosa within the cystic fibrosis lung and its effects on antibiotic resistance[J]. FEMS Microbiol Lett, 2018, 365(6): fny026. DOI:10.1093/femsle/fny026 |
| [2] |
YU X Y, XU Y, GU Y, et al. Characterization and genomic study of "phiKMV-like" phage PAXYB1 infecting Pseudomonas aeruginosa[J]. Sci Rep, 2017, 7(1): 13068. DOI:10.1038/s41598-017-13363-7 |
| [3] |
PABARY R, SINGH C, MORALES S, et al. Antipseudomonal bacteriophage reduces infective burden and inflammatory response in murine lung[J]. Antimicrob Agents Chemother, 2015, 60(2): 744-751. DOI:10.1128/AAC.01426-15 |
| [4] |
吴芷莹, 金泽源, 李万霞, 等. PB1样铜绿假单胞菌噬菌体PHW2的生物学特性[J]. 微生物学通报, 2021, 48(9): 3205-3217. WU Z Y, JIN Z Y, LI W X, et al. Characterization of a PB1-like phage PHW2 infecting Pseudomonas aeruginosa[J]. Microbiol China, 2021, 48(9): 3205-3217. |
| [5] |
张琳, 乐率, 卢曙光, 等. 铜绿假单胞菌噬菌体PaP4的分离与鉴定[J]. 微生物学通报, 2013, 40(4): 609-616. ZHANG L, LE S, LU S G, et al. Isolation and identification a novel Pseudomonas aeruginosa phage PaP4[J]. Microbiol China, 2013, 40(4): 609-616. |
| [6] |
HARPER D R, ENRIGHT M C. Bacteriophages for the treatment of Pseudomonas aeruginosa infections[J]. J Appl Microbiol, 2011, 111(1): 1-7. DOI:10.1111/j.1365-2672.2011.05003.x |
| [7] |
HORCAJADA J P, MONTERO M, OLIVER A, et al. Epidemiology and treatment of multidrug-resistant and extensively drug-resistant Pseudomonas aeruginosa infections[J]. Clin Microbiol Rev, 2019, 32(4): e00031-e00019. DOI:10.1128/CMR.00031-19 |
| [8] |
DEBARBIEUX L, LEDUC D, MAURA D, et al. Bacteriophages can treat and Prevent Pseudomonas aeruginosa lung infections[J]. J Infect Dis, 2010, 201(7): 1096-1104. DOI:10.1086/651135 |
| [9] |
ALEMAYEHU D, CASEY P G, MCAULIFFE O, et al. Bacteriophages φMR299-2 and φNH-4 can eliminate Pseudomonas aeruginosa in the murine lung and on cystic fibrosis lung airway cells[J]. mBio, 2012, 3(2): e00029-e00012. DOI:10.1128/mBio.00029-12 |
| [10] |
FUKUDA K, ISHIDA W, UCHIYAMA J, et al. Pseudomonas aeruginosa keratitis in mice: effects of topical bacteriophage KPP12 administration[J]. PLoS One, 2012, 7(10): e47742. DOI:10.1371/journal.pone.0047742 |
| [11] |
HAGENS S, HABEL A, VON AHSEN U, et al. Therapy of experimental pseudomonas infections with a nonreplicating genetically modified phage[J]. Antimicrob Agents Chemother, 2004, 48(10): 3817-3822. DOI:10.1128/AAC.48.10.3817-3822.2004 |
| [12] |
WATANABE R, MATSUMOTO T, SANO G, et al. Efficacy of bacteriophage therapy against gut-derived sepsis caused by Pseudomonas aeruginosa in mice[J]. Antimicrob Agents Chemother, 2007, 51(2): 446-452. DOI:10.1128/AAC.00635-06 |
| [13] |
ROSE T, VERBEKEN G, VOS D D, et al. Experimental phage therapy of burn wound infection: difficult first steps[J]. Int J Burns Trauma, 2014, 4(2): 66-73. |
| [14] |
WRIGHT A, HAWKINS C H, ÄNGGÅRD E E, et al. A controlled clinical trial of a therapeutic bacteriophage preparation in chronic otitis due to antibiotic-resistant Pseudomonas aeruginosa; a preliminary report of efficacy[J]. Clin Otolaryngol, 2009, 34(4): 349-357. DOI:10.1111/j.1749-4486.2009.01973.x |
| [15] |
JAMAL M, BUKHARI S M A U S, ANDLEEB S, et al. Bacteriophages: an overview of the control strategies against multiple bacterial infections in different fields[J]. J Basic Microbiol, 2019, 59(2): 123-133. DOI:10.1002/jobm.201800412 |
| [16] |
陈邬锦, 周静茹, 李瑞, 等. 一株分离自重症肺炎患者痰液标本的铜绿假单胞菌噬菌体生物学特性及基因分析[J]. 中国病原生物学杂志, 2020, 15(5): 546-550, 579. CHEN W J, ZHOU J R, LI R, et al. Biological characteristics and gene analysis of a Pseudomonas aeruginosa phage isolated from sputum samples of patients with severe pneumonia[J]. J Pathog Biol, 2020, 15(5): 546-550, 579. DOI:10.13350/j.cjpb.200510 |
| [17] |
CHAN B K, ABEDON S T, LOC-CARRILLO C. Phage cocktails and the future of phage therapy[J]. Future Microbiol, 2013, 8(6): 769-783. DOI:10.2217/fmb.13.47 |
| [18] |
WEINBAUER M G. Ecology of prokaryotic viruses[J]. FEMS Microbiol Rev, 2004, 28(2): 127-181. DOI:10.1016/j.femsre.2003.08.001 |
| [19] |
PIRES D P, VILAS BOAS D, SILLANKORVA S, et al. Phage therapy: a step forward in the treatment of Pseudomonas aeruginosa infections[J]. J Virol, 2015, 89(15): 7449-7456. DOI:10.1128/JVI.00385-15 |
| [20] |
CAO Z H, ZHANG J C, NIU Y D, et al. Isolation and characterization of a "phiKMV-like" bacteriophage and its therapeutic effect on mink hemorrhagic pneumonia[J]. PLoS One, 2015, 10(1): e0116571. DOI:10.1371/journal.pone.0116571 |
| [21] |
FREDRIKSSON N J, HERMANSSON M, WILÉN B M. The choice of PCR primers has great impact on assessments of bacterial community diversity and dynamics in a wastewater treatment plant[J]. PLoS One, 2013, 8(10): e76431. DOI:10.1371/journal.pone.0076431 |
| [22] |
WANG C, LI P Y, NIU W K, et al. Protective and therapeutic application of the depolymerase derived from a novel KN1 genotype of Klebsiella pneumoniae bacteriophage in mice[J]. Res Microbiol, 2019, 170(3): 156-164. DOI:10.1016/j.resmic.2019.01.003 |
| [23] |
ROSSI E, LA ROSA R, BARTELL J A, et al. Pseudomonas aeruginosa adaptation and evolution in patients with cystic fibrosis[J]. Nat Rev Microbiol, 2021, 19(5): 331-342. DOI:10.1038/s41579-020-00477-5 |
| [24] |
GALLET R, KANNOLY S, WANG I N. Effects of bacteriophage traits on plaque formation[J]. BMC Microbiol, 2011, 11: 181. DOI:10.1186/1471-2180-11-181 |
| [25] |
CHIBEU A, LINGOHR E J, MASSON L, et al. Bacteriophages with the ability to degrade uropathogenic Escherichia coli biofilms[J]. Viruses, 2012, 4(4): 471-487. DOI:10.3390/v4040471 |
| [26] |
ZHAO Y L, WANG K, JIAO N Z, et al. Genome sequences of two novel phages infecting marine roseobacters[J]. Environ Microbiol, 2009, 11(8): 2055-2064. DOI:10.1111/j.1462-2920.2009.01927.x |
| [27] |
SHI X J, ZHAO F Y, SUN H Z, et al. Characterization and complete genome analysis of Pseudomonas aeruginosa bacteriophage vB_PaeP_LP14 belonging to genus litunavirus[J]. Curr Microbiol, 2020, 77(9): 2465-2474. DOI:10.1007/s00284-020-02011-5 |
| [28] |
YANG M, DU C T, GONG P J, et al. Therapeutic effect of the YH6 phage in a murine hemorrhagic pneumonia model[J]. Res Microbiol, 2015, 166(8): 633-643. DOI:10.1016/j.resmic.2015.07.008 |
| [29] |
李莎莎, 范航, 安小平, 等. 采用高通量测序技术分析尾病毒目噬菌体基因组末端序列特点[J]. 病毒学报, 2013, 29(1): 39-43. LI S S, FAN H, AN X P, et al. Utility of high throughput sequencing technology in analyzing the terminal sequence of caudovirales bacteriophage genome[J]. Chin J Virol, 2013, 29(1): 39-43. |
| [30] |
邢少贞, 张湘莉兰, 舒鹏, 等. 粪肠球菌噬菌体vB_E.faecalis_IME196的生物学特性及其全基因组分析[J]. 微生物学通报, 2016, 43(9): 2040-2048. XING S Z, ZHANGXIANG L L, SHU P, et al. Characterization and complete genome sequence analysis of Enterococcus faecalis bacteriophage v B_E. faecalis_IME196[J]. Microbiol China, 2016, 43(9): 2040-2048. |
| [31] |
杨雨卉, 乐率. 铜绿假单胞菌噬菌体治疗的研究进展[J]. 中国抗生素杂志, 2017, 42(10): 814-820. YANG Y H, LE S. Progress on Pseudomonas aeruginosa bacteriophage therapy[J]. Chin J Antibiot, 2017, 42(10): 814-820. |
| [32] |
KIFELEW L G, WARNER M S, MORALES S, et al. Efficacy of lytic phage cocktails on Staphylococcus aureus and Pseudomonas aeruginosa in mixed-species planktonic cultures and biofilms[J]. Viruses, 2020, 12(5): 559. DOI:10.3390/v12050559 |




