2. 610083 成都, 西部战区总医院:重症医学科;
3. 610083 成都, 西部战区总医院:检验科;
4. 610083 成都, 西部战区总医院:急诊科
2. Department of Critical Care Medicine, the General Hospital of Western Theater Command, Chengdu, Sichuan Province, 610083, China;
3. Department of Clinical Laboratory, the General Hospital of Western Theater Command, Chengdu, Sichuan Province, 610083, China;
4. Department of Emergency, the General Hospital of Western Theater Command, Chengdu, Sichuan Province, 610083, China
肺炎克雷伯菌(Klebsiella pneumoniae)是一种广泛存在于环境中的非运动、有荚膜的革兰阴性杆菌[1]。其能够定植于健康人胃肠道、呼吸道及皮肤上,引起尿路感染、呼吸道感染、软组织感染等,严重时可导致患者死亡[2]。随着抗生素的广泛使用和滥用,包括碳青霉烯类耐药肺炎克雷伯菌(carbapenem-resistant Klebsiella pneumonia,CRKP)在内的细菌耐药问题日趋严重[3]。由于新型抗生素的开发停滞不前,研发新型抗菌制剂已成为亟待解决的重大公共卫生问题。
噬菌体分为温和及毒性噬菌体两类,其中毒性噬菌体能在宿主菌内增殖并释放子代噬菌体,从而导致宿主菌死亡[4]。1919年,d’Hérelle用噬菌体制剂成功治疗了4例细菌性痢疾患者,首次证实噬菌体制剂具有临床应用价值[5]。既往研究发现,噬菌体在杀灭细菌方面具有较多优点,如大多数噬菌体具有高度特异性和低自然毒性等[6]。虽然噬菌体的高度特异性可降低治疗中不良反应的风险,但由于宿主谱较窄,因而需要不断地筛选噬菌体。一项研究发现,裂解性噬菌体vB_kpnM_17-11不仅可以在体外抑制CRKP的生长,而且可以在体内降低小鼠腹腔中CRKP的含量,从而提高脓毒症小鼠的存活率[7]。这与CAO等[8]、GENG等[9]的研究一致,为噬菌体疗法在体外和体内的应用提供了更多依据。随着研究的不断深入,还发现噬菌体疗法具有可通过口服和局部等多种途径给药,在体内和抗生素具有协同效应等特点[10-12]。同时,噬菌体疗法在临床研究中也显示出了良好的应用前景。BAO等[13]报道了因泛耐药肺炎克雷伯菌而发生复发性尿路感染(urinary tract infection,UTI)的63岁女性患者病例,磺胺甲噁唑-甲氧苄啶与噬菌体的联合使用抑制了噬菌体抗性突变体的出现, 最终成功治愈了患者的UTI。但目前噬菌体疗法的研究仍不足,缺乏可供临床应用和推广的大样本噬菌体库和大样本的临床研究证据。因此,深入研究噬菌体疗法对耐药菌感染的防治具有重要意义。
本研究拟以分离自西部战区总医院重症医学科患者的CRKP1作为宿主菌,从西部战区总医院未处理的污水中分离出噬菌体,并研究其生物学特性和全基因组信息,以期为噬菌体制剂的开发和CRKP感染患者的治疗提供参考。
1 材料与方法 1.1 菌株和样品23株碳青霉烯类耐药肺炎克雷伯菌(CRKP1-23)、1株大肠埃希菌(Eco01)、1株沙雷氏菌(Serratia01)、1株阴沟肠杆菌(Ecl01)、1株产气肠杆菌(Eae01)共27株,均由西部战区总医院检验科提供,并通过VITEK2-compact系统鉴定,根据临床和实验室标准协会药敏试验执行标准(CLSI2018)测定抗生素最小抑菌浓度(minimal inhibitory concentration,MIC)。其中宿主菌CRKP1对替卡西林/克拉维酸、哌拉西拉/他唑巴坦、头孢哌酮/舒巴坦、头孢他啶、头孢吡肟、氨曲南、亚胺培南、美罗培南、阿米卡星、妥布霉素、环丙沙星、左旋氧氟沙星、多西环素、米诺环素、替加环素耐药,对多粘菌素B和复方新诺明敏感。污水样品采自西部战区总医院污水处理站。
1.2 主要试剂及仪器SM缓冲液、PEG8000、蛋白胨、琼脂粉、氯化钠等试剂购自北京索莱宝科技有限公司;固体、半固体、LB液体培养基等以标准方法配制。液体LB培养基的配制: 酵母提取物0.5 g,胰蛋白胨1 g,氯化钠1 g,ddH2O 100 mL;半固体LB培养基的配制: 每升液体培养基加7 g的琼脂粉;固体LB培养基的配制: 每升液体培养基加15 g的琼脂粉。
生化培养箱Blue pard购自上海一恒科学仪器有限公司;恒温金属浴LEGEND Micro21R购自杭州博日科技有限公司;高通量生长曲线分析仪SPECTRO star Nano购自德国BMGL abtech公司。
1.3 噬菌体的分离纯化及效价的测定将50 mL未处理的污水与2×LB培养基等体积混合,然后接种于200 μL对数生长期的宿主菌CRKP1,混匀后置于摇床(37 ℃,200 r/min)过夜培养。将培养液离心(4 ℃,10 000 r/min,10 min)后,取上清用0.22 μm的滤器过滤,滤液为噬菌体原液,4 ℃保存。采用双层琼脂平板法[14]纯化噬菌体,用枪头扎单个噬菌斑与含宿主菌的LB培养基在摇床(37 ℃,200 r/min) 过夜培养,离心后过滤上清液,滤液倍比稀释后用双层琼脂平板法分离,上述步骤重复5次,直到平板上出现形状大小均一的噬菌斑,即为纯化的噬菌体vB_KpnP1。
噬菌体效价测定:将100 μL纯化的噬菌体液倍比稀释后使用双层琼脂平板法测噬菌体效价,选择噬菌斑在30~300个的平板,噬菌体的滴度(PFU/mL)=噬菌斑数×稀释倍数×10。
1.4 噬菌体的浓缩及透射电镜观察采用NaCL-PEG8000进行噬菌体颗粒的浓缩[15],将20 μL浓缩后噬菌体液(1×1011 PFU/mL)滴在碳涂层铜网上孵育2 min,取10 μL 2%磷钨酸负染90 s,用滤纸吸取多余的染料,然后用透射电镜在80 kV下观察噬菌体vB_KpnP1的形态。
1.5 噬菌体的裂解谱测定噬菌体裂解谱通过斑点实验(spot test assay)[16]和双层平板法进行。斑点实验: 将200 μL对数生长期的待测菌液与5 mL半固体LB混匀后倒入固体LB上,将5 μL纯化后的噬菌体液(108 PFU/mL)滴在双层琼脂培养基的中心,待吸收后倒置,37 ℃过夜培养。双层琼脂平板法: 将噬菌体液梯度稀释(10倍稀释),将稀释成不同浓度的噬菌体液分别与27株细菌混合后用双层琼脂平板法检测vB_KpnP1能否裂解细菌,可同时获得vB_KpnP1对细菌的裂解能力即成斑率(efficiency of plating,EOP),将vB_KpnP1与宿主菌形成的噬菌斑的数量作为标准,测定并计算出测试菌株与vB_KpnP1形成的噬菌斑数量,两者相比即为EOP[17]。
1.6 噬菌体的氯仿稳定性将1 mL纯化的噬菌体液(1×108 PFU/mL)分别与20、200 μL氯仿均匀混合,振荡30 s后于37 ℃静置1 h。离心(4 ℃,5 000×g,5 min)后取出10 μL上清液,梯度稀释后测效价。实验重复3次,用等量的SM缓冲液作阴性对照组。
1.7 噬菌体的温度和pH稳定性参照张洋等[18]描述的方法,将1 mL纯化后的噬菌体液(1×108 PFU/mL),在不同温度(10、20、30、40、50、60、70 ℃)下分别孵育20、40、60 min后取样,然后立即用双层琼脂平板法测效价,实验重复3次,取平均数。
用盐酸和NaOH溶液将SM缓冲液调到不同的pH值(1~13),用0.22 μm的滤器过滤除菌,将100 μL纯化后的噬菌体液(1×108 PFU/mL)与900 μL不同pH值(1~13)的SM缓冲液混合后37 ℃恒温孵育2 h,用双层琼脂平板法测效价,实验重复3次,取平均值。
1.8 噬菌体的最佳感染复数(multiplicity of infection,MOI)参照PU等[19]描述的方法,将100 μL培养至对数生长期的CRKP1的连续梯度稀释液与等体积的不同效价的噬菌体液按表 2的比例混合后,将200 μL不同MOI混合物加入1 mL新鲜LB培养基中,摇床(37 ℃,220 r/min)中培养3~5 h,离心(4 ℃,10 000×g,10 min),0.22 μm的滤器过滤后,用双层琼脂平板法测定噬菌体效价,最高滴度为噬菌体的最佳MOI。实验重复3次,取平均值。
1.9 噬菌体的一步生长曲线参照石云龙[20]描述的方法,将噬菌体vB_KpnP1与宿主菌CRKP1以最佳感染复数(MOI=0.000 1)的比例混合后加入5 mL液体LB培养基中,37 ℃下孵育15 min,离心(4 ℃,13 000×g,30 s)后弃上清,沉淀用液体LB重悬2次,将重悬后的混合物加入37 ℃预热的5 mL液体LB培养基中,在37 ℃、220 r/min摇床上震荡培养120 min。每10分钟取样1次,共取样13次,双层平板法测定其滴度。实验重复3次,取平均值。爆发量=爆发末期噬菌体滴度/感染初期宿主菌浓度[21]。
1.10 噬菌体的吸附实验将噬菌体液(1×106 PFU/mL)与宿主菌(1×108 CFU/mL)以MOI=0.01比例混合,然后在37 ℃孵育,分别于0、2、4、6、8、10、12、14、16、18、20 min取100 μL混合液与900 μL SM缓冲液混匀后,立即离心(4 ℃,13 000×g,1 min),用双层平板法测游离噬菌体效价,噬菌体的吸附率为每个时间点吸附的噬菌体效价相对于总噬菌体效价的百分比[22]。实验重复3次,取平均值。
1.11 噬菌体的体外溶菌活性测定将宿主菌培养至对数生长期,噬菌体液用SM缓冲液倍比稀释,将噬菌体与细菌各取100 μL分别按MOI为10、1、0.1、0.01的比例混合,加入96孔板中;阴性对照组为100 μL SM缓冲液加100 μL LB液体培养基;阳性对照组为100 μL菌悬液加100 μL LB液体培养基。加样完成后,将96孔板放入全自动生长曲线分析仪中,37 ℃孵育24 h,每隔10 min测定光密度值[D(600)]。实验重复3次,取平均值。
1.12 噬菌体的全基因组测序及生物信息学分析采用平衡酚-氯仿-异戊醇法进行DNA提取[23],并通过1%的琼脂糖凝胶电泳检测基因组的完整性。DNA样品送至成都生命基线科技有限公司进行二代测序和全基因组测序分析。
使用RAST(http://rast.nmpdr.org/)对噬菌体全基因组进行注释,使用在线BLAST工具(http://www.ncbi.nlm.nih.gov/BLAST)进行比对分析,预测基因组功能。使用在线抗生素耐药基因和毒力因子库(https://cge.cbs.dtu.dk/services/Res Finder/和https://cge.cbs.dtu.dk/services/Virulence Finder/)筛选假定的抗生素耐药基因和毒力因子。用tRNA scan-SE v.2.0软件(http://lowelab.ucsc.edu/tRNAscan-SE/)识别tRNA编码序列,使用CG View Server软件(http://cgview.ca/)绘制全基因组图谱。采用MEGA 11.0软件,用最大似然法构建全基因组、末端酶大亚基和DNA聚合酶基因的系统发育进化树,bootstrap检验1 000次。采用Easyfig 2.2.3软件进行比较基因分析。
1.13 数据及图像处理使用GraphPad Prism 8.0软件进行制图及数据分析。计量资料以x±s表示。使用Photoshop软件进行添加标尺等图像处理。
2 结果 2.1 噬菌体的分离及透射电镜观察成功从西部战区总医院污水中分离出1株CRKP噬菌体,噬菌斑为2~3 mm透亮的圆形,周围有6~8 mm半透明的晕环(图 1A),随着时间的延长,晕环逐渐增大。电镜下发现该噬菌体的头部为正二十面体,大小约68.2 nm×64.3 nm,有1条不可收缩的短尾,尾部长约11 nm(图 1B)。通过透射电镜观察和全基因组测序分析,该噬菌体属于有尾噬菌体目,自复制短尾噬菌体科(Autographiviridae)。按照国际病毒分类委员会(The International Committee on Taxonomy of Viruses,ICTV)分类与命名原则,将噬菌体命名为vB_KpnP1。
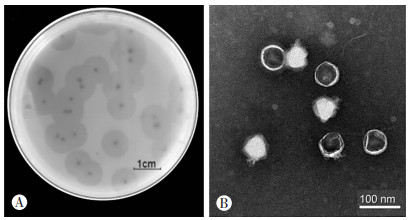
|
| A:噬菌体vB_KpnP1的噬菌斑;B:噬菌体vB_KpnP1的电镜图 图 1 噬菌体vB_KpnP1的形态鉴定 |
2.2 噬菌体的裂解谱测定
如表 1所示,通过斑点实验和双层平板法测定噬菌体vB_KpnP1的宿主谱,斑点实验提示噬菌体对碳青霉烯类耐药肺炎克雷伯菌裂解率为59.09%(13/22),双侧琼脂平板法提示噬菌体对碳青霉烯类耐药肺炎克雷伯菌裂解率为54.55%(12/22);在12株可以被噬菌体感染的肺炎克雷伯菌中,9株具有高效的成斑率,2株具有中效的成斑率,1株为低效成斑率。以上实验结果表明,斑点实验得到的宿主谱要广于双层琼脂平板法。vB_KpnP1不能裂解大肠埃希菌、沙雷氏菌、阴沟肠杆菌、产气肠杆菌。
| 菌株名称 | 斑点实验 | 成斑率 | 菌株名称 | 斑点实验 | 成斑率 | |
| CRKP02 | + | 1.24 | CRKP15 | - | N | |
| CRKP03 | + | 0.925 | CRKP16 | + | 0.84 | |
| CRKP04 | + | 0.715 | CRKP17 | + | 0.795 | |
| CRKP05 | - | N | CRKP18 | - | N | |
| CRKP06 | - | N | CRKP19 | + | 0.53 | |
| CRKP07 | - | N | CRKP20 | - | N | |
| CRKP08 | + | 0.45 | CRKP21 | + | 0.29 | |
| CRKP09 | + | N | CRKP22 | + | 1.085 | |
| CRKP10 | - | N | CRKP23 | + | 1.505 | |
| CRKP11 | - | N | Eco01 | - | N | |
| CRKP12 | - | N | Serratia01 | - | N | |
| CRKP13 | + | 0.05 | Ecl01 | - | N | |
| CRKP14 | + | 0.5 | Eae01 | - | N | |
| +: 裂解;-:没有裂解;N: 没有噬菌斑形成;噬菌体成斑率分为高效(EOP≥0.5)、中效(0.1≤EOP < 0.5)、低效(0.001 < EOP < 0.1)和无效(EOP≤0.001) | ||||||
2.3 噬菌体的氯仿稳定性
噬菌体效价经20、200 μL的氯仿作用后由9.9× 107 PFU/mL分别变为9.75×107、9.5×107 PFU/mL,表明该噬菌体对氯仿不敏感,推测其无囊膜。
2.4 噬菌体的温度和pH稳定性如图 2A所示,噬菌体vB_KpnP1在10~50 ℃有较高的活性,在60 ℃时作用1 h,噬菌体完全失活,在70 ℃时作用40 min,噬菌体也完全失活。
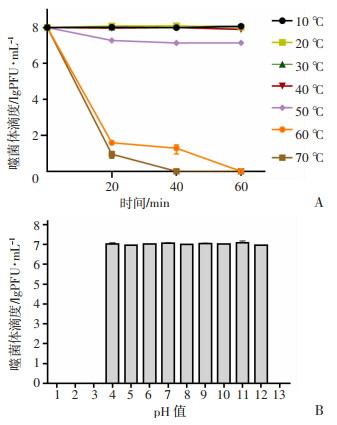
|
| A:温度稳定性;B:pH稳定性 图 2 噬菌体vB_KpnP1的温度和酸碱度稳定性 |
如图 2B所示,噬菌体vB_KpnP1在pH值为4~12时活性变化不大,其效价基本稳定,在强酸或强碱环境中(pH值为1~3或13)被灭活,其结果表明该噬菌体对酸碱有一定的耐受性,但不耐强酸强碱。
2.5 噬菌体的最佳感染复数如表 2所示,当感染复数为0.000 1时,噬菌体vB_KpnP1效价最高,为6.53× 108 PFU/mL,表明该噬菌体的最佳感染复数为0.000 1。
| 组号 | 细菌数/CFU·mL-1 | 噬菌体数/PFU·mL-1 | 感染复数 | vB_KpnP1滴度/PFU·mL-1 |
| 1 | 1×107 | 1×109 | 100 | 2.10×107 |
| 2 | 1×108 | 1×109 | 10 | 2.47×107 |
| 3 | 1×108 | 1×108 | 1 | 4.80×107 |
| 4 | 1×108 | 1×107 | 0.1 | 1.37×108 |
| 5 | 1×108 | 1×106 | 0.01 | 3.73×108 |
| 6 | 1×108 | 1×105 | 0.001 | 3.63×108 |
| 7 | 1×108 | 1×104 | 0.000 1 | 6.53×108 |
| 8 | 1×108 | 1×103 | 0.000 01 | 3.10×108 |
2.6 噬菌体的一步生长曲线
由图 3可知,噬菌体vB_KpnP1的潜伏期为10 min,在此期间,噬菌体的效价非常缓慢地升高,在感染后10~40 min,噬菌体效价明显上升,此后较稳定,表示裂解期为30 min,裂解末期该噬菌体的滴度为2.1× 108 PFU/mL。根据爆发量的计算公式,计算如下: 2.1×108÷(8× 106)=26 PFU/cell。
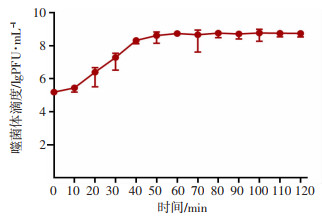
|
| 图 3 噬菌体vB_KpnP1的一步生长曲线 |
2.7 噬菌体吸附曲线试验
如图 4所示,当噬菌体vB_KpnP1和宿主菌混合孵育4 min就有60%左右的噬菌体吸附到宿主上,10 min时就有80%左右的噬菌体吸附到宿主菌中,然后趋于稳定。
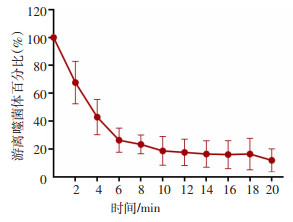
|
| 图 4 噬菌体vB_KpnP1的吸附曲线 |
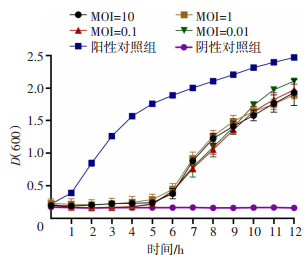
2.8 噬菌体体外抑菌实验
如图 5所示,阳性对照组光密度值不断增长,即细菌在不断生长;与阳性对照组相比,加入不同感染复数的实验组在噬菌体vB_KpnP1的作用下,光密度值低于阳性对照组,直到5 h后,细菌浓度迅速增长,随之光密度值增高,推测为噬菌体抗性菌株的产生。
|
| 图 5 噬菌体vB_KpnP1在不同感染复数下的体外抑菌实验 |
2.9 噬菌体的基因组学分析
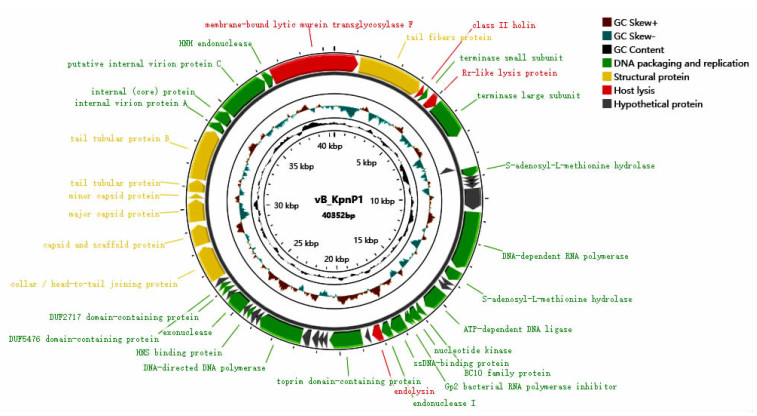
噬菌体vB_KpnP1基因组为双链DNA,全基因组长度为40 352 bp,基因组中4种碱基的含量分别为A=25.18%、T=21.73%、C=25.26%和G=27.83%,GC含量为53.09%,低于肺炎克雷伯菌株平均GC的含量(57.7%)。通过注释和比对后,该基因组共比对到50个编码序列(coding sequence, CDS)。其中46个CDS以ATG作为起始密码子,3个CDS以GTG作为起始密码子,1个CDS以TTG作为起始密码子。tRNAscan-SE v.2.0分析vB_KpnP1全基因组无tRNA结构,也未预测出已知的耐药基因和毒力因子,说明该噬菌体在基因水平上具有安全性。
噬菌体vB_KpnP1基因组共预测到50个CDS,其中49个CDS在正义链,1个CDS在反义链上。在50个CDS中,有32个(64%)CDS编码功能蛋白(图 6),其余的18个CDS为功能未知的假定蛋白。根据基因的功能将32个CDS分为3个模块: 噬菌体DNA复制及装配调控、噬菌体结构蛋白和宿主细胞溶解。其中约21个CDS与噬菌体DNA复制及装配有关,如terminase large subunit,DNA-dependent RNA polymerase、ATP-dependent DNA ligase和BC10 family protein等;7个CDS与噬菌体的结构有关,如tail fibers protein,collar/head-to-tail joining protein、capsid and scaffold protein、major capsid protein和tail tubular protein等;4个CDS为class Ⅱ holin、endolysin、Rz-like lysis protein和membrane-bound lytic murein transglycosylase F与宿主菌溶解有关。
|
| 图 6 噬菌体vB_KpnP1基因组图谱 |
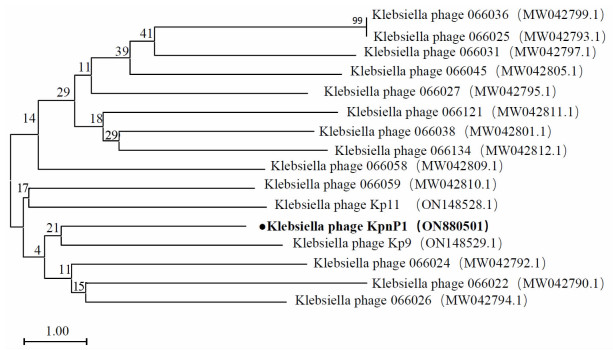
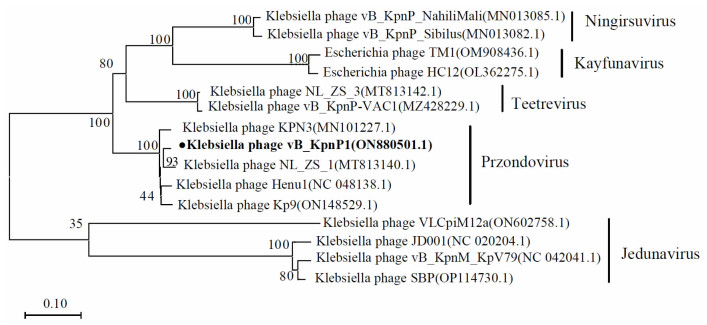
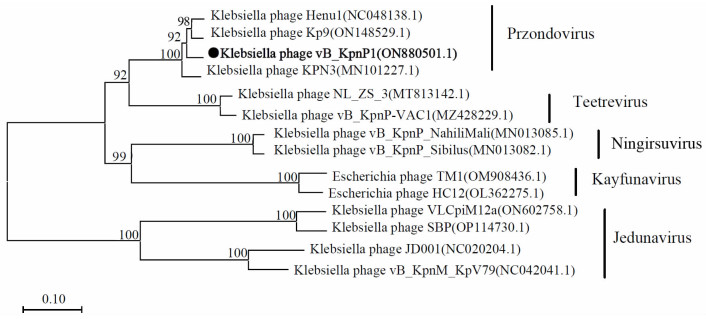
用MEGA11软件,采用Neighbor-joining method法,针对16株噬菌体的全基因组构建进化树。如图 7所示,vB_KpnP1与肺炎克雷菌噬菌体Kp9、066024、066022、066026亲缘关系较近,与BLASTn结果一致,这4种噬菌体与vB_KpnP1具有核苷酸相似性(一致度:93.93%~97.69%,覆盖度:86%~97%)。基于噬菌体末端酶大亚基和DNA聚合酶构建系统进化树如图 8、9所示,噬菌体vB_KpnP1与Przondovirus属的肺炎克雷伯菌噬菌体NL_ZS_1、KPN3、Henu1和Kp9处于同一分支,推测该噬菌体属于Autographiviridae科,Studiervirinae亚科,Przondovirus属,未分类的Przondovirus种(unclassified Przondovirus)。该噬菌体与同一亚科的Ningirsuvirus、Kayfunavirus和Teetrevirus属的肺炎克雷伯菌噬菌体密切相关,有一定的亲缘关系。
|
| 图 7 基于噬菌体全基因组构建的同源进化树 |
|
| 图 8 基于噬菌体末端酶大亚基构建的同源进化树 |
|
| 图 9 基于噬菌体DNA聚合酶构建的同源进化树 |
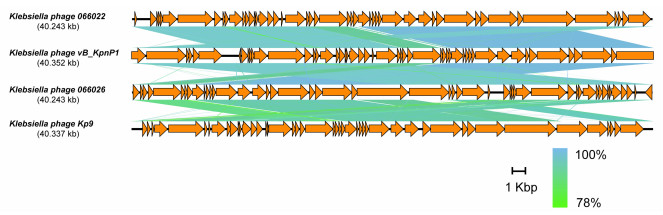
用Easyfig 2.2.3软件,选择与vB_KpnP1同属的噬菌体KP9(Przondovirus属的噬菌体)以及与其在公共核苷酸数据库中最接近的噬菌体Klebsiella phage 066022和066026进行比较。如图 10所示,vB_KpnP1与肺炎克雷伯菌噬菌体Klebsiella phage 066022关系最密切(一致度:97.7%,覆盖度:97%)。它是一种新发现的噬菌体,也是Autographiviridae科,Studiervirinae亚科的成员。
|
| 黄色箭头:CDS;蓝色连线:相似度较高;绿色连线:相似度较低 图 10 噬菌体vB_KpnP1的比较基因组分析 |
3 讨论
据世界卫生组织估计,到2050年,耐药性细菌感染每年可能导致约1 000万人死亡[23]。其中,CRKP感染可导致患者治疗失败、住院时间延长、死亡率升高[24],而CRKP是造成这一严重公共卫生危机的重要细菌之一[25]。最近的一项系统评价和Meta分析指出,肺炎克雷伯菌对厄他培南和美罗培南的耐药率分别为44.4%和36.1%,然而更令人担忧的是肺炎克雷伯菌对粘菌素耐药率也明显升高[26]。因此,迫切需要针对耐药菌感染研发新的抗菌制剂和治疗策略。
本研究成功分离出1株烈性噬菌体vB_KpnP1,进一步拓展了噬菌体样本库,为后续研究奠定了基础。研究发现选择宿主谱较广的噬菌体进行治疗时,不仅能够杀灭更多的细菌,而且与窄谱的噬菌体相比更不容易产生噬菌体抗性突变体。然而,既往的许多研究并没有区分点滴法和双层琼脂平板法所获得的宿主谱的差别。点滴法的宿主谱常大于50%,有时还能裂解其他属的细菌,通常高估了噬菌体的毒力和宿主范围[27]。这主要有以下两个方面的原因:①噬菌体液中含有可杀死细菌的细菌素;②噬菌体本身的“流产感染”和“自外裂解”[28]。因此噬菌体库中噬菌体的选择和鉴定不能使用点滴法,应用成斑率测定结果代替。双层琼脂平板法表明该噬菌体在75%(9/12)的肺炎克雷伯菌中可形成高效的成斑率,略高于噬菌体KPP-5,它在73.68%(14/19)的肺炎克雷伯菌中具有高效的成斑率[29]。因此,应尽量选择双层琼脂平板法中具有高效成斑率的噬菌体进行治疗,有利于更好地杀灭细菌。
噬菌体的稳定性与储存、运输和治疗密切相关,其中,稳定性较好的噬菌体才具有进一步应用的潜力。JAMALLUDEEN等[30]研究发现,大多数的噬菌体在生理条件的pH值(5~9)中能够保持正常的结构和稳定性。该噬菌体在运用的常见环境下的温度(10~50 ℃) 和酸碱度(pH值为4~12)中保持稳定,表明它有进一步运用的潜力。该噬菌体的最佳感染复数为0.000 1,表明此时噬菌体的增殖效率最高,单位噬菌体所产生的后代最多。因此在制备大量噬菌体时,常常选择最佳MOI培养噬菌体,从而降低生产成本并且增加利润。在噬菌体中潜伏期短、爆发量大的噬菌体可更快和更多地产生后代,有利于噬菌体快速和大量扩增。噬菌体vB_KpnP1与既往报道的噬菌体ϕBO1E的潜伏期相同均为10 min,但爆发量远远低于ϕBO1E的爆发量300 PFU/cell[31]。vB_KpnP1的爆发量低可能成为今后应用的一个限制。
经过全基因组测序注释后,将噬菌体序列上传至genbank (genbank登录号: ON880501)。该噬菌体的50个CDS中有18个CDS是未知功能的基因,表明其基因组序列有待进一步研究。实验发现噬菌斑周围的光晕随着时间的延长不断增大,表明它编码多糖解聚酶,多糖解聚酶通常是短尾和自复制短尾噬菌体科中的尾刺或尾纤维蛋白[32],可通过降解细菌荚膜多糖和胞外聚合物使噬菌体更易接近宿主,这有利于更好地杀灭细菌。vB_KpnP1与kpssk3都为Autographiviridae的成员,其中vB_KpnP1的CDS2与kpssk3的尾纤维蛋白有很高的核苷酸相似性(一致度:98.45%,覆盖度:99%)。因此本研究将CDS2注释为尾纤维蛋白,并且推测它与kpssk3的尾纤维蛋白相似,它们既充当结构蛋白同时兼具多糖解聚酶的功能[19]。噬菌体功能注释中发现噬菌体vB_KpnP1采用穿孔素-裂解酶系统来裂解细菌。一般来讲,在基因组中编码穿孔素和裂解酶的基因相邻,然而在vB_KpnP1基因组中却发现它们相距很远,这种独特的遗传结构已在SH-KP152226、P13和vB_Kox_ZX8噬菌体中报道过[33]。噬菌体侵染革兰阴性菌时首先为裂解蛋白逐渐积累,其次穿孔素在宿主内膜形成跨膜孔,使裂解酶能够从胞质中到细胞壁肽聚糖(peptidoglycan,PG)层从而裂解PG,最后spanins破坏宿主外膜导致细菌裂解[34]。研究发现噬菌体vB_KpnP1与Przondovirus属的噬菌体基因组中都含有Rz-like lysis protein,它作为一种连接宿主内膜和外膜的信号锚定蛋白,可介导宿主内膜和外膜的融合,从而导致宿主完全裂解[35]。噬菌体的末端酶大亚基和DNA聚合酶的系统发育树均与大肠杆菌噬菌体有关,表明噬菌体vB_KpnP1有可能具有裂解大肠杆菌的能力,有待进一步研究。
| [1] |
HERRIDGE W P, SHIBU P, O'SHEA J, et al. Bacterio-phages of Klebsiella spp., their diversity and potential therapeutic uses[J]. J Med Microbiol, 2020, 69(2): 176-194. |
| [2] |
PODSCHUN R, ULLMANN U. Klebsiella spp as nosocomial pathogens: epidemiology, taxonomy, typing methods, and pathogenicity factors[J]. Clin Microbiol Rev, 1998, 11(4): 589-603. |
| [3] |
CHANG D, SHARMA L, DELA CRUZ C S, et al. Clinical epidemiology, risk factors, and control strategies of Klebsiella pneumoniae infection[J]. Front Microbiol, 2021, 12: 750662. |
| [4] |
SALMOND G P, FINERAN P C. A century of the phage: past, present and future[J]. Nat Rev Microbiol, 2015, 13(12): 777-786. |
| [5] |
D'HERELLE F. Studies upon Asiatic cholera[J]. Yale J Biol Med, 1929, 1(4): 195-219. |
| [6] |
LOC-CARRILLO C, ABEDON S T. Pros and cons of phage therapy[J]. Bacteriophage, 2011, 1(2): 111-114. |
| [7] |
BAI J W, ZHANG F Y, LIANG S, et al. Isolation and characterization of vB_kpnM_17-11, a novel phage efficient against carbapenem-resistant Klebsiella pneumoniae[J]. Front Cell Infect Microbiol, 2022, 12: 897531. |
| [8] |
CAO F, WANG X T, WANG L H, et al. Evaluation of the efficacy of a bacteriophage in the treatment of pneumonia induced by multidrug resistance Klebsiella pneumoniae in mice[J]. Biomed Res Int, 2015, 2015: 752930. |
| [9] |
GENG H J, ZOU W, ZHANG M X, et al. Evaluation of phage therapy in the treatment of Staphylococcus aureus-induced mastitis in mice[J]. Folia Microbiol (Praha), 2020, 65(2): 339-351. |
| [10] |
DHUNGANA G, REGMI M, PAUDEL P, et al. Therapeutic efficacy of bacteriophage therapy to treat carbapenem resistant Klebsiella pneumoniae in mouse model[J]. J Nepal Health Res Counc, 2021, 19(1): 76-82. |
| [11] |
FAYEZ M S, HAKIM T A, AGWA M M, et al. Topically applied bacteriophage to control multi-drug resistant Klebsiella pneumoniae infected wound in a rat model[J]. Antibiotics (Basel), 2021, 10(9): 1048. |
| [12] |
OECHSLIN F, PICCARDI P, MANCINI S, et al. Synergistic interaction between phage therapy and antibiotics clears pseudomonas aeruginosa infection in endocarditis and reduces virulence[J]. J Infect Dis, 2017, 215(5): 703-712. |
| [13] |
BAO J, WU N N, ZENG Y G, et al. Non-active antibiotic and bacteriophage synergism to successfully treat recurrent urinary tract infection caused by extensively drug-resistant Klebsiella pneumoniae[J]. Emerg Microbes Infect, 2020, 9(1): 771-774. |
| [14] |
CHHIBBER S, KAUR P, GONDIL V S. Simple drop cast method for enumeration of bacteriophages[J]. J Virol Methods, 2018, 262: 1-5. |
| [15] |
单文雅, 王兆飞, 杨登辉, 等. 一株可裂解阴沟肠杆菌的T7样噬菌体的分离鉴定及基因组分析[J]. 上海交通大学学报(农业科学版), 2019, 37(1): 74-80, 86. SHAN W Y, WANG Z F, YANG D H, et al. Isolation, identification and genomic analysis of a T7-like bacteriophage that can lyse enterobacter cloacae[J]. J Shanghai Jiaotong Univ Agric Sci, 2019, 37(1): 74-80, 86. |
| [16] |
DONG Z X, XING S Z, LIU J, et al. Isolation and characterization of a novel phage Xoo-sp2 that infects Xanthomonas oryzae pv. oryzae[J]. J Gen Virol, 2018, 99(10): 1453-1462. |
| [17] |
ZHANG W J, ZHANG R R, HU Y J, et al. Biological characteristics and genomic analysis of a stenotrophomonas maltophilia phage vB_SmaS_BUCT548[J]. Virus Genes, 2021, 57(2): 205-216. |
| [18] |
张洋, 贾凯翔, 张秀文, 等. 噬菌体PZL-Ah1的生物学特性及其治疗嗜水气单胞菌感染的效果评价[J]. 中国预防兽医学报, 2021, 43(6): 602-608. ZHANG Y, JIA K X, ZHANG X W, et al. Biological characteristics of bacteriophage PZL-Ah1 and evaluation of its efficacy in the treatment of aeromonas hydrophila infection[J]. Chin J Prev Vet Med, 2021, 43(6): 602-608. |
| [19] |
PU M F, HAN P J, ZHANG G Y, et al. Characterization and comparative genomics analysis of a new bacteriophage BUCT610 against Klebsiella pneumoniae and efficacy assessment in Galleria mellonella larvae[J]. Int J Mol Sci, 2022, 23(14): 8040. |
| [20] |
石云龙. 碳青霉烯类耐药肺炎克雷伯菌噬菌体的分离鉴定与应用研究[D]. 重庆: 中国人民解放军陆军军医大学, 2021. SHI Y L. Isolation, identification and application of carbapenem-resistant Klebsiella pneumoniae phages[D]. Chongqing: Army Medical University, 2021. |
| [21] |
WANG R H, CONG Y, MI Z Q, et al. Characterization and complete genome sequence analysis of phage GP4, a novel lytic Bcep22-like podovirus[J]. Arch Virol, 2019, 164(9): 2339-2343. |
| [22] |
HYMAN P, ABEDON S T. Practical methods for determining phage growth parameters[J]. Methods Mol Biol, 2009, 501: 175-202. |
| [23] |
HAN P J, HU Y J, AN X P, et al. Biochemical and genomic characterization of a novel bacteriophage BUCT555 lysing Stenotrophomonas maltophilia[J]. Virus Res, 2021, 301: 198465. |
| [24] |
TAATI MOGHADAM M, KHOSHBAYAN A, CHEGINI Z, et al. Bacteriophages, a new therapeutic solution for inhibiting multidrug-resistant bacteria causing wound infection: lesson from animal models and clinical trials[J]. Drug Des Devel Ther, 2020, 14: 1867-1883. |
| [25] |
XU L F, SUN X X, MA X L. Systematic review and meta analysis of mortality of patients infected with carbapenem-resistant Klebsiella pneumoniae[J]. Ann Clin Microbiol Antimicrob, 2017, 16(1): 18. |
| [26] |
UZAIRUE L I, RABAAN A A, ADEWUMI F A, et al. Global prevalence of colistin resistance in Klebsiella pneumoniae from bloodstream infection: a systematic review and meta analysis[J]. Pathogens, 2022, 11(10): 1092. |
| [27] |
KHAN MIRZAEI M, NILSSON A S. Isolation of phages for phage therapy: a comparison of spot tests and efficiency of plating analyses for determination of host range and efficacy[J]. PLoS One, 2015, 10(3): e0118557. |
| [28] |
HYMAN P, ABEDON S T. Bacteriophage host range and bacterial resistance[J]. Adv Appl Microbiol, 2010, 70: 217-248. |
| [29] |
SOFY A R, EL-DOUGDOUG N K, REFAEY E E, et al. Characterization and full genome sequence of novel KPP-5 lytic phage against Klebsiella pneumoniae responsible for recalcitrant infection[J]. Biomedicines, 2021, 9(4): 342. |
| [30] |
JAMALLUDEEN N, JOHNSON R P, FRIENDSHIP R, et al. Isolation and characterization of nine bacteriophages that lyse O149 enterotoxigenic escherichia coli[J]. Vet Microbiol, 2007, 124(1/2): 47-57. |
| [31] |
D'ANDREA M M, MARMO P, HENRICI DE ANGELIS L, et al. φBO1E, a newly discovered lytic bacteriophage targeting carbapenemase-producing Klebsiella pneumoniae of the pandemic Clonal Group 258 clade Ⅱ lineage[J]. Sci Rep, 2017, 7(1): 2614. |
| [32] |
PIRES D P, OLIVEIRA H, MELO L D, et al. Bacteriophage-encoded depolymerases: their diversity and biotechnological applications[J]. Appl Microbiol Biotechnol, 2016, 100(5): 2141-2151. |
| [33] |
LI P, ZHANG Y H, YAN F H, et al. Characteristics of a bacteriophage, vB_Kox_ZX8, isolated from clinical Klebsiella oxytoca and its therapeutic effect on mice bacteremia[J]. Front Microbiol, 2021, 12: 763136. |
| [34] |
CAHILL J, YOUNG R. Phage Lysis: multiple genes for multiple barriers[J]. Adv Virus Res, 2019, 103: 33-70. |
| [35] |
BERRY J, SUMMER E J, STRUCK D K, et al. The final step in the phage infection cycle: the Rz and Rz1 lysis proteins link the inner and outer membranes[J]. Mol Microbiol, 2008, 70(2): 341-351. |




