2. 335000 江西 鹰潭,中国人民武装警察部队江西总队鹰潭支队
2. Yingtan Detachment, Jiangxi Corps, Chinese People's Armed Police Force, Yingtan, Jiangxi Province, 335000, China
汉坦病毒(Hantan virus)是属于布尼亚病毒科正汉坦病毒属的负性RNA病毒[1-2],是一种危害严重的人畜共患病原体。其中汉滩病毒(Hantaan virus,HTNV)主要引起肾综合征出血热(hemorrhagic fever with renal syndrome,HFRS),以发热、出血和急性肾功能损伤为主要临床症状。我国陕西、黑龙江等地区是HTNV引起HFRS的主要疫区。根据中国疾病预防控制局(http://www.nhc.gov.cn/jkj/s3578/202204/4fd88a291d914abf8f7a91f6333567e1.shtml)统计结果显示,2021年全国法定传染病中,HFRS患者死亡率位于乙类传染病死亡数前5位,全年发病数为9 187例,死亡数为64人,对我国公共卫生安全造成重大威胁。
HTNV三股单负链RNA分为小(S)、中(M)和大(L)3个片段,M段编码病毒表面糖蛋白(GP)[3]。GP可水解得到Gn和Gc分子,在HTNV GP中,Gn主要负责宿主细胞吸附,Gc主要介导膜融合[4]。病毒侵入细胞后,大量糖基化的Gn经过裂解和加工进而调节Gc的膜融合作用。在低pH环境下,汉坦病毒的Gc仍能保持稳定的α螺旋折叠结构,这为膜融合提供了驱动力[5],提示Gc在病毒免疫过程中发挥重要的结合作用。JIANG等[6]发现用编码HTNV Gc(pVAX-Gc)的质粒免疫小鼠可产生具有中和HTNV感染效果的特异性抗体。免疫接种后小鼠脾脏淋巴细胞IFN-γ释放增强,提示Gc分子能够引起适应性免疫反应。
抗原表位在机体抗HTNV感染免疫中具有重要作用。MA等[7]预测并验证了HTNV GP衍生的一个小鼠H2-Kb限制性CTL表位。TANG等[8]验证了HTNV GP衍生的HLA-A*0201限制性表位,发现免疫保护表位LL9(aa358-aa366, LIWTGMIDL)。Gc主要介导膜融合,在病毒免疫中发挥重要的结合作用。上述研究结果表明,Gc的表位预测具有靶向性、特异性、安全性等独特优势,具有很大的研究潜力。与此同时MHC-Ⅰ和MHC-Ⅱ两种表位的共性和差异亟待进一步阐明,CD4+CD8+T细胞反应的规律仍待进一步探索。
本研究基于HTNV Gc抗原肽开展表位相关的预测及评估,并进一步比较了Gc相关MHC-Ⅰ和MHC-Ⅱ之间的差异。以比较免疫学和结构免疫学为基础,利用分子生物信息学的原理,采取多种预测工具,探究HTNV Gc上高亲和力、强免疫原性和种间种内均保守的9肽、15肽优势表位,预测表位覆盖率超过全球98%人群[9]。通过模拟肽-MHC分子对接,探究其在体内可能的对接方式。双向聚类与MHC分子共亲和区段探究分析HTNV Gc表位跨地区、跨种群、跨物种之间的相互作用。多方面数据支持和多维度过程探索,为HTNV Gc特异性群体免疫性的表位疫苗研制工作奠定了理论基础。
1 材料与方法 1.1 Gc蛋白序列获取从NCBI GenBank检索获得HTNV 76~118株Gc蛋白序列(P08668#PRO_0000036817)[10]。
1.2 Gc蛋白表位亲和力预测 1.2.1 预测工具的选取利用5种预测算法包括IEDB[11](https://www.iedb.org/),SMMPMBEC[12](https://github.com/ykimbiology/smmpmbec),NetMHCpan 4.1[13](https://services.healthtech.dtu.dk/service.php?NetMHCpan-4.1),SYFPEITHI[14](http://www.syfpeithi.de/bin/MHCServer.dll/EpitopePrediction.htm),Rankpep[15](http://imed.med.ucm.es/Tools/rankpep.html)全面无偏倚地获得高亲和力的表位候选肽,并对HTNV Gc蛋白序列进行序列寡肽分割,计算MHC-Ⅰ类分子与9肽表位之间的亲和力;对于MHC-Ⅱ类分子与15肽表位之间亲和力的预测,利用4种预测算法包括IEDB、NetMHCIIpan3.2[16](https://services.healthtech.dtu.dk/service.php?NetMHCIIpan-3.2)、SYFPEITHI、Rankpep。
1.2.2 MHC-Ⅰ/Ⅱ类限制性表位预测与筛选对IEDB推荐的人类主要白细胞抗原HLA-Ⅰ类亚型等位基因、Allel Frequency网站[17](http://www.allelefrequencies.net/)中各地区基因频率>10%的HLA基因型(表 1)以及小鼠H-2基因型H2-Db、H2-Dd、H2-Kb、H2-Kd、H2-Kk、H2-Ld选定进行亲和力分析,其中HLA-Ⅰ基因型覆盖了超过99.3%人群的MHC-Ⅰ类亚型,HLA-Ⅱ基因型覆盖了超过98%人群的MHC-Ⅱ类亚型。选取每种算法结果前2%的预测表位为候选优势表位,MHC-Ⅰ类限制性表位选择至少3种预测算法结果中出现的表位作为优势表位,MHC-Ⅱ类限制性表位选择至少2种预测算法结果中出现的表位作为优势表位。
| HLA家族 | HLA超家族 | HLA基因型 |
| HLA-Ⅰ | HLA-A1 | HLA-A*0101、-A*2601、-A*3001、-A*3002 |
| HLA-A2 | HLA-A*0201、-A*0203、-A*0206、-A*0207、-A*0212、-A*6802 | |
| HLA-A3 | HLA-A*0301、-A*1101、-A*3001、-A*3101、-A*3301、HLA-A*3303、-A*6801 | |
| HLA-A24 | HLA-A*2301、-A*2402、-A*2407、-A*3201 | |
| HLA-B7 | HLA-B*0702、-B*3501、-B*5101、-B*5301 | |
| HLA-B8 | HLA-B*0801 | |
| HLA-B15 | HLA-B*1501、-B*1502、-B*1506、-B*1512、-B*1521 | |
| HLA-B44 | HLA-B*4001、-B*4002、-B*4006、-B*4402、-B*4403、-B*4404 | |
| HLA-B58 | HLA-B*5701、-B*5801 | |
| outlier | HLA-A*3401、HLA-B*1301、HLA-B*1405、HLA-B*1807、HLA-B*3508、HLA-B*3903、HLA-B*4601、HLA-B*5001、HLA-B*5110、HLA-B*5201、HLA-B*5502、HLA-B*5601 | |
| DRB1 | DRB1*0101,DRB1*0301,DRB1*0304,DRB1*0401,DRB1*0405,DRB1*0407,DRB1*0412,DRB1*0701,DRB1*0704,DRB1*0802,DRB1*0803,DRB1*0901,DRB1*1101,DRB1*1104,DRB1*1201,DRB1*1202,DRB1*1301,DRB1*1302,DRB1*1304,DRB1*1401,DRB1*1402,DRB1*1408,DRB1*1409,DRB1*1501,DRB1*1502,DRB1*1503,DRB1*1602 | |
| HLA-Ⅱ | DRB3/4/5 | DRB3*0101,DRB3*0202,DRB4*0101,DRB5*0101 |
| DQA1/DQB1 | DQA1*0501/DQB1*0201,DQA1*0501/DQB1*0301,DQA1*0301/DQB1*0302,DQA1*0401/DQB1*0402,DQA1*0101/DQB1*0501,DQA1*0102/DQB1*0602 | |
| DPB1 | DPB1*0101,DPB1*0201,DPB1*0401,DPB1*0402,DPB1*0501,DPB1*1401 |
1.3 HTNV Gc MHC-Ⅰ/Ⅱ类限制性表位免疫原性分析
高亲和力肽可能不能充分诱导免疫应答,因此优势表位除了具有免疫反应性外,还应具有免疫原性。免疫原性由氨基酸序列所决定。VaxiJen-2.0[18](http://www.ddg-pharmfac.net/vaxijen/VaxiJen/VaxiJen.html)用于计算9肽、15肽表位的免疫原性。视评分>0.5为阳性标准,即该表位被视为强免疫原性;否则被视为不具有免疫原性。在同等条件下可以认为免疫原性强的表位更有可能充分诱导免疫应答。
1.4 HTNV Gc MHC分子间的共亲和区段分析为了进一步探究具有高亲和力和强免疫原性的优势表位其跨人群、跨物种的交叉免疫反应性,对比分析了不同MHC分子对应优势表位所在的区段。计数重复区段后利用Excel绘制柱形图,可以直观得出不同基因型之间的交叉免疫反应性。其中通过数据库NetMHCpan得到的MHC-Ⅱ类限制性15肽表位的核心9肽视为该15肽所在区段。
1.5 HTNV Gc MHC-Ⅰ/Ⅱ类限制性表位保守性预测为了确定HTNV 76~118毒株Gc序列中优势抗原的进化保守性,使用BLASTP[19](https://blast.ncbi.nlm.nih.gov/Blast.cgi)工具预测高亲和力HTNV Gc 9肽和15肽的种间种内保守性。其中,种间保守的评价标准为正汉坦病毒(Orthohantavirus, taxid:1980442),而除外汉滩病毒(HTNV, taxid:1980471)、人(human, taxid: 9606)、鼠(mouse, taxid: 10088)。种内保守评价标准为汉滩病毒(Hantaan hantavirus, taxid:1980471),而除外汉滩病毒76~118株(Hantaan virus 76~118, taxid:11602)、人(human, taxid: 9606)、鼠(mouse, taxid: 10088)。在分析结果中,若E值小于10-5,则认为保守。因此,根据保守性结果统计不同MHC基因型对应的种间种内均保守的优势表位数量。
1.6 双向层次聚类分析MHC分子的多态性和表位肽氨基酸序列的多样性构成了pMHC相互作用的差异性。为使其中的关系可视化,利用TBtools[20]对MHC-Ⅰ、Ⅱ类超家族和HTNV Gc相关肽的亲和力排名进行双向层次聚类。亲和力排序数据用以2为底的对数和Z-Score处理之后,用Complete方法进行欧氏距离的双向层次聚类。分析包含57个MHC-Ⅰ类分子与HTNV Gc 9肽相互作用及51个MHC-Ⅱ类分子与HTNV Gc 15肽表位相互作用,绘制热图表示。
1.7 模拟pMHC分子对接 1.7.1 HTNV Gc 9肽与MHC-Ⅰ类分子模拟对接HPEPDOCK(http://huanglab.phys.hust.edu.cn/hpepdock/)[21]是一种新型的网络服务器,它通过输入9肽优势表位序列与MHC-Ⅰ类分子RDB格式文件即可模拟分子对接,从而获得对接模型。使用RCSB PDB数据库(https://www.rcsb.org/)即可获得相关MHC-Ⅰ类分子3D结构数据:HLA-A0206(3OXR)、HLA-A2402(5WWJ)、HLA-A0101(4NQX)、HLA-B5301(1A1N)、HLA-B4001(6IEX)、HLA-B5801(5V5L)、HLA-B0801(3X13)、H2Kb(6JQ3)、H2-Ld (6L9M)。与表位相对接后,结果提供前10种可能的对接形式及相应的对接分数。将对接分数数据标准化后,绝对值高于总体中位数者被认为是可结合的,称为可结合核心数;其中,对接分数绝对值最高者称为最优核心结合力。将上述2种数据制成气泡图。
1.7.2 HTNV Gc 15肽与MHC-Ⅱ类分子模拟对接使用EpiDOCK(www.ddg-pharmfac.net/epidock/EpiDockPage.html)[22]工具模拟15肽与MHC-Ⅱ类分子对接。输入15肽优势表位序列后选择对应的HLA-Ⅱ类亚型进行对接模拟。每条15肽被认为均由7个9肽核心与对应的MHC-Ⅱ类分子进行对接,随后得出每种情况与HLA-Ⅱ类分子的结合得分及对应的对接阈值。结合分数高于或等于给定阈值的情况被认为是可结合,否则不是。将结合分数数据标准化后,高于总体中位数者被认为是可结合的,称为可结合核心数;其中,结合力最强者称为最优核心结合力。将上述2种数据制成气泡图。
1.8 DNA疫苗和灭活疫苗二价HFRS灭活疫苗(HANPUWEIⓇ)由中国长春生物制品研究所有限公司生产。该疫苗由仓鼠肾细胞中灭活和纯化的HTNV和SEOV制成(在中国没有单价HFRS疫苗)。质粒pVAX-Gc疫苗为本课题组先前研究所合成[5]。
1.9 动物和免疫无特定病原体的近交系8周龄雌性BALB/c小鼠购自空军军医大学实验动物中心。BALB/c小鼠位于MHC单倍型H2-d内,分成2组,每组10只小鼠。第1组小鼠接种50 μL 1 ∶5稀释的二价灭活疫苗(参照生产厂家说明书确定免疫剂量),第2组小鼠注射50 μL质粒pVAX-Gc疫苗,疫苗每3周注射1次,共注射2次。第1次免疫后5周,处死免疫小鼠,进行ELISpot实验,评估细胞免疫反应性。所有动物饲养在无病原体的动物实验室中,饲养条件安静。用标准饲料和过滤水喂养动物,每周更换垫料,确保10 h以上的连续夜间时间以维持昼夜节律。所有实验操作佩戴无菌手套。通过吸入二氧化碳处死动物。由空军军医大学动物中心伦理委员会批准动物护理和使用的整个程序,遵守所有适用的关于动物伦理使用的机构和政府法规。
1.10 Elispot实验验证由于其生产中固有的困难,对HTNV Gc上2个9肽核心与所预测出的H-2d限制性15肽相同的表位以及2条非预测优势表位和2个非H-2d限制性表位进行合成(ChinaPeptides,上海),用20 μg/mL的PBS稀释单个肽用于INF-γ ELISpot分析,在4 ℃条件下放置在包被有IFN-γ特异性捕获抗体的ELISpot板上过夜。处死小鼠并解剖脾脏,然后研磨至单细胞悬液。红细胞裂解后,冲洗脾细胞并重新悬浮。在室温下用含有10% 胎牛血清RPMI1640培养基封闭ELISpot板2 h后,将1×106个脾细胞添加到每个孔中,并用最终浓度为5 μg/mL的合成Gc肽刺激脾细胞。将平板在37 ℃、5% CO2培养箱中培养24 h。阴性对照为完整的培养基。阳性对照为10 μg/mL的刀豆蛋白A(Con A)。孵育后,将培养板用H2O和PBST洗涤,洗涤后在孔中加入100 μL终浓度为2 μg/mL生物素化抗IFN-γ抗体并在室温孵育2 h。用PBST(0.05% Tween-20的PBS)洗涤后加入链霉亲和素-辣根过氧化物酶(streptavidin-HRP)室温孵育1 h。用PBST洗涤后加入AEC(DAKEWEI,China)底物显色并用水洗涤终止反应。待96孔板风干后使用CTL ELISpot读取器(CTL,US)对板中斑点进行计数。所有结果显示为每106个脾细胞的斑点形成单位(SFU)平均值。条形图采用GraphPad Prism 9绘制。
2 结果 2.1 HTNV Gc上鼠H-2和人HLA-Ⅰ、Ⅱ类限制性优势表位的亲和力分析表 1列举出了预测工具对小鼠H-2类亚型及人HLA-Ⅰ、Ⅱ类主要亚型等位基因。重新计算排除重复9肽和15肽后,分析产生的优势表位的数目。在MHC-Ⅰ类亚型中得到103个优势表位,MHC-Ⅱ类亚型中得到114个优势表位。在H-2类亚型中,H2-Kd优势表位数目最多,为7个9肽表位。在HLA-Ⅰ类亚型中,HLA-B15优势表位数目最多,为24个9肽表位。在HLA-Ⅱ类亚型中,DRB1优势表位数目最多,为41个15肽表位。
2.2 HTNV Gc上优势表位免疫原性分析在同等条件下免疫原性强的表位更有可能充分诱导免疫应答。表 1列出了小鼠和人对Gc的强免疫原性表位数目。在H-2亚型中获得了10个强免疫原性优势表位,在MHC-Ⅰ类亚型中获得了74个强免疫原性优势表位,在MHC-Ⅱ类亚型中获得了27个强免疫原性优势表位。
2.3 HTNV Gc MHC分子间的共亲和区段分析图 1柱状图显示,对于HTNV Gc而言,HLA-A24、HLA-B15、HLA-B44、HLA-B46、HLA-B58等基因超家族与其他MHC基因超家族有较为普遍的交叉反应区段,提示不同人群之间可能存在跨群体的交叉反应性;HLA-B46与HLA-B15存在最多交叉反应区段,HLA-B46为HLA-B15进化而来的等位基因(https://en.wikipedia.org/wiki/HLA-B46),提示具有较多相同高亲和区段的MHC分子可能在进化上存在一定联系。
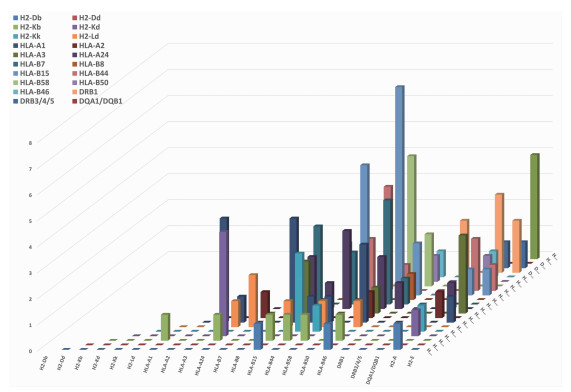
|
| 图 1 MHC分子间共亲和区段分析结果 |
2.4 HTNV Gc上优势表位的保守性分析
使用BLASTP工具预测上述强免疫原性的HTNV GC 9肽和15肽优势表位的种间种内保守性,获得高亲和力、强免疫原性和种间种内保守的MHC-Ⅰ/Ⅱ类限制性优势表位,结果如表 2。其中上述MHC-Ⅰ、Ⅱ类限制性表位中存在7条交叉表位;MHC-Ⅱ类限制性表位均为种间种内保守;交叉表位在MHC-Ⅰ、Ⅱ类分子中均为强免疫原性表位。
| MHC家族 | MHC基因型 | Gc优势表位数 | 强免疫原性表位数 | 种间种内保守表位数 |
| MHC-Ⅰ | H2-Db | 5 | 2 | 0 |
| H2-Dd | 5 | 3 | 0 | |
| H2-Kb | 3 | 0 | 0 | |
| H2-Kd | 7 | 2 | 0 | |
| H2-Kk | 4 | 2 | 0 | |
| H2-Ld | 3 | 1 | 0 | |
| HLA-A1 | 8 | 4 | 2 | |
| HLA-A2 | 13 | 6 | 2 | |
| HLA-A3 | 19 | 12 | 3 | |
| HLA-A24 | 13 | 6 | 4 | |
| HLA-B7 | 12 | 5 | 3 | |
| HLA-B8 | 2 | 1 | 0 | |
| HLA-B15 | 24 | 11 | 2 | |
| HLA-B40 | 7 | 5 | 2 | |
| HLA-B44 | 12 | 7 | 2 | |
| HLA-B46 | 10 | 4 | 1 | |
| HLA-B50 | 10 | 6 | 4 | |
| HLA-B58 | 12 | 7 | 1 | |
| DRB1 | 41 | 22 | 22 | |
| DQA1/DQB1 | 1 | 1 | 1 | |
| MHC-Ⅱ | DRB3/4/5 | 7 | 5 | 5 |
| H2-A | 48 | 27 | 27 | |
| H2-E | 40 | 25 | 25 |
2.5 双向层次聚类分析显示HTNV Gc表位与MHC分子的相互作用关系 2.5.1 HTNV Gc 9肽与MHC-Ⅰ类分子的相互作用关系
双向聚类分析结果如图 2所示,显示不同MHC-Ⅰ类分子在提呈HTNV Gc 9肽的差异与关系。其中,57个MHC-Ⅰ类分子被分为3组,包括HLA-Ⅰ亚型组以及交叉反应组(HLA主要)、交叉反应组(H2主要)。
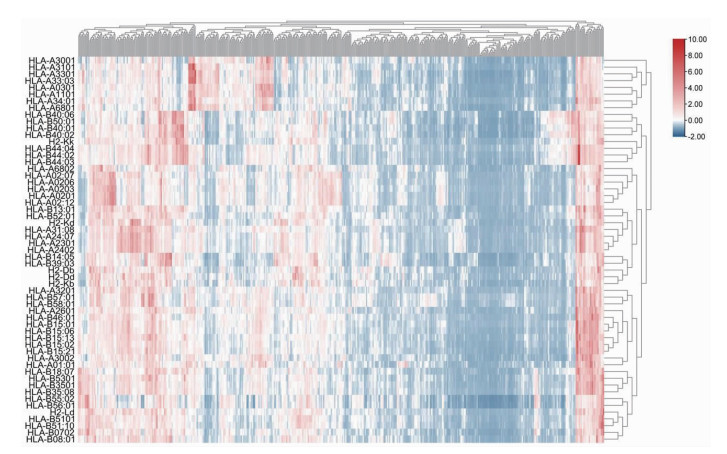
|
| 图 2 HTNV Gc MHC-Ⅰ类双向层次聚类分析 |
在HLA-Ⅰ组中,HLA-B4006与HLA-B5001、HLA-A6801与HLA-A3401分别归属于同一类别,2个基因型得分相似;HLA-A3201与HLA-B58(HLA-B5701、HLA-B5801)得分更为相似,提示HLA-A3201和HLA-B58基因型在提呈HTNV Gc抗原过程中展现出相似特征;HLA-B15(-B1501,-B1502,-B1506,-B1513)与HLA-A1(-A3002,-A0101)两类基因型得分相似,提示其具有相似的特征。对于交叉反应组(H2主要),H2-Db、H2-Dd和H2-Kb得分与HLA-B1405、HLA-B3903相似。在交叉反应组(HLA主要)中,H2-Kk得分与HLA-B44(-B4402、-B4403、-B4404)相似,H2-Kd与HLA-A3108、HLA-A2407、HLA-A2301得分相似。
2.5.2 HTNV Gc 15肽与MHC-Ⅱ类分子的相互作用关系图 3显示了不同MHC-Ⅱ亚型间的差异。51个MHC-Ⅱ类分子被分为3组,包括HLA-Ⅱ亚型组以及交叉反应组(HLA主要)、交叉反应组(H2主要)。在HLA-Ⅱ亚型组中,HLA-DQA1*0101-DQB1*0501、HLA-DQA1*0501-DQB1*0201、HLA-DQA1*0301-DQB1*0302、HLA-DQA1*0401-DQB1*0402提呈抗原的亲和力相近;HLA-DPA1*0103(-DPB1*0101,-DPB1*0402,-DPB1*0201,-DPB1*0401)和DRB1类的基因型提呈抗原能力相近;HLA-DPA1*0103-DPB1*0501与DRB1类的基因型得分相近。对于交叉反应组(H2主要),H2-IAd得分与HLA-DPA1*0103-DPB1*1401相似,体现出在人种和鼠种间跨物种提呈HTNV Gc的相似性; DRB3*0202与H2-IAu、H2-IAb、H2-IAk具有相似的抗原呈递能力。在交叉反应组(HLA主要)中,H2-IEK得分与DRB1*1101相近。
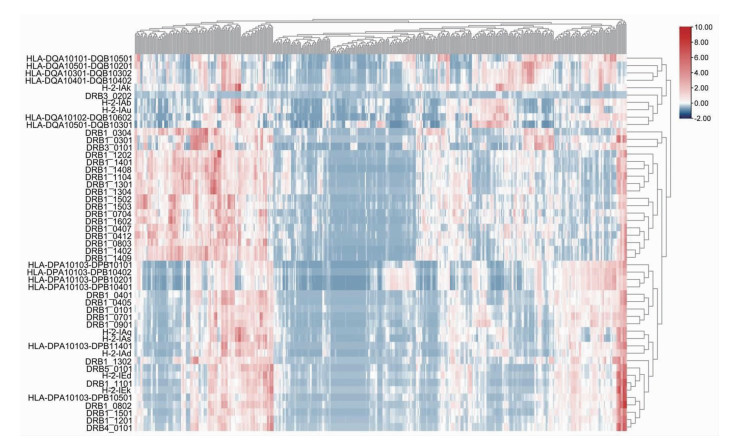
|
| 图 3 HTNV Gc MHC-Ⅱ类聚类分析 |
2.6 MHC分子与优势表位的模拟分子对接 2.6.1 HTNV Gc 9肽表位与MHC-Ⅰ类分子对接
使用HPEPDOCK工具对具有高亲和力、强免疫原性和种间种内均保守的8条9肽优势表位与MHC-Ⅰ类分子模拟分子对接,如图 4所示为每个肽与MHC对接后的可结合核心数及最优核心结合力的气泡图,表位IEWKDPDGM、FTDERIEWK与各个基因型的最优核心结合力普遍偏高;表位GWFVKSGEW与各个基因型的最优核心结合力较低,其中H2-Ld、HLA-B0801、HLA-B5301的最优核心结合力较低;表位HFTDERIEW与HLA-B5801最优核心结合力较低。其中,对接得分越低,表明肽-MHC对接越好。另一方面,H2-Ld、HLA-A0206与优势表位对接时可结合核心数多;H2-Ld的对接得分普遍低于HLA-B5301,提示这些表位在与人类HLA发生结合时可能更加稳定。
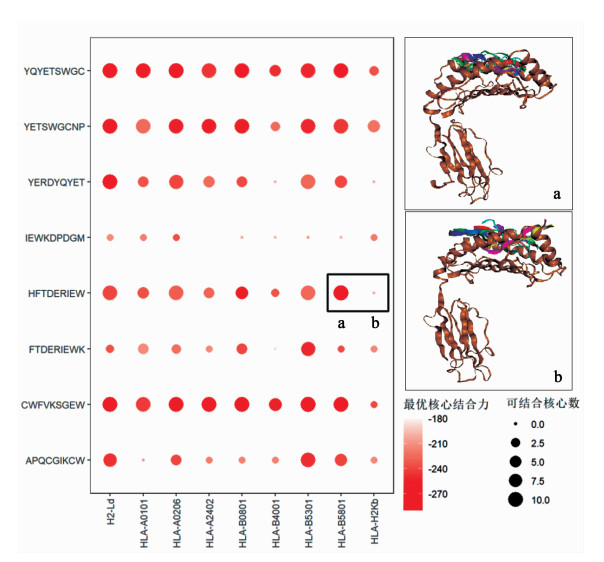
|
| 图 4 HTNV Gc 9肽优势表位与MHC-Ⅰ类分子对接分数气泡图 |
在气泡图中选取表位HFTDERIEW与HLA-B5801为对接得分较高的典例,选取表位HFTDERIEW与H2-Kb为对接得分较低的典例,通过相应的对接模拟结构可以看出表位HFTDERIEW与HLA-B5801的10种对接模拟结构中,9肽均位于MHC分子抗原结合槽内;而表位HFTDERIEW与H2-Kb的10种对接模拟结构中,多数9肽位于MHC分子抗原结合槽外。MHC与表位对接结构进一步解释了对接时可能的状态。
2.6.2 HTNV Gc 15肽优势表位与MHC-Ⅱ类分子模拟对接使用EpiDOCK对具有高亲和力、强免疫原性和种间种内保守的8条人鼠共优势表位与MHC-Ⅱ类分子结合亲和力进行评估。利用气泡图反映8个15肽对应的最优结合核心数和对接分数。结果如图 5所示,表位SVPMHTDLELDFSLT与各个基因型的最优核心结合力普遍偏低;表位FHCYGACTKYEYPWH与各个基因型的最优核心结合力较高,其中DRB10404、DRB10405、DRB10701、DRB10802的最优核心结合力较好;表位FHCYGACTKYEYPWH与DRB10701最优核心结合力较好。另一方面,DQA1*0102/DQB1*0602、DQA1*0301/DQB1*0302可结合核心数多;YKITIRYSRRVCVQ和SVPMHTDLELDFSLT等表位的对接得分较低。
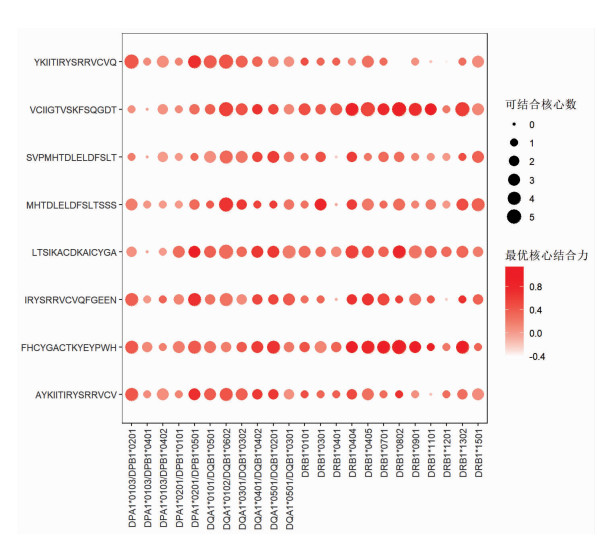
|
| 图 5 HTNV Gc 15肽优势表位与MHC-Ⅰ类分子对接分数气泡图 |
2.7 ELISpot实验评估15肽优势表位细胞免疫反应性
对HTNV Gc上2个9肽核心与所预测出的H-2d限制性15肽相同的表位以及2条非H-2d限制性优势表位进行合成,用于ELISpot实验验证。用上述肽分别刺激小鼠脾细胞,检测IFN-γ的分泌水平。如图 6所示,结果为每106个脾细胞所分泌的IFN-γ斑点形成单位(SFU)平均值。从图中可以看出灭活疫苗组和pVAX-Gc组中,2条H-2d限制性优势表位均可引起脾细胞分泌IFN-γ,且SFU平均值高于对照组,证明优势表位的筛选用于促进细胞免疫应答确实行之有效。同时,表位IITIRYSRRVCVQFG在pVAX-Gc组小鼠中的细胞免疫应答较灭活疫苗组强,提示上述表位在HTNV Gc上具有更加显著的优势。GVGSVPMHTDLELDF和HTDLELDFSLTSSSK为非预测优势表位, 2组实验中的SFU平均值均未明显高于对照组。
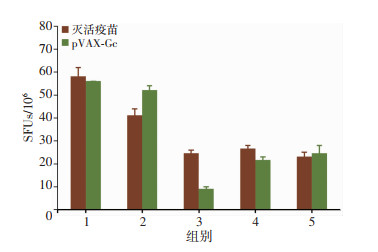
|
| 1:PVGSAYKIITIRYSR;2:IITIRYSRRVCVQFG;3:GVGSVP-MHTDLELDF;4:HTDLELDFSLTSSSK;5:空白对照组 图 6 HTNV Gc合成肽的细胞免疫反应性实验结果 |
3 讨论
本研究以比较免疫学和结构免疫学为基础,利用分子生物信息学的原理,采用多种预测工具探究HTNV Gc上高亲和力、强免疫原性和种间种内均保守的9肽、15肽优势表位,预测表位覆盖率超过全球98%人群。通过亲和力预测,在MHC-Ⅰ类亚型中得到103个优势表位,MHC-Ⅱ类亚型中得到114个优势表位;继而根据免疫原性分析,获得了10个强免疫原性的H-2限制性优势表位,在HLA-Ⅰ类和Ⅱ类亚型中分别获得了74和27个强免疫原性优势表位,并对其交叉免疫反应性进行了分析;使用BLASTP工具预测上述强免疫原性的HTNV Gc 9肽和15肽优势表位的种间种内保守性,获得高亲和力、种间种内保守、强免疫原性的MHC-Ⅰ类和Ⅱ类限制性优势表位分别为8和80个,其中MHC-Ⅱ类人鼠共同优势表位8个。基于Gc肽和MHC之间的相互作用进行了双向分层聚类分析,揭示了HTNV Gc上的9肽和15肽表位跨地区、跨种群、跨物种之间的相似性。通过ELISpot实验鉴定,在非优势表位对比下实验结果表明,优势表位可刺激鼠脾细胞分泌更多的IFN-Y,提示优势表位有较好的免疫反性与免疫原性,与预测结果相符。使用上述方法进行精准预测出HTNV Gc上能够引起细胞免疫的优势表位,在短时间内找到更多具有潜在疫苗作用靶点的抗原表位,极大地节省实验成本。
CD4+T细胞是适应性免疫中指导和增强固有淋巴细胞、B淋巴细胞和CD8+T细胞的功能,以抵御不同的病原体的关键细胞[23]。在HTNV免疫中,MHC-Ⅱ处理抗原后有效地启动CD4+T细胞,而CD4+T细胞对细胞、体液和长期免疫反应至关重要[24]。目前的研究表明,疫苗诱导的中和抗体水平与临床保护作用密切相关[25]。高滴度的中和抗体证明疫苗的高有效性,故MHC-Ⅱ类限制性优势表位对未来疫苗研制具有重大意义。在本研究中我们发现9肽优势表位与15肽优势表位在部分HTNV Gc蛋白位点上高度重合。在8个MHC-Ⅰ类限制性优势表位中有6个表位与MHC-Ⅱ类限制性优势表位的位点重合。这提示,这些区段的表位可以同时激活CD8+T细胞和CD4+T细胞,针对这些区段表位的疫苗可能产生更加全面的免疫保护。
ELISPOT实验已被广泛用于检测免疫细胞在抗原刺激后细胞因子的分泌。在各种类型的ELISPOT中,基于IFN-γ的检测是该技术最常见的应用,经常用于跟踪特定的CD8+T细胞反应[26]。IFN-γ在多种疾病的免疫应答中起重要免疫防御作用,如艾滋病、疟疾、结核病等[27-28],同时也为评价SARS-CoV-2等病原体疫苗保护效力的主要细胞因子[29-30]。本研究通过ELISPOT实验检测了优势表位的特异性细胞免疫反应性,验证了免疫反应性与免疫原性分析结果。但表位真实的疾病保护效力仍需要进一步的血清学实验验证。
用于表位预测的生物信息学方法可用于协助后续的免疫学实验和基于表位的疫苗设计[31-32],但此过程中仍存在一定的局限性。亲和力预测过程中预测肽在蛋白抗原中的位置与亲和力强弱的关系仍需研究,优势肽表位对于人体是否存在毒性反应和过敏反应等预测仍需补充。本研究提供了一种筛选优势表位的先进方法,并且可服务于当下在全球大流行的SARS-CoV-2和猴痘病毒等的防治研究。
| [1] |
WANG Q W, YUE M, YAO P P, et al. Epidemic trend and molecular evolution of HV family in the main hantavirus epidemic areas from 2004 to 2016, in P.R. China[J]. Front Cell Infect Microbiol, 2021, 19: 584814. DOI:10.3389/fcimb.2020.584814 |
| [2] |
The problem with neoantigen prediction[J]. Nat Biotechnol, 2017, 35(2): 97. DOI: 10.1038/nbt.3800.
|
| [3] |
MUYANGWA M, MARTYNOVA E V, KHAIBOULLINA S F, et al. Hantaviral proteins: structure, functions, and role in hantavirus infection[J]. Front Microbiol, 2015, 6: 1326. DOI:10.3389/fmicb.2015.01326 |
| [4] |
丁国永, 王志玉, 高璐, 等. 埃博拉病毒包膜糖蛋白研究进展[J]. 病毒学报, 2013, 29(2): 233-237. DING G Y, WANG Z Y, GAO L, et al. Research progress on Ebola virus glycoprotein[J]. Chin J Virol, 2013, 29(2): 233-237. |
| [5] |
刘硕, 张黎, 王玉琳, 等. 埃博拉病毒包膜糖蛋白变异及抗原表位研究进展[J]. 病毒学报, 2019, 35(5): 801-812. LIU S, ZHANG L, WANG Y L, et al. Research progress in the envelope glycoprotein variation and epitope of Ebola virus[J]. China Ind Econ, 2019, 35(5): 801-812. DOI:10.13242/j.cnki.bingduxuebao.003581 |
| [6] |
JIANG D B, ZHANG J P, CHENG L F, et al. Hantavirus Gc induces long-term immune protection via LAMP-targeting DNA vaccine strategy[J]. Antivir Res, 2018, 150: 174-182. DOI:10.1016/j.antiviral.2017.12.011 |
| [7] |
MA R X, CHENG L F, YING Q K, et al. Screening and identification of an H-2Kb-restricted CTL epitope within the glycoprotein of Hantaan virus[J]. Front Cell Infect Microbiol, 2016, 6: 151. DOI:10.3389/fcimb.2016.00151 |
| [8] |
TANG K, CHENG L F, ZHANG C M, et al. Novel identified HLA-A*0201-restricted Hantaan virus glycoprotein cytotoxic T-cell epitopes could effectively induce protective responses in HLA-A2.1/K(b) transgenic mice may associate with the severity of hemorrhagic fever with renal syndrome[J]. Front Immunol, 2017, 8: 1797. |
| [9] |
GREENBAUM J, SIDNEY J, CHUNG J, et al. Functional classification of class Ⅱ human leukocyte antigen (HLA) molecules reveals seven different supertypes and a surprising degree of repertoire sharing across supertypes[J]. Immunogenetics, 2011, 63(6): 325-335. DOI:10.1007/s00251-011-0513-0 |
| [10] |
SERRIS A, STASS R, BIGNON E A, et al. The Hantavirus surface glycoprotein lattice and its fusion control mechanism[J]. Cell, 2020, 183(2): 442-456. e16. DOI:10.1016/j.cell.2020.08.023 |
| [11] |
VITA R, MAHAJAN S, OVERTON J A, et al. The immune epitope database (IEDB): 2018 update[J]. Nucleic Acids Res, 2019, 47(D1): D339-D343. DOI:10.1093/nar/gky1006 |
| [12] |
KIM Y, SIDNEY J, PINILLA C, et al. Derivation of an amino acid similarity matrix for peptide: MHC binding and its application as a Bayesian prior[J]. BMC Bioinformatics, 2009, 10: 394. DOI:10.1186/1471-2105-10-394 |
| [13] |
REYNISSON B, ALVAREZ B, PAUL S, et al. NetMHCpan-4.1 and NetMHCⅡpan-4.0: improved predictions of MHC antigen presentation by concurrent motif deconvolution and integration of MS MHC eluted ligand data[J]. Nucleic Acids Res, 2020, 48(W1): W449-W454. DOI:10.1093/nar/gkaa379 |
| [14] |
RAMMENSEE H G, BACHMANN J, EMMERICH N P N, et al. SYFPEITHI: database for MHC ligands and peptide motifs[J]. Immunogenetics, 1999, 50(3): 213-219. DOI:10.1007/s002510050595 |
| [15] |
RECHE P A, GLUTTING J P, ZHANG H, et al. Enhancement to the RANKPEP resource for the prediction of peptide binding to MHC molecules using profiles[J]. Immunogenetics, 2004, 56(6): 405-419. DOI:10.1007/s00251-004-0709-7 |
| [16] |
JENSEN K K, ANDREATTA M, MARCATILI P, et al. Improved methods for predicting peptide binding affinity to MHC class Ⅱ molecules[J]. Immunology, 2018, 154(3): 394-406. DOI:10.1111/imm.12889 |
| [17] |
GONZALEZ-GALARZA F F, MCCABE A, SANTOS E J M D, et al. Allele frequency net database (AFND) 2020 update: gold-standard data classification, open access genotype data and new query tools[J]. Nucleic Acids Res, 2020, 48(D1): D783-D788. DOI:10.1093/nar/gkz1029 |
| [18] |
DOYTCHINOVA I A, FLOWER D R. VaxiJen: a server for prediction of protective antigens, tumour antigens and subunit vaccines[J]. BMC Bioinform, 2007, 8(1): 1-7. DOI:10.1186/1471-2105-8-4 |
| [19] |
ZHANG J, MADDEN T. PowerBLAST: a new network BLAST application for interactive or automated sequence analysis and annotation[J]. Genome Res, 1997, 7(6): 649-656. DOI:10.1101/gr.7.6.649 |
| [20] |
CHEN C J, CHEN H, ZHANG Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Mol Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [21] |
ZHOU P, JIN B W, LI H, et al. HPEPDOCK: a web server for blind peptide-protein docking based on a hierarchical algorithm[J]. Nucleic Acids Res, 2018, 46(W1): W443-W450. DOI:10.1093/nar/gky357 |
| [22] |
ATANASOVA M, PATRONOV A, DIMITROV I, et al. EpiDOCK: a molecular docking-based tool for MHC class Ⅱ binding prediction[J]. Protein Eng Des Sel, 2013, 26(10): 631-634. DOI:10.1093/protein/gzt018 |
| [23] |
NGUYEN Q P, DENG T Z, WITHERDEN D A, et al. Origins of CD4+ circulating and tissue-resident memory T-cells[J]. Immunology, 2019, 157(1): 3-12. DOI:10.1111/imm.13059 |
| [24] |
KAECH S M, WHERRY E J, AHMED R. Effector and memory T-cell differentiation: implications for vaccine development[J]. Nat Rev Immunol, 2002, 2(4): 251-262. DOI:10.1038/nri778 |
| [25] |
LIU J Y, MAO Q Y, WU X, et al. Considerations for the feasibility of neutralizing antibodies as a surrogate endpoint for COVID-19 vaccines[J]. Front Immunol, 2022, 13: 814365. |
| [26] |
SLOTA M, LIM J B, DANG Y S, et al. ELISpot for measuring human immune responses to vaccines[J]. Expert Rev Vaccines, 2011, 10(3): 299-306. DOI:10.1586/erv.10.169 |
| [27] |
HILL P C, BROOKES R H, FOX A, et al. Longitudinal assessment of an ELISPOT test for Mycobacterium tuberculosis infection[J]. PLoS Med, 2007, 4(6): e192. DOI:10.1371/journal.pmed.0040192 |
| [28] |
KESTER K E, CUMMINGS J F, OCKENHOUSE C F, et al. Phase 2a trial of 0, 1, and 3 month and 0, 7, and 28 day immunization schedules of malaria vaccine RTS, S/AS02 in malaria-naive adults at the Walter Reed Army Institute of Research[J]. Vaccine, 2008, 26(18): 2191-2202. DOI:10.1016/j.vaccine.2008.02.048 |
| [29] |
ZHU F C, LI Y H, GUAN X H, et al. Safety, tolerability, and immunogenicity of a recombinant adenovirus type-5 vectored COVID-19 vaccine: a dose-escalation, open-label, non-randomised, first-in-human trial[J]. Lancet, 2020, 395(10240): 1845-1854. DOI:10.1016/S0140-6736(20)31208-3 |
| [30] |
FOLEGATTI P M, EWER K J, ALEY P K, et al. Safety and immunogenicity of the ChAdOx1 nCoV-19 vaccine against SARS-CoV-2: a preliminary report of a phase 1/2, single-blind, randomised controlled trial[J]. Lancet, 2020, 396(10249): 467-478. DOI:10.1016/S0140-6736(20)31604-4 |
| [31] |
MICHEL-TODÓ L, BIGEY P, RECHE P A, et al. Design of an epitope-based vaccine ensemble for animal trypanosomiasis by computational methods[J]. Vaccines, 2020, 8(1): 130. DOI:10.3390/vaccines8010130 |
| [32] |
RUSSO G, RECHE P, PENNISI M, et al. The combination of artificial intelligence and systems biology for intelligent vaccine design[J]. Expert Opin Drug Discov, 2020, 15(11): 1267-1281. DOI:10.1080/17460441.2020.1791076 |




