骨肉瘤是儿童和青少年最常见的骨原发恶性肿瘤,约15%的患者在就诊时就出现了转移或微转移。随着手术和化疗联合治疗方式的引入,其5年生存率从17%提高至60%~70%[1-2]。近40年来,骨肉瘤的治疗陷入了瓶颈,远处转移仍然是骨肉瘤患者预后不理想的主要原因之一[3]。研究表明,高表达黏附分子的活化血管内皮细胞可以通过与肿瘤细胞结合抑制其转移; 而在某些具有高转移特性的肿瘤中,肿瘤血管内皮可以分泌一种叫Biglycan的富含亮氨酸的多聚糖蛋白来刺激肿瘤转移[4-5]。在骨肉瘤的生长及转移过程中,血管内皮细胞也是重要的微环境成分之一,然而其与骨肉瘤之间的相互作用关系及机制尚未完全阐明。因此,深入挖掘内皮细胞-骨肉瘤相互作用中的关键分子并探讨其作用及分子机制至关重要。
富亮氨酸胶质瘤失活蛋白(leucine-rich glioma inactivated proteins,LGI)属于大的富亮氨酸重复蛋白家族,家族包含4个高度保守的成员(LGI1~4),在脊椎动物神经系统的发育和功能中起重要作用,例如突触传递和髓鞘形成等[6-7]。LGI作为一种分泌型糖蛋白,由富含亮氨酸的重复(LRR)结构域和癫痫相关表位蛋白(epitempin,EPTP)结构域组成[8]。LGI1是LGI家族的首个被发现的成员,在恶性胶质瘤中表达缺失,随后被证实是一个典型的肿瘤抑制基因[9-10]。LGI2与犬家族性少年良性癫痫相关[11], LGI3在脂肪组织、大脑和皮肤中表达,是一种多功能细胞因子和促炎脂肪因子[12]。LGI4与一系列神经系统疾病包括癫痫和边缘性脑炎有关[13]。LGI4还参与周围神经髓鞘形成的旁分泌信号通路,即由施万细胞合成和分泌的LGI4与轴突分离整合素和金属蛋白酶22(adisintegrin and metalloproteinase 22, Adam22)结合,从而驱动施万细胞的分化和髓鞘形成[14]。在整个周围神经系统发育过程中,LGI4/Adam22信号通路还可调节胶质形成,促进神经胶质祖细胞的增殖,促进胶质细胞和卫星细胞在DRGs和交感神经节的生成和分化[15]。LGI1和LGI3均被报道与肿瘤生长转移相关,如胶质瘤、肝癌等。但关于LGI4在肿瘤中的作用研究较少,有研究表明,在浸润性乳腺癌中Adam23阴性细胞可产生LGI4和一氧化氮,促进邻近Adam23阳性细胞的增殖和侵袭,从而促进肿瘤生长和转移[16]。
本课题组前期通过转录组测序筛选出一个骨肉瘤表达差异基因-LGI4。鉴于LGI4在骨肉瘤发生发展中的作用尚不清楚,本研究围绕LGI4在骨肉瘤中的功能展开研究,以期为骨肉瘤的发生发展机制提供新的资料。
1 材料与方法 1.1 实验材料人骨肉瘤细胞系143B和人脐静脉内皮细胞(human umbilical vein endothelial cells, HUVECs)购自广州赛库生物公司,MTF细胞系由本课题组用人永生化成骨细胞系Hfob1.19恶性转化并保存。高糖DMEM培养基、胰酶、PBS缓冲液均购自Gibco公司,胎牛血清购自ZETA LIFE公司,青-链霉素购自Biosharp公司。6孔板和0.4 μm以及8 μm的Transwell小室、Matrigel基质胶购自美国康宁公司,蛋白裂解液、蛋白印迹配胶试剂盒购自上海碧云天公司,PVDF膜购自瑞士罗氏公司,ECL显影液购自美国Pico公司,anti-GAPDH、anti-NOTCH3和anti-NOTCH4均购自美国CST,anti-LGI4购自Proteintech公司,DAPT购自Selleck公司,LGI4质粒由上海生工合成,siRNA购自美国赛默飞,质粒小量抽提试剂盒购自上海碧云天公司。
1.2 实验方法 1.2.1 细胞培养将骨肉瘤细胞系143B、MTF和HUVECs培养于含10% FBS及1%双抗的DMEM培养基中,置于5%CO2、37 ℃培养箱恒温培养,143B每天更换培养基,MTF和HUVECs隔天更换。
1.2.2 质粒提取、细胞转染及构建稳定LGI4表达细胞系按碧云天试剂盒操作提取质粒,得到浓度为160 ng/μL的LGI4质粒。将生长状态良好的骨肉瘤细胞系143B及MTF分别按每孔2×105和3×105个接种于6孔板中。细胞汇合度达50%~60%后,按Opti-MEM培养基200 μL+质粒2 μg配置体系,静置5 min;随后在体系内加入脂质体LipofectamineTM 2000 6 μL,静置15 min,将上述转染复合物加入6孔板中置于培养箱培养。12 h后用含1 μg/mL嘌呤霉素的培养基筛选并构建稳定细胞系,获得骨肉瘤细胞系143B- LGI4及MTF-LGI4。将143B-LGI4细胞按2.5×105个/孔接种于6孔板中,第2天更换无血清DMEM培养基,按20 nmol/L添加siRNA,并加入ribobio-FECTTMCP协助转染,48 h后收集样本用于后续实验。干扰序列如下:5′-GATTGAGGACGATGCATTTGC-3′。
1.2.3 共培养体系的构建将HUVECs每孔1.5×105个细胞接种于Transwell上室,143B按分组每孔2.5×105个细胞,MTF每孔3×105个细胞接种于下室,12 h后将HUVECs上室移入下室共培养,共培养48 h后收集骨肉瘤细胞系143B加入适量Trizol裂解后进行全转录组测序,并于0、24、48 h拍照记录骨肉瘤细胞系形态变化。将构建的共培养体系和稳定LGI4表达细胞系分为5组,①阴性对照组:正常培养的骨肉瘤细胞,②共培养组:HUVECs与骨肉瘤细胞共培养细胞,③共培养基组:采用HUVECs培养基与骨肉瘤细胞共培养,④载体对照组:采用过表达对照载体转染细胞,⑤LGI4组:采用过表达LGI4载体转染骨肉瘤细胞。在验证LGI4下游信号通路实验中,加入DAPT抑制骨肉瘤细胞株的NOTCH信号,分为3组:siRNA对照组、siRNA干扰组及加入DAPT的siRNA抑制剂组。
1.2.4 骨肉瘤细胞系LGI4基因及蛋白表达的检测Trizol法提取各个组的RNA,使用试剂盒反转录后进行RT-qPCR检测LGI4表达;收集各组细胞分别加入RIPA裂解液提取总蛋白,用BCA法测定蛋白浓度。用提取的蛋白行Western blot, 先恒压90 V,30 min,之后120 V,1 h进行SDS-PAGE凝胶电泳,恒流150 mA 2 h转膜,脱脂牛奶封闭1 h,加入按抗体说明书配制的一抗(anti-GAPDH、anti-LGI4、anti-NOTCH3和anti-NOTCH4)在摇床4℃孵育过夜。次日用PBST洗膜5次,每次5 min,然后放入1 ∶5 000稀释的二抗中室温孵育1.5 h,再次用PBST洗膜后进行显影。使用Image J分析蛋白条带。
1.2.5 Transwell迁移及侵袭实验共培养48 h后,消化收集各组细胞,用无血清培养基重悬计数,迁移实验143B每孔2×104个,MTF每孔4×104个;侵袭实验预先在上室中铺Matrigel基质胶(1 ∶8稀释),待凝固后吸出残留液体,按143B每孔4×104个,MTF每孔6×104个接种到上室,下室加入10% FBS DMEM培养基600 μL。分别于24、48 h后,取出143B和MTF小室,弃其液体,PBS洗3次后用4%多聚甲醛固定30 min,置于结晶紫中染色10 min,用棉签擦掉小室内侧细胞,再次PBS洗3次,拍照。
1.2.6 划痕实验用记号笔在6孔板背后每隔1cm画一道横线,横穿过孔。各组细胞按每孔8×106个接种到6孔板,第2天汇合度达90%,弃旧培养基,使用10 μL枪头垂直于六孔板底部,每孔作2条划痕,再用PBS洗涤2次,加入无血清培养基后,置入培养箱培养,于0、24、48 h后拍照观察。
1.2.7 裸鼠转移模型12只5周龄雌性裸鼠饲养于新桥医院中心实验室动物房。实验分为2组,分别收集对数生长期的143B骨肉瘤细胞(对照组)以及稳定转染的143B-LGI4骨肉瘤细胞(模型组),裸鼠尾静脉注射。每5天观察裸鼠生长状态,于第30天处死裸鼠,收集各脏器并拍照后,加入10%福尔马林固定,制成组织蜡块,HE染色观察肿瘤形态及脏器转移情况。实验中所用裸鼠来自于北京维通利华公司,并经陆军军医大学实验动物福利伦理审查委员会批准(批件号:AMUWEC2019178)。
1.3 统计学分析采用SPSS 22.0和GraphPad 8.3进行数据分析,计量资料以x±s表示,两组比较采用t检验,多组间比较采用单因素方差分析,检验水准α=0.05。
2 结果 2.1 骨肉瘤细胞系143B和MTF与HUVECs共培养48 h后LGI4表达降低共培养模型显示:共培养组较阴性对照组细胞密度显著增加(图 1A);通过全转录组测序筛选出在骨肉瘤细胞系143B与HUVECs共培养后表达差异倍数前十位的基因分别为:CCL20、SNORA14B、PTPRN、KDF1、NMRAL2P、NEURL1B、LGI4、AKR1C1、ABCC2、AL161431.1(表 1、图 1B)。为了进一步验证测序结果,将人骨肉瘤细胞系143B和MTF与HUVECs及其培养基共培养48 h,RT-qPCR和Western blot检测结果显示,与HUVECs共培养后,骨肉瘤细胞中的LGI4表达均明显下调(P < 0.05,图 1C~E)。
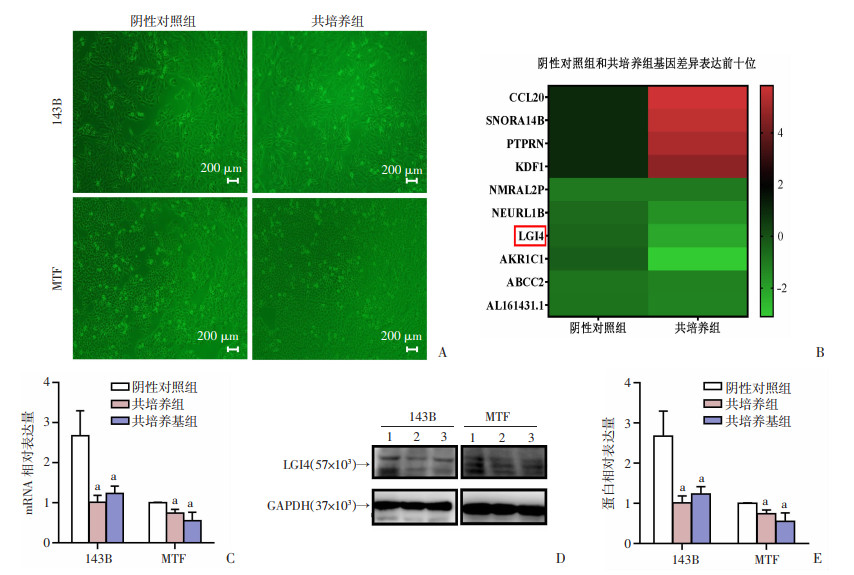
|
| A:阴性对照组和共培养组143B及 MTF 细胞形态;B:阴性对照组和共培养组基因差异表达前十位基因;C:RT-qPCR 检测 LGI4 的表达变化;D、E:Western blot 检测 LGI4 的表达变化 1:阴性对照组;2:共培养组;3:共培养基组;a: P < 0.05,与阴性对照组比较 图 1 骨肉瘤细胞143B和MTF与HUVECs共培养48 h后LGI4表达(n=3,x±s) |
| 基因名称 | 差异倍数 | 功能 |
| CCL20 | 5.818 058 | 巨噬细胞炎症蛋白 |
| SNORA14B | 5.545 233 | 小核仁RNA,胰腺癌诊断 |
| PTPRN | 5.174 403 | 提示肺癌不良预后 |
| KDF1 | 4.660 147 | 调控增殖、转移的抑癌基因 |
| NMRAL2P | -1.002 93 | LncRNA,促癌基因 |
| NEURL1B | -1.603 65 | 抑癌基因 |
| LGI4 | -2.156 89 | 促进髓鞘生成 |
| AKR1C1 | -3.102 51 | 肿瘤相关免疫细胞 |
| ABCC2 | -1.197 74 | 转运蛋白 |
| AL161431.1 | -1.234 55 | LncRNA,促癌基因 |
2.2 构建稳定表达的143B-LGI4和MTF-LGI4细胞系
与载体对照组相比,LGI4组的细胞密度明显较低(图 2A),进一步采用RT-qPCR和Western blot检测共培养体系及过表达细胞中LGI4的表达,结果表明成功构建LGI4过表达骨肉瘤稳定细胞系143B-LGI4和MTF-LGI4(P < 0.05,图 2B~D)。
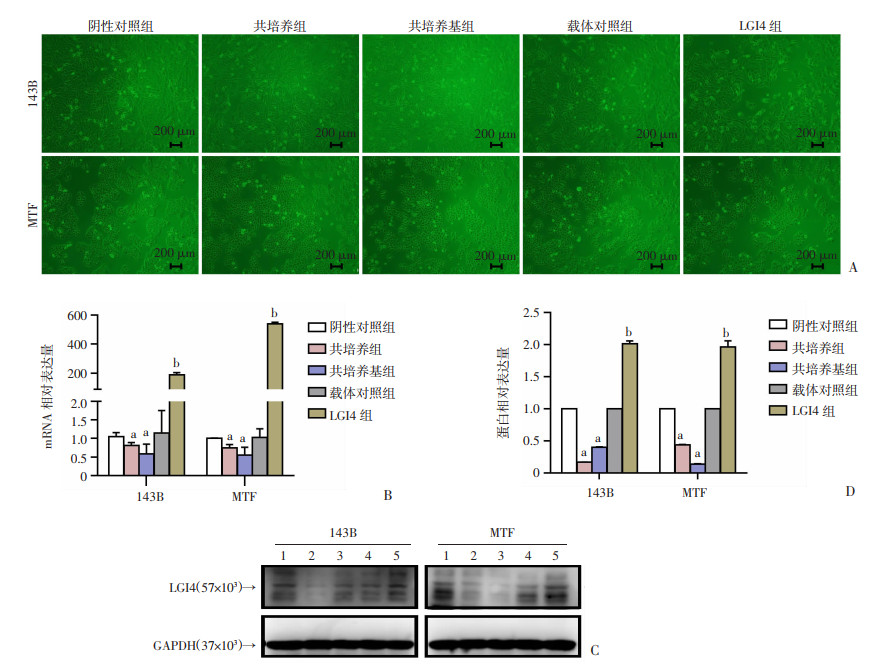
|
| A:143B 及 MTF 各组细胞形态; B:RT-qPCR 检测 LGI4 的表达变化;C、D:Western blot 检测LGI4的表达变化 1: 阴性对照组; 2:共培养组; 3:共培养基组; 4:载体对照组; 5:LGI4 组 a:P < 0.05,与阴性对照组比较;b:P < 0.05,与载体对照组比较 图 2 各组骨肉瘤细胞形态及LGI4的mRNA和蛋白表达变化(n=3,x±s) |
2.3 LGI4抑制了骨肉瘤细胞系143B和MTF的迁移、侵袭能力
划痕实验显示,骨肉瘤细胞系143B和MTF的共培养组和共培养基组的48 h迁移率均明显高于阴性对照组(P < 0.05,图 3A、B)。Transwell迁移及侵袭实验中骨肉瘤细胞系143B和MTF的共培养组和共培养基组的迁移、侵袭能力强于阴性对照组,143B和MTF LGI4组的迁移、侵袭能力均受到明显抑制(P < 0.05,图 3C~E)。结果表明,将143B和MTF与HUVECs共培养后其迁移、侵袭能力增加,而143B和MTF过表达LGI4后,其迁移、侵袭能力都显著降低,提示LGI4在骨肉瘤中起抑制细胞迁移和侵袭的作用。
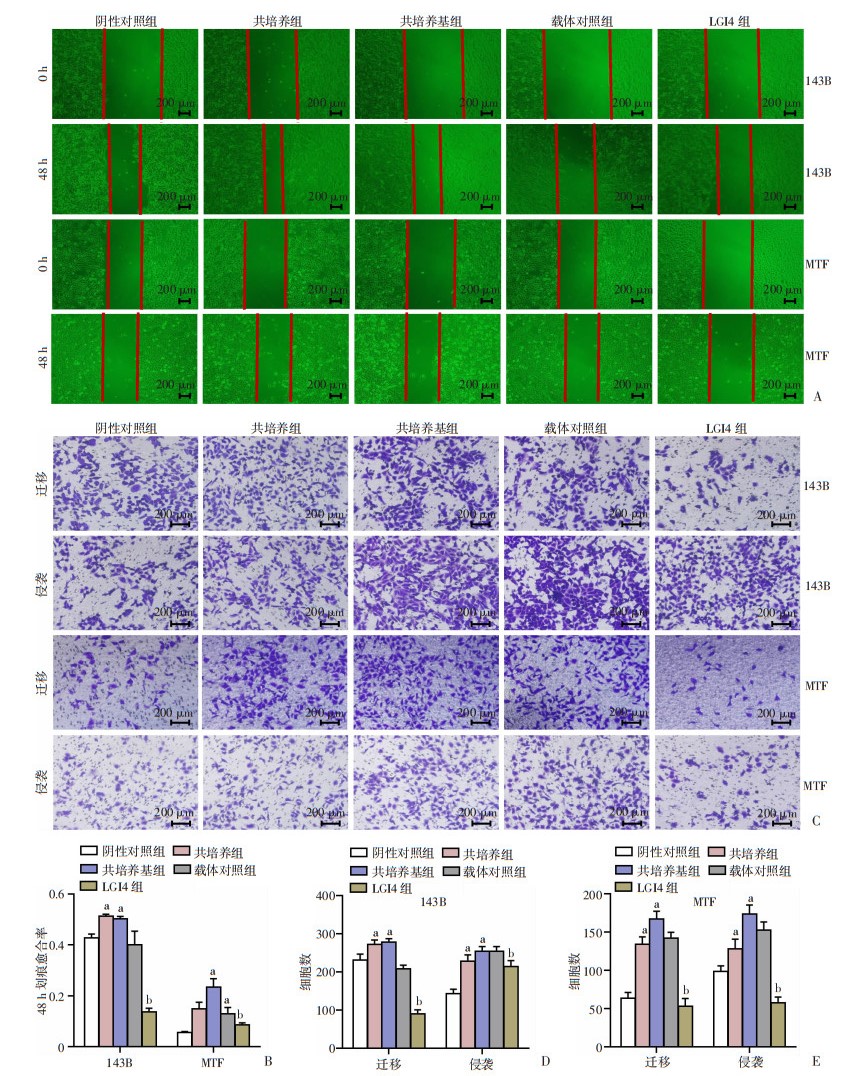
|
| A、B:143B 及 MTF 各组细胞的划痕愈合率;C、D、E:143B 及 MTF 各组细胞的迁移及侵袭能力 a:P < 0.05,与阴性对照组比较;b:P < 0.05,与载体对照组比较 图 3 LGI4对骨肉瘤细胞迁移及侵袭能力的影响(n=3,x±s) |
2.4 LGI4抑制了骨肉瘤细胞143B在裸鼠脏器的转移
在裸鼠转移模型尾静脉注射骨肉瘤细胞30天后观察到裸鼠状态不佳,随即处死裸鼠并取材心、肝、脾、肺和双肾后进行大体观察,发现143B对照组肝组织表面有少量坏死,其余脏器表面未见明显转移灶或坏死灶(图 4A)。HE染色(图 4B)显示,裸鼠模型发生了肝转移和肺转移,低倍镜下对转移灶进行计数,对照组和实验组肺组织内转移灶的数量分别为(42.83± 8.38)、(30.8±9.20)个,差异有统计学意义(P < 0.05);肝组织内转移灶的数量分别为(97.50±20.78)、(14.00±3.61)个,差异有统计学意义(P < 0.05)。
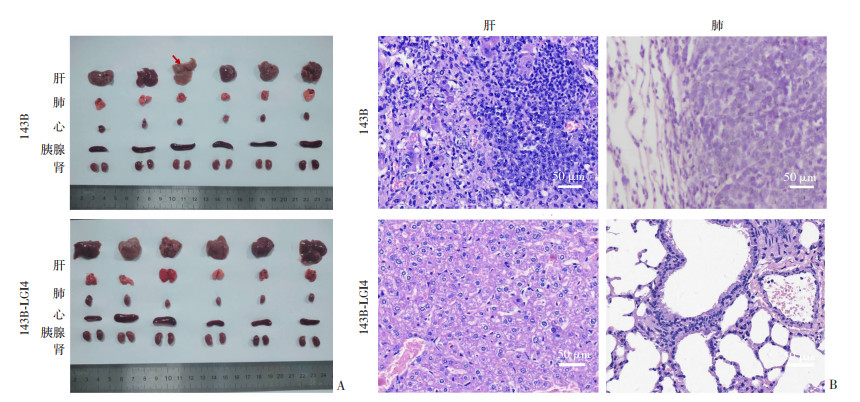
|
| A:裸鼠转移模型脏器转移大体观察;B: 裸鼠转移模型肝和肺转移病理学观察 图 4 裸鼠转移模型比较143B组和143B-LGI4组的转移情况(n=6,x±s) |
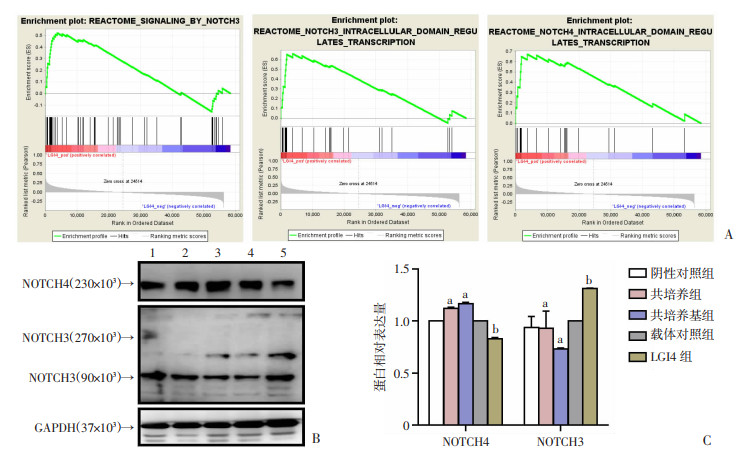
2.5 NOTCH4是LGI4的下游靶信号分子
为了探索LGI4的下游靶信号通路,根据转录组测序数据进行GSEA(gene set enrichment analysis)分析,骨肉瘤细胞与HUVECs共培养后与LGI4相关的下游信号通路基因见表 2。结果显示NOTCH3和NOTCH4在LGI4下游富集(图 5A),Western blot检测结果显示随着LGI4上调,NOTCH4上调,而NOTCH3下调(图 5B、C)。
| 下游信号路径 | 富集得分 | 校正后的富集分数 | 名义P值 | 误报率q值 | 错误发现率P值 |
| NEGATIVE_REGULATION_OF_ NOTCH4_SIGNALING | -0.603 5 | -2.275 9 | 0.010 8 | 0 | 0.008 4 |
| SIGNALING_BY_NOTCH | -0.500 8 | -1.330 4 | 0.014 2 | 0.217 9 | 0.338 9 |
| SIGNALING_BY_NOTCH4 | 0.521 1 | -1.693 3 | 0.021 0 | 0.287 7 | 0.424 0 |
| NOTCH1_INTRACELLULAR_DOMAIN_REGULATES_TRANSCRIPTION | 0.670 8 | -1.723 3 | 0.023 6 | 0.429 1 | 0.534 7 |
| NOTCH3_INTRACELLULAR_DOMAIN_REGULATES_TRANSCRIPTION | 0.659 6 | 1.814 1 | 0.010 8 | 1 | 0.565 |
| SIGNALING_BY_NOTCH3 | 0.521 1 | 1.693 3 | 0.021 0 | 1 | 0.791 |
| NOTCH4_INTRACELLULAR_DOMAIN_REGULATES_TRANSCRIPTION | 0.670 8 | 1.723 3 | 0.023 6 | 1 | 0.748 |
|
| A: GSEA 分析富集 REACTOME 中 LGI4 相关信号通路;B、C:Western blot 检测 NOTCH3 和 NOTCH4 的表达 1: 阴性对照组;2:共培养组;3:共培养基组;4:载体对照组;5:LGI4组;a:P < 0.05,与阴性对照组比较;b:P < 0.05,与载体对照组比较 图 5 LGI4和NOTCH信号通路的关联性分析(n=3,x±s) |
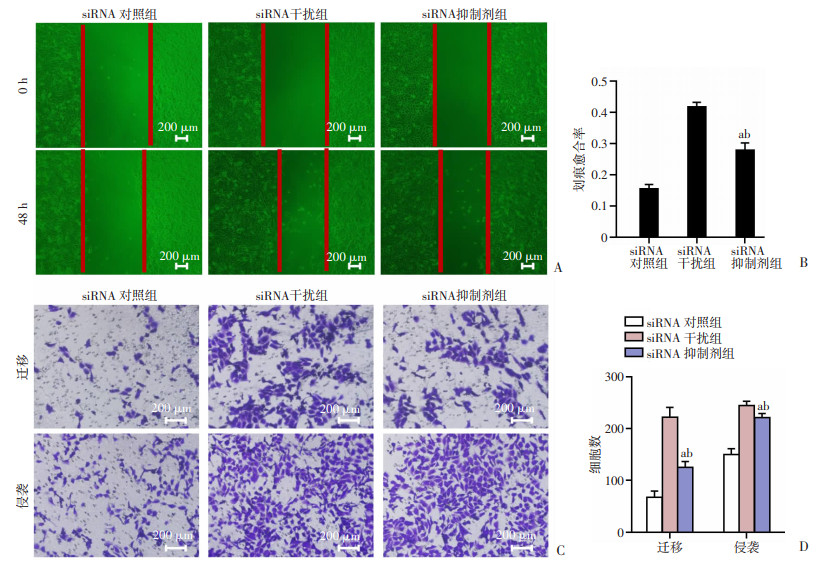
2.6 抑制LGI4下游NOTCH4信号通路抑制了骨肉瘤细胞143B的迁移及侵袭能力
为了明确NOTCH4是否参与了LGI4调控骨肉瘤细胞转移的过程,使用NOTCH抑制剂对NOTCH4活性抑制后进行功能回复实验,发现抑制下游NOTCH4信号通路,siRNA抑制剂组划痕愈合更快(P < 0.05,图 6A、B);同时在迁移和侵袭实验中,迁移侵袭能力相较于siRNA对照组更强(P < 0.05,图 6C、D)。说明在骨肉瘤细胞中LGI4可能通过激活NOTCH4信号通路抑制骨肉瘤的转移。
|
| A、B:各组细胞的划痕愈合情况;C、D:各组细胞的迁移及侵袭能力 a:P < 0.05,与siRNA干扰组比较;b:P < 0.05,与siRNA对照组比较 图 6 抑制NOTCH4信号通路对骨肉瘤细胞迁移及侵袭能力的影响(n=3,x±s) |
3 讨论
骨肉瘤作为常见的骨原发恶性肿瘤,具有极高的肿瘤异质性。尽管其发病率不高,但患者往往在确诊时已是临床晚期,因此骨肉瘤患者的预后极差[17]。骨肉瘤作为血管极其丰富的肿瘤,血管内皮细胞是其重要的微环境成分之一[18],内皮细胞除了形成肿瘤血管这一主要功能外,还可以分泌细胞因子,如VEGF、TGF等[19]。本研究采用全转录组测序、RT-qPCR和Western blot发现骨肉瘤细胞与HUVECs共培养后LGI4表达明显下调,为进一步探讨LGI4在骨肉瘤中的作用,本研究首先通过体外实验,建立稳定的LGI4过表达骨肉瘤细胞系143B-LGI4和MTF-LGI4,并证实骨肉瘤细胞143B-LGI4和MTF-LGI4的迁移、侵袭功能受到抑制。在体内实验中,骨肉瘤细胞143B-LGI4裸鼠转移模型组小鼠肝和肺发生的转移明显少于对照组143B,表明LGI4在骨肉瘤中发挥了抑癌基因的作用。LGI家族已被发现在多种肿瘤中不同程度地表达,例如恶性黑色素瘤、胶质瘤、结肠癌等[20]。此前,LGI4仅在少数肿瘤中作为一个预后相关标记被报道,并未深入探讨其影响肿瘤预后的机制。生物信息学研究表明,同正常结肠组织相比,结肠癌组织中LGI4的水平显著下调[21]。另有生物信息学研究通过对肝癌耐药细胞株基因测序,发现LGI4的高表达,并运用Ualcan数据库分析LGI4在肿瘤组织与正常组织中的表达发现,在淋巴结转移患者中LGI4上调,且LGI4下调患者生存期延长[22],目前仅有研究表明LGI4参与乳腺癌进展及转移[16]。而本研究证明LGI4可能参与骨肉瘤的进展及转移。
如前所述,LGI4参与了骨肉瘤的转移过程,但其下游的靶信号通路尚不清楚。结合生物信息学和Western blot分析,发现骨肉瘤细胞与HUVECs共培养后LGI4与NOTCH3和NOTCH4都具有关联性。鉴于课题组前期实验证明NOTCH3在骨肉瘤中发挥促癌作用[23],与LGI4的抑制转移作用相悖,因而NOTCH3可能不是LGI4的潜在下游信号通路。而NOTCH4在结肠癌和乳腺癌中都具有抑制肿瘤转移的作用[24-25],与LGI4作用一致,因此,LGI4是否是通过激活NOTCH4实现抑制骨肉瘤转移的作用呢?NOTCH4在多种肿瘤中发挥抑癌作用,为进一步探索NOTCH4和LGI4的关系,本研究采用NOTCH抑制剂DAPT,抑制NOTCH4信号通路后,143B-LGI4的迁移侵袭能力得到一定程度的恢复。表明LGI4可能通过NOTCH4信号通路发挥作用,为下一步探索LGI4对骨肉瘤的分子机制以及同血管内皮的相互作用关系奠定了基础。
综上所述,本研究通过细胞学实验、裸鼠模型及生物信息学分析发现,LGI4可能是血管内皮和骨肉瘤相互作用的关键信号,并作为抑癌基因参与了对抗骨肉瘤转移,LGI4可能通过NOTCH4信号通路发挥作用,但关于内皮细胞是如何调控骨肉瘤细胞LGI4的表达以及LGI4如何调控NOTCH4信号通路的分子机制还需进一步深入研究。
| [1] |
GILL J, GORLICK R. Advancing therapy for osteosarcoma[J]. Nat Rev Clin Oncol, 2021, 18(10): 609-624. |
| [2] |
YANG B S, LI L T, TONG G, et al. Circular RNA circ_001422 promotes the progression and metastasis of osteosarcoma via the miR-195-5p/FGF2/PI3K/Akt axis[J]. J Exp Clin Cancer Res, 2021, 40(1): 235. |
| [3] |
LIU Y, HUANG N G, LIAO S J, et al. Current research progress in targeted anti-angiogenesis therapy for osteosarcoma[J]. Cell Prolif, 2021, 54(9): e13102. |
| [4] |
MAISHI N, ANNAN D A, KIKUCHI H, et al. Tumor endo thelial heterogeneity in cancer progression[J]. Cancers (Basel), 2019, 11(10): E1511. |
| [5] |
MAISHI N, HIDA K. Tumor endothelial cells accelerate tumor metastasis[J]. Cancer Sci, 2017, 108(10): 1921-1926. |
| [6] |
HERRANZ-PÉREZ V, OLUCHA-BORDONAU F E, MORANTE-REDOLAT J M, et al. Regional distribution of the leucine-rich glioma inactivated (LGI) gene family transcripts in the adult mouse brain[J]. Brain Res, 2010, 1307: 177-194. |
| [7] |
KEGEL L, AUNIN E, MEIJER D, et al. LGI proteins in the nervous system[J]. ASN Neuro, 2013, 5(3): 167-181. |
| [8] |
GU W L, WEVERS A, SCHRÖDER H, et al. The LGI1 gene involved in lateral temporal lobe epilepsy belongs to a new subfamily of leucine-rich repeat proteins[J]. FEBS Lett, 2002, 519(1/2/3): 71-76. |
| [9] |
SAGANE K, ISHIHAMA Y, SUGIMOTO H. LGI1 and LGI4 bind to ADAM22, ADAM23 and ADAM11[J]. Int J Biol Sci, 2008, 4(6): 387-396. |
| [10] |
CHERNOVA O B, SOMERVILLE R P, COWELL J K. A novel gene, LGI1, from 10q24 is rearranged and downregulated in malignant brain tumors[J]. Oncogene, 1998, 17(22): 2873-2881. |
| [11] |
JOKINEN T S, METSÄHONKALA L, BERGAMASCO L, et al. Benign familial juvenile epilepsy in Lagotto Romagnolo dogs[J]. J Vet Intern Med, 2007, 21(3): 464-471. |
| [12] |
KIM D S, KWON N S, YUN H Y. Leucine rich repeat LGI family member 3: Integrative analyses reveal its prognostic association with non-small cell lung cancer[J]. Oncol Lett, 2019, 18(3): 3388-3398. |
| [13] |
BOOTH D G, KOZAR N, BRADLEY S, et al. Characterizing the molecular etiology of arthrogryposis multiplex congenita in patients with LGI4 mutations[J]. Glia, 2021, 69(11): 2605-2617. |
| [14] |
XUE S F, MALUENDA J, MARGUET F, et al. Loss-of-function mutations in LGI4, a secreted ligand involved in schwann cell myelination, are responsible for arthrogryposis multiplex congenita[J]. Am J Hum Genet, 2017, 100(4): 659-665. |
| [15] |
NISHINO J, SAUNDERS T L, SAGANE K, et al. LGI4 promotes the proliferation and differentiation of glial lineage cells throughout the developing peripheral nervous system[J]. J Neurosci, 2010, 30(45): 15228-15240. |
| [16] |
COSTA E T, BARNABÉ G F, LI M, et al. Intratumoral heterogeneity of ADAM23 promotes tumor growth and metastasis through LGI4 and nitric oxide signals[J]. Oncogene, 2015, 34(10): 1270-1279. |
| [17] |
FIORAMONTI M, FAUSTI V, PANTANO F, et al. Cabozantinib affects osteosarcoma growth through a direct effect on tumor cells and modifications in bone microenvironment[J]. Sci Rep, 2018, 8(1): 4177. |
| [18] |
CORRE I, VERRECCHIA F, CRENN V, et al. The osteosarcoma microenvironment: a complex but targetable ecosystem[J]. Cells, 2020, 9(4): E976. |
| [19] |
PELELI M, MOUSTAKAS A, PAPAPETROPOULOS A. Endothelial-tumor cell interaction in brain and CNS malignancies[J]. Int J Mol Sci, 2020, 21(19): 7371. |
| [20] |
ROSSI M R, HUNTOON K, COWELL J K. Differential expression of the LGI and SLIT families of genes in human cancer cells[J]. Gene, 2005, 356: 85-90. |
| [21] |
LUO Y B, SUN F L, PENG X W, et al. Integrated bioinformatics analysis to identify abnormal methylated differentially expressed genes for predicting prognosis of human colon cancer[J]. Int J Gen Med, 2021, 14: 4745-4756. |
| [22] |
马博, 田志华, 曲莉, 等. 索拉菲尼诱导的肝癌耐药细胞株的建立及基因表达分析[J]. 北京大学学报(医学版), 2020, 52(2): 207-213. MA B, TIAN Z H, QU L, et al. Establishment and gene expression analysis of drug-resistant cell lines in hepatocellular carcinoma induced by sorafenib[J]. J Peking Univ Heal Sci, 2020, 52(2): 207-213. |
| [23] |
TANG X F, CAO Y, PENG D B, et al. Overexpression of Notch3 is associated with metastasis and poor prognosis in osteosarcoma patients[J]. Cancer Manag Res, 2019, 11: 547-559. |
| [24] |
ZHANG Z X, BU X, YANG J Y, et al. NOTCH4 regulates colorectal cancer proliferation, invasiveness, and determines clinical outcome of patients[J]. J Cell Physiol, 2018, 233(10): 6975-6985. |
| [25] |
LI W J, XIE X X, BAI J, et al. Increased expression of miR-1179 inhibits breast cancer cell metastasis by modulating Notch signaling pathway and correlates with favorable prognosis[J]. Eur Rev Med Pharmacol Sci, 2018, 22(23): 8374-8382. |




