神经母细胞瘤(neuroblastoma,NB)是儿童最常见的实体肿瘤,该疾病约占儿童癌症相关死亡率的15%[1]。大多数病例在5岁之前确诊,最常见的发病年龄段在18~22个月[1]。在美国,每年约有700名儿童被诊断为NB,占所有类型儿童癌症的8%~10%,占儿童癌症死亡人数的15%[2]。目前对高危NB的治疗标准包括诱导、巩固和维持[3],而NB表现出的遗传和临床的异质性限制了标准治疗方法的疗效[4]。约一半的高危NB患者对标准治疗方案无反应或在治疗后的两年内复发,长期生存率仍低于50%[5-8]。最近,TSVETKOV等[9]发现了一种新的细胞死亡途径,并将其命名为铜死亡。铜死亡与现有已知的细胞死亡途径不同,主要依赖细胞中铜离子的积累参与线粒体代谢活性。同时,为了探索调节铜死亡过程的关键基因,研究人员使用全基因组CRISPR-Cas9功能丧失筛选,再进行个体敲除实验。最终确定了10个与铜死亡过程密切相关的调节基因。关于铜死亡在癌症中的作用,特别是在儿童NB预后中的作用仍不清楚。
本研究基于公开的基因表达谱数据,对NB样本中铜死亡相关基因(cuproptosis-related genes,CRGs)的表达特征及预后情况进行分析。通过LASSO-Cox回归在3个独立队列中构建并验证铜死亡相关预后模型的临床价值和风险基因的表达和功能。
1 材料与方法 1.1 NB数据集的准备和处理从公开的ArrayExpress数据库、基因表达综合数据库(gene expression omnibus,GEO)和开放有效治疗方法的治疗性研究数据库(therapeutically applicable research to generate effective treatments,TARGET)数据库中收集具有相关完整临床病理变量的基因表达数据。本研究总共获得了3个独立的数据集,包括来自GEO数据库的GSE49711队列、来自TARGET数据库的TARGET-NB队列和来自ArrayExpress数据库的E-MTAB-8248队列。缺失随访信息的患者被排除,最终纳入968例患者进行后续数据分析。相关临床变量包括年龄、性别、种族、DNA倍体、美国儿童肿瘤协作组(Children’s Oncology Group,COG)风险、组织学、有丝分裂核碎裂指数(mitosis-karyorrhexis index,MKI)、染色体1p缺失(1pdel/im)、谷丙转氨酶状态(ALT status)、MYCN基因状态(MYCN status)、国际神经母细胞瘤分期(International Neuroblastoma Staging System,INSS)和生存信息。基于最大的样本量,选择GSE49711队列作为“训练集”,开发预后基因模型。将TARGET-NB和E-MTAB-8248队列作为“验证集”,检测预后模型的可靠性。所有的分析数据都是公开的,因此不需要伦理批准。
1.2 基于铜死亡相关基因构建预后模型从已发表的研究中确定了10个与“铜死亡”密切相关的调节基因,包括:PDHA1、GLS、LIAS、MTF1、PDHB、CDKN2A、DLAT、LIPT1、FDX1和DLD。本研究使用来自GSE49711队列的数据作为训练队列。采用单因素Cox回归分析初步筛选与预后相关的铜死亡相关基因(cuproptosis-related genes,CRGs)。基于预后相关的CRGs,采用LASSO-Cox比例风险模型(迭代10次)和R包“glmnet”鉴定最佳预后多基因模型[10]。通过将选定的基因表达水平以线性方式组合,并根据它们的LASSO-Cox回归系数进行加权,构建了铜死亡相关风险模型。公式:风险评分=Σin(Coefi×Xi)可以简洁地描述所开发的预后模型,其中X代表每个基因的表达水平,Coefi代表模型中相应基因的加权系数。风险评分公式可计算每个病人的风险分数,中位数被定义为高风险组和低风险组之间的分界值。Kaplan-Meier生存曲线和时间依赖的受试者工作特征(receiver operating characteristic,ROC)曲线用于评估该特征的预测能力。
1.3 铜死亡相关预后模型的验证为了验证该风险模型的临床价值,本研究评估了不同风险亚组中的临床病理特征分布,并用热图展示。为了验证风险得分的预测能力是否独立于传统的临床特征,本研究使用多变量Cox回归分析,并利用独立变量建立列线图。ROC曲线、校准曲线和由R软件包“rmda”计算的决策曲线(decision curve analysis,DCA)用来衡量列线图的准确性。
1.4 RT-qPCR和功能实验风险基因在NB细胞系[SH-SY5Y和BE(2)-C]中的表达水平通过RT-qPCR进行验证。使用Simply P Total RNA Extraction Kit(中国BioFlux公司)提取细胞总RNA。使用NanodropOne(美国赛默飞公司)测定RNA浓度。使用PrimeScriptTM RT试剂盒(日本TaKaRa公司)对mRNA进行反转录,并使用TB GreenⓇ Premix Ex TaqTMⅡ试剂盒(日本TaKaRa公司)进行扩增和定量。RT-qPCR在CFX Connect Real-Time PCR Detection System上操作。每个样本包含3个技术重复。GAPDH用于mRNA表达的标准化。目标基因的表达基于2-△△Ct公式计算。使用的引物见表 1。
| RNA | 前引物(5'→3') | 后引物(5'→3') |
| MTF1 | AGCACAATTTCACCAGCAATCAT | TGACTGACTCTGCATCACCATTAA |
| GLS | ACCAAAGTTCCCTTCTGTCTTCA | TGAAGTCACAACAATTGCTCCAG |
| PDHB | GGATAGAGGACACGACCAAGATG | GCTCCTCATCCATACCCTGATTT |
| PDHA1 | AGGGCCAGATATTCGAAGCTTAC | AGGAATGAAATCGCCTCTCTTGT |
| LIAS | AGTATGTGAGGAAGCTCGATGTC | GGAGGAGGATTTCTTGCAGTCTTA |
| GAPDH | CCTTCCTGGGCATGGAGTC | TGATCTTCATTGTGCTGGGTG |
PDHA1作为肿瘤抑制剂或致癌基因在多种恶性肿瘤中发挥了关键作用。本研究进一步探索PDHA1的潜在功能。使用BE(2)-C细胞转染PDHA1 siRNA(由擎科设计合成),进行功能丧失实验(用于沉默PDHA1的序列见表 2)。使用PCR和Western blot来验证PDHA1的沉默效率。分别采用CCK-8实验,伤口愈合实验和Transwell实验检测细胞增殖、迁移和侵袭能力。使用BD检测试剂盒和流式细胞仪检测细胞周期和细胞凋亡比例。
| siRNA | 序列(5'→3') |
| Si-PDHA1-1-ss | GCAGAGCUUACAGGACGAATT |
| Si-PDHA1-1-as | UUCGUCCUGUAAGCUCUGCTT |
| Si-PDHA1-2-ss | CCAGUGUGGAAGAACUAAATT |
| Si-PDHA1-2-as | UUUAGUUCUUCCACACUGGTT |
| Si-PDHA1-3-ss | GAGAAUAAUCGCUAUGGAATT |
| Si-PDHA1-3-as | UUCCAUAGCGAUUAUUCUCTT |
1.5 统计学分析
使用GraphPad Prism 8.0软件分析实验结果。所有的生物信息学分析和R包都使用R软件。P < 0.05认为差异具有统计学意义。
2 结果 2.1 基于GSE49711队列构建铜死亡相关预后模型本研究选择GSE49711数据集作为发现队列。单因素生存分析显示有6个CRGs具有预后能力(图 1A)。使用LASSO-Cox回归分析确定惩罚参数的最佳值并建立最优化的预后特征。针对对数λ序列生成了一个系数曲线图,最佳的λ显示了5个非零系数(图 1B、C)。最终得到一个达到最佳预测效率的铜死亡相关5基因模型。利用各基因的独立回归系数构建5基因风险特征,风险评分公式:(1.573)×PDHA1+(-0.561)×GLS+(0.320)×LIAS+(0.088)×MTF1+(0.301)×PDHB。根据风险评分公式,测算每个患者的风险分数,并根据风险分数将患者分成高风险组和低风险组。每位患者的风险基因表达如图 1D所示。主成分分析(principal component analysis,PCA)显示,根据这5个风险基因的表达水平,可以明显区分两个风险亚组(图 1E)。时间依赖的ROC曲线显示3、5、7年生存率的曲线下面积(area under the subject curve,AUC)分别为0.80、0.80和0.81(图 1F)。生存分析显示高风险亚组的NB患者的生存率显著低于低风险亚组(P < 0.001,图 1G)。
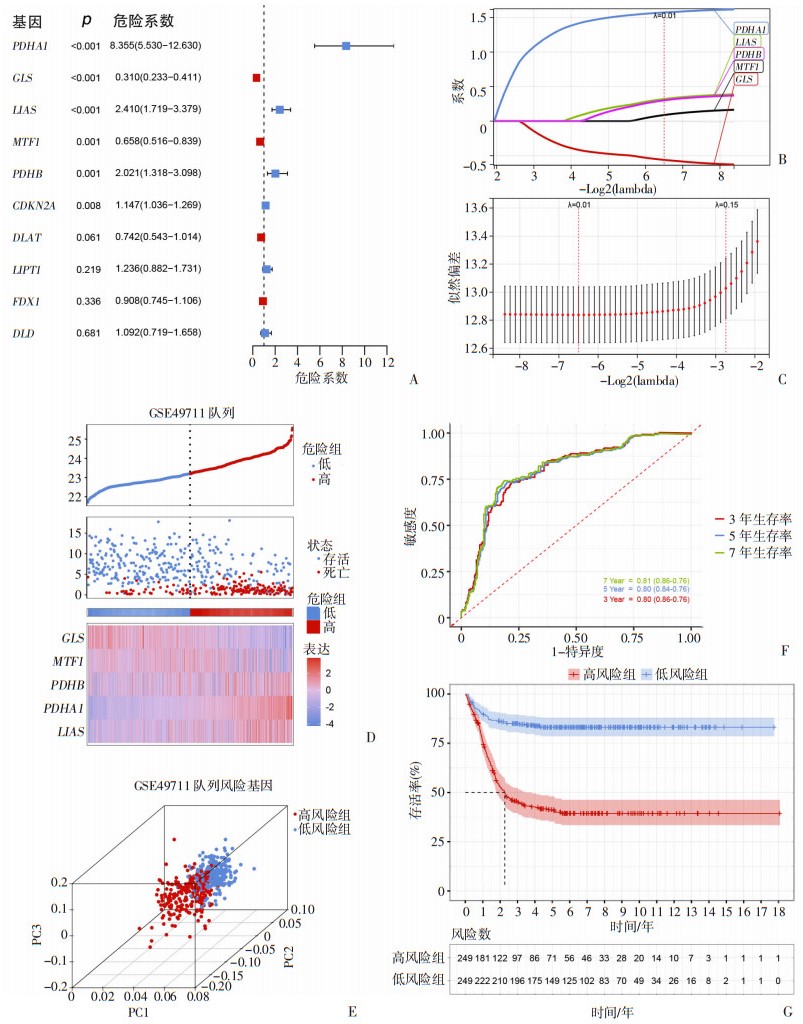
|
| A:6个铜死亡相关基因与NB患者预后相关;B:LASSO回归分析基因轨迹横轴:示自变量的对数值,纵轴:示自变量的系数;C:变量的部分似然偏差红点:示偏差值的部分可能性,灰线:示标准误差,左右两条垂直虚线:示最小标准和1-SE标准的最佳值;D:基因特征的风险评分分布、相关生存数据和基因表达热图;E:PCA分析不同风险组患者的区别;F:预后模型在3、5和7年生存率中的预测性能;G:高风险组患者和低风险组患者的生存分析 图 1 基于GSE49711队列构建铜死亡相关预后模型 |
2.2 基于TARGET-NB和E-MTAB-8248队列验证预后模型的准确性
为进一步验证预后模型在NB患者中的适用性,将在GSE49711队列中建立的预后模型应用于其他两个独立队列。根据建立的风险模型计算的TARGET-NB和E-MTAB-8248队列中风险分数绘制风险分数分布图(图 2A、B)。根据相应的中位得分,将患者分层为高风险组和低风险组,PCA可将这两个风险组明显区分开(图 2C、D)。生存分析表明,在TARGET-NB(P=0.011)和E-MTAB-8248队列(P=0.008 7)中高风险组的患者比低风险组的患者的预后更差(图 2E、F)。
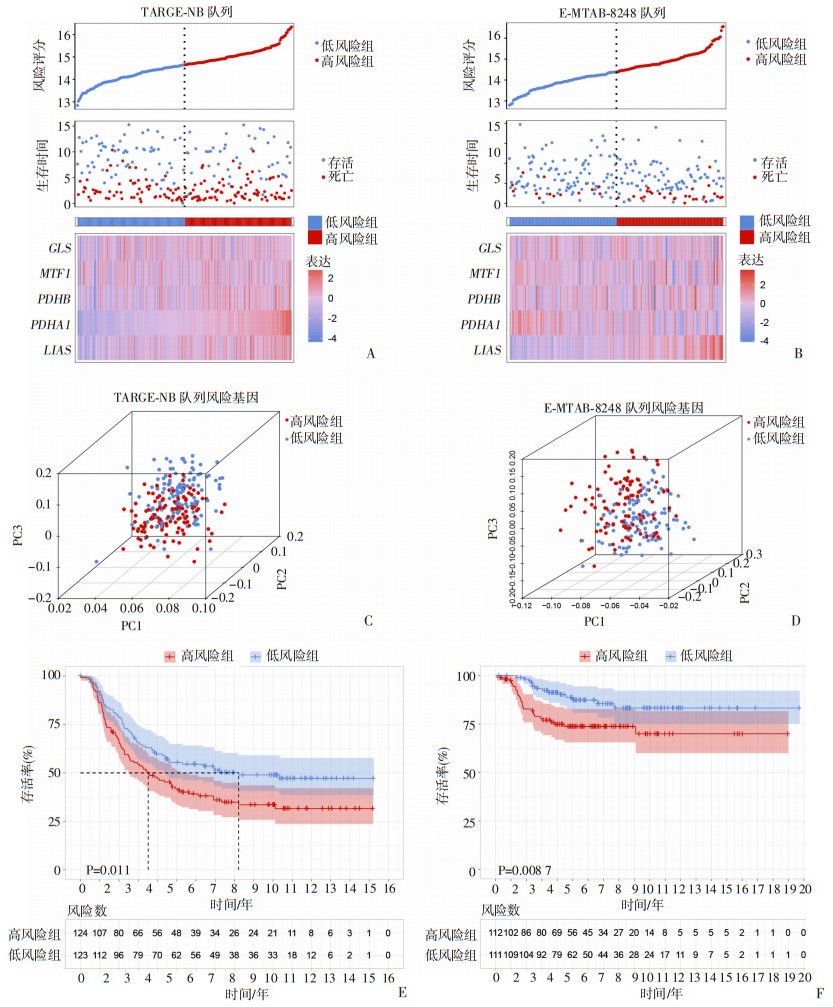
|
| A:基于TARGET-NB队列的风险评分、生存数据和风险基因表达热图;B:基于E-MTAB-8248队列的风险评分、生存数据和风险基因表达热图;C:基于TARGET-NB队列中风险基因表达的PCA分析;D:基于E-MTAB-8248队列中风险基因表达的PCA分析;E:TARGET-NB队列中高风险组患者和低风险组患者的生存分析;F:E-MTAB-8248队列中高风险组患者和低风险组患者的生存分析 图 2 基于两个独立的NB队列的预后模型准确性 |
2.3 风险分层与临床指标的相关性
本研究评估了不同风险组与临床病理特征之间的关联。复合热图显示了风险组和临床病理因素之间的相关性(图 3A~C)。在高风险组和低风险组之间,GSE49711队列中患者的年龄、COG风险、MYCN状态和INSS阶段,TARGET-NB队列中患者的年龄、性别、MYCN状态、INSS阶段、DNA倍体、组织学、COG风险和MKI以及E-MTAB-8248队列中患者的年龄、1p del/im和MYCN状态的差异均有统计学意义(P < 0.05)。在GSE49711队列的单变量Cox回归分析中,发现该预后模型的风险分数与患者的无病生存率显著相关(P < 0.001,图 3D)。在多变量Cox回归分析中,在调整了传统的临床预后变量(年龄、性别、COG风险、MYCN状态和INSS阶段)后,该模型依然可以作为NB患者的独立预测因子,表明模型稳定性较好(P < 0.001,图 3E)。
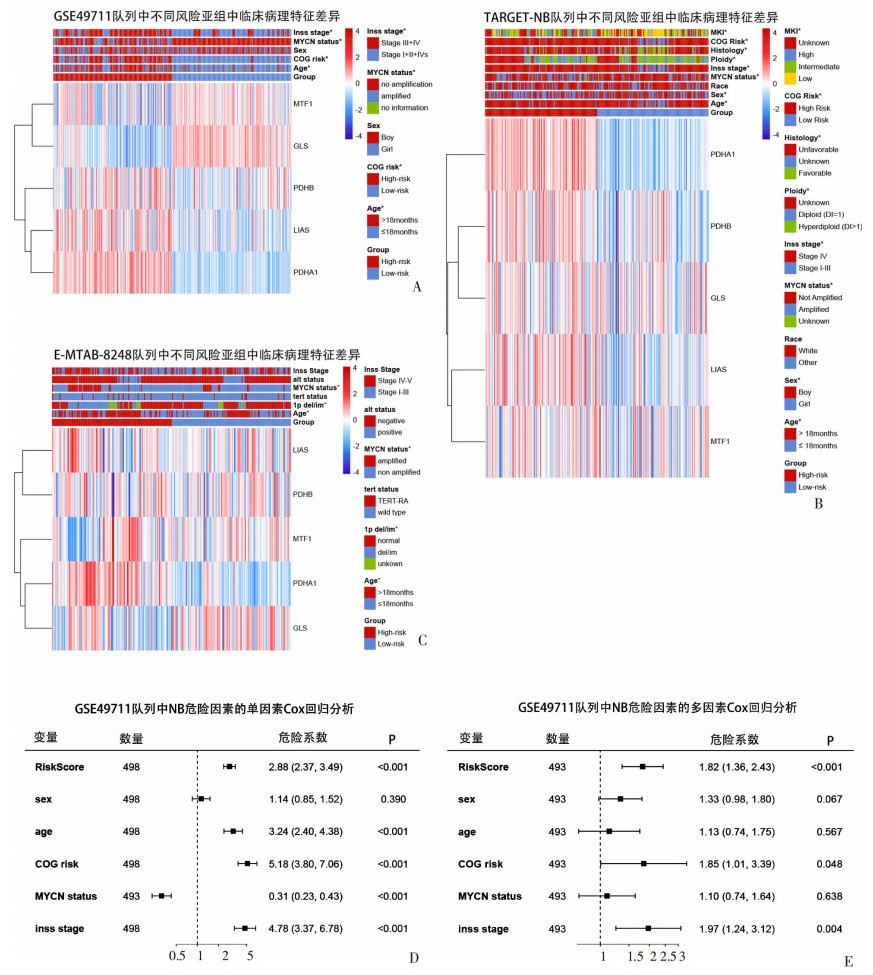
|
| A~C:分别为GSE49711、TARGET-NB、E-MTAB-8248队列中的不同风险亚组中临床病理特征的差异;D:GSE49711队列中NB危险因素的单因素Cox回归分析;E:GSE49711队列中NB危险因素的多因素Cox回归分析 图 3 不同风险组间的临床病理特征差异和风险评分的预后意义 |
2.4 基于风险评分和临床预后指标构建列线图及验证
GSE49711队列的预后分析表明,COG风险、MYCN状态和预后模型风险评分可作为NB患者的独立预后指标(P < 0.05)。结合上述风险模型评分和临床指标,本研究构建列线图以扩大临床可用性(图 4A)。通过将每个风险因素的加权分数相加,给每个病人分配一个风险分数,总分越高,对应的生存结果就越差。预测3、5、7年生存率的AUC值分别为0.83、0.83和0.84(图 4B)。校准曲线显示预测模型的性能良好(图 4C)。DCA显示,列线图具有良好的临床利用率(列线图的C指数为0.736),与单独的预后模型或单独的临床模型相比,联合列线图模型获得更多的净收益(图 4D)。
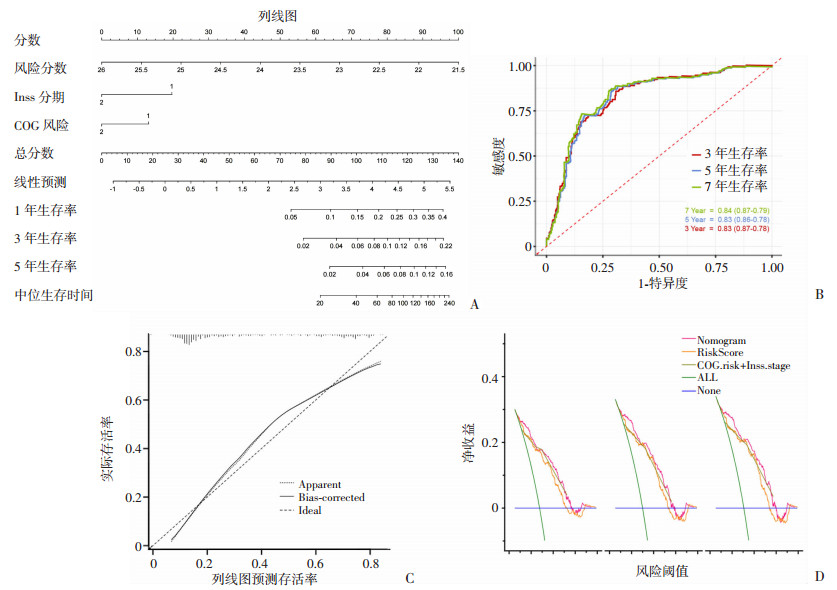
|
| A:联合风险评分和临床病理指标构建的列线图;B:依赖时间的ROC曲线评估列线图在3年、5年和7年生存期中的预测性能;C:评估列线图预测准确性的校准曲线;D:评估列线图临床收益的DCA曲线 图 4 联合风险评分和临床病理指标构建列线图,并验证其准确性 |
2.5 风险基因的表达和功能分析
PCR分析显示五个风险基因在SH-SY5Y(MYCN未扩增)和BE(2)-C(MYCN扩增)细胞系之间存在显著的表达差异(P < 0.05,图 5A)。为了探索PDHA1在NB中的功能作用,本研究设计了3个siRNA来沉默该基因,最终选择沉默效率最高的siRNA-3进行后续实验(P < 0.05,图 5B)。与阴性对照相比,siRNA-3显著降低了BE(2)-C细胞的蛋白水平(P < 0.05,图 5C)。功能丧失实验表明沉默PDHA1可以显著抑制了NB细胞的增殖、迁移和侵袭能力(P < 0.05,图 5D~F)。流式分析表明,沉默PDHA1基因促进NB细胞阻滞在S期,并增加细胞凋亡比例(P < 0.05,图 5G、H)。

|
| A:RT-qPCR检测5个风险基因在SH-SY5Y和BE(2)-C细胞中的mRNA表达水平;B:RT-qPCR检测3个不同siRNA的沉默效率;C:Western blot检测siRNA-3的蛋白敲底水平;D:CCK-8检测沉默PDHA1基因后的细胞增殖能力变化;E:划痕实验检测沉默PDHA1基因后的细胞迁移能力变化;F:Transwell实验检测沉默PDHA1基因后的细胞侵袭能力变化;G:流式细胞术检测沉默PDHA1基因后的细胞周期分布变化;H:流式细胞术检测沉默PDHA1基因后的细胞凋亡比例变化a:P < 0.05,与SH-SY5Y比较;b:P < 0.01,与Si-PDHA1-NC比较;c:P < 0.001,与Si-NC比较 图 5 风险模型相关基因的表达验证和功能验证 |
3 讨论
近来,研究者们已经注意到NB多样化和戏剧性的临床表现:一部分肿瘤会自行消退,而另一部分则持续进展[11]。自最初发现MYCN基因突变以来,研究人员已经发现较多NB的预后生物标志物,包括组织病理学分类、肿瘤分期、MYCN扩增、肿瘤细胞DNA倍体和分段染色体畸变等[12-14]。基于证据的NB风险分层在患者治疗和预后上取得了重大进展,但高危NB患者的预后仍需改善。使用微阵列、RNA-seq或其他技术对NB进行的遗传和分子分析正越来越多地被用来确定预测患者预后的遗传特征。
已有研究表明铜离子在NB细胞系SH-SY5Y通过多种途径如线粒体损伤和氧化应激诱导细胞凋亡[15-18]。因此,铜死亡相关基因可能在NB中发挥潜在的作用。最近,一个新的术语“cuproptosis”即“铜死亡”被用来表示由铜离子依赖的一种新的细胞死亡形式。该研究揭示铜的毒性来自于某些线粒体代谢酶的破坏,导致一种独特的细胞死亡机制[19]。该机制可以解释与铜超载疾病有关的病理现象,并提出一种新的癌症治疗方式[16]。同时,该研究团队确定了促进铜诱导的细胞死亡的关键调节基因。本研究旨在分析铜死亡相关基因在NB预后中的作用,对预后预测和风险调整的个体化管理具有潜在的临床意义。
本研究采用LASSO-Cox回归分析基于铜死亡相关的基因,建立铜死亡相关的多基因预后模型,并构建风险评分系统。利用风险评分将所有患者分为高风险组和低风险组。该模型在预测生存风险和临床病理特征方面均显示出良好的预后价值。单变量和多变量Cox回归分析显示,风险分数是NB患者的独立预后指标,表明该预后模型可以作为一个可靠的预测工具,同时,以上分析结果在另外两个独立队列中得到验证。为给临床提供预测NB患者预后的定量方法,本研究将临床特征与风险评分结合来构建组合列线图模型,可更准确地预测NB患者的短期和长期生存情况。
这些风险基因在肿瘤的预后和恶性进展中起着至关重要的作用。CAI等[20]通过一项全面的泛癌症分析表明,LIAS可能在各种癌症的进展中具有潜在意义,也可以预测癌症患者的免疫疗效。PDHB可作为多种非编码RNA的调控靶点调节肿瘤细胞的进展[21-23]。GLS是谷氨酰胺代谢的关键酶,在肿瘤发生中具有多种功能[24]。MTF1是哺乳动物细胞中金属调节系统的重要组成部分,敲除MTF1抑制卵巢癌细胞上皮-间质转化[25]。研究发现PDHA1通过代谢调控参与较多恶性肿瘤的发生和进展[26-27]。在一些肿瘤类型中,如肝细胞癌、胆管癌、卵巢癌或食管鳞癌,该基因通过调节Warburg效应或代谢重编程来调节肿瘤进展[26, 28-32]。此外,PDHA1可能是多种癌症的预后和免疫相关的生物标志物[33]。因此,PDHA1被认为是抗癌治疗的关键靶点,该基因被选作进一步的功能验证。功能丧失实验表明,沉默PDHA1能明显抑制NB细胞的增殖、迁移和侵袭能力,同时促进细胞周期阻滞和细胞凋亡比例。以上结果提示PDHA1在NB发生发展中的重要作用。
综上所述,本研究结果表明铜死亡相关预后模型在NB患者中的预后价值,该模型可以帮助风险调整的个性化治疗。研究结果突出了铜死亡相关基因的重要临床意义,有助于为NB进展的分子机制提供新的见解。
| [1] |
NEWMAN E A Jr. Update on neuroblastoma[J]. J Pediatr Surg, 2019, 54(3): 383-389. |
| [2] |
SIEGEL D A, KING J, TAI E, et al. Cancer incidence rates and trends among children and adolescents in the United States, 2001-2009[J]. Pediatrics, 2014, 134(4): e945-e955. |
| [3] |
PINTO N R, APPLEBAUM M A, VOLCHENBOUM S L, et al. Advances in risk classification and treatment strategies for neuroblastoma[J]. J Clin Oncol, 2015, 33(27): 3008-3017. |
| [4] |
WHITTLE S B, SMITH V, DOHERTY E, et al. Overview and recent advances in the treatment of neuroblastoma[J]. Expert Rev Anticancer Ther, 2017, 17(4): 369-386. |
| [5] |
BERLANGA P, CAÑETE A, CASTEL V. Advances in emerging drugs for the treatment of neuroblastoma[J]. Expert Opin Emerg Drugs, 2017, 22(1): 63-75. |
| [6] |
DE BERNARDI B, NICOLAS B, BONI L C, et al. Disseminated neuroblastoma in children older than one year at diagnosis: comparable results with three consecutive high-dose protocols adopted by the Italian Co-Operative Group for Neuroblastoma[J]. J Clin Oncol, 2003, 21(8): 1592-1601. |
| [7] |
BERTHOLD F, FALDUM A, ERNST A, et al. Extended induction chemotherapy does not improve the outcome for high-risk neuroblastoma patients: results of the randomized open-label GPOH trial NB2004-HR[J]. Ann Oncol, 2020, 31(3): 422-429. |
| [8] |
MATTHAY K K, VILLABLANCA J G, SEEGER R C, et al. Treatment of high-risk neuroblastoma with intensive chemotherapy, radiotherapy, autologous bone marrow transplantation, and 13-Cis-retinoic acid. Children's Cancer Group[J]. N Engl J Med, 1999, 341(16): 1165-1173. |
| [9] |
TSVETKOV P, COY S, PETROVA B, et al. Copper induces cell death by targeting lipoylated TCA cycle proteins[J]. Science, 2022, 375(6586): 1254-1261. |
| [10] |
ENGEBRETSEN S, BOHLIN J. Statistical predictions with glmnet[J]. Clin Epigenetics, 2019, 11(1): 123. |
| [11] |
MARIS J M, HOGARTY M D, BAGATELL R, et al. Neuroblastoma[J]. Lancet, 2007, 369(9579): 2106-2120. |
| [12] |
COHN S L, PEARSON A D, LONDON W B, et al. The International Neuroblastoma Risk Group (INRG) classification system: an INRG task force report[J]. J Clin Oncol, 2009, 27(2): 289-297. |
| [13] |
MONCLAIR T, BRODEUR G M, AMBROS P F, et al. The International Neuroblastoma Risk Group (INRG) staging system: an INRG Task Force report[J]. J Clin Oncol, 2009, 27(2): 298-303. |
| [14] |
MARIS J M. Recent advances in neuroblastoma[J]. N Engl J Med, 2010, 362(23): 2202-2211. |
| [15] |
MATIAS A C, et al. Diethyldithiocarbamate induces apoptosis in neuroblastoma cells by raising the intracellular copper level, triggering cytochrome c release and caspase activation[J]. Toxicol In Vitro, 2013, 27(1): 349-357. |
| [16] |
LOMBARDO M F, CIRIOLO M R, ROTILIO G, et al. Prolonged copper depletion induces expression of antioxidants and triggers apoptosis in SH-SY5Y neuroblastoma cells[J]. Cell Mol Life Sci, 2003, 60(8): 1733-1743. |
| [17] |
FILOMENI G, CARDACI S, DA COSTA FERREIRA A M, et al. Metabolic oxidative stress elicited by the copper(Ⅱ) complex[Cu(isaepy)2]triggers apoptosis in SH-SY5Y cells through the induction of the AMP-activated protein kinase/p38MAPK/p53 signalling axis: evidence for a combined use with 3-bromopyruvate in neuroblastoma treatment[J]. Biochem J, 2011, 437(3): 443-453. |
| [18] |
ARCIELLO M, ROTILIO G, ROSSI L. Copper-dependent toxicity in SH-SY5Y neuroblastoma cells involves mitochondrial damage[J]. Biochem Biophys Res Commun, 2005, 327(2): 454-459. |
| [19] |
LI S R, BU L L, CAI L L. Cuproptosis: lipoylated TCA cycle proteins-mediated novel cell death pathway[J]. Signal Transduct Target Ther, 2022, 7(1): 158. |
| [20] |
CAI Y, HE Q C, LIU W, et al. Comprehensive analysis of the potential cuproptosis-related biomarker LIAS that regulates prognosis and immunotherapy of pan-cancers[J]. Front Oncol, 2022, 12: 952129. |
| [21] |
ZHU Y Z, WU G, YAN W F, et al. miR-146b-5p regulates cell growth, invasion, and metabolism by targeting PDHB in colorectal cancer[J]. Am J Cancer Res, 2017, 7(5): 1136-1150. |
| [22] |
XU D X, GUO J J, ZHU G Y, et al. miR-363-3p modulates cell growth and invasion in glioma by directly targeting pyruvate dehydrogenase B[J]. Eur Rev Med Pharmacol Sci, 2018, 22(16): 5230-5239. |
| [23] |
WANG G Q, YE Q W, NING S J, et al. LncRNA MEG3 promotes endoplasmic Reticulum stress and suppresses proliferation and invasion of colorectal carcinoma cells through the MEG3/miR-103a-3p/PDHB CeRNA pathway[J]. Neoplasma, 2021, 68(2): 362-374. |
| [24] |
MASISI B K, EL ANSARI R, ALFARSI L, et al. The role of glutaminase in cancer[J]. Histopathology, 2020, 76(4): 498-508. |
| [25] |
JI L, ZHAO G N, ZHANG P, et al. Knockout of MTF1 inhibits the epithelial to mesenchymal transition in ovarian cancer cells[J]. J Cancer, 2018, 9(24): 4578-4585. |
| [26] |
LI D, WANG C Q, MA P F, et al. PGC1α promotes cholangiocarcinoma metastasis by upregulating PDHA1 and MPC1 expression to reverse the Warburg effect[J]. Cell Death Dis, 2018, 9(5): 466. |
| [27] |
CHEN J J, GUCCINI I, MITRI D D, et al. Compartmentalized activities of the pyruvate dehydrogenase complex sustain lipogenesis in prostate cancer[J]. Nat Genet, 2018, 50(2): 219-228. |
| [28] |
ZHUANG L W, ZHANG B B, LIU X M, et al. Exosomal miR-21-5p derived from cisplatin-resistant SKOV3 ovarian cancer cells promotes glycolysis and inhibits chemosensitivity of its progenitor SKOV3 cells by targeting PDHA1[J]. Cell Biol Int, 2021, 45(10): 2140-2149. |
| [29] |
SUN J H, LI J J, GUO Z X, et al. Overexpression of pyruvate dehydrogenase E1α subunit inhibits Warburg effect and induces cell apoptosis through mitochondria-mediated pathway in hepatocellular carcinoma[J]. Oncol Res, 2019, 27(4): 407-414. |
| [30] |
LIU Z, YU M, FEI B Y, et al. miR-21-5p targets PDHA1 to regulate glycolysis and cancer progression in gastric cancer[J]. Oncol Rep, 2018, 40(5): 2955-2963. |
| [31] |
LIU L, CAO J, ZHAO J, et al. PDHA1 gene knockout in human esophageal squamous cancer cells resulted in greater Warburg effect and aggressive features in vitro and in vivo[J]. Onco Targets Ther, 2019, 12: 9899-9913. |
| [32] |
KWAK C H, JIN L, HAN J H, et al. Ilimaquinone induces the apoptotic cell death of cancer cells by reducing pyruvate dehydrogenase kinase 1 activity[J]. Int J Mol Sci, 2020, 21(17): 6021. |
| [33] |
DENG L M, JIANG A Q, ZENG H Q, et al. Comprehensive analyses of PDHA1 that serves as a predictive biomarker for immunotherapy response in cancer[J]. Front Pharmacol, 2022, 13: 947372. |




