肾透明细胞癌(kidney renal clear cell carcinoma,KIRC)是肾细胞癌中最常见的类型,占所有肾细胞癌患者的75%~85%,其发病原因与遗传、吸烟、肥胖及高血压等有关。临床上多数KIRC患者接受化疗、放疗或靶向治疗后均产生较为明显的副作用,手术切除仍是目前的主要治疗手段[1-2]。KIRC患者与其他肾癌亚型患者相比,具有更高的肿瘤复发率和转移率,且与其不良预后高度相关[3-4],探索潜在的生物标志物用于指导KIRC的术后治疗意义重大。
代谢为细胞提供能量来源,是生命活动的基本特征之一。代谢特征异常改变是肿瘤细胞快速增殖的显著特征[5-6]。包括KIRC在内的肾细胞癌被普遍认为是一种代谢性疾病。肾细胞癌的一个明显特征是代谢相关基因(metabolism-related genes,MRGs)发生表观遗传改变,导致有氧糖酵解、脂肪酸代谢和色氨酸谷氨酰胺利用等代谢功能异常[7]。异常MRGs表达是否可用于KIRC患者术后预后评估尚不明确。
本研究通过分析MRGs表达与KIRC患者预后间的关系,构建风险预后模型考察其对KIRC患者预后的预测价值,以期为KIRC临床诊断和治疗提供参考。
1 材料与方法 1.1 数据来源与处理利用癌症基因图谱(Cancer Genome Atlas Program, TCGA)数据库(https://portal.gdc.cancer.gov/)下载获取KIRC转录组RNA-seq (Workflow Type: HTSeq-counts)和相应患者临床资料,数据收录的时间截至2021年12月。收录的数据包含537个KIRC和71个癌旁组织的RNA-seq信息。目前已被证实的编码人代谢酶及相关转运体的MRGs共有2 752个[8]。从中比对并筛选KIRC组织中存在差异表达的MRGs,构建风险评分(riskscore)模型。
1.2 差异表达MRGs鉴定通过R软件limma程序包对TCGA数据KIRC数据标准化和差异分析,按照阈值为log2 fold change≥1和P < 0.05来筛选差异表达基因(differentially expressed genes,DEGs)。然后与MRGs取交集,获得差异表达的MRGs,绘制相应的热图和火山图。
1.3 富集分析利用clusterProfiler包对差异表达MRGs进行基因本体功能(gene ontology,GO)和京都基因与基因组百科全书通路(Kyoto Encyclopedia of Genes and Genomes pathway,KEGG pathway)富集分析,使用GOplot包进行结果可视化。
1.4 预后相关MRGs筛选及预后风险评分模型构建剔除TCGA数据库537例KIRC样本中病历及随访资料不完整样本,共有237例KIRC患者信息用于预后相关MRGs筛选及模型构建。纳入标准:①年龄≥18岁;②随访时间≥30 d;③病理组织学类型:肾透明细胞癌。患者临床病理特征见表 1。基于差异表达MRGs在237例KIRC患者癌组织中的表达量及对应生存数据,利用R语言survival包进行单因素Cox回归分析,以P < 0.001筛选出与KIRC预后相关MRGs,计算MRGs风险比(hazard ratio,HR)及95%CI。随后采用R语言glmet包进行LASSO回归分析进一步筛选预后相关MRGs,以避免单因素Cox回归分析可能存在过度拟合的情况。最后采用逐步多因素Cox回归计算每个靶基因的相关系数(β)建立风险评分模型。计算每例患者的风险评分,依据中位数临界值将患者分为低风险和高风险组。对两个亚组进行K-M生存分析绘制生存曲线,利用R语言survival和timeROC包计算1、3、5年总生存率的ROC曲线下面积(area under the curve,AUC)。
| 临床病理特征 | 例数 |
| 年龄 | |
| <60岁 | 103 |
| ≥60岁 | 134 |
| 性别 | |
| 男性 | 146 |
| 女性 | 91 |
| Grade分级 | |
| 1 | 2 |
| 2 | 101 |
| 3 | 97 |
| 4 | 37 |
| Stage分期 | |
| Ⅰ | 96 |
| Ⅱ | 32 |
| Ⅲ | 66 |
| Ⅳ | 43 |
| T分期 | |
| 1 | 100 |
| 2 | 40 |
| 3 | 90 |
| 4 | 7 |
| N分期 | |
| 0 | 223 |
| 1 | 14 |
| M分期 | |
| 0 | 195 |
| 1 | 42 |
| 生存状态 | |
| 存活 | 147 |
| 死亡 | 90 |
1.5 Hub基因的验证 1.5.1 细胞培养
人肾透明细胞癌细胞系A498以及人肾小管上皮细胞HK-2,均购自美国典型培养物保藏中心。用含10%FBS的RPMI DMEM培养液中培养,取对数生长期细胞,胰酶消化收集细胞。
1.5.2 主要仪器和试剂Nanodrop2000微量分光光度计(Thermo)、普通PCR仪(Bio-rad)、实时荧光定量PCR仪(ABI 7500)、高速离心机(Thermo);RNAiso Plus(TaKaRa, 9109)、PrimeScript RT reagent Kit with gDNA Eraser (TaKaRa, RR047A)、TB Green Premix Ex Taq(TaKaRa, RR820A)。
1.5.3 总RNA提取与定量PCR利用TRIzol法提取总RNA,Nanodrop2000微量分光光度计检测总RNA浓度及纯度,取吸光度D(260)/D(280)在1.8~2.0的总RNA样本,置-80 ℃保存。取2 μg总RNA样本,去除基因组DNA并反转录成cDNA,样本置-80 ℃保存。PCR引物由擎科生物公司合成,引物序列见表 2。PCR反应程序为预变性:94 ℃ 5 min;循环反应:(95 ℃ 10 s;60 ℃ 30 s)×40;溶解曲线:95 ℃ 15 s,60 ℃ 60 s,95 ℃ 15 s。基因相对表达水平采取2-△△Ct相对定量法计算。
| 基因 | 引物序列(5′→3′) | 产物大小/bp |
| ATP2A1 | 上游:CGGCGGTGTGATGATGACT 下游:CTTTGCACTGACTTGCGGTC |
235 |
| CHST6 | 上游:CTTGGTCACCGCCAACTCT 下游:CTGCAACGCTGATCACAAATC |
296 |
| PLA2G4D | 上游:ACTACTCCCTATCTGCGCCC 下游:CATGTTGGACAGGGTGTAGGG |
296 |
| KCNH1 | 上游:GGGTAGCAGCCAGAGGGA 下游:GGCCAGTCCACTATCTGAGC |
279 |
| KCNT1 | 上游:ACCGGACCTTCGAGTTTGAC 下游:GGAGAAGTTGAACAGCCGGA |
149 |
| CACNA1I | 上游:CATCCTGGTCAACACCGTCA 下游:TCAGGATCATCTCCAGGGCA |
122 |
| CNGB1 | 上游:TGACTCCCCTGGGATATGTGA 下游:AGCTCTCCCAGATTCCCTTCT |
87 |
1.6 预后模型验证
结合KIRC患者的年龄、性别、病理分期等临床相关信息,通过单因素及多因素Cox回归分析评价预后风险评分模型,以验证riskscore是否独立于其他临床因素,作为独立的预后预测因子。
1.7 统计学分析数据以x±s表示,两组间比较采用t检验,代谢相关基因与KIRC患者预后的关系采用Cox回归分析,KIRC患者风险评分与预后的关系采用Log-rank检验,并进行生存分析。P<0.05表示差异有统计学意义。
2 结果 2.1 差异表达MRGs的筛选通过用R语言EdgeR包进行差异分析,与已证实的2 752个代谢相关基因对比,在KIRC与癌旁组织间有875个MRGs存在差异表达(图 1A),其中分别有375和500个MRGs表达在KIRC癌组织表达上调和下调(图 1B)。
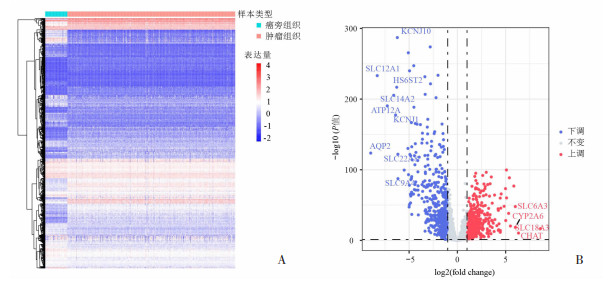
|
| A:差异表达MRGs热图;B:差异表达MRGs火山图 图 1 代谢相关基因在癌与癌旁组织中的差异表达 |
2.2 差异代谢相关基因的富集分析结果
GO富集分析结果显示,875个差异表达MRGs主要富集在小分子分解、脂肪酸、羧酸分解、有机酸运输、钠离子跨膜转运等生物过程(biological process, BP),以及转运蛋白及离子通道复合物等细胞组分(cell component, CC)。在分子功能(molecular function, MF)方面,主要参与了金属离子跨膜转运、离子通道、门控通道等活性调节(图 2A)。KEGG通路富集分析结果显示,差异表达MRGs主要与视黄醇及花生四烯酸代谢、胆汁分泌、糖酵解/糖异生、氨基酸生物合成等通路相关(图 2B)。
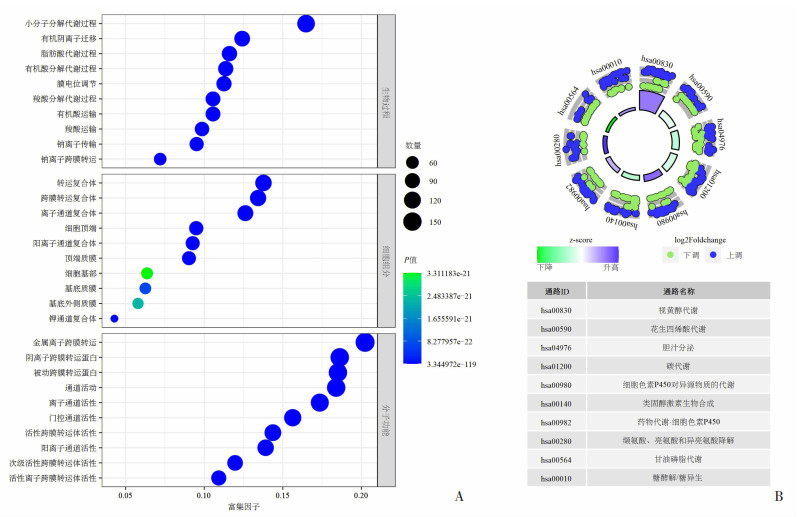
|
| A:GO富集分析的气泡图;B:KEGG通路分析的圈图 图 2 875个差异表达代谢相关基因GO和KEGG富集分析结果 |
2.3 预后相关代谢基因的筛选及预测模型构建
为减少采样偏倚,将总生存期(OS)或随访时间超出3个月的KIRC患者纳入分析。通过单因素Cox回归分析,以P < 0.001为筛选条件,共发现38个与预后相关的MRGs(表 3)。采用LASSO回归对筛选出来的MRGs进行再次过滤(图 3A),并使用交叉验证(图 3B)。最终,通过逐步多因素Cox回归分析进一步筛选得到7个与患者预后相关的风险基因(P < 0.001,图 3),构建风险评分预测模型:risk score=CNGB1表达量×0.130 3+CACNA1I×0.244 2+KCNT1×0.290 7+KCNH1×0.968 2+PLA2G4D×1.093 2+CHST6×1.842 6+ATP2A1×2.835 7。
| 基因名称 | r | HR | 95%CI | P | 基因名称 | r | HR | 95%CI | P | |
| CYP51A1 | -0.279 | 0.757 | 0.652~0.879 | < 0.001 | KCNH1 | 2.208 | 9.098 | 3.499~23.66 | < 0.001 | |
| CACNB1 | 0.298 | 1.347 | 1.214~1.495 | < 0.001 | GGTLC1 | -2.581 | 0.076 | 0.019~0.307 | < 0.001 | |
| CNGB1 | 1.258 | 3.518 | 2.043~6.059 | < 0.001 | GDPD5 | 0.121 | 1.129 | 1.060~1.202 | < 0.001 | |
| TRPM3 | -0.206 | 0.814 | 0.744~0.890 | < 0.001 | PLA2G4D | 0.262 | 1.300 | 1.155~1.463 | < 0.001 | |
| P2RX5 | 0.339 | 1.404 | 1.196~1.648 | < 0.001 | CBS | 1.047 | 2.848 | 1.921~4.221 | < 0.001 | |
| L2HGDH | -0.124 | 0.884 | 0.830~0.941 | < 0.001 | CYP3A7 | -0.390 | 0.677 | 0.564~0.812 | < 0.001 | |
| XYLB | -0.167 | 0.846 | 0.777~0.921 | < 0.001 | TTYH1 | 0.311 | 1.364 | 1.159~1.606 | < 0.001 | |
| CACNA1I | 1.750 | 5.756 | 3.025~10.955 | < 0.001 | TRPV3 | 0.744 | 2.103 | 1.592~2.778 | < 0.001 | |
| KCNT1 | 3.103 | 22.266 | 6.841~72.474 | < 0.001 | ADAL | -0.205 | 0.815 | 0.728~0.912 | < 0.001 | |
| SLC25A2 | 0.923 | 2.517 | 1.566~4.046 | < 0.001 | CHRNA5 | 0.395 | 1.485 | 1.246~1.770 | < 0.001 | |
| SLC12A5 | 0.525 | 1.691 | 1.304~2.193 | < 0.001 | KCNAB3 | 0.381 | 1.463 | 1.228~1.743 | < 0.001 | |
| AANAT | 0.531 | 1.700 | 1.307~2.211 | < 0.001 | CATSPER1 | 0.321 | 1.378 | 1.205~1.576 | < 0.001 | |
| SLC7A10 | 0.452 | 1.572 | 1.259~1.962 | < 0.001 | B3GNTL1 | 0.384 | 1.468 | 1.278~1.686 | < 0.001 | |
| SLC26A10 | 0.483 | 1.620 | 1.314~1.998 | < 0.001 | KCNJ14 | 0.333 | 1.396 | 1.19~1.637 | < 0.001 | |
| SLC9A5 | 0.380 | 1.463 | 1.221~1.752 | < 0.001 | CHST6 | 0.273 | 1.313 | 1.129~1.528 | < 0.001 | |
| SLCO5A1 | 1.183 | 3.265 | 1.732~6.154 | < 0.001 | ST6GALNAC3 | -0.121 | 0.886 | 0.828~0.948 | < 0.001 | |
| RDH16 | 0.488 | 1.630 | 1.288~2.061 | < 0.001 | ATP2A1 | 0.238 | 1.269 | 1.120~1.438 | < 0.001 | |
| CACNA1A | 0.582 | 1.790 | 1.461~2.193 | < 0.001 | ACSM4 | 0.712 | 2.038 | 1.455~2.856 | < 0.001 | |
| CACNG8 | 0.547 | 1.728 | 1.348~2.216 | < 0.001 | ACAD11 | -0.141 | 0.868 | 0.810~0.931 | < 0.001 |
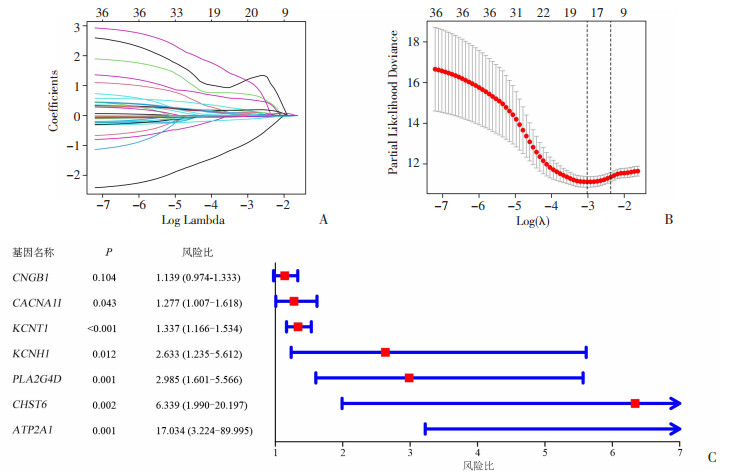
|
| A:LASSO筛选预后相关MRGs;B:LASSO回归交叉验证结果;C:逐步多因素Cox回归分析的森林图 图 3 预后风险MRGs筛选 |
2.4 RT-qPCR验证
为了进一步验证数据挖掘结果,通过RT-qPCR验证了预后相关代谢基因在人肾透明细胞癌细胞系A498与人肾小管上皮细胞HK-2中的表达水平。结果显示:ATP2A1、PLA2G4D、CNGB1在A498细胞中表达显著高于HK-2细胞(P < 0.01),CHST6表达轻微上调,但无统计学意义(P>0.05,图 4)。本研究未检测到KCNH1、CACNA1I、KCNT1的表达,可能与其在上述两种细胞中表达水平较低有关。

|
| a:P < 0.01 图 4 RT-qPCR验证4个预后关键基因在肾透明细胞癌A498细胞及HK-2细胞中的mRNA表达 |
2.5 预后模型评价
基于风险评分模型计算KIRC患者风险评分,以风险评分中位值为cutoff值,将患者分成高风险组和低风险组(图 5A、B)。采用Log-rank检验对两组患者进行K-M分析,结果显示两组之间的总生存预后差异显著(P<0.05,图 5D)。以AUC来评估风险评分模型预测效能(图 5E),该风险评分模型预测患者1、3和5年总生存率的AUC分别为0.728、0.775和0.792,说明该模型具有良好的预测能力。
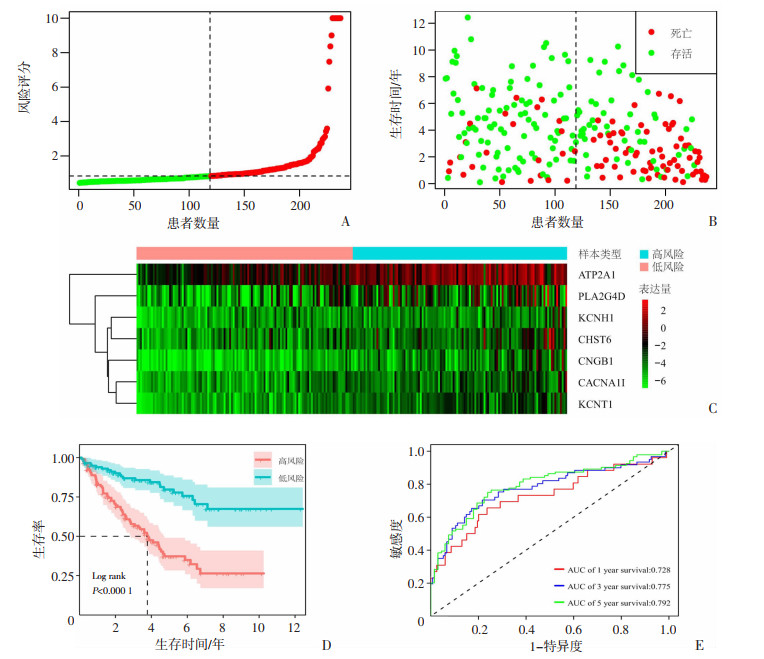
|
| A:风险曲线图 黑色虚线将患者分为低、高风险2组;B:随访终点低、高风险组患者生存状态;C:低、高风险KIRC患者中风险基因的表达热图;D:低、高风险组患者生存曲线;E:KIRC患者1、3、5年生存预测的ROC曲线 图 5 风险评分模型的建立与评价 |
2.6 预后模型的临床应用
为验证上述风险评分能否作为预测KIRC患者预后的独立因子,联合患者临床特征及风险评分进行Cox回归分析。单因素Cox分析结果显示,患者肿瘤分级、TNM分期和risk score均与KIRC患者总生存期显著相关(P<0.05,图 6A)。然而多因素Cox分析结果显示,仅有肿瘤M分期和risk score与KIRC患者总生存期显著相关,可作为患者预后的独立风险因子(图 6B),但risk score与患者的不良预后关系更密切(HR=4.671,P < 0.001)。
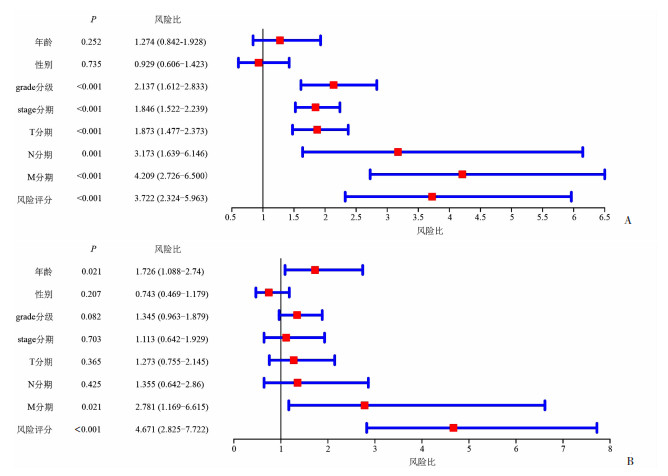
|
| A:单因素Cox回归分析;B:多因素Cox回归分析 图 6 患者临床参数Cox回归分析的森林图 |
3 讨论
准确预测癌症患者预后和风险分级关乎治疗方案的选择,更对治疗效果有直接影响。临床上通常将肾癌分为肾透明细胞型肾癌(KIRC)、肾颗粒细胞型肾癌、肾混合细胞型肾癌、肾未分化细胞型肾癌4种类型。KIRC是恶性程度较低的一种肾癌,但是KIRC通过血道途径转移的概率极高,约有60%的患者存在转移可能[2]。肿瘤细胞为了适应其对能量和物质的需求,通常会改变其代谢特征,代谢相关基因表达水平异常、突变、异位等引起肿瘤代谢异常,通过研究代谢相关基因的变化来判断肿瘤的特征和预后被越来越多的人所重视[9]。
本研究采用生物信息学方法筛选TCGA数据库KIRC患者和对照样本的基因转录组表达数据,找到875个有差异表达的代谢相关基因。通过Cox回归结合LASSO回归分析,最终筛选出其中7个基因建立了风险评分模型。7个基因风险评分是KIRC患者总生存期的独立危险因素,可用于KIRC患者预后判断。
CNGB1基因编码环核苷酸门控通道(CNGs)β1亚基。CNGs属于电压门控离子通道超家族,通过控制阳离子流入来响应信号诱导的胞内cGMP或cAMP水平改变,是信号转导的关键组成部分[10]。文献报道[11]侵袭性肉瘤样膀胱癌携带CNGB1的频繁突变,且CNGB1异常表达与肌肉浸润性膀胱癌化疗敏感性密切相关。
离子通道参与了几乎所有的细胞过程,在细胞增殖、分化和凋亡调控中发挥关键作用,其表达及功能异常与癌细胞的恶性程度有关[12]。CACNA1I基因编码T型电压门控钙通道α1亚单位,在调控钙离子选择孔形成中发挥重要作用。T型钙通道参与细胞增殖和分化,在大多数肿瘤中过表达并参与钙介导的细胞死亡,已成为一种癌症治疗的潜在靶点。CACNA1I在乳腺癌、肉瘤和食管癌等多种肿瘤中的表达显著上调,在肾癌中的作用机制尚不清楚[13]。KCNT1基因编码钠激活型钾通道蛋白KCa4.1,在心脏、肾脏、睾丸及大脑中均有表达,但其在肾癌中的作用尚不清楚。
磷脂酶A2家族(phospholipase A2,PLA2)是体内前列腺素合成的限速酶。PLA2G4D是IVD亚型PLA2蛋白的编码基因,与精神分裂症、自闭症、银屑病和自发性早产有关[14-15]。PLA2G4D高表达与肺鳞癌的不良预后正相关,与其他肿瘤相关的研究较少[16]。CHST6基因位于人染色体16q22,编码碳水化合物磺基转移酶(carbohydrate sulfotransferases,CHST),分布在高尔基体内腔膜上。CHST6参与细胞糖酵解过程,负责催化硫酸角质素中葡糖胺的硫酸化,其位点突变与角膜营养不良及相关病变相关[17]。
ATP2A1是肌浆网ATP酶家族(sarcoplasmic reticulum Ca2+ATPases,SERCA)的3个编码基因之一。胞内钙稳态与细胞生存、凋亡和自噬调控密切相关,SERCA负责将胞浆内的Ca2+泵出胞外或储存在肌浆网内,对维持细胞钙稳态尤为关键[18]。SERCA表达异常与细胞癌变及凋亡抑制密切相关。SERCA在多种癌细胞中过表达,姜黄素等SERCA抑制剂可抑制其活性导致胞内Ca2+浓度增加,进而促进卵巢癌等肿瘤细胞发生凋亡[19-20]。
目前,对于风险评分预后模型中的7个基因与肿瘤相关文献报道较少,仅有KCNH1基因表达上调与肾癌化疗耐药的报道,而CNGB1、CACNA1I、KCNT1、PLA2G4D、CHST6和ATP2A1在肾癌病情进展中的作用及对KIRC患者预后的影响均未见报道。PLA2G4D、CHST6和ATP2A1是对KIRC预后影响最大的3个代谢相关基因,这些基因在KIRC发生发展中的分子机制非常值得深入研究。
综上所述,本研究基于TCGA数据库,采用生物信息学方法分析代谢相关基因对KIRC患者预后的影响。对研究代谢相关基因在KIRC进展过程中的作用及机制,有一定的潜在参考价值。同时建立了一个具有良好预测性能的7个基因特征相关风险评分预测模型,可作为KIRC患者预后评估的独立指标,为KIRC患者预后评估及个性化治疗方案制定提供参考。这7个风险基因及相关信号通路在KIRC发生发展中的具体作用机制尚未阐明,需要进一步研究和验证。
| [1] |
SIEGEL R L, MILLER K D, FUCHS H E, et al. Cancer statistics, 2022[J]. CA Cancer J Clin, 2022, 72(1): 7-33. |
| [2] |
MOTZER R J, JONASCH E, AGARWAL N, et al. Kidney cancer, Version 2.2017, NCCN Clinical Practice Guidelines in Oncology[J]. J Natl Compr Canc Netw, 2017, 15(6): 804-834. |
| [3] |
DONG D, MU Z Y, WEI N, et al. Long non-coding RNA ZFAS1 promotes proliferation and metastasis of clear cell renal cell carcinoma via targeting miR-10a/SKA1 pathway[J]. Biomed Pharmacother, 2019, 111: 917-925. |
| [4] |
LJUNGBERG B, ALBIGES L, ABU-GHANEM Y, et al. European Association of Urology guidelines on renal cell carcinoma: The 2019 Update[J]. Eur Urol, 2019, 75(5): 799-810. |
| [5] |
VANDER HEIDEN M G, DEBERARDINIS R J. Understanding the intersections between metabolism and cancer biology[J]. Cell, 2017, 168(4): 657-669. |
| [6] |
MARTÍNEZ-REYES I, CHANDEL N S. Cancer metabolism: looking forward[J]. Nat Rev Cancer, 2021, 21(10): 669-680. |
| [7] |
LUCARELLI G, LOIZZO D, FRANZIN R, et al. Metabolomic insights into pathophysiological mechanisms and biomarker discovery in clear cell renal cell carcinoma[J]. Expert Rev Mol Diagn, 2019, 19(5): 397-407. |
| [8] |
POSSEMATO R, MARKS K M, SHAUL Y D, et al. Functional genomics reveal that the serine synthesis pathway is essential in breast cancer[J]. Nature, 2011, 476(7360): 346-350. |
| [9] |
YIN D, XU Y, ZHEN Y J, et al. Relationship between the methylation of folic acid metabolism-related genes and the incidence and prognosis of esophageal cancer among ethnic kazakhs[J]. J Cancer, 2018, 9(16): 2865-2875. |
| [10] |
NASSISI M, SMIRNOV V M, HERNANDEZ C S, et al. CNGB1-related rod-cone dystrophy: A mutation review and update[J]. Hum Mutat, 2021, 42(6): 641-666. |
| [11] |
HEPBURN A C, LAZZARINI N, VEERATTERAPILLAY R, et al. Identification of CNGB1 as a predictor of response to neoadjuvant chemotherapy in muscle-invasive bladder cancer[J]. Cancers, 2021, 13(15): 3903. |
| [12] |
PREVARSKAYA N, SKRYMA R, SHUBA Y. Ion channels in cancer: are cancer hallmarks oncochannelopathies?[J]. Physiol Rev, 2018, 98(2): 559-621. |
| [13] |
PHAN N N, WANG C Y, CHEN C F, et al. Voltage-gated calcium channels: Novel targets for cancer therapy[J]. Oncol Lett, 2017, 14(2): 2059-2074. |
| [14] |
LIU G J, HE J R, KUANG Y S, et al. Associations of maternal PLA2G4C and PLA2G4D polymorphisms with the risk of spontaneous preterm birth in a Chinese population[J]. Mol Med Rep, 2017, 15(6): 3607-3614. |
| [15] |
TEN BERGEN L L, PETROVIC A, AAREBROT A K, et al. Current knowledge on autoantigens and autoantibodies in psoriasis[J]. Scand J Immunol, 2020, 92(4): e12945. |
| [16] |
KE D, GUO Q, FAN T Y, et al. Analysis of the role and regulation mechanism of hsa-miR-147b in lung squamous cell carcinoma based on the Cancer Genome Atlas Database[J]. Cancer Biother Radiopharm, 2021, 36(3): 280-291. |
| [17] |
SAFARI I, BARADARAN-RAFⅡ A, ISSAZADEH-NAVIKAS S, et al. CHST6 mutations identified in Iranian MCD patients and CHST6 mutations reported worldwide identify targets for gene editing approaches including the CRISPR/Cas system[J]. Int Ophthalmol, 2020, 40(9): 2223-2235. |
| [18] |
CHEMALY E R, TRONCONE L, LEBECHE D. SERCA control of cell death and survival[J]. Cell Calcium, 2018, 69: 46-61. |
| [19] |
COGHI P, NG J P L, KADIOGLU O, et al. Synthesis, computational docking and biological evaluation of celastrol derivatives as dual inhibitors of SERCA and P-glycoprotein in cancer therapy[J]. Eur J Med Chem, 2021, 224: 113676. |
| [20] |
CHRISTODOULOU P, YIALLOURIS A, MICHAIL A, et al. Altered SERCA expression in breast cancer[J]. Medicina (Kaunas), 2021, 57(10): 1074. |




