2. 400054 重庆,重庆市第七人民医院检验科
2. Department of Clinical Laboratory, the Seventh People's Hospital of Chongqing, Chongqing, 400054, China
解脲脲原体(ureaplasma urealyticum,Uu)是引起人类泌尿生殖系统感染的主要病原体之一,可单独感染,也可与人型支原体混合感染[1]。它与非淋菌性尿道炎、泌尿结石、不孕不育、不良妊娠结局、早产及产后新生儿肺发育不良等疾病密切相关[2-3]。Uu常侵犯尿道、前庭大腺及宫颈, 可引起相关部位炎症;上行感染时, 可引起输卵管炎、子宫内膜炎以及盆腔炎等, 尤其以输卵管炎最多见,均可导致女性不孕[4]。男性不育患者的精液Uu检出率是5%~58%,Uu在男性下生殖道的定植被认为是男性不育症的主要原因之一[5]。国内研究发现不孕夫妇中男性和女性患者感染的Uu血清型分布有显著差异,且Uu有可能在夫妇之间传播。
Uu分为UPA和UUR两个生物群,共14个血清型。目前临床上常用于Uu感染治疗的抗生素主要包括氟喹诺酮类和四环素类。近年来,关于各种抗生素的耐药性均有报道,氟喹诺酮类药物作为临床一线用药,耐药率的增长尤其明显[6]。耐药机制主要由拓扑异构酶gyrA、gyrB、parC和parE等基因的突变引起[7-8],多种病原体的gyrA、gyrB、parC和parE碱基突变所致氨基酸变异与氟喹诺酮类药物耐药相关[9]。
目前关于Uu对氟喹诺酮类药物耐药率和耐药机制的研究发现,不同地区、不同人群之间差异较大[10],随着时间的推移,耐药谱和耐药率也在动态变化,特别是针对不孕不育人群感染Uu的研究非常有限,参考既往研究进行经验用药存在一定的局限性。本研究通过对不孕不育患者的临床菌株进行拓扑异构酶基因gyrA、gyrB、parC和parE的QRDR进行扩增与测序,探讨这4个基因与氟喹诺酮类药物耐药机制的关系,可初步掌握本地区不孕不育人群Uu的耐药率、耐药谱和耐药机制等流行病学资料,对于指导临床合理用药具有重要意义。
1 材料与方法 1.1 材料 1.1.1 主要试剂解脲脲原体和人型支原体选择分离培养药敏试剂盒、解脲脲原体和人型支原体选择分离固体培养基及支原体肉汤基础均购自众爱生河北生物科技有限公司,药敏试验测试板购自天津市金章科技发展有限公司,DNA提取试剂盒购自北京天根生化科技有限公司,PCR试剂购自上海生工生物工程有限公司。
1.1.2 标准菌株解脲脲原体标准菌株ATCC 27618由众爱生河北生物科技有限公司惠赠。
1.2 方法 1.2.1 临床资料解脲脲原体的筛选标本来自2019年12月至2022年1月就诊于重庆医科大学第一附属医院生殖中心的不孕不育患者。其中男性898名,女性1 900名。男性取尿道分泌物,女性取阴道分泌物,采用支原体鉴定及药敏检测试剂盒进行初步鉴定及药敏。液体及固体培养基变成樱桃红色,同时在显微镜下观测到固体培养基上有褐色海胆样菌落判定为阳性,挑取单个菌落进行纯培养。
1.2.2 菌株鉴定将纯化后的菌株复苏后用试剂盒提取DNA, 运用16SrRNA测序技术进行鉴定。引物[11]515f Modified (5′-GTGYCAGCMGCCGCGGTAA-3′),806r Modified(5′-GGACTACNVGGG-TWTCTAAT-3′),由上海生工生物工程股份有限公司合成。PCR反应产物送公司测序,测序结果经NCBI的GenBank数据库中BLAST与标准菌株进行比对。GenBank在线检索解脲脲原体ATCC标准菌株14种血清型的基因组序列,通过BLAST程序比对,发现共有47个UPA特异性基因和45个UUR特异性基因。UU295和UUR10_0588这两个基因分别在UPA和UUR中表现出非常保守的性质,能够区分出UPA、UUR及同时包含UPA和UUR的标本。根据这两个特异性基因,设计引物UU295-F/R和UUR10_0588-F/R用于区分解脲脲原体与微小脲原体[12],上下游引物分别是UU295-F(5′-GCCAAGAAAACATTTAATCGCT-3′),UU295-R(5′-CTGATATTGTCCGCTGCTCATT-3′), UUR10_0588-F(5′-AAAGTTAAAGAGTCTTGTGGA-3′),UUR10_0588-R(5′-AATAGGTAATAGCCTCTTTGAT-3′)。利用两对引物对184株临床菌株进行PCR扩增,产物用1%凝胶电泳进行检测,根据电泳有无条带进行鉴别。
1.2.3 最低抑菌浓度(minimum inhibitory concentration, MIC)的测定首先将纯化后的菌株采用颜色变化单位法测Uu的最大代谢活性,将剩余菌株置于-80 ℃冰箱备用。依据CLSI M43-A(2011版)标准,采用微量肉汤稀释法测定4种氟喹诺酮类药物对所有临床菌株的MIC值。将环丙沙星、左氧氟沙星、司帕沙星和莫西沙星倍比稀释成12个浓度(0.25~128 μg/mL),同时设计两孔做阴性和阳性对照,由天津市金章科技发展有限公司完成药物的包被和冻干。阴性对照加100 μL的支原体液体培养基,其余各孔分别加100 μL含104CFU/mL的Uu培养物,每个批次都加做标准菌株ATCC 27618作为质控。每孔滴加一滴液态石蜡,加盖后置含5%CO2的35 ℃孵箱孵育48 h,由黄色变为樱桃红为阳性,观测并记录结果。药敏结果根据CLSI M43-A(2011版)进行判读,左氧氟沙星≥4 μg/mL为耐药,莫西沙星≥4 μg/mL为耐药,环丙沙星和司帕沙星没有折点。
1.2.4 PCR扩增拓扑异构酶基因扩增测序针对解脲脲原体gyrA、gyrB、parC、parE基因的QRDR设计引物[13]。PCR扩增反应条件:94 ℃预变性5 min,94 ℃变性30 s,50 ℃退火30 s,72 ℃延伸1 min,构成35循环。扩增产物送上海生工生物工程有限公司进行测序。
1.2.5 基因序列及氨基酸突变分析测序结果采用DNASTAR Lasergene分析并与解脲脲原体标准菌株进行比对。根据文献[14]标准菌株为:UPA采用ATCC 700970(GenBank AF222894),UUR采用ATCC 33699(GenBank CP001184.1)。找出gyrA、gyrB、parC、parE 4个基因的碱基突变位点及相应的氨基酸改变。
1.2.6 同源建模使用Swiss-Model同源建模服务器(http://swissmodel.expasy.org/),用一株同时出现parC S83L与gyrB R523E双突变的58号高耐药菌株,以喹诺酮类药物(左氧氟沙星)-肺炎链球菌Ⅳ型拓扑异构酶DNA裂解复合物(PDB代码3RAE)作为模板,进行同源建模,来模拟解脲脲原体的拓扑异构酶基因parC和gyrB的突变模型。
2 结果 2.1 解脲脲原体的生物群分布与氟喹诺酮类药物的敏感性分析共分离到临床菌株184株,其中9株(4.89%)来自男性尿道分泌物,175株(95.11%)来自女性阴道分泌物。173株属于UPA(94.02%),8株为UUR(4.35%),3株同时包含UPA和UUR(1.63%)。在173株UPA临床菌株中,环丙沙星、左氧氟沙星、司帕沙星及莫西沙星的MIC90分别是64、8、8、2 μg/mL。UPA、UUR、UPA+UUR 3组菌株对左氧氟沙星的耐药率分别是80.4%、50%、66.7%,对莫西沙星耐药率依次是2.2%、0、33.3%(表 1)。
| Uu菌株和抗菌药物 | MIC(μg/mL) | 耐药率(%) | ||
| 范围 | 50% | 90% | ||
| UPA(菌株数n=173) | ||||
| 环丙沙星 | 0.5~128 | 32 | 64 | - |
| 左氧氟沙星 | 0.25~64 | 4 | 8 | 80.4 |
| 司帕沙星 | 0.25~16 | 4 | 8 | - |
| 莫西沙星 | 0.25~16 | 1 | 2 | 2.2 |
| UUR(菌株数n=8) | ||||
| 环丙沙星 | 4~64 | 16 | 64 | - |
| 左氧氟沙星 | 1~8 | 4 | 8 | 50.0 |
| 司帕沙星 | 0.5~4 | 2 | 4 | - |
| 莫西沙星 | 0.5~2 | 1 | 2 | 0 |
| UPA+UUR(菌株数n=3) | ||||
| 环丙沙星 | 8~128 | 16 | 128 | - |
| 左氧氟沙星 | 1~64 | 2 | 64 | 66.7 |
| 司帕沙星 | 0.5~16 | 2 | 16 | - |
| 莫西沙星 | 0.5~16 | 1 | 16 | 33.3 |
2.2 解脲脲原体拓扑异构酶QRDR碱基突变与对应的氨基酸变异
将184株解脲脲原体临床菌株测序结果与标准菌株的理论基因进行比对,结果显示4株gyrA基因发生L176F变异导致亮氨酸变为苯丙氨酸,变异率为2.2%;3株发生S174Q变异导致丝氨酸变为精氨酸,变异率为1.6%;21株gyrB基因发生R523E变异导致精氨酸变为苯丙氨酸,变异率为11.4%;157株parC基因发生S83L变异导致丝氨酸变为亮氨酸,变异率为85.3%;13株发生L127P突变,导致亮氨酸变为脯氨酸,变异率为7.1%;2株parE基因发生L408I变异导致亮氨酸变为异亮氨酸,为新发现的突变,变异率为1.1%(表 2)。
| 蛋白 | 位点 | 碱基突变 | 氨基酸变异 | 变异株(n) | 变异率(%) |
| gyrA | 176 | TTA→TTC | L→F | 4 | 2.2 |
| 174 | AGT→CAA | S→Q | 3 | 1.6 | |
| gyrB | 523 | AGA→GAA | R→E | 21 | 11.4 |
| parC | 83 | TCA→TTA | S→L | 157 | 85.3 |
| 127 | CTC→CCC | L→P | 13 | 7.1 | |
| parE | 408 | CTT→ATT | L→I | 2 | 1.1 |
| L:亮氨酸;F:苯丙氨酸;S:丝氨酸;Q:谷氨酰胺;R:精氨酸;E:谷氨酸;P:脯氨酸;I:异亮氨酸 | |||||
2.3 QRDR氨基酸变异与氟喹诺酮类药物的MIC值的相关性
根据测序结果对184株Uu临床菌株gyrA、gyrB、parC、parE的QRDRs扩增产物的碱基和氨基酸序列与标准菌株进行比对,在gyrA蛋白的112位置、parC的125位置及parE的417位置发现氨基酸变异,但有文献报道这些位点与物种特异性或血清型特异的基因多态性有关[15]。排除这些干扰,总结了所测临床菌株的氟喹诺酮类药物的MIC值与QRDR中对应的氨基酸变化(表 3)。可以看出,对左氧氟沙星耐药的解脲脲原体菌株均发生了parC S83L的变异,其变异率为85.3%。parC S83L的变异分别导致解脲脲原体对左氧氟沙星MIC90从1 μg/mL升高至8 μg/mL,对环丙沙星MIC90从4 μg/mL升高至64 μg/mL,莫西沙星MIC90从0.5 μg/mL升高至2 μg/mL;11株同时发生parC S83L与parC L127P变异的菌株导致环丙沙星和左氧氟沙星MIC50分别由16、2 μg/mL升高至32、4 μg/mL;21株同时发生parC S83L与gyrB R523E双突变的菌株相较于单独发生parC S83L变异的菌株左氧氟沙星MIC50由2 μg/mL升高至4 μg/mL。有2株同时发生parC S83L、parC L127P和gyrB R523E 3个氨基酸变异的菌株。
| 菌株 | 数量 | 环丙沙星 | 左氧氟沙星 | 司帕沙星 | 莫西沙星 | gyrA | gyrB | parC | parE | |||||||||||
| 50% | 90% | Rang | 50% | 90% | Rang | 50% | 90% | Rang | 50% | 90% | Rang | |||||||||
| 标准菌株ATCC 27618 | 2 | 1 | 0.5 | 0.5 | N | N | N | N | ||||||||||||
| UPA | 1 | 32 | 4 | 2 | 1 | L176F, S174Q | S83L | L408I* | ||||||||||||
| 2 | 64 | 8 | 8 | 1 | L176F | S83L | ||||||||||||||
| 1 | 16 | 4 | 4 | 1 | L176F, S174Q | R523E | S83L | |||||||||||||
| 1 | 64 | 4 | 4 | 2 | S174R | S83L | L408I* | |||||||||||||
| 21 | 32 | 64 | 32~128 | 4 | 8 | 4~8 | 2 | 8 | 2~8 | 1 | 2 | 1~2 | R523E | S83L | ||||||
| 2 | 16 | 4 | 4 | 1 | R523E | S83L, L127P | ||||||||||||||
| 112 | 16 | 64 | 8~64 | 2 | 8 | 1~32 | 2 | 8 | 2~8 | 1 | 2 | 1~8 | S83L | |||||||
| 11 | 32 | 64 | 16~64 | 4 | 8 | 2~8 | 4 | 4 | 2~4 | 1 | 1 | 1~2 | S83L, L127P | |||||||
| 22 | 2 | 4 | 1~4 | 0.5 | 1 | 0.25~2 | 0.25 | 0.5 | 0.25~2 | 0.25 | 0.5 | 0.25~1 | N | N | N | N | ||||
| UUR | 3 | 16 | 64 | 16~64 | 4 | 8 | 4~8 | 2 | 4 | 2~4 | 1 | 2 | 1~2 | S83L | ||||||
| 5 | 4 | 16 | 4~16 | 1 | 2 | 1~4 | 0.5 | 2 | 0.5~2 | 0.5 | 1 | 0.5~1 | N | N | N | N | ||||
| UPA+UUR | 2 | 8 | 128 | 8~128 | 1 | 64 | 1~64 | 0.5 | 16 | 0.5~16 | 0.5 | 16 | 0.5~16 | S83L | ||||||
| 1 | 64 | 4 | 2 | 1 | R523E | S83L | ||||||||||||||
| *:新发现突变 | ||||||||||||||||||||
2.4 对拓扑异构酶基因突变parC S83L及gyrB R523E进行同源建模
根据测序结果,发现184份解脲脲原体培养阳性标本的QRDRs中存在最多的氨基酸替换是parC S83L,丝氨酸(TCA)变为亮氨酸(TTA)(157份);其次是gyrB R523E,精氨酸(AGA)到谷氨酸(GAA)(21份)。在58号样本中同时检测到parC S83L与gyrB R523E的双突变,4种喹诺酮药物MIC值分别为环丙沙星128 μg/mL、左氧氟沙星64 μg/mL、司帕沙星16 μg/mL、莫西沙星16 μg/mL。拓扑异构酶QRDR的氨基酸序列比对如图 1所示,为了评价ParC S83L和gyrB R523E的氨基酸替换的结构重要性,对标准菌株ATCC 700970和58号临床高耐药菌株进行同源建模。发现喹诺酮的C-3羧酸和C-4羰基以及二价镁离子对拓扑异构酶复合物的结合作用很重要。从模型中可以看出,S83L的侧链占据了左氧氟沙星C-3羧酸的喹诺酮结合袋。这些侧链S83L突变会通过空间位阻干扰左氧氟沙星的正常结合,导致左氧氟沙星耐药。
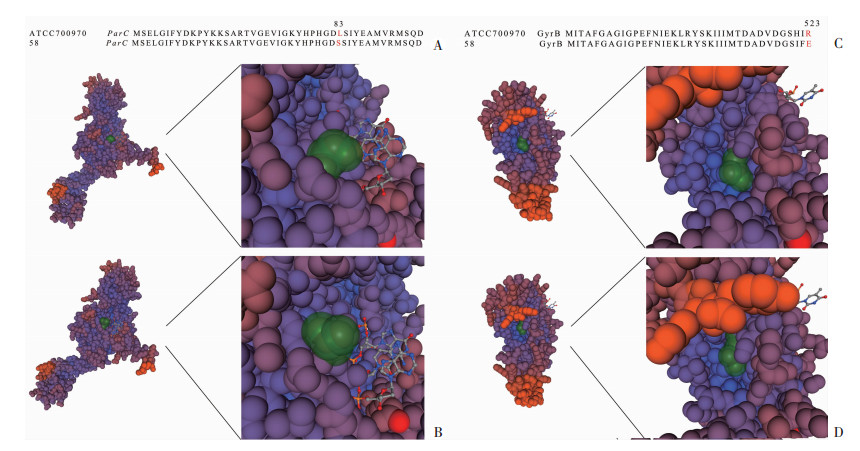
|
| A、C:分别为标准菌株ATCC 700970parC及gyrB的表面模型;B:58号临床菌株parC S83L突变通过同源建模后的表面模型;D:58号临床菌株gyrB R523E突变通过同源建模后的表面模型;绿色气泡为突变位点对应的氨基酸 图 1 标准菌株ATCC 700970与58号临床菌株QRDR突变的模型结构 |
3 讨论
本研究从不孕不育患者分离的Uu临床菌株对左氧氟沙星耐药率较高,对氟喹诺酮类药物耐药的主要机制是拓扑异构酶基因parC S83L突变,通过空间位阻干扰左氧氟沙星的正常结合。3组Uu的左氧氟沙星的耐药率均超过50%,UPA组耐药率超过80%。ZHAO等[16]对来自320名男性不育患者的临床Uu菌株进行分析,左氧氟沙星耐药率为47.5%,与本研究一致。在普通人群的研究中,YANG等[15]报告UPA及UUR对左氧氟沙星耐药率均超过80%;而FERNÁNDEZ等[17]报告来自美国明尼苏达州的250株临床菌株,左氧氟沙星耐药率为6.4%(UPA),5.2%(UUR);美国佛罗里达州报告[10]在大学生年龄女性人群中,左氧氟沙星的耐药率分别为1.6% (UPA)、0%(UUR);瑞士的研究[18]显示Uu对左氧氟沙星耐药率为9.7%;德国的研究报道[19]Uu对氟喹诺酮类药物的耐药率为7.1%。可能由于不同国家和地区抗生素管理政策,以及使用习惯不同导致了耐药率的差异。本研究显示莫西沙星耐药率较低,比环丙沙星、左氧氟沙星具有更强的抗解脲脲原体活性。司帕沙星同环丙沙星及左氧氟沙星同为第3代喹诺酮类药物,但目前临床上应用较少,耐药率较低,属于可供选择的药物。
氟喹诺酮类药物通过抑制微生物Ⅱ型拓扑异构酶活性阻碍DNA复制,对解脲脲原体发挥抗菌作用。Ⅱ型拓扑异构酶包括DNA促旋酶和拓扑异构酶Ⅳ。DNA促旋酶由gyrA和gyrB基因分别编码的两个A亚基和两个B亚基组成四聚体A2B2;拓扑异构酶Ⅳ由parC及parE基因编码而成的两个C亚基和两个E亚基组成四聚体C2E2[20]。解脲脲原体与氟喹诺酮类药物耐药相关的突变有gyrA 100和104位点,gyrB 462位点,parC 82、83、87、88位点,parE 457位点[21-23]。本研究中变异率最高的是parC基因83位点发生的碱基突变,导致丝氨酸(TCA)变异为亮氨酸(TTA)。parC S83L变异的112株Uu菌株对左氧氟沙星MIC值升高了8倍,环丙沙星MIC值升高了16倍。意大利的PICCINELLI等[24]报告在85株耐药菌株中发现10株parC S83L突变,1株parC E87Q突变。HAMASUNA等[25]发现解脲脲原体parC基因发生S83L突变,与莫西沙星耐药也密切相关。本研究发生parC S83L变异的112株Uu菌株导致莫西沙星MIC值增加了4~32倍;有3株莫西沙星耐药的菌株均发生了parC S83L的变异,莫西沙星的MIC值增加了32~64倍。本研究有11株parC S83L与parC L127P双突变,21株parC S83L与gyrB R523E双突变,相比parC S83L单独变异的菌株,左氧氟沙星MIC50升高2倍,提示gyrB R523E与parC L127P的氨基酸变异可能对氟喹诺酮类药物耐药有叠加的作用,目前没有关于这两个突变的功能研究报道,有待进一步证实。本研究还发现了1个新突变parE L408I,但并没有引起氟喹诺酮类药物耐药,可能为无义突变。22株对氟喹诺酮类药物均敏感的解脲脲原体菌株及标准菌株ATCC 27618则无任何氨基酸变异发生,表明QRDR氨基酸变异是氟喹诺酮类药物耐药的主要机制。用标准菌株ATCC 700970和58号临床高耐药菌株进行同源建模显示,parC S83L位于喹诺酮药物结合袋,突变会通过空间位阻干扰喹诺酮类药物的正常结合,导致喹诺酮类药物耐药。gyrB R523E突变的位点与喹诺酮药物结合袋距离较远,与氟喹诺酮类药物耐药的相关性还有待进一步研究。
综上所述,在不孕不育人群分离的解脲脲原体中,左氧氟沙星的敏感性明显下降,莫西沙星与司帕沙星有较好的抗菌活性。解脲脲原体对氟喹诺酮类药物的耐药主要与拓扑异构酶基因parC S83L突变导致的氨基酸变异密切相关,可以考虑针对该常见的突变位点设计杂交探针,从而快速检测解脲脲原体对氟喹诺酮类药物的耐药性。为预防和减少抗生素耐药,需要加强与临床沟通,用药前进行抗支原体抗生素敏感性试验,交替使用抗生素,注重Uu耐药监测[26],同时探索特异性新型支原体抗生素。
| [1] |
PAIRA D A, MOLINA G, TISSERA A D, et al. Results from a large cross-sectional study assessing Chlamydia trachomatis, Ureaplasma spp. and Mycoplasma hominis urogenital infections in patients with primary infertility[J]. Sci Rep, 2021, 11(1): 13655. |
| [2] |
KLETZEL H H, ROTEM R, BARG M, et al. Ureaplasma urealyticum: the role as a pathogen in women's health, a systematic review[J]. Curr Infect Dis Rep, 2018, 20(9): 33. |
| [3] |
KAWAI Y, NAKURA Y, WAKIMOTO T, et al. In vitro activity of five quinolones and analysis of the quinolone resistance-determining regions of gyrA, gyrB, parC, and parE in Ureaplasma parvum and Ureaplasma urealyticum clinical isolates from perinatal patients in Japan[J]. Antimicrob Agents Chemother, 2015, 59(4): 2358-2364. |
| [4] |
杨英杰, 谈笑. 解脲脲原体感染研究进展[J]. 医学信息, 2018, 31(3): 61-63. YANG Y J, TAN X. Advances in Ureaplasma urealyticum infection[J]. Med Inf, 2018, 31(3): 61-63. |
| [5] |
BEETON M L, PAYNE M S, JONES L. The role of Ureaplasma spp. in the development of nongonococcal urethritis and infertility among men[J]. Clin Microbiol Rev, 2019, 32(4): e00137-e00118. |
| [6] |
郭炽鹏, 陆春, 朱国兴. 解脲脲原体耐药性研究进展[J]. 微生物与感染, 2018, 13(4): 245-249. GUO C P, LU C, ZHU G X. Research progress on drug resistance of Ureaplasma urealyticum[J]. J Microbes Infect, 2018, 13(4): 245-249. |
| [7] |
SINGH P, JAIN A, DIXIT P, et al. Prevalence of gyrA and B gene mutations in fluoroquinolone-resistant and-sensitive clinical isolates of Mycobacterium tuberculosis and their relationship with MIC of ofloxacin[J]. J Antibiot (Tokyo), 2015, 68(1): 63-66. |
| [8] |
BEETON M L, CHALKER V J, KOTECHA S, et al. Comparison of full gyrA, gyrB, parC and parE gene sequences between all Ureaplasma parvum and Ureaplasma urealyticum serovars to separate true fluoroquinolone antibiotic resistance mutations from non-resistance polymorphism[J]. J Antimicrob Chemother, 2009, 64(3): 529-538. |
| [9] |
AVALOS E, CATANZARO D, CATANZARO A, et al. Frequency and geographic distribution of gyrA and gyrB mutations associated with fluoroquinolone resistance in clinical Mycobacterium tuberculosis isolates: a systematic review[J]. PLoS One, 2015, 10(3): e0120470. |
| [10] |
VALENTINE-KING M A, BROWN M B. Antibacterial resistance in Ureaplasma species and Mycoplasma hominis isolates from urine cultures in college-aged females[J]. Antimicrob Agents Chemother, 2017, 61(10): e01104-e01117. |
| [11] |
WALTERS W, HYDE E R, BERG-LYONS D, et al. Improved bacterial 16S rRNA gene (V4 and V4-5) and fungal internal transcribed spacer marker gene primers for microbial community surveys[J]. mSystems, 2016, 1(1): e00009-e00015. |
| [12] |
ZHANG J, KONG Y, FENG Y, et al. Development of a multilocus sequence typing scheme for Ureaplasma[J]. Eur J Clin Microbiol Infect Dis, 2014, 33(4): 537-544. |
| [13] |
BEBEAR C M, RENAUDIN H, CHARRON A, et al. In vitro activity of trovafloxacin compared to those of five antimicrobials against mycoplasmas including Mycoplasma hominis and Ureaplasma urealyticum fluoroquinolone-resistant isolates that have been genetically characterized[J]. Antimicrob Agents Chemother, 2000, 44(9): 2557-2560. |
| [14] |
KAMIYA Y, SHIMADA Y, ITO S, et al. Analysis of the quinolone-resistance determining region of the gyrA gene and the analogous region of the parC gene in Ureaplasma parvum and Ureaplasma urealyticum detected in first-void urine of men with non-gonococcal urethritis[J]. J Antimicrob Chemother, 2013, 68(2): 480-482. |
| [15] |
YANG T, PAN L L, WU N N, et al. Antimicrobial resistance in clinical Ureaplasma spp. and Mycoplasma hominis and structural mechanisms underlying quinolone resistance[J]. Antimicrob Agents Chemother, 2020, 64(6): e02560-e02519. |
| [16] |
ZHAO L H, LIU A H, LI R Y, et al. Antimicrobial resistance, genetic characterization, and molecular epidemiology of Ureaplasma species in males with infertility[J]. Eur J Clin Microbiol Infect Dis, 2020, 39(11): 2177-2183. |
| [17] |
FERNÁNDEZ J, KARAU M J, CUNNINGHAM S A, et al. Antimicrobial susceptibility and clonality of clinical Ureaplasma isolates in the United States[J]. Antimicrob Agents Chemother, 2016, 60(8): 4793-4798. |
| [18] |
SCHNEIDER S C, TINGUELY R, DROZ S, et al. Antibiotic susceptibility and sequence type distribution of Ureaplasma species isolated from genital samples in Switzerland[J]. Antimicrob Agents Chemother, 2015, 59(10): 6026-6031. |
| [19] |
DUMKE R. Antimicrobial resistance in clinical isolates of Ureaplasma spp. from samples in Germany[J]. Antimicrob Agents Chemother, 2021, AAC.02342-AAC.02320. |
| [20] |
王春燕, 杜江, 吴森林, 等. gyrA和parC基因突变与解脲支原体喹诺酮类药物耐药相关性研究[J]. 医学研究杂志, 2012, 41(10): 63-66. WANG C Y, DU J, WU S L, et al. Correlation of mutation patterns in gyrA and parC genes in Ureaplasma urealyticum isolates with quinolones resistance[J]. J Med Res, 2012, 41(10): 63-66. |
| [21] |
BEETON M L, CHALKER V J, MAXWELL N C, et al. Concurrent titration and determination of antibiotic resistance in Ureaplasma species with identification of novel point mutations in genes associated with resistance[J]. Antimicrob Agents Chemother, 2009, 53(5): 2020-2027. |
| [22] |
XIAO L, CRABB D M, DUFFY L B, et al. Chromosomal mutations responsible for fluoroquinolone resistance in Ureaplasma species in the United States[J]. Antimicrob Agents Chemother, 2012, 56(5): 2780-2783. |
| [23] |
DUFFY L, GLASS J, HALL G, et al. Fluoroquinolone resistance in Ureaplasma parvum in the United States[J]. J Clin Microbiol, 2006, 44(4): 1590-1591. |
| [24] |
PICCINELLI G, GARGIULO F, BISCARO V, et al. Analysis of mutations in DNA gyrase and topoisomerase Ⅳ of Ureaplasma urealyticum and Ureaplasma parvum serovars resistant to fluoroquinolones[J]. Infect Genet Evol, 2017, 47: 64-67. |
| [25] |
HAMASUNA R, LE P T, KUTSUNA S, et al. Mutations in ParC and GyrA of moxifloxacin-resistant and susceptible Mycoplasma genitalium strains[J]. PLoS One, 2018, 13(6): e0198355. |
| [26] |
YATOO M I, PARRAY O R, BHAT R A, et al. Emerging antibiotic resistance in Mycoplasma microorganisms, designing effective and novel drugs/therapeutic targets: current knowledge and futuristic prospects[J]. J Pure Appl Microbiol, 2019, 13(1): 27-44. |




