阿尔兹海默症(Alzheimer’s disease,AD)是一种神经退行性疾病,也是导致老年痴呆的最常见原因,占所有痴呆患者的60%~80%[1]。AD病程缓慢且发生积累性病变,最终表现为认知缺失、行为异常、语言障碍等严重影响患者正常生活的症状[2]。AD患者大脑中先出现神经元外的β-淀粉样蛋白(β-amyloid, Aβ)的积累和神经元内过度磷酸化的Tau蛋白组成的神经纤维缠结(neurofibrillary tangles,NFT),之后神经元大量死亡,脑组织发生结构性损伤[3-4]。因此,同时抑制Tau蛋白的过度磷酸化和减少Aβ的积累是治疗AD的一个重要研究方向[5]。
肽基脯氨酰顺/反异构酶NIMA互作蛋白1(peptidyl-prolylcis/trans isomerase,NIMA-inter- acting1,PIN1)属于肽基脯氨酸顺反异构酶家族中的一员[6]。PIN1由163个氨基酸残基构成,有2个功能结构域:氨基末端的双色氨酸结构域(WW结构域)和羧基末端的催化结构域(PPIase结构域),专一性地催化细胞内的丝/苏-脯氨酸基序发生顺/反异构,影响蛋白的磷酸化过程并参与细胞内多种代谢过程和信号传导调节[6]。研究报道,PIN1蛋白在AD患者大脑中包括海马区在内的多个区域的表达量是降低的[7-8]。当PIN1蛋白表达水平降低或活性受到抑制时,将导致顺式pT231-Tau含量上升和Aβ合成增加,并最终导致AD患者大脑中NFT和Aβ斑块的出现[9-10]。因此,内源性PIN1蛋白缺失与AD患者典型病理特征的出现具有明显的相关性。
构建与AD患者临床症状相似性高的动物模型不仅对AD发病机制的研究至关重要,而且可以为筛选治疗药物提供临床前受试对象。随着基因工程技术的不断发展,基因修饰的AD模型鼠得到了广泛应用[11]。据报道,与野生型同窝小鼠相比,PIN1基因敲除(KO)小鼠出现年龄依赖性的NFT和Aβ斑块[12]。目前,多种转基因小鼠被广泛用于研究Tau蛋白和Aβ斑块。然而,它们与人类AD患者脑生理结构、认知能力和行为学等方面均有一定差异,因此构建与人类疾病表型相似性更高的AD动物模型将极大地推动AD发病机制的剖析和治疗药物的开发[13]。猪是除灵长类动物外和人类进化关系最近的物种之一,其在大脑皮层结构、器官形态和大小及疾病发生与发展等方面均与人类十分相似。因此,PIN1基因缺陷的猪模型可能为阿尔兹海默症的药物研发提供更理想的受试对象[14]。
本研究通过分析人与猪PIN1蛋白的进化距离,然后对它们的蛋白二级结构组成和三级空间结构进行预测分析,验证人和猪PIN1蛋白的高度同源性。以长白猪胎儿原代成纤维细胞(porcine fetal fibroblasts,PFFs)为原材料,结合CRISPR/Cas9技术,构建PIN1基因敲除的单克隆细胞系,为进一步利用体细胞核移植(somatic cell nuclear transfer,SCNT)技术构建阿尔茨海默症猪模型提供了原材料。
1 材料与方法 1.1 材料1月龄长白猪PFFs原代细胞;DH5α感受态细胞和质粒抽提试剂盒(天根);pX330质粒(Addgene #42230)、pHY54_SV40-Neo质粒、BbsⅠ限制性内切酶、T4 DNA连接酶(NEB公司,美国);Basic NucleofectorTM Kits转染试剂盒和细胞电转仪(Lonza公司,德国);RNA-easyTM Isolation Reagent,HiScript® Ⅲ RT SuperMix,AceQ® qPCR SYBR Green Master Mix(Vazyme Biotech,中国);DMEM培养基、胎牛血清、胰蛋白酶、G418、青链霉素双抗和DPBS缓冲液(Gibco公司,美国);引物由生工生物工程(上海)股份有限公司合成。
1.2 方法 1.2.1 人/猪PIN1蛋白的同源性分析在NCBI数据库中检索包括人和猪在内的19个物种的PIN1蛋白的氨基酸序列,利用MEGA X软件和ITOL在线工具(https://itol.embl.de/)绘制系统进化树。采用DNAMAN软件对人和猪PIN1蛋白的氨基酸序列进行比对,再使用在线工具BLAST中的Global Align计算它们的一致性。
1.2.2 人/猪PIN1蛋白的结构比较利用DNAstar软件分析人和猪PIN1蛋白的二级结构,使用Chou Fasman算法预测人和猪PIN1蛋白的α螺旋、β折叠、β转角的比例。使用Swiss Model对人和猪PIN1蛋白进行三维建模,然后利用pymol软件比较两者三维结构的相似度,得出均方根偏差(RMSD)值。
1.2.3 长白猪PIN1基因序列验证和靶向sgRNAs的设计根据猪PIN1基因的序列,利用在线工具Primer-Blast,在第2外显子两侧设计PCR产物约500 bp的引物(上游引物:5′-CACTGGGGACTTGC-TGAATGA-3′;下游引物:5′-TGCTGGACAGGAAAA-GAAGGG-3′),对长白猪PFFs的基因组DNA进行PCR扩增和测序鉴定。以测序鉴定后的序列作为模板,利用CRISPR靶点在线设计工具Guide Design Resources(http://crispor.tefor.net/)设计2对sgRNAs,分别命名为2_sgRNA1(上游引物:5′-CACCGTTGAAGTAGTACACCCGGCC-3′;下游引物:5′-AAACGGCCGGGTGTACTACTTCAAC-3′;PAM序列:TGG)和2_sgRNA2(上游引物:5′-CACCGGAGA-AGATCACCCGGACTA-3′;下游引物:5′-AAACTA-GTCCGGGTGATCTTCTCC-3′;PAM序列:AGG),5′末端磷酸化修饰,交由公司合成。
1.2.4 CRISPR/Cas9打靶质粒的构建用高压灭菌的去离子水将2对Oligos稀释成100 pmol/μL,然后进行退火:37 ℃,30 min,95 ℃,5 min,以5 ℃/min的速度降至25 ℃。将利用BbsⅠ酶切线性化后的pX330载体与双链sgRNAs进行连接。把连接产物转化至DH5α中,涂板后挑取单菌落进行测序鉴定。将构建完成的打靶质粒命名为2_sgRNA1-pX330和2_sgRNA2-pX330。
1.2.5 PFFs培养与转染将原代长白猪PFFs复苏在6孔培养板内, 用含16% FBS的全培养基培养约24 h,条件为38.5 ℃恒温,5% CO2,至细胞汇合度达80%左右。用DPBS清洗3次后使用0.05% 胰酶消化2 min,1 200 r/min离心4 min得到细胞沉淀。配制核转液(体系为Nucleofector:82 μL;Supplement:18 μL;2_sgRNA1-pX330:2.5 μg;2_sgRNA2-pX330:2.5 μg;pHY54_SV40-neo:2 μg),将构建的2个打靶质粒与Neomycin抗性质粒利用核转染程序U-023转染。转染结束后,用培养基重悬细胞并分盘置于恒温培养箱中培养。
1.2.6 单细胞克隆的筛选与基因型鉴定转染后培养24 h,用含1 mg/mL G418药物的全培养基并进行换液。根据细胞状态适度降低药物浓度,转染后8 d左右,挑选适宜单细胞克隆并作标记。待其生长至最佳状态时,将细胞消化并转移至24孔板培养。24 h后用含300 μg/mL G418的全培养基进行换液,待细胞长满后传代至12孔板(24孔板中留少量细胞用于鉴定),待12孔板中的细胞长满后,冻存备用。
1.2.7 单细胞克隆基因型的鉴定将24孔板中的半数细胞用于提取基因组DNA。配制NP-40裂解液(10 mL):NP-40,45 μL;Proteinase K,2 mL;10×PCR Buffer,1 mL;ddH2O,7 mL。取适量NP-40裂解液重悬细胞沉淀,进行裂解程序:55 ℃,60 min;95 ℃,10 min;4 ℃低温保存。利用引物2_primer F和2_primer R进行PCR扩增。对PCR产物进行测序鉴定,根据测序结果确定PIN1基因敲除的阳性单细胞克隆的基因型。
1.2.8 PIN1基因敲除的细胞克隆的表达分析PIN1 mRNA表达分析:将PIN1双等位基因敲除的单细胞克隆培养至6孔板,使用RNA-easyTM Isolation Reagent提取总RNA,在检测其纯度和完整性后进行逆转录获得cDNA,进而进行荧光实时定量PCR分析。反应体系为:2×AceQ qPCR SYBR Green Master Mix,10 μL;Primer1 (10 μmol/L),0.4 μL;Primer2 (10 μmol/L),0.4 μL;cDNA,1 μg;补ddH2O至20 μL。PIN1蛋白表达分析:分别提取PIN1双等位基因敲除的PFFs和野生型PFFs的总蛋白,变性后上样,采用100 V恒压电泳100 min后转移至PVDF膜上,封闭40 min,加一抗(兔源抗PIN1,1∶1 000稀释),4 ℃过夜孵育,PBST清洗后加入羊抗兔的二抗(1∶5 000),室温孵育1 h后清洗3次,加入500 μL显色液,对蛋白Marker和PIN1蛋白条带曝光显影,显影后用PBST清洗并孵育一抗GAPDH(1∶20 000),室温孵育1 h后曝光并保存结果。
1.3 统计学分析利用GraphPad Prism 5.0软件对数据进行分析,数据以x±s表示,两组间比较采用独立样本t检验,检验水准:α=0.05。
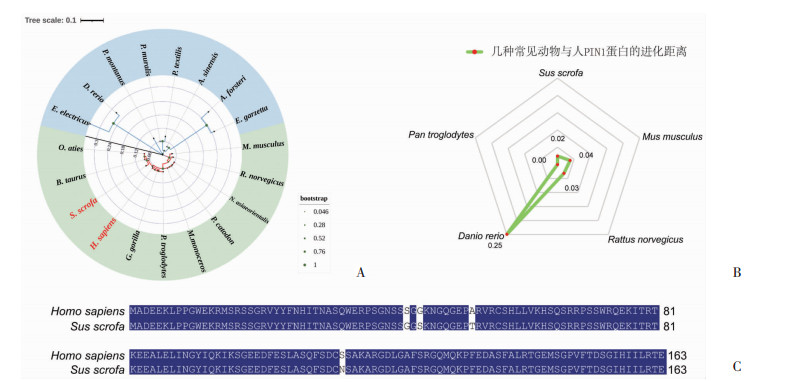
2 结果 2.1 人/猪PIN1的同源性分析系统进化树构建结果表明人和猪PIN1蛋白遗传距离较近(图 1A);根据绘制的系统进化树计算几种常见动物与人类PIN1氨基酸序列的进化距离并绘制雷达图(图 1B);人与猪PIN1蛋白的氨基酸序列比对结果显示两者高度一致,二者一致性和相似性均为98%(图 1C)。以上结果表明与小鼠等物种相比,猪和人PIN1蛋白在进化上更为接近。
|
| A:不同物种PIN1蛋白的系统进化树淡蓝色区域为非哺乳动物,淡绿色区域为哺乳动物;分支部位的绿色圆点大小代表自展值,自展检验次数为1 500次;B:几种常见动物与人的PIN1蛋白进化距离越接近中心表明这个物种与人PIN1蛋白进化距离越近;C:人和猪PIN1蛋白氨基酸序列一致性比对深色区域是相同氨基酸,浅色为不同氨基酸 图 1 人/猪PIN1蛋白同源性分析 |
2.2 人/猪PIN1蛋白结构预测与比对
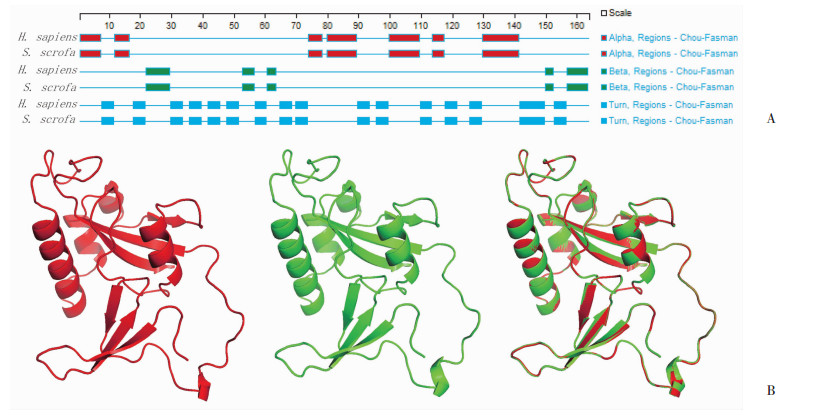
人和猪PIN1蛋白的α螺旋,β折叠,β转角的比例相似度很高:人PIN1的α螺旋占32.5%,β折叠占15.3%,β转角占41.7%;猪PIN1的α螺旋占32.5%,β折叠占12.9%,β转角占44.2%(图 2A)。对人和猪PIN1进行三维建模并比较两者三维结构的相似度,得出RMSD值为0.014,表明两者在三维结构上亦具有极高的相似性(图 2B)。以上结果表明,人/猪PIN1具有高度的同源性,因此可以推测人/猪PIN1在相应组织或器官内应具有相似的生理功能。
|
| A:人(上)和猪(下)的PIN1蛋白二级结构对比红色表示α螺旋;绿色表示β折叠;蓝色表示β转角;B:人/猪PIN1三级结构比较红色为人PIN1三维建模图;绿色为猪PIN1三维建模图;二者RMSD=0.014 图 2 人/猪PIN1蛋白质二级结构和三级结构分析 |
2.3 CRISPR/Cas9打靶质粒的构建
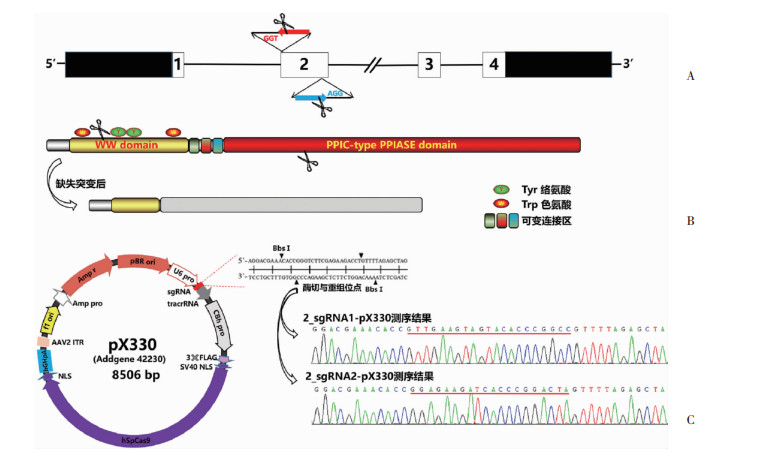
PIN1基因有4个外显子,其中第2外显子序列长213 bp,编码PIN1蛋白识别底物功能的WW结构域和底物催化结构域的部分序列。依据测序确认的长白猪PIN1第2外显子序列,利用在线工具设计2对sgRNAs,靶向的编码序列位置分别为c.113-132和c.280-299(图 3A),预期能切除PIN1的WW部分区域,可变连接区域以及部分底物催化结构域(图 3B)。测序结果(图 3C)表明这2个sgRNAs成功克隆至pX330质粒的预定位点。
|
| A:长白猪PIN1基因示意图黑色区域为非编码区;白色框内数字代表外显子位置;红/蓝色箭头所指的序列为靶序列位置与方向,与靶序列相邻的序列为Cas9识别的PAM序列;B:长白猪PIN1蛋白结构以及预测突变结果示意图黄色区域为WW domain,即双色氨酸结构域,作为底物识别和结合位点,红色区域为PPIC-type PPIASE结构域,负责催化磷酸化底物的顺反异构,二者中间为可变连接区域;C:pX330载体示意图和重组载体的测序结果 图 3 PIN1基因靶点位置和重组载体测序 |
2.4 单细胞克隆的获得和鉴定
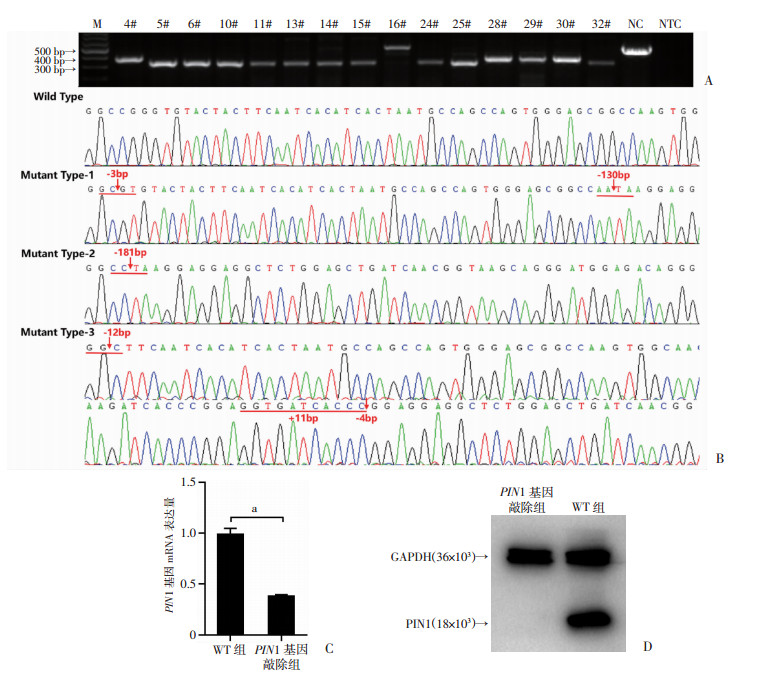
野生长白猪胚胎成纤维细胞转染重组的打靶质粒和抗性质粒后,经过G418筛选,最终得到34个单细胞克隆,其中有15个发生纯合的双等位基因突变(表 1),电泳鉴定结果显示有片段缺失(图 4A)。测序结果显示共有3种突变型(mutant type,MT):MT1在发生130 bp的大片段缺失的基础上发生移码突变并引入终止密码子;MT2则在缺失181 bp的同时发生移码突变并引入终止密码子;MT3在sgRNA1靶向区域缺失了WW结构域底物结合位点处的酪氨酸,其在sgRNA2靶向的区域发生小片段缺失,但在其上游插入突变区引入终止密码子(图 4B)。选用状态较好的单细胞克隆(MT2,5#)进行冻存和PIN1的表达鉴定。qRT-PCR结果显示5#克隆仍能检测到PIN1 mRNA,但其表达显著低于野生型对照(图 4C)。将野生型和5#克隆的PIN1蛋白进行Western blot分析,结果显示其PIN1蛋白的表达已经被完全破坏(图 4D)。
| 序号 | 测序区域序列 | 突变类型 |
| WT | GGCCGGGTGTACTACTTCAATCA//GATCACCCGGACTAAGGAGGAGGCTCT | WT |
| 4# | GGC---GTGTACTACTTCAA//AGTGGGAGCGGCCAA--//--TAAGGAGGAG | -3,-130 bp |
| 5# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| 6# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| 10# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| 11# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| 13# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| 14# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| 15# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| 16# | GGC------------TTCAAT//GGAGGTGATCACCC----GGAG//CAACGG | -12,+11, -4 bp |
| 24# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| 25# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| 28# | GGC---GTGTACTACTTCAA//AGTGGGAGCGGCCAA--//--TAAGGAGGAG | -3,-130 bp |
| 29# | GGC---GTGTACTACTTCAA//AGTGGGAGCGGCCAA--//--TAAGGAGGAG | -3,-130 bp |
| 30# | GGC---GTGTACTACTTCAA//AGTGGGAGCGGCCAA--//--TAAGGAGGAG | -3,-130 bp |
| 32# | GGCC--------//--------TAAGGAGGAGGCTCTGGAGCTGATCAACGGT | -181 bp |
| WT:野生型;+:碱基插入;-:碱基缺失;标红字母为插入的碱基序列 | ||
3 讨论
AD是一种退行性神经疾病,因病程漫长,前期症状不明显而难以对其进行早期诊断和治疗。我国已成为全世界患AD人数最多的国家,但目前仅有5种药物获批用于治疗AD,且疗效有限。抑制Tau蛋白的过度磷酸化,并减少Aβ的积累被认为是治疗AD的一个重要研究方向[5]。而PIN1可通过降低Tau蛋白的过度磷酸化和减少APP的产生以防止AD典型症状的出现,这表明PIN1可能是探索AD发生、发展的分子机制以及对AD进行诊断和治疗的新型靶点[15]。
AD临床药物获批种类少的原因之一是缺乏良好的动物模型,而对潜在候选药物的筛选效果在很大程度上取决于动物模型与人类在疾病方面的相似度。相较于小鼠,猪作为一种与人类临床相关性更高的大动物模型,更适宜用于AD发病机制研究与治疗药物研发。另外,与ZFN和TALEN等基因编辑技术相比,CRISPR/Cas9与SCNT相结合可对内源性猪基因进行更加高效的遗传修饰。本课题组已利用此技术成功构建载脂蛋白E (apolipoprotein E,ApoE)基因敲除猪、结节性硬化复合体1(tuberous sclerosis complex1,TSC1)基因敲除猪等多种猪模型[16-17],显示了CRISPR/Cas9技术在构建基因编辑大动物模型研究中的优越性。
本研究以原代长白猪胎儿成纤维细胞为原材料,通过生物同源性分析证明人和猪PIN1蛋白氨基酸序列、二级结构组成和三维空间结构方面均具有极高的相似性,可以推测猪PIN1基因突变后能够产生与人类似的遗传效应和病理表型。此外,猪PIN1基因2号外显子区编码氨基酸序列较长,并且会参与形成PIN1蛋白两个重要的功能结构域及它们的连接区。为了确保PIN1蛋白质结构被完全破坏并尽可能提高编辑效率,本研究在PIN1的2号外显子区设计2个方向相反且相距3n+1个碱基的sgRNAs[18]。T7EN1酶切实验证明,设计的sgRNAs均能以较高效率切割目的基因序列。对筛选得到的单细胞克隆进行测序,结果显示在PIN1靶点位置出现3种不同形式突变,其中占比最高的突变类型是MT2,其发生181 bp的大片段缺失并造成移码突变。PCR测序结果显示随机选取sgRNAs的13个潜在的脱靶位点均未检测到脱靶。这表明利用2个sgRNAs进行敲除能够有效提高基因编辑效率,同时也有很好的靶向特异性。因此,利用CRISPR/Cas9进行基因敲除时,设计2个sgRNAs同时进行编辑可能是更为理想的策略。qRT-PCR结果显示MT2突变类型的细胞克隆中仍能检测到PIN1 mRNA,但其表达水平显著低于野生型对照,显示PIN1的基因编辑对PIN1 mRNA的转录或者转录后的稳定性产生了影响。本研究获得了3种突变类型的PIN1敲除的单细胞克隆。虽然仅利用Western blot证实了MT2突变类型的细胞克隆中无PIN1蛋白的表达,但由于本研究获得的PIN1双等位基因敲除的细胞克隆均在靶点处发生了移码突变或引入了终止密码子,我们推测3种突变类型的细胞克隆中PIN1蛋白均被完全破坏。
综上所述,本研究利用CRISPR/Cas9技术成功构建了PIN1双等位基因敲除的长白猪胎儿成纤维细胞系,为后续体细胞核移植和胚胎移植提供了核供体细胞,为构建人类阿尔茨海默症猪模型提供了重要的原材料。
| [1] |
Alzheimer's Association. 2020 Alzheimer's disease facts and figures[J]. Alzheimers Dement, 2020, 16(3): 391-460. |
| [2] |
LANE C A, HARDY J, SCHOTT J M. Alzheimer's disease[J]. Eur J Neurol, 2018, 25(1): 59-70. |
| [3] |
GALLARDO G, HOLTZMAN D M. Amyloid-β and tau at the crossroads of Alzheimer's disease[J]. Adv Exp Med Biol, 2019, 1184: 187-203. |
| [4] |
BAZZARI F H, ABDALLAH D M, EL-ABHAR H S. Pharmacological interventions to attenuate Alzheimer's disease progression: the story so far[J]. Curr Alzheimer Res, 2019, 16(3): 261-277. |
| [5] |
WERWEIN E, CIBIS H, HESS D, et al. Activation of the oncogenic transcription factor B-Myb via multisite phosphorylation and prolyl Cis/trans isomerization[J]. Nucleic Acids Res, 2019, 47(1): 103-121. |
| [6] |
ANGELUCCI F, HORT J. Prolyl isomerase Pin1 and neurotrophins: a loop that may determine the fate of cells in cancer and neurodegeneration[J]. Ther Adv Med Oncol, 2017, 9(1): 59-62. |
| [7] |
LANKE V, MOOLAMALLA S T R, ROY D, et al. Integrative analysis of hippocampus gene expression profiles identifies network alterations in aging and Alzheimer's disease[J]. Front Aging Neurosci, 2018, 10: 153. |
| [8] |
LIOU Y C, SUN A Y, RYO A, et al. Role of the prolyl isomerase Pin1 in protecting against age-dependent neurodegeneration[J]. Nature, 2003, 424(6948): 556-561. |
| [9] |
PARK J S, LEE J, JUNG E S, et al. Brain somatic mutations observed in Alzheimer's disease associated with aging and dysregulation of tau phosphorylation[J]. Nat Commun, 2019, 10(1): 3090. |
| [10] |
HODSON R. Alzheimer's disease[J]. Nature, 2018, 559(7715): S1. |
| [11] |
DRUMMOND E, WISNIEWSKI T. Alzheimer's disease: experimental models and reality[J]. Acta Neuropathol, 2017, 133(2): 155-175. |
| [12] |
PASTORINO L, SUN A Y, LU P J, et al. The prolyl isomerase Pin1 regulates amyloid precursor protein processing and amyloid-beta production[J]. Nature, 2006, 440(7083): 528-534. |
| [13] |
MA Y L, WEI J L, ZHANG Q, et al. A genome scan for selection signatures in pigs[J]. PLoS One, 2015, 10(3): e0116850. |
| [14] |
LI M Z, CHEN L, TIAN S L, et al. Comprehensive variation discovery and recovery of missing sequence in the pig genome using multiple de novo assemblies[J]. Genome Res, 2017, 27(5): 865-874. |
| [15] |
CORTES-HERNANDEZ P, DOMÍNGUEZ-RAMÍREZ L. Role of Cis-trans proline isomerization in the function of pathogenic enterobacterial periplasmic binding proteins[J]. PLoS One, 2017, 12(11): e0188935. |
| [16] |
FANG B, REN X Y, WANG Y, et al. Apolipoprotein E deficiency accelerates atherosclerosis development in miniature pigs[J]. Dis Model Mech, 2018, 11(10): dmm036632. |
| [17] |
LI X X, HU T D, LIU J Y, et al. A Bama miniature pig model of monoallelic TSC1 mutation for human tuberous sclerosis complex[J]. J Genet Genomics, 2020, 47(12): 735-742. |
| [18] |
GUO T, FENG Y L, XIAO J J, et al. Harnessing accurate non-homologous end joining for efficient precise deletion in CRISPR/Cas9-mediated genome editing[J]. Genome Biol, 2018, 19(1): 170. |