2. 400038 重庆, 陆军军医大学(第三军医大学)药学与检验医学系临床检验与野战检验教研室
2. Department of Clinical and Military Laboratory Medicine, Faculty of Pharmacy and Laboratory Medicine, Army Medical University (Third Military Medical University), Chongqing, 400038, China
DNA甲基化是一种重要的表观遗传机制,在染色质结构和基因表达调控中起重要作用[1]。现今,国内外大量研究发现DNA甲基化与心血管疾病[2]、糖尿病[3]、肺癌[4]、肝癌[5]和宫颈癌[6]等多种疾病相关。因此,准确检测DNA甲基化水平对甲基化修饰紊乱类疾病的诊断和病理机制研究至关重要。光电化学(photoelectrochemistry, PEC)传感技术因背景噪声低、成本低、操作简便和响应速度快等特点被广泛应用于DNA甲基化检测[7-8],但是目前大多数生物传感器是通过对DNA甲基转移酶活性的检测从而间接推算DNA甲基化水平[9-10],不是对DNA甲基化水平进行直接检测。因此,建立一种准确、特异的DNA甲基化位点分析方法已成为表观遗传疾病诊断的迫切需求。
有研究表明纳米传感器有限的界面面积限制了固定化探针分子的有效杂交数量,从而降低了探针与靶分子之间碰撞和结合的概率[11]。为突破传感器界面面积的限制,有研究者应用四面体DNA框架纳米结构控制螺旋ssDNA之间的横向距离,提高探针与靶分子之间的结合率,以提高DNA杂交效率和杂交动力学,从而提高传感器的灵敏度和特异性[12]。基于上述理论基础,本研究拟构建一种新型3D-TPN,该探针以具有三维立体结构的DNA三棱柱(TP)为纳米框架,连接探针为纳米机械手柄,将ssDNA生物探针拉伸并固定在生物传感界面,以提高与靶序列的结合率、杂交效率和检测特异性。
本研究利用三维刚性DNA桥式纳米探针联合AuNPs/TiO2@CdTe QDs双信号放大体系,探索建立一种能直接、准确分析DNA甲基化丰度的光电化学传感器,为DNA甲基化的定量检测提供一种新方法。
1 材料与方法 1.1 材料 1.1.1 主要试剂L-抗坏血酸(AA)、二氧化钛(TiO2)、氯化镉(CdCl2)、亚碲酸钠(NaTeO3)、巯基丙酸(MPA)购自上海阿拉丁生化科技股份有限公司;四氯金酸三水合物(HAuCl4·3H2O)、硼氢化钠(NaBH4)购自西格玛奥德里奇贸易有限公司;5-甲基胞嘧啶(5-mC)抗体购自美国Abcam公司。所有化学试剂为分析纯,实验用水为超纯水(电阻率18 MΩ/cm)。实验所用DNA寡核苷酸序列,均由上海生工生物工程技术服务有限公司合成并经过高效液相色谱法纯化(表 1)。
| 名称 | 序列(5'→3') |
| C1 | TCGCTGAGTATTTTGCCTGGCCTTGGTCCATTTGTTTTGCAAGTGTGGGCACGCACACTTTTCGCACCGCGACTGCGAGGACTTTTCACAAATCTG |
| C2 | CACTGGTCAGTTTTCCACCAGCTAGATGTTGAAGTTTTTACTCAGCGACAGATTTGTGTTTTCGCTCTTCTATACTGGCGGATTTTGGTTTGCTGA |
| C3 | CCACACTTGCTTTTGTGCCTGGCCTTCCAGTTTGTTTTCTGACCAGTGTCAGCAAACCTTTTGCGACCGCGAGGACGTCCTGTTTTGTGTGCGTGCTCC |
| linker strand 1(L1) | TCCTCCTCCTTTTTTTTTTCAAATGGACCAAGGCCAGGC |
| linker strand 2(L2) | TCCTCCTCCTCCTTTTTTTTTTCTTCAACATCTAGCTGGTGG |
| linker strand 3(L3) | TCCTCCTCCTCCTTTTTTTTTTCAAACTGGAAGGCCAGGCAC |
| anchor strand 1(A1) | GTCCTCGCAGTCGCGGTCCG |
| anchor strand 2(A2) | TCCGCCAGTATAGAAGAGAG |
| anchor strand 3(A3) | CAGGACGTCCTCGCGGTCGC |
| probe | GGAGGAGGAGGATTTTTTTTTTCGCGTTGACCGCGGGGTCCGACATGATGGCTGGTGGGCAGCGGGTCGCGCGTTTTTTTTTTGGAGGAGGAGGA |
| Target 1 | C/i5MedC/GCGCGACCCGCTGCCCACCAGCCATCATGTCGGACCCCGCGGTCAACGCG |
| Target 2 | C/i5MedC/GC/i5MedC/GCGACCCGCTGCCCACCAGCCATCATGTCGGACCCCGCGGTCAACGCG |
| Target 3 | C/i5MedC/GC/i5MedC/GC/i5MedC/GACCCGCTGCCCACCAGCCATCATGTCGGACCCCGCGGTCAACGCG |
| Target 4 | C/i5MedC/GC/i5MedC/GC/i5MedC/GACCC/i5MedC/GCTGCCCACCAGCCATCATGTCGGACCCCGCGGTCAACGCG |
| Target 5 | C/i5MedC/GC/i5MedC/GC/i5MedC/GACCC/i5MedC/GCTGCCCACCAGCCATCATGTC/i5MedC/GGACCCCGCGGTCAACGCG |
| Target 6 | C/i5MedC/GC/i5MedC/GC/i5MedC/GACCC/i5MedC/GCTGCCCACCAGCCATCATGTC/i5MedC/GGACCCC/i5MedC/GCGGTCAACGCG |
| Target 7 | C/i5MedC/GC/i5MedC/GC/i5MedC/GACCC/i5MedC/GCTGCCCACCAGCCATCATGTC/i5MedC/GGACCCC/i5MedC/GC/i5MedC/GGTCAACGCG |
| M1 | C/i5MedC/GGGCGACCCGCTGCCCACCAGCCATCATGTCGGACCCCGCGGTCAACGCG |
| M3 | C/i5MedC/GGGCGACCCGCTGCCCACCAGCCATCATGTCGGACCCCGCGGTCAACGCG |
| M5 | C/i5MedC/GGGCGACCCGCTGCGCACCAGCGATCATGTCGGACCCGGCGGTGAACGCG |
| Mn | C/i5MedC/GGGCGACACGTTGCGCATCAGCGATGATGTCGGAACCGGCGGTGAACGCG |
1.1.2 主要仪器
CHI660D电化学工作站(上海辰华);CompactStat Potentiostat光电化学工作站(荷兰Ivium);Bruker原子力显微镜(德国布鲁克公司);Spectra S透射电子显微镜(荷兰赛默飞);Zeiss扫描电子显微镜(德国蔡司)。
1.2 方法 1.2.1 3D-TPN的制备各取C1、C2和C3序列(100 μmol/L)1 μL加入97 μL Tris-Mg(OAc)2缓冲液[40 mmol/L Tris,1 mmol/L EDTA,12.5 mmol/L Mg(OAc)2],振荡混匀后于PCR仪中按以下程序退火处理:95 ℃/5 min,4 ℃/30 s,通过热变性法自组装成TP结构。使用自动PCR热循环仪将连接序列和锚定序列依次加载到TP上,退火程序如下:60 ℃/30 min,50 ℃/30 min,37 ℃/30 min,4 ℃/60 min。最后,将500 nmol/L的探针与上述产物混合于37 ℃水浴中组装成三维刚性桥式DNA纳米探针,存于4 ℃备用,原理见图 1。
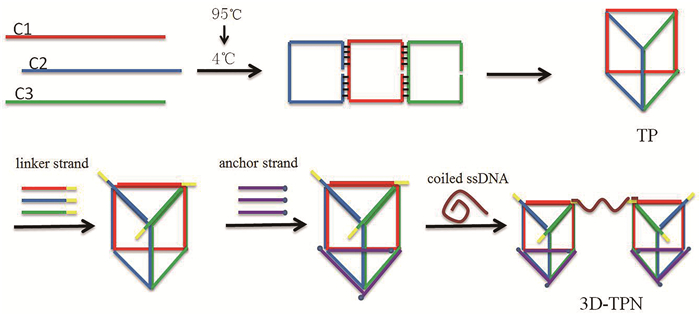
|
| 图 1 三维刚性桥式DNA纳米探针构建原理 |
1.2.2 Ab-CdTe QDs生物偶联物的制备
根据文献[13]制备羧基化CdTe量子点。取0.11 g CdCl2和87 μL MPA溶于120 mL超纯水,超声溶解,继续加入120 mg NaBH4和13.3 mg NaTeO3,搅拌混匀,100 ℃加热回流6 h,离心洗涤得到CdTe QDs。用含20 mmol/L EDC和40 mmol/L NHS的混合液活化CdTe QDs,将5-甲基胞嘧啶抗体加入CdTe QDs溶液中,37 ℃反应1 h,离心弃上清,重悬于10 mmol/L PBS,标记产物为Ab-CdTe QDs生物偶联物,放于4 ℃冰箱保存。
1.2.3 PEC生物传感器的制备用丙酮、乙醇和去离子水依次超声清洗ITO导电玻璃10 min,氮气中干燥,滴加10 μL TiO2悬浮液(2 mg/mL)于电极表面,晾干,将电极置于马弗炉450 ℃煅烧30 min,得到TiO2/ITO电极;再将电极浸入HAuCl4溶液(0.025 mol/L) 进行电沉积(电位:-0.2 V,时间:120 s),超纯水冲洗,氮气吹干,制得AuNPs/TiO2/ITO电极;10 μL三维刚性DNA桥式纳米探针滴加到电极表面,置于4 ℃冰箱过夜。超纯水充分冲洗,得到TPN/AuNPs/TiO2/ITO电极;滴加10 μL 2 mmol/L MCH,室温放置1 h,以阻断非特异性位点,超纯水清洗,标记为MCH/TPN/AuNPs/TiO2/ITO,即制得实验所用传感器。
1.2.4 PEC传感器检测原理取10 μL靶DNA序列滴加到制备好的电极表面,37 ℃孵育2 h,去离子水冲洗,10 μL Ab-CdTe QDs偶联物(10 μg/mL)滴加到电极表面,37 ℃孵育1 h后冲洗。5-甲基胞嘧啶抗体特异性识别5-甲基胞嘧啶位点,将Ab-CdTe QDs偶联物加载到靶序列上,CdTe QDs对AuNPs/TiO2的敏化作用使得光电信号产生变化,从而实现对DNA甲基化的定量分析,检测原理如图 2所示。
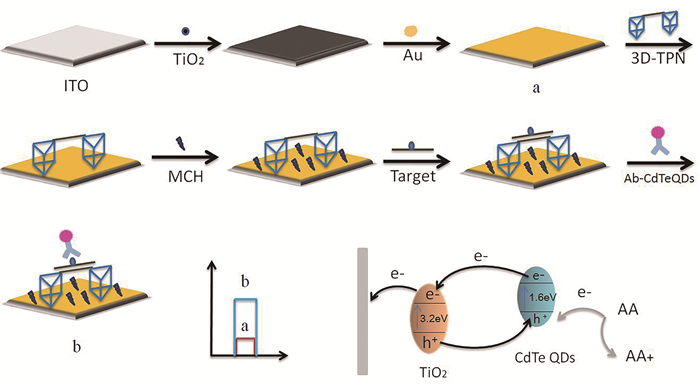
|
| a:AuNPs/TiO2的光电流响应;b:PEC传感器组装完成后的光电流响应 图 2 基于三维刚性桥式纳米探针的DNA甲基化光电化学传感器构建原理图 |
1.2.5 光电化学检测
PEC检测采用标准三电极体系,辅助电极为铂丝电极,参比电极为Ag/AgCl电极,ITO电极为工作电极;电解质溶液为0.1 mol/L PBS缓冲液(含10 mmol/L AA),测试光源为蓝色发光二极管(15 mW/cm2),偏置电压为0 V,开关光间隔时间为10 s。
1.2.6 观察指标① 采用电化学阻抗谱(EIS)对该传感器的逐层组装过程进行表征;②PEC生物传感器实验条件优化,包括TiO2悬浮液浓度和5-甲基胞嘧啶抗体浓度;③DNA甲基化丰度分析:分别对DNA靶序列Target1~Target7进行检测,观察PEC光电响应与DNA甲基化位点数量的关系;④PEC传感体系重复性、稳定性和特异性检测。
2 结果 2.1 TP的电泳表征图 3A示各单链、2条链组合和3条链组合通过一步热变性法合成后,在10%聚丙烯酰胺凝胶中的电泳结果。从图中可以看出:条带的迁移速度随着DNA碱基数和结构复杂程度的增加而减慢,泳道8为DNA三棱柱结构,由于其分子量最大且为三维立体结构,因此迁移速率最慢。图 3B是连接序列和锚定探针在TP上逐步组装的聚丙烯酰胺凝胶电泳结果,泳道2至泳道7迁移速率逐渐减慢,表明连接序列和锚定探针已成功固定于TP上。三维刚性桥式DNA纳米合成结果用1%琼脂糖进行表征,如图 3C所示:泳道4是3D-TPN产物,由于它的碱基数目最多、三维结构最复杂,导致其迁移速度最慢。电泳结果证明TP和3D-TPN结构均组装成功。
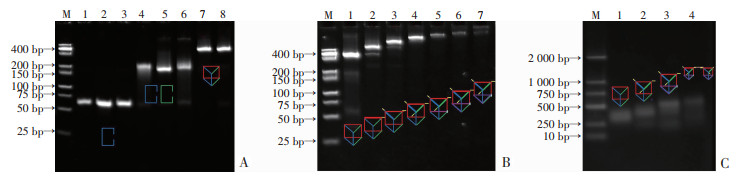
|
| A: DNA三棱柱合成过程 M:500 bp DNA marker;1:C1;2:C2;3:C3;4:C1+C2;5:C2+C3;6:C1+C3;7:C1+C2+C3;8:C1+C2+C3(热循环); B: DNA纳米框架探针机械臂组装过程 M:500 bp DNA marker;1:TP;2:TP+L1;3:TP+L1+L2;4:TP+L1+L2+L3;5:TP+L1+L2+L3+A1;6:TP+L1+L2+L3+A1+A2;7:TP+L1+L2+L3+A1+A2+A3; C: 三维刚性DNA桥式纳米探针组装过程 M:2 000 bp DNA marker;1:TP;2:TP+L1+L2+L3;3:TP+L1+L2+L3+A1+A2+A3;4:3D-TPN 图 3 3D-TPN合成的电泳表征图 |
2.2 3D-TPN的原子力显微镜表征
采用原子力显微镜对3D-TPN结构进行表征。如图 4所示:3D-TPN具有桥式链接空间结构,表示桥式DNA纳米结构探针合成成功。

|
| 图 4 3D-TPN的原子力显微镜表征图 |
2.3 CdTe纳米微粒的表征
采用透射电镜对CdTe QDs进行表征,如图 5所示:CdTe QDs为颗粒状,形态清晰,尺寸约为4 nm。

|
| 图 5 CdTe QDs的透射电镜表征图 |
2.4 AuNPs/TiO2/ITO电极的表征
采用扫描电镜对AuNPs/TiO2修饰的ITO电极进行形貌表征,如图 6A所示:TiO2微粒较小,在ITO电极表面分布均匀;由图 6B可见:覆盖于TiO2/ITO电极表面的金纳米粒子均匀分布,粒径较大,表明AuNPs和TiO2均已成功修饰到ITO电极表面。

|
| A: TiO2修饰ITO电极; B: AuNP修饰TiO2/ITO电极 图 6 AuNPs/TiO2修饰ITO电极的扫描电镜表征图 |
2.5 传感器组装的电化学表征
采用EIS在含有5 mmol/L[Fe(CN)6]3-/4-的0.1 mol/L KCl电解液中对电极逐层组装过程进行表征。结果如图 7所示:与裸ITO电极(曲线a)相比,TiO2纳米膜覆盖后电极(曲线b)阻抗值增大,这是由于TiO2阻碍了电子转移;沉积AuNPs后电极(曲线c)的阻抗值减小,是因为AuNPs可以加快电子的传递速率;TPN修饰后的电极(曲线d)所产生的阻抗值增大;表明TPN已成功固定到电极表面,随后依次进行MCH的封闭、靶序列的杂交和Ab-CdTe QDs复合物的组装,电极产生的阻抗值逐渐增大,是由于这些生物分子修饰在电极表面后导致空间位阻逐渐增大,影响了电荷在电解液和电极表面之间的转移和扩散。EIS表征结果表明,光电化学传感器的每一步组装均是成功的。
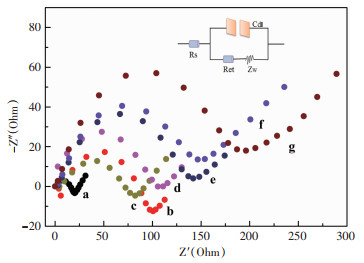
|
| a:裸ITO;b:TiO2/ITO;c:AuNPs/TiO2/ITO;d:TPN/AuNPs/TiO2/ITO;e:MCH/TPN/AuNPs/TiO2/ITO;f:DNA/MCH/TPN/AuNPs/TiO2/ITO;g:Ab-CdTe QDs/DNA/MCH/TPN/AuNPs/TiO2/ITO 图 7 PEC传感器的电化学阻抗谱表征 |
2.6 TiO2悬浮液浓度的优化
TiO2悬浮液的浓度直接影响PEC传感体系的检测性能,为达到最佳检测效果,对TiO2悬浮液浓度(1、2、3、4、5 mg/mL)进行优化(图 8A)。由图所示:TiO2溶液在低浓度时,光电信号较低,在浓度为4 mg/mL时,响应电流值达到最大,继续增大TiO2溶液浓度,光电响应变小。因此,选择4 mg/mL作为TiO2悬浮液的最佳浓度。
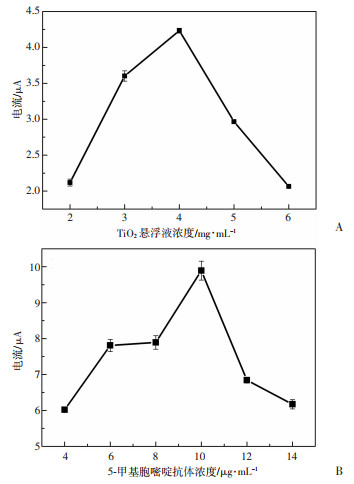
|
| A: TiO2悬浮液浓度对PEC电流响应值的影响; B: 5-甲基胞嘧啶浓度对PEC电流响应值的影响 图 8 不同实验因素对PEC电流响应值的影响 |
2.7 5-甲基胞嘧啶抗体的优化
本研究中Ab-CdTe QDs偶联物作为信号放大单元,抗体浓度直接影响光电流信号的产生,通过控制5-甲基胞嘧啶抗体浓度这一单一变量研究其对传感器响应的影响。如图 8B所示:当抗体浓度从3 μg/mL增加到10 μg/mL,光电流响应值逐渐增大,在10 μg/mL达到峰值,但抗体浓度大于10 μg/mL,光电流响应逐渐下降,说明抗体浓度已达到饱和,故10 μg/mL为本研究中5-甲基胞嘧啶抗体最佳浓度。
2.8 PEC生物传感器对DNA甲基化位点的定量分析在最适实验条件下,利用该PEC传感器分别对100 mmol/L的Target1~Target7进行检测,PEC信号响应随着DNA甲基化位点数量的增加而增大,在1~7个甲基化位点,PEC信号响应值与DNA甲基化位点数量呈线性关系,并计算得回归方程ΔI=-0.004 7+4.011 4×N,决定系数R2为0.999 7,见图 9。
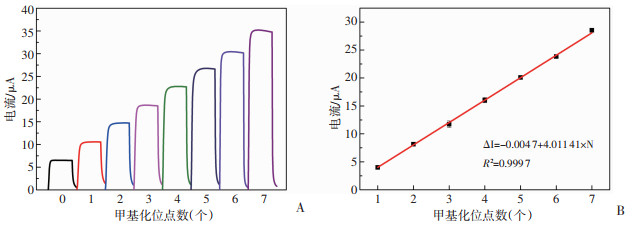
|
| A:不同数量甲基化位点的PEC响应;B:PEC信号强度与甲基化DNA位点数的相关曲线 图 9 PEC生物传感器的检测性能 |
2.9 PEC传感器的重复性、稳定性和特异性研究
在相同实验条件下,同时制备5支电极对100 mmol/L的Target4目标序列进行光电信号检测,每根电极重复测量3次,结果如图 10A所示:计算所得光电信号相对标准差为1.8%,证明该传感器具有良好的重复性。将制备好的电极置于4 ℃冰箱保存,每周重新检测其光电信号响应值,如图 10B所示:4周后光电流仍保持初始值的92.5%,该传感器稳定性较佳。
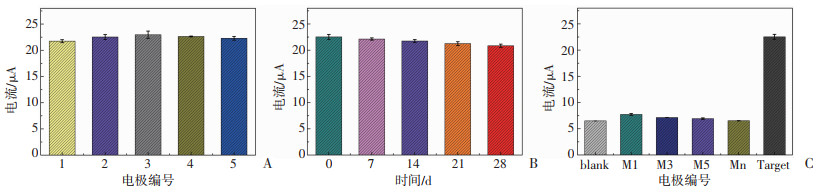
|
| A:重复性检测;B:稳定性分析;C:特异性研究 blank:未加互补序列;M1:一碱基错配序列;M3:三碱基错配序列;M5:五碱基错配序列;Mn:非互补序列;Target:Target 4 图 10 PEC传感器分析体系的性能 |
为评估该传感器的特异性,将TPN修饰后的工作电极分别与浓度为100 mmol/L的一碱基错配序列(M1)、三碱基错配序列(M3)、五碱基错配序列(M5)和非互补序列(Mn)杂交,检测其光电流信号。从图 10C可见:目标序列Target4的光电流响应值远高于其他干扰DNA序列,结果表明该传感器对目标序列的检测有较高的特异性。
3 讨论TiO2具有良好的载流子传输性能、高稳定性和低毒性等优点,被广泛应用于光电化学生物传感,但其带隙较宽(3.2 eV),仅在387.5 nm下有紫外活性,导致光转换率低[14]。金纳米粒子是一种常见的贵金属,可产生表面等离子体共振效应从而提高TiO2的光电转换效率[15]。为扩大吸收范围,引入AuNPs对TiO2进行修饰,可有效抑制电子-空穴对的复合,提高光电转换效率,从而产生强而稳定的光电流信号[16]。同时,本研究采用共敏化放大策略来增强实验中的光电流信号。CdTe QDs具有优异的光物理特性,可充分利用光能,能有效促进载流子分离[17]。由于CdTe QDs带隙比AuNPs/TiO2窄,CdTe QD对AuNPs/TiO2有敏化作用,可增强传感器的光电流信号响应。
与细胞内DNA杂交不同,体外DNA检测中,ssDNA的无序构象限制DNA的杂交过程,导致杂交动力学低、结合效率低和特异性低[18]。近年来,纳米机械力的应用促进了体外DNA分析设计的发展,如生物传感器和生物芯片可灵敏地检测生物分子,被广泛用于精准医学、生物研究和诊断[19]。本研究采用DNA三棱柱框架作为纳米机械手臂拉伸捕获探针,形成三维刚性DNA桥式纳米探针,可极大地提高生物传感器检测的准确性和特异性,为体外DNA甲基化丰度检测提供一种新方法。
目前检测DNA甲基化的经典方法是亚硫酸盐转化法和甲基化敏感的限制性内切酶消化(MSRE)法。亚硫酸氢盐测序法检测结果较为准确,但易出现假阳性结果,且目前无法统一认证检测结果。MSREs可识别和切割DNA甲基化特异性序列,但目前只有少数MSRE(例如HpaII和DpnI)可用,仅能检测特定的DNA甲基序列[20]。并且,目前大多数生物传感器是基于DNA甲基转移酶活性间接推算DNA甲基化水平,不能直接、准确地量化DNA甲基化水平。本研究采用三维刚性桥式DNA纳米探针为捕获探针,AuNPs/TiO2联合CdTe QDs为光活性材料构建光电化学传感器并用于DNA甲基化的定量检测。当靶序列存在时,三维刚性桥式DNA纳米探针通过碱基互补配对将其固定在电极表面,Ab-CdTe QDs偶联物通过5-甲基胞嘧啶抗体特异性识别5-甲基胞嘧啶连接到电极表面,CdTe QDs共敏化AuNPs/TiO2复合材料可促进电子从抗坏血酸电解质向电极表面转移,提高AuNPs/TiO2的光电流响应。在最佳实验条件下,随着甲基化位点数目的增多,结合到电极表面的Ab-CdTe QDs偶联物也增多,光电信号响应值随之增高,且变化值与DNA甲基化位点数量成正比,可对DNA甲基化位点数量进行检测分析,更换捕获探针序列后可检测其他未知的甲基化位点。所构建的光电化学传感器可对多甲基化位点进行分析,且具备准确性高、稳定性好和检测时间短等优点,有望应用于表观遗传类疾病和相关肿瘤的早期临床诊断。
| [1] |
HUANG J, ZHANG S, MO F, et al. An electrochemical DNA biosensor analytic technique for identifying DNA methylation specific sites and quantify DNA methylation level[J]. Biosens Bioelectron, 2019, 127: 155-160. |
| [2] |
PRASHER D, GREENWAY S C, SINGH R B. The impact of epigenetics on cardiovascular disease[J]. Biochem Cell Biol, 2020, 98(1): 12-22. |
| [3] |
ZHANG Y H, CHEN Y Y, QU H M, et al. Methylation of HIF3A promoter CpG Islands contributes to insulin resistance in gestational diabetes mellitus[J]. Mol Genet Genomic Med, 2019, 7(4): e00583. |
| [4] |
WEI B, WU F X, XING W Q, et al. A panel of DNA methylation biomarkers for detection and improving diagnostic efficiency of lung cancer[J]. Sci Rep, 2021, 11(1): 16782. |
| [5] |
ZHENG Y C, HUANG Q Q, DING Z J, et al. Genome-wide DNA methylation analysis identifies candidate epigenetic markers and drivers of hepatocellular carcinoma[J]. Brief Bioinform, 2018, 19(1): 101-108. |
| [6] |
SALTA S, MAIA-MOÇO L, ESTEVÃO-PEREIRA H, et al. Performance of DNA methylation-based biomarkers in the cervical cancer screening program of northern Portugal: a feasibility study[J]. Int J Cancer, 2021, 149(11): 1916-1925. |
| [7] |
WANG H Y, YIN H S, HUANG H, et al. Dual-signal amplified photoelectrochemical biosensor for detection of N6-methyladenosine based on BiVO4-110-TiO2 heterojunction, Ag+-mediated cytosine pairs[J]. Biosens Bioelectron, 2018, 108: 89-96. |
| [8] |
XIE Y Z, ZHANG M, BIN Q Y, et al. Photoelectrochemical immunosensor based on CdSe@BiVO4 co-sensitized TiO2 for carcinoembryonic antigen[J]. Biosens Bioelectron, 2020, 150: 111949. |
| [9] |
HOU T, XU N N, WANG W X, et al. Label-free and immobilization-free photoelectrochemical biosensing strategy using methylene blue in homogeneous solution as signal probe for facile DNA methyltransferase activity assay[J]. Biosens Bioelectron, 2019, 141: 111395. |
| [10] |
HAQUE M H, GOPALAN V, ISLAM M N, et al. Quantification of gene-specific DNA methylation in oesophageal cancer via electrochemistry[J]. Anal Chim Acta, 2017, 976: 84-93. |
| [11] |
LIN M H, WANG J J, ZHOU G B, et al. Programmable engineering of a biosensing interface with tetrahedral DNA nanostructures for ultrasensitive DNA detection[J]. Angew Chem Int Ed Engl, 2015, 54(7): 2151-2155. |
| [12] |
LI F Q, MAO X H, LI F, et al. Ultrafast DNA sensors with DNA framework-bridged hybridization reactions[J]. J Am Chem Soc, 2020, 142(22): 9975-9981. |
| [13] |
DING M L, WANG K, FANG M, et al. MPA-CdTe quantum dots as "on-off-on" sensitive fluorescence probe to detect ascorbic acid via redox reaction[J]. Spectrochim Acta A Mol Biomol Spectrosc, 2020, 234: 118249. |
| [14] |
JIAO W, ZHU J X, LING Y, et al. Photoelectrochemical properties of MOF-induced surface-modified TiO2 photoelectrode[J]. Nanoscale, 2018, 10(43): 20339-20346. |
| [15] |
LIU X P, CHEN J S, MAO C J, et al. Enhanced photoelectrochemical DNA sensor based on TiO2/Au hybrid structure[J]. Biosens Bioelectron, 2018, 116: 23-29. |
| [16] |
SHI H M, ZHANG S, ZHU X P, et al. Uniform gold- nanoparticle-decorated{001}-faceted anatase TiO2 nanosheets for enhanced solar-light photocatalytic reactions[J]. ACS Appl Mater Interfaces, 2017, 9(42): 36907-36916. |
| [17] |
SUN B, DONG J, CUI L, et al. A dual signal-on photoelectrochemical immunosensor for sensitively detecting target avian viruses based on AuNPs/g-C3 N4 coupling with CdTe quantum dots and in situ enzymatic generation of electron donor[J]. Biosens Bioelectron, 2019, 124/125: 1-7. |
| [18] |
GUO C L, HARRIS N C, WIJERATNE S S, et al. Multiscale mechanobiology: mechanics at the molecular, cellular, and tissue levels[J]. Cell Biosci, 2013, 3(1): 25. |
| [19] |
BLANCHARD A T, SALAITA K. Emerging uses of DNA mechanical devices[J]. Science, 2019, 365(6458): 1080-1081. |
| [20] |
ZHANG Q, WU Y X, XU Q F, et al. Recent advances in biosensors for in vitro detection and in vivo imaging of DNA methylation[J]. Biosens Bioelectron, 2021, 171: 112712. |




