心肌致密化不全(left ventricular noncompaction,LVNC)是儿童常见的一种心肌病,由心脏发育早期时心肌致密化过程迟滞或停顿造成,以心室内突出的肌小梁和众多深陷的小梁隐窝为病理特征[1],主要临床表现为进行性心力衰竭、心律失常和栓塞形成,严重者甚至发生猝死[2]。该病是公认的遗传性心肌病,但遗传学发生机制尚未阐明。
Tafazzin (TAZ/G4.5)是文献报道的第一个LVNC的致病基因[3]。而近年来随着对LVNC研究的深入,越来越多与LVNC致病相关的基因被发现,包括肌节编码基因βMHC/MYH7、MYBPC3、ACTC1、MYL2、MYL3、Dtna、TNNT2、TNNI3、LMNA、ZASP/LBD3、DTNA、XIRP1等[4-5],编码离子通道的基因SCN5A、HCN4等[6-7],但这些基因对疾病的解释度仅为20%~30%[8-9]。
LVNC被定义为异质性的遗传性心肌病,诸多未知基因参与该疾病的发生发展过程。本研究通过临床收集的3个具有典型临床表现的LVNC核心家系,对其血液样本进行全外显子测序(whole-exome sequencing,WES),试图扩展致病基因的范围和发生机制;经过生物信息学分析,筛选出3个家系均可能致病基因的交集,缩小可能致病基因范围,利用已成功构建的LVNC患者和正常人来源的诱导多能干细胞-心肌细胞(induced pluripotent stem cells-cardiomyocytes,iPSC-CMs)提取RNA[10-11],采用qPCR技术对这些基因在心肌分化过程中的表达量及表达差异进行验证,为下一步在分子细胞和模式动物水平进行验证并阐述发生机制奠定基础。
1 材料与方法 1.1 病例来源3个LVNC家系来自2018年1月至2020年12月于重庆医科大学附属儿童医院门诊就诊的患儿。第1例女性(即KC2018-C078),16岁,以心肌致密化不全就诊,并伴有肺动脉高压;第2例男性(即KC2020-E160),6岁,以心肌致密化不全就诊,并伴有慢性心衰、频发室性早搏;第3例女性(即KC2020-E353),3岁,以心肌致密化不全就诊,并伴有慢性心衰。诊断标准:Jenni超声心动图:①双层心肌,收缩末期有多个突出的小梁。②NC/C>2 ∶1。③彩色多普勒显示与小梁间隙之间相互联通。④同时没有并存的心脏异常[12]。STEFFEN等[13]提出的MRI:①两个不同心肌层的视觉表现——致密的心外膜层和非致密的心内膜层。②非致密层内存在粗大小梁和深陷的小梁隐窝。③在舒张末期测量非致密心肌:致密心肌>2.3。分别收集LVNC患儿及其父母兄妹的外周血液。本研究通过重庆医科大学伦理委员会审查[(2021)年伦审(预)第(19)号]。
1.2 全外显子测序将上述3个LVNC家系的外周血液送检武汉康测科技有限公司进行WES。根据测序流程,将这3个家系外周血的gDNA制备外显子组富集文库,再通过Illumina Hiseq平台,以2×150 bp双端测序模式进行高通量测序,获得FastQ的原始数据;对原始数据进行质量控制,运用BWA软件与正常基因组进行序列(人类基因组https://hgdownload.soe.ucsc.edu/downloads.html)比对,Picard软件去除低质量测序数据及重复数据,并使用GATK软件进行碱基校正,获得可进行分析的比对序列;采用GATK HaplotypeCaller软件(https://software.broadinstitute.org/gatk/best-practices/)检测每个样品的SNV/InDel,按照软件推荐的筛选方案进行过滤,建立SNV库,通过ANNOVAR软件(http://annovar.openbioinformatics.org/en/latest/) 将所有的SNV/InDel位点与最新发布的群体数据库、功能数据库、以及疾病数据库等已知信息进行比对分析,评估这些SNV/InDel位点的变异频率、功能特征、保守性、致病性等信息,并与多态性数据库进行变异过滤后,对这3个LVNC家系的基因取交集,3个家系均筛选出的可能致病基因。
1.3 主要试剂和设备基质胶、消化液、PGM1培养基、心肌诱导分化试剂盒、心肌消化液和纯化培养基均购自北京赛贝公司;1640基础培养基购自Sigma公司;RNA提取试剂盒购自北京博迈斯科技发展有限公司;反转录试剂盒购自湖南艾瑞科生物工程公司;qPCR八联管购自赛默飞(Thermo)公司;引物购自上海生工生物工程股份有限公司;Sybre购自美国Bimake生物科技有限公司。实验室常用设备由重庆医科大学附属儿童医院儿科研究所提供。
1.4 细胞培养为了模拟心肌细胞分化的过程,采用课题组前期构建的LVNC患者来源的iPSC-CMs(此LVNC患者具有心脏增大、心功能不全的临床表现,心脏彩超提示有粗大的肌小梁和深陷的隐窝,非致密层/致密层比值大于2.0,完全符合LVNC的诊断标准)和正常人来源的iPSC-CMs [10],分别记为疾病组和正常组。使用PGM1培养基和干细胞消化液、分化培养基、纯化培养基等进行培养、传代、分化[11];收集诱导分化过程中0(未进行诱导前的iPSCs)、10、20、30 d分化成熟的心肌细胞进行下一步实验。
1.5 RNA提取收集正常组、疾病组0、10、20、30 d的细胞分别进行RNA提取,提取过程参照RNA提取试剂盒(北京博迈斯科技发展有限公司)说明书进行,提取后RNA放入-80 ℃保存。
1.6 反转录将提取后的不同时间点的正常组和患者组的RNA进行浓度检测,提取产物检测D(260/280)值在1.8~2.1最佳,反转录操作步骤参照艾瑞克公司提供的PrimeScript RT reagent Kit with gDNA Eraser说明书进行。
1.7 qPCR检测基因mRNA的表达水平进入NCBI GenBank数据库,搜索所需基因mRNA序列,将mRNA序列发送至上海生工公司网站,合成引物序列后,通过NCBI primer Blast进行引物特异性验证,验证成功后由生工合成引物,引物序列见表 1。反应条件:95 ℃预变性2 min,95 ℃变性15 s,55~60 ℃退火30 s(根据引物不同,具体温度不同),72 ℃延伸30 s,上述步骤重复39个循环。
| 基因 | 引物序列(5′→3′) |
| ABCC8 | 正义: GGTGACCGAATCCCACCATC 反义: CAGGGCAATTAGCAGCTTGG |
| CACN2D1 | 正义: ACGCAGCAGTCCATATTCCTA 反义: GCCACAATAATGAAGGGTCTTCC |
| COL6A3 | 正义: CATAACCGCTGTGCGGAAAAT 反义: TCATCTAGGGACTTACCACCTG |
| CPAMD8 | 正义: TTAACTCTCCAAGGGAAGTCACG 反义: GGTCTGGTTGTGAAAGAGGGG |
| DCHS1 | 正义: GACATCAACGACCATGCTCCA 反义: GTGCCAAAAGCTGTATGCTCA |
| FAT1 | 正义: CATCCTGTCAAGATGGGTGTTT 反义: TCCGAGAATGTACTCTTCAGCTT |
| MUC4 | 正义: CCCTACCTCTGTGCCCTGTA 反义: AGTCCCCCAGCCCATTGAA |
| NEB | 正义: CACCAAGGATAAGTACCTGCTTC 反义: AGTTCCGGGCTATCATTATAGGG |
| PCDH15 | 正义: CTTCCTTGTGTCCTTGTGCC 反义: GCTTGGATTGGTGGCGTAAC |
| PDE4DIP | 正义: CTGCTTTCGCACGTCTTGG 反义: CAATCCGGGCAATAACTGTGT |
| PRUNE2 | 正义: CAACGCGCCAAATCTAAACTG 反义: GTTCAGCACTGGTAAACACAGA |
| TTN | 正义: CCCCATCGCCCATAAGACAC 反义: CCACGTAGCCCTCTTGCTTC |
| USH2A | 正义: AGATTGGGAGGACTGGCAATA 反义: TCCAGGTGTCAGGATGCTAAA |
| ZFHX4 | 正义: AAAACGGATGAGCGCAAAAGT 反义: TGGTAAACGATCTCCCCTGTTAG |
| GAPDH | 正义: CCCATCACCATCTTCCAGG 反义: CATCACGCCACAGTTTCCC |
1.8 统计学分析
以GAPDH基因作为内参,以2-ΔΔCt作为mRNA量化数值,计算基因的相对mRNA水平。每组实验至少重复3次,采用GraphPad Prism 8.3.0软件进行独立样本t检验并制图,P < 0.05为差异有统计学意义。
2 结果 2.1 LVNC家系外显子测序结果将收集到的3个LVNC家系[3个家系样本号分别为KC2018-C078(记为C078)、KC2020-E160(记为E160)、KC2020-E353(记为E353)]外周血样送检武汉康测科技有限公司进行WES,利用生物信息学分别对这3个家系所检测到的SNP(单核苷酸多态性)进行GO分析,包括生物过程(biological process,BP)、细胞组成(cellular component,CC)、分子功能(molecular function,MF)及KEGG通路分析。结果见图 1、2、3。
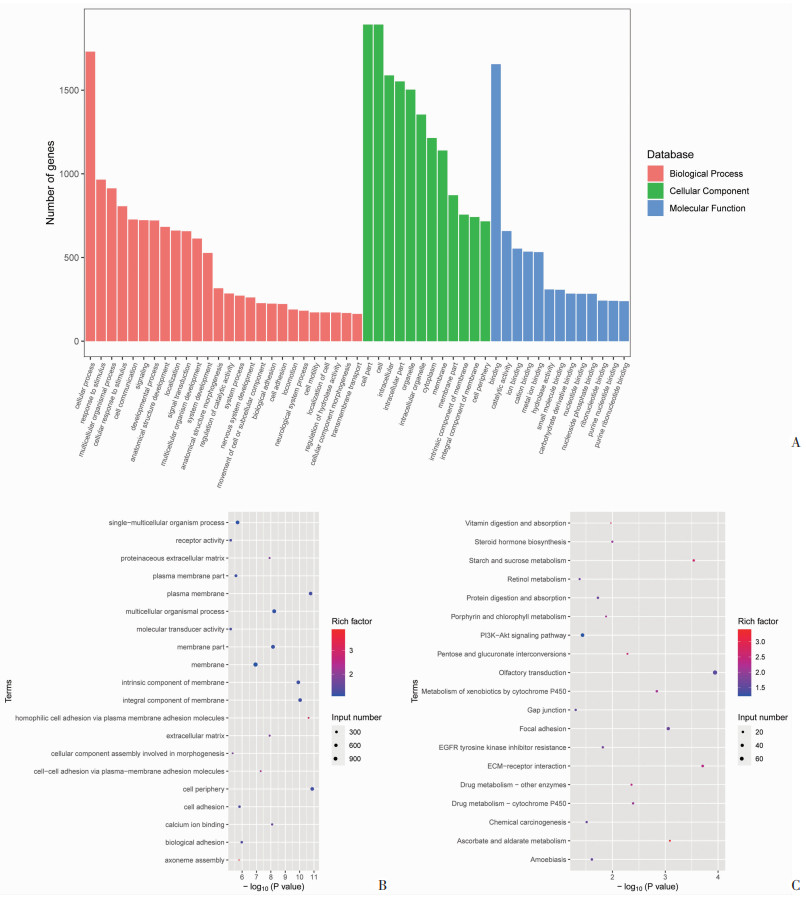
|
| A:GO分析显示生物过程、细胞组成、分子功能的基因富集情况;B:GO分析前20名富集基因的功能;C:KEGG通路分析显示前20名与富集基因关系密切的信号通道 图 1 C078家系WES的GO分析及KEGG通路基因富集情况 |
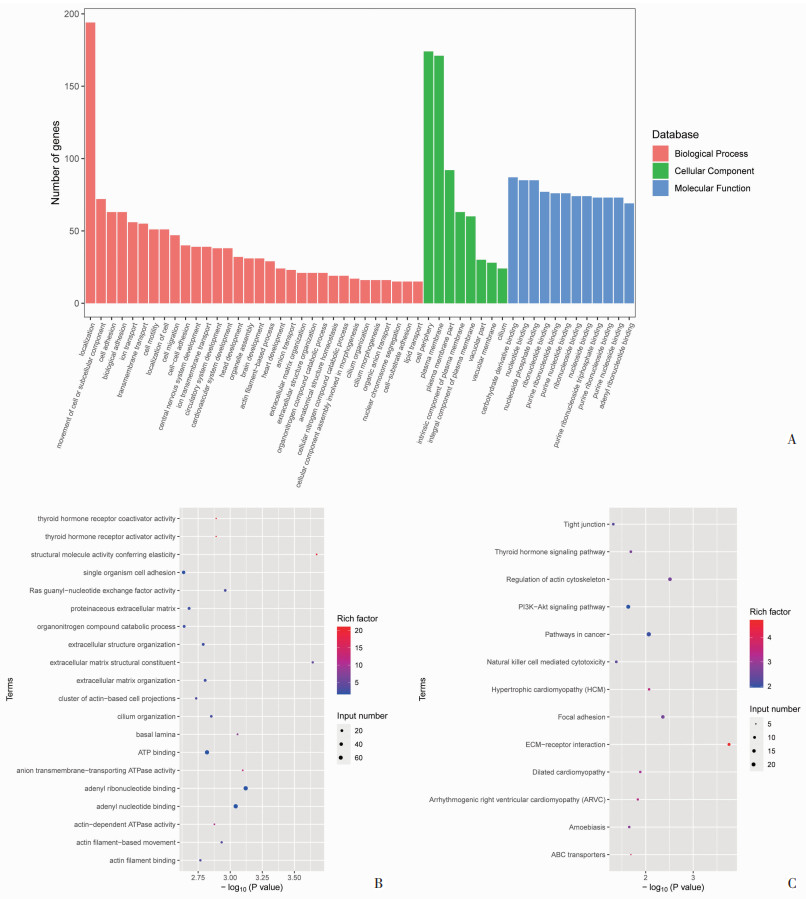
|
| A:GO分析显示生物过程、细胞组成、分子功能的基因富集情况;B:GO分析前20名富集基因的功能;C:KEGG通路分析显示前20名与富集基因关系密切的信号通道 图 2 E160家系WES的GO分析及KEGG通路基因富集情况 |
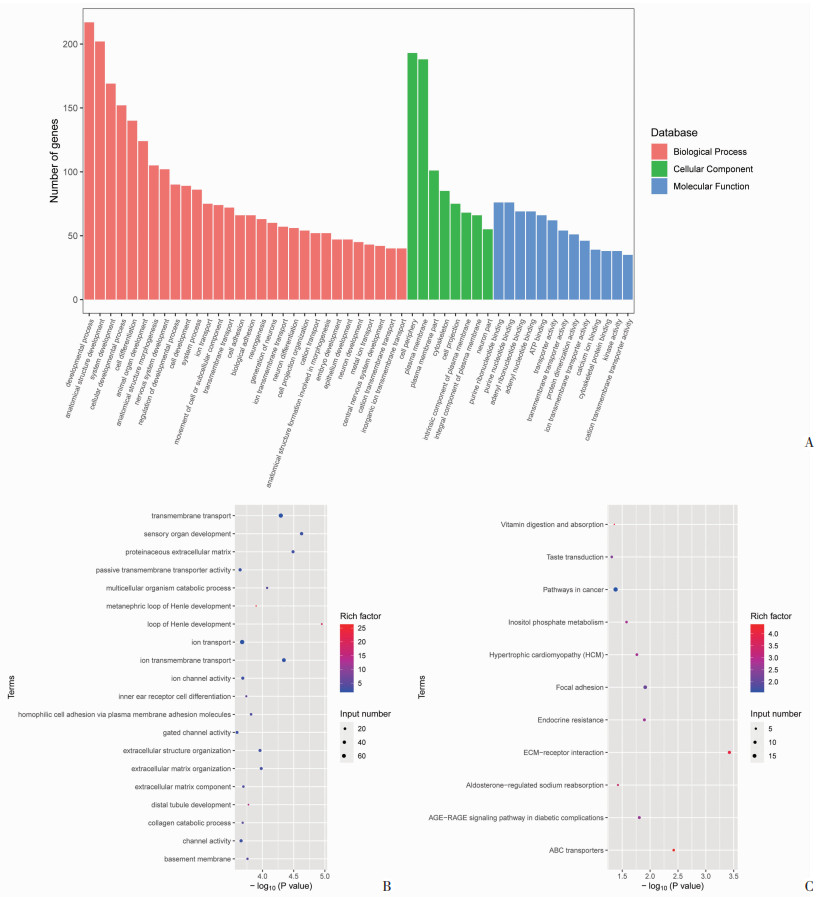
|
| A:GO分析显示生物过程、细胞组成、分子功能的基因富集情况;B:GO分析前20名富集基因的功能;C:KEGG通路分析显示前20名与富集基因关系密切的信号通道 图 3 E353家系WES的GO分析及KEGG通路基因富集情况 |
C078家系共检测到2 519个可能致病基因;E160家系共检测到585个可能致病基因;E353家系共检测到622个可能致病基因;三者可能致病基因列表取交集,制作韦恩(Venn)图(图 4),获得14个基因,分别为:ABCC8、CACNA2D1、COL6A3、CPAMD8、DCHS1、FAT1、MUC4、NEB、PCDH15、PDE4DIP、PRUNE2、TTN、USH2A、ZFHX4。
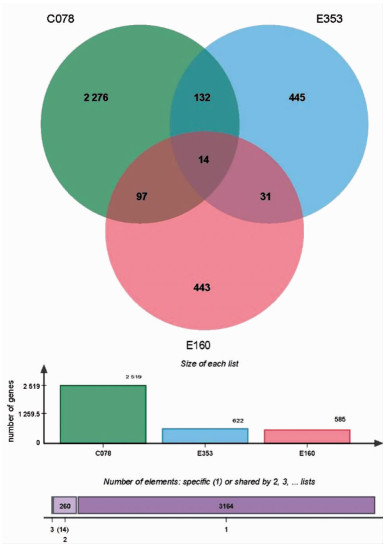
|
| 图 4 心肌致密化不全疾病三个家系可能致病基因交集韦恩(Venn)图 |
2.2 qPCR检测基因mRNA表达量差异
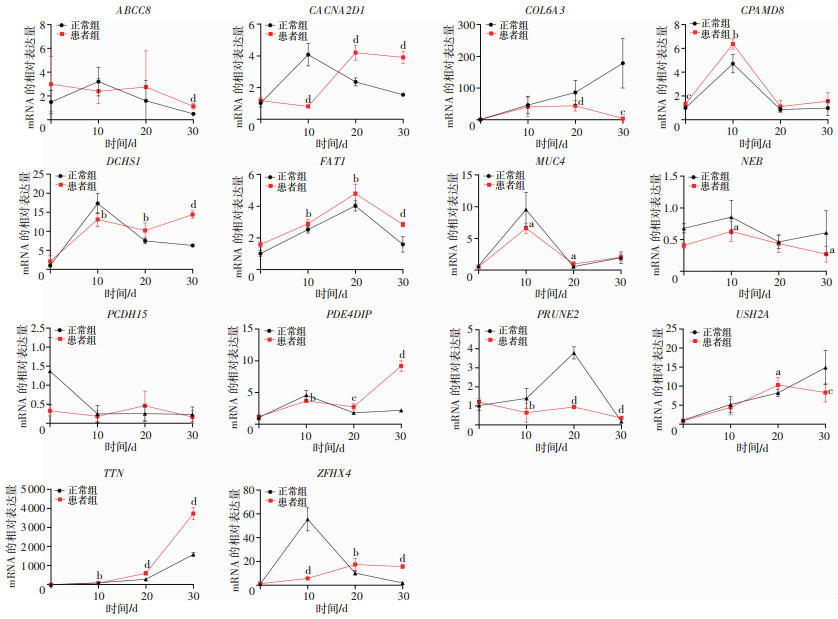
分别取正常组、疾病组的iPSCs到iPSC-CMs诱导过程中不同时间点(0、10、20、30 d)的细胞,提取RNA反转录为cDNA,对上述14个基因采用qPCR技术进行检测,各基因检测结果及统计分析如下所示(表 2),绘制折线图(图 5)。
| 基因名称 | 0 d | 10 d | 20 d | 30 d |
| ABCC8 | P=0.1229 | P=0.1499 | P=0.3384 | P < 0.0001 |
| CACNA2D1 | P=0.1621 | P < 0.0001 | P < 0.0001 | P < 0.0001 |
| COL6A3 | P=0.2165 | P=0.9653 | P < 0.0001 | P=0.0002 |
| CPAMD8 | P=0.0001 | P=0.0011 | P=0.1965 | P=0.0857 |
| DCHS1 | P=0.0455 | P=0.0014 | P=0.0015 | P < 0.0001 |
| FAT1 | P < 0.0001 | P=0.0054 | P=0.0054 | P < 0.0001 |
| MUC4 | P=0.1085 | P=0.0283 | P=0.0123 | P=0.8844 |
| NEB | P≤0.0001 | P=0.0455 | P=0.6465 | P=0.0208 |
| PCDH15 | P=0.0073 | P=0.5175 | P=0.1776 | P=0.4604 |
| PDE4DIP | P=0.4176 | P=0.0076 | P=0.0003 | P < 0.0001 |
| PRUNE2 | P=0.0930 | P=0.0065 | P < 0.0001 | P < 0.0001 |
| TTN | P=0.0506 | P=0.0011 | P < 0.0001 | P < 0.0001 |
| USH2A | P=0.2758 | P=0.4494 | P=0.0188 | P=0.0002 |
| ZFHX4 | P=0.0005 | P < 0.0001 | P=0.0028 | P < 0.0001 |
|
| a:P < 0.05,b:P < 0.01,c:P < 0.001, d:P < 0.0001,与正常组比较 图 5 14个基因在心肌致密化不全患者组与正常组在诱导多能干细胞-心肌细胞不同时间点mRNA相对表达量的变化折线图 |
结果显示,在诱导分化不同时间的基因表达方面,ABCC8、CACNA2D1、DCHS1、FAT1、PRUNE2、ZFHX4等基因随着分化时间延长,其mRNA的表达量先增加后降低;CPAMD8、MUC4、NEB、PDE4DIP等基因随着分化时间延长其mRNA的表达量先增加后降低再增加;COL6A3、PCDH15、USH2A等基因随着分化时间延长其mRNA的表达量增加;TTN基因随着分化时间延长其mRNA的表达量先降低后保持不变。在不同组别的基因表达量差异方面,ABCC8、CPAMD8基因正常组与疾病组的mRNA表达量无明显差异;MUC4、NEB、PCDH15等基因正常组与疾病组的mRNA表达量稍有差异;COL6A3、PRUNE2、USH2A等基因正常组与疾病组的mRNA表达量有明显或显著差异,且正常组表达量高于疾病组;CACNA2D1、DCHS1、PDE4DIP、ZFHX4等基因正常组与疾病组的mRNA表达量有明显或显著差异,且随着分化时间,正常组表达量先高于疾病组后低于疾病组;FAT1、TTN基因正常组与疾病组的mRNA表达量有明显或显著差异,且疾病组表达量高于正常组。以上qPCR验证结果提示CACNA2D1、COL6A3、DCHS1、FAT1、PDE4DIP、PRUNE2、TTN、USH2A、ZFHX4等基因在正常组及疾病组不同时间点mRNA表达量差异性较大,提示与心脏发育密切相关,是下一步验证LVNC发生机制的新的可能靶点基因。
3 讨论LVNC是小儿常见的原发性遗传性心肌病,年发病率为0.5%~2.4%。患者多年少发病,临床症状重、预后差、治疗手段局限,是威胁儿童健康的严重心血管疾病,对其病因和诊疗的探索已成为研究的热点和难点。但该病具体的遗传发生机制不明,既往基于全基因组关联分析或全外显子组关联分析的LVNC遗传学研究已发现数十个疾病相关的易感基因,包括与肌小节相关的基因、与离子通道相关的基因和与线粒体相关的基因等[8-9, 14]。由于LVNC为遗传异质性疾病,仍有许多的基因引起疾病的发生未被证实。
本研究通过外显子测序筛选出14个共同可能致病基因并进行qPCR验证,发现疾病组与正常组相比,上述基因mRNA在心肌诱导分化的不同时间点存在表达量的差异,可能为LVNC新的致病基因。查阅文献,发现PDE4DIP、NEB、ZFHX4、TTN与细胞骨架有关,其中PDE4DIP、TTN心脏表达量较高,NEB、ZFHX4心脏表达量较低。PDE4DIP的异常可能与扩张性心肌病、慢性房颤的发生相关[15-17];TTN的突变可能导致家族性扩张型心肌病[18];ZFHX4家族蛋白ZFHX3可能引起先天性心脏病[19];NEB编码的蛋白nebulin,是细胞骨架基质的一种巨大蛋白质成分,与骨骼肌肌节内的粗细丝共存[19]。CACNA2D1、DCHS1、FAT1、PCDH15参与钙离子的调节,与心脏传导系统相关,其异常可引起心律失常。CACNA2D1、DCHS1、FAT1在心脏中表达量较高,CACNA2D1的异常可能跟心室颤动[20]、J波综合征、短QT综合征等相关[21];FAT1可能是心肌缺血再灌注的标志物之一[22]及室间隔缺损的潜在致病基因[23];DCHS1异常可能跟细胞平面极性相关[24],它的突变可导致二尖瓣脱垂[25]。PCDH15在心脏表达量较低,但有文献报道此基因的异常可能跟扩张型心肌病相关[17]。ABCC8在心脏中表达量较低,该基因编码的蛋白质是ATP结合盒(ABC)转运蛋白超家族的成员,该蛋白起调节ATP敏感性钾通道和胰岛素释放的作用,文献报道它的缺失可能引起肺动脉高压和慢性心衰有关[26-27]。PRUNE2基因编码的蛋白质属于B细胞CLL/淋巴瘤2和腺病毒E1B 19 kDa相互作用家族,与抑制致癌细胞转化有关;其心脏表达量较少,但也有研究表明与心脏能量代谢相关[28]。MUC4编码细胞粘连蛋白4,在心脏表达量较低,有文献报道此基因是扩张型心肌病最常见的突变基因[29];还可能通过抑制细胞凋亡来促进肿瘤生长[30]。COL6A3编码α-3链,是Ⅵ型胶原蛋白的3个α链之一,目前主要发现它的突变跟Bethlem肌病和肌张力障碍有关[31],与心脏无明显关系,但该基因在心脏表达量较高。CPAMD8基因编码的蛋白是蛋白酶抑制剂I39蛋白家族的成员之一,在心脏表达量较低,主要跟先天性及获得性免疫相关,其突变可导致眼睛发育障碍[32]。USH2A基因在心脏表达量较低,编码的蛋白质包含层粘连蛋白EGF基序,存在于基底膜中,可能对内耳、视网膜的发育和体内稳态起重要作用[33-34]。
在心脏发生发育过程中,Wnt信号通路是关键调控因子之一。Wnt信号通路对斑马鱼、小鼠胚胎和小鼠胚胎干细胞的心脏发育具有双相作用,表现为早期激活Wnt信号通路,促进干细胞向中胚层发育;后期抑制Wnt信号通路,促进中胚层向心肌细胞分化[35]。我们在培养iPSC-CMs时,模拟此过程,先激活Wnt信号,后抑制Wnt信号,完成心肌分化过程[36]。在上述14个可能致病基因不同时间点mRNA表达量的检测中,随着心肌诱导分化时间的改变,表达量也随之改变,且正常组与疾病组存在差异,这可能表明这些基因可能影响心脏的发生发育引起LVNC的发生,且可能与Wnt通路有关,需要通过下一步的功能验证明确。
综上所述,3个LVNC家系的WES筛选出的14个共同可能致病基因,在iPSC-CMs分化过程中随着时间变化而改变,正常组与疾病组存在差异,且既往文献报道中涉及细胞骨架、细胞极性、钙离子及钾离子的调节、能量代谢等方面,因此这些基因的异常可能通过改变其涉及的细胞功能,影响心脏的发生发育最终导致LVNC的发生,需进一步进行基因位点改变及体内外功能实验验证。
| [1] |
LIU Y, CHEN H Y, SHOU W N. Potential common pathogenic pathways for the left ventricular noncompaction cardiomyopathy (LVNC)[J]. Pediatr Cardiol, 2018, 39(6): 1099-1106. |
| [2] |
UEDA Y, KAMIYA C A, NAKANISHI A, et al. Cardiomyopathy phenotypes and pregnancy outcomes with left ventricular noncompaction cardiomyopathy[J]. Int Heart J, 2018, 59(4): 862-867. |
| [3] |
HIRONO K, HATA Y, NAKAZAWA M, et al. Clinical and echocardiographic impact of tafazzin variants on dilated cardiomyopathy phenotype in left ventricular non-compaction patients in early infancy[J]. Circ J, 2018, 82(10): 2609-2618. |
| [4] |
TAKASAKI A, HIRONO K, HATA Y, et al. Sarcomere gene variants act as a genetic trigger underlying the development of left ventricular noncompaction[J]. Pediatr Res, 2018, 84(5): 733-742. |
| [5] |
MISZALSKI-JAMKA K, JEFFERIES J L, MAZUR W, et al. Novel genetic triggers and genotype-phenotype correlations in patients with left ventricular noncompaction[J]. Circ Cardiovasc Genet, 2017, 10(4): e001763. |
| [6] |
ISHIKAWA T, OHNO S, MURAKAMI T, et al. Sick sinus syndrome with HCN4 mutations shows early onset and frequent association with atrial fibrillation and left ventricular noncompaction[J]. Heart Rhythm, 2017, 14(5): 717-724. |
| [7] |
ALONSO-FERNÁNDEZ-GATTA M, GALLEGO-DELGADO M, CABALLERO R, et al. A rare HCN4 variant with combined sinus bradycardia, left atrial dilatation, and hypertrabeculation/left ventricular noncompaction phenotype[J]. Rev Esp Cardiol (Engl Ed), 2021, 74(9): 781-789. |
| [8] |
TOWBIN J A, LORTS A, JEFFERIES J L. Left ventricular non-compaction cardiomyopathy[J]. Lancet, 2015, 386(9995): 813-825. |
| [9] |
TOWBIN J A. Inherited cardiomyopathies[J]. Circ J, 2014, 78(10): 2347-2356. |
| [10] |
刘奥怡, 张蕾, 龚梦嘉, 等. 心肌致密化不全患者特异性诱导多潜能干细胞及心肌样细胞的构建[J]. 第三军医大学学报, 2019, 41(24): 2401-2408. LIU A Y, ZHANG L, GONG M J, et al. Generation of induced pluripotent stem cells and cardiac myocyte-like cells from a patient with noncompaction of ventricular myocardium[J]. J Third Mil Med Univ, 2019, 41(24): 2401-2408. |
| [11] |
郭芸, 葛梅, 刘奥怡, 等. 细胞骨架及极性在心肌致密化不全患者来源心肌样细胞中的改变[J]. 第三军医大学学报, 2021, 43(9): 822-829. GUO Y, GE M, LIU A Y, et al. Changes of cytoskeleton and polarity in cardiomyocyte-like cells derived from left ventricular noncompaction patients[J]. J Third Mil Med Univ, 2021, 43(9): 822-829. |
| [12] |
JENNI R, OECHSLIN E, SCHNEIDER J, et al. Echocardiographic and pathoanatomical characteristics of isolated left ventricular non-compaction: a step towards classification as a distinct cardiomyopathy[J]. Heart, 2001, 86: 666-671. |
| [13] |
STEFFEN E, PETERSEN, JOSEPH B, et al. Left ventricular non-compaction: insights from cardiovascular magnetic resonance imaging[J]. Am Coll Cardiol, 2005, 46(1): 101-105. |
| [14] |
UDEOJI D U, PHILIP K J, MORRISSEY R P, et al. Left ventricular noncompaction cardiomyopathy: updated review[J]. Ther Adv Cardiovasc Dis, 2013, 7(5): 260-273. |
| [15] |
LOMBARDO B, D'ARGENIO V, MONDA E, et al. Genetic analysis resolves differential diagnosis of a familial syndromic dilated cardiomyopathy: a new case of Alström syndrome[J]. Mol Genet Genomic Med, 2020, 8(7): e1260. |
| [16] |
OSOEGAWA K, IOVANNISCI D M, LIN B, et al. Identification of novel candidate gene loci and increased sex chromosome aneuploidy among infants with conotruncal heart defects[J]. Am J Med Genet A, 2014, 164A(2): 397-406. |
| [17] |
ABOU ZIKI M D, BHAT N, NEOGI A, et al. Epistatic interaction of PDE4DIP and DES mutations in familial atrial fibrillation with slow conduction[J]. Hum Mutat, 2021, 42(10): 1279-1293. |
| [18] |
BUONOCORE F, CLIFFORD-MOBLEY O, KING T F J, et al. Next-generation sequencing reveals novel genetic variants (SRY, DMRT1, NR5A1, DHH, DHX37) in adults with 46, XY DSD[J]. J Endocr Soc, 2019, 3(12): 2341-2360. |
| [19] |
KISS B, GOHLKE J, TONINO P, et al. Nebulin and Lmod2 are critical for specifying thin-filament length in skeletal muscle[J]. Sci Adv, 2020, 6(46): eabc1992. |
| [20] |
BLANCARD M, DEBBICHE A, KATO K, et al. A loss-of-function CACNA1C variant, p.T1787M, associated with risk of ventricular fibrillation in Africans[J]. Arch Cardiovasc Dis Suppl, 2018, 10(2): 194. |
| [21] |
HANCOX J C, WHITTAKER D G, DU C Y, et al. Emerging therapeutic targets in the short QT syndrome[J]. Expert Opin Ther Targets, 2018, 22(5): 439-451. |
| [22] |
ZHENG A H, CAO L, QIN S, et al. Exenatide regulates substrate preferences through the p38γ MAPK pathway after ischaemia/reperfusion injury in a rat heart[J]. Heart Lung Circ, 2017, 26(4): 404-412. |
| [23] |
FU F, DENG Q, LEI T Y, et al. Clinical application of SNP array analysis in fetuses with ventricular septal defects and normal karyotypes[J]. Arch Gynecol Obstet, 2017, 296(5): 929-940. |
| [24] |
RAO-BHATIA A, ZHU M, YIN W C, et al. Hedgehog-activated Fat4 and PCP pathways mediate mesenchymal cell clustering and villus formation in gut development[J]. Dev Cell, 2020, 52(5): 647-658. |
| [25] |
VAN WIJNGAARDEN A L, HIEMSTRA Y L, KOOPMANN T T, et al. Identification of known and unknown genes associated with mitral valve prolapse using an exome slice methodology[J]. J Med Genet, 2020, 57(12): 843-850. |
| [26] |
BOHNEN M S, MA L J, ZHU N, et al. Loss-of-function ABCC8 mutations in pulmonary arterial hypertension[J]. Circ Genom Precis Med, 2018, 11(10): e002087. |
| [27] |
STRUTYNSKYI R B, VORONKOV L G, NAGIBIN V S, et al. Changes of the echocardiographic parameters in chronic heart failure patients with Ile337val, Glu23lys, and Ser1369ala polymorphisms of genes encoding the ATP-sensitive potassium channels subunits in the Ukrainian population[J]. Ann Hum Genet, 2018, 82(5): 272-279. |
| [28] |
SONG Y, AHN J, SUH Y, et al. Identification of novel tissue-specific genes by analysis of microarray databases: a human and mouse model[J]. PLoS One, 2013, 8(5): e64483. |
| [29] |
LIU L, HUANG J J, LIU Y, et al. Multiomics analysis of transcriptome, epigenome, and genome uncovers putative mechanisms for dilated cardiomyopathy[J]. Biomed Res Int, 2021, 2021: 6653802. |
| [30] |
LEE S R, MUN J Y, JEONG M S, et al. Thymoquinone-induced tristetraprolin inhibits tumor growth and metastasis through destabilization of MUC4 mRNA[J]. Int J Mol Sci, 2019, 20(11): E2614. |
| [31] |
BAZRAFSHAN S, KUSHLAF H, KAKROO M, et al. Genetic modifiers of hereditary neuromuscular disorders and cardiomyopathy[J]. Cells, 2021, 10(2): 349. |
| [32] |
WIGGS J L. CPAMD8, a new gene for anterior segment dysgenesis and childhood glaucoma[J]. Ophthalmology, 2020, 127(6): 767-768. |
| [33] |
VILLANUEVA A, BISWAS P, KISHABA K, et al. Identification of the genetic determinants responsible for retinal degeneration in families of Mexican descent[J]. Ophthalmic Genet, 2018, 39(1): 73-79. |
| [34] |
MAURIAC S A, GÉLÉOC G S G. A hop, skip, and a jump to evade USH2A deaf-blindness mutations[J]. Mol Ther, 2021, 29(8): 2391-2393. |
| [35] |
ZHAO M, TANG Y W, ZHOU Y, et al. Deciphering Role of Wnt Signalling in Cardiac Mesoderm and Cardiomyocyte Differentiation from Human iPSCs: four-dimensional control of Wnt pathway for hiPSC-CMs differentiation[J]. Sci Rep, 2019, 9(1): 19389. |
| [36] |
ZHANG X Y, YE L, XU H, et al. NRF2 is required for structural and metabolic maturation of human induced pluripotent stem cell-derived ardiomyocytes[J]. Stem Cell Res Ther, 2021, 12(1): 208. |




