2. 400037 重庆,陆军军医大学(第三军医大学)第二附属医院肿瘤科;
3. 400010 重庆,重庆医科大学附属第二医院肿瘤科;
4. 401120 重庆,重庆医科大学附属第三医院肿瘤科
2. Department of Oncology, Second Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400037;
3. Department of Oncology, the Second Affiliated Hospital of Chongqing Medical University, Chongqing, 400010;
4. Department of Oncology, the Third Affiliated Hospital of Chongqing Medical University, Chongqing, 401120, China
鼻咽癌好发于东南亚,是一种具有明显地域分布差异的恶性肿瘤。我国的鼻咽癌发病率占世界的47%,居头颈部恶性肿瘤首位;南方省份发病率较高,北方省份发病率相对较低[1]。由于鼻咽紧邻颅底、位置较深、邻近结构复杂,鼻咽肿瘤难以通过手术完整切除,所以放射治疗成为鼻咽癌根治性治疗的主要手段。近年,精准调强放射治疗(intensity modulated radiation therapy,IMRT),已成为鼻咽癌标准治疗方式[2-3]。因此,精确的靶区勾画对于鼻咽癌患者治疗疗效起着至关重要的作用。然而,鼻咽癌临床靶区(clinical target volume,CTV)勾画范围广,且多数为重复性勾画,耗时费力[4]。
随着对肿瘤放射治疗认识的不断提高,我国开展肿瘤放疗的医疗机构剧增,基层医院亦开始普及放疗设备配置。但基层医院所配置的放疗设备多为国产直线加速器,因技术参差不齐,不同医生或同一医生不同时间勾画的靶区存在差异,导致放疗效果相差较大[5-9]。近年,国内外关于鼻咽癌放疗靶区和危及器官的自动勾画研究越来越多,自动勾画能提高靶区勾画的准确性和一致性,从而提升鼻咽癌放疗效率[10-14]。本研究旨在通过小样本人工智能(artificial intelligence,AI) 技术训练一套鼻咽癌临床靶区自动勾画模型(即AI+CTV模型),以期减少不同层级医疗机构、不同技术层次放疗医生间鼻咽癌CTV勾画的差异,缩短放疗医生鼻咽癌CTV勾画时间,实现不同层级放疗单位鼻咽癌临床靶区勾画的规范化、标准化、同质化,提高工作效率。
1 材料与方法 1.1 入组条件收集陆军特色医学中心肿瘤科放疗中心自2020年9月至2021年5月收治的鼻咽癌患者,筛选出分期较早(T1~2N2)且淋巴结较小(≤2 cm)的30例患者,其中男性20例,女性10例;中位年龄52岁。本研究已获得陆军特色医学中心伦理委员会批准[批文号:2021第181号]。
1.2 定位患者均采用头颈肩热塑膜和合适的U型枕固定,头稍向后仰,双手置于体侧,采用飞利浦大孔径模拟CT定位。扫描范围:头顶至胸锁关节下2 cm;扫描层厚:3 mm。
1.3 临床靶区规范勾画依据以2013年颈部淋巴结新分区[15]、2017年鼻咽癌CTV勾画国际指南[16]和头颈部肿瘤放射治疗图谱[17]作为鼻咽癌放疗靶区勾画规范。由1名有经验的医师(10年头颈部放疗靶区勾画经验)按以上指南在Monaco上进行规范勾画CTV,包括高危CTV(CTV1)和低危CTV(CTV2)的勾画。CTV1包括鼻咽原发灶的CTV[即GTVnx及其周围5 mm范围的亚临床病灶区域以及双侧圆孔、卵圆孔、破裂孔、岩尖1/2、后组筛窦(包括梨骨)、鼻腔和上颌窦后部、翼腭窝、咽旁间隙、蝶窦下1/2和斜坡前1/3]和颈部淋巴结的CTV(咽后、Ⅱ、Ⅲ、Ⅴa淋巴引流区)。CTV2为需预防照射的淋巴引流区即Ⅳ和Ⅴb淋巴引流区[18]。30例手动勾画CTV最后经3名有经验的头颈部肿瘤放疗医师(15年以上头颈部放疗靶区勾画经验)一致审核同意。分别选取本院10例(男性7例,女性3例;中位年龄54岁)和3所教学医院6例[病例1(男,35岁)、病例2(女,47岁)来自陆军军医大学第二附属医院,病例3(男,56岁)、病例4(女,45岁)来自重庆医科大学附属第二医院,病例5(男,58岁)、病例6(男,37岁)来自重庆医科大学附属第三医院]分期较早(T1~2N2)且淋巴结较小(≤2 cm)的鼻咽癌,按以上规范勾画并审核。
1.4 AI+CTV勾画模型训练将手动勾画完成的30例靶区传输至AccuLearning深度学习自主训练平台,按8 ∶1 ∶1的比例随机分为训练集(24例)、验证集(3例)和测试集(3例)。在训练之前,AccuLearning将检查所有转换后的数据并对数据进行预处理。然后采用UNet网络模型和Dice损失函数作为AccuLearning深度学习训练方式,进入训练循环,随机选择一个案例,依次执行数据增强和窗口采样以获得样本,将样本输入到神经网络中进行正向传播,然后计算损失函数并进行优化。每经过一定次数的迭代,训练器将进行验证并根据验证损失选择最佳模型。迭代次数达到预设值后,训练终止,并输出最佳模型,即AI+CTV模型。
1.5 内部验证和外部验证随后将训练好的模型传至AccuContour平台,分别对本院10例手动勾画患者进行AI勾画,对比分析AI勾画与手动勾画的各项指标。包括:①相似性系数(dice similarity coefficient,DSC),即预测体积和参考体积的重叠部分的dice系数,1.0为最佳值,0为最差值。②豪斯多夫距离(Hausdorff distance,HD),即预测体积和参考体积之间的最近点的最大距离,0为最佳,无限大(infinity,INF)为最差。③平均表面距离(average symmetric surface distance,ASSD)即预测表面点集中所有点到参考表面点集的最小欧氏距离,并计算所有距离的平均值,0为最佳,INF为最差。④最大表面距离(maximum symmetric surface distance,MSSD),即预测表面点集中所有点到参考表面点集的最小欧氏距离,并计算出所有距离的最大值,0为最佳,INF为最差。⑤相对/绝对体积比(relative absolute volume difference,RAVD)即预测体积和参考体积的非重叠部分的相对系数,0为最佳,100%为最差[18]。以评估AI勾画的可行性。再分别对三所教学医院的6例鼻咽癌进行AI勾画,并对比分析以上指标。
1.6 统计学方法5个指标均采用x±s表示,采用配对t检验分析5个指标在CTV1与CTV2间的差异。6例教学医院手动勾画验证结果采用Kruskal-Wallis H检验。P < 0.05为差异具有统计学意义。
2 结果 2.1 模型训练结果该AI+CTV模型的平均DSC值为0.865,其中测试患者1的CTV1和CTV2 DSC值分别为0.906和0.866,测试患者2的CTV1和CTV2 DSC值分别为0.886和0.737,测试患者3的CTV1和CTV2 DSC值分别为0.922和0.871。将该AI+CTV勾画模型应用于临床鼻咽癌患者勾画时,发现其在解剖结构识别方面表现出明显的优势(图 1),比如对颅底孔道如圆孔(图 1C)和卵圆孔(图 1E)、肩胛舌骨肌(图 1F)及静脉角(图 1G)等解剖结构的识别较人工勾画好;但该勾画模型对于较大原发灶(图 2A)、较大淋巴结(>2 cm)(图 2B)识别仍不够,且在环状软骨交界层面存在CTV1和CTV2共存现象(图 2C)。
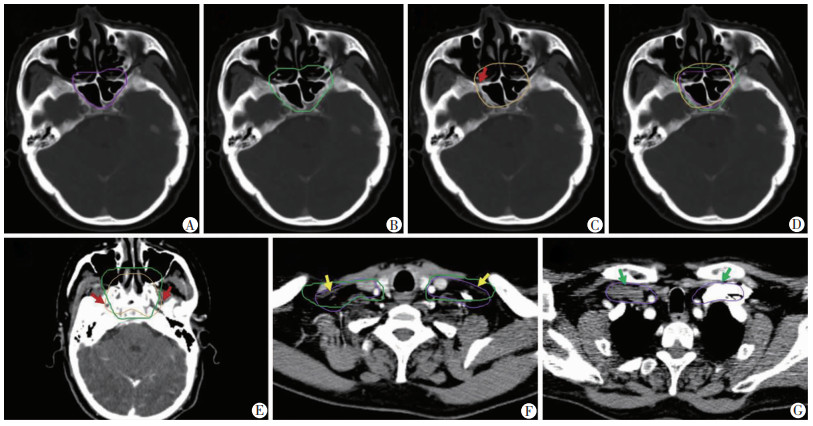
|
| A:人工勾画CTV1 (紫色示右外侧界与右侧圆孔重叠);B:人工勾画CTV2 (绿色示右外侧界在右侧圆孔外3 mm);C:AI+CTV勾画CTV1 (浅黄色示右外侧界壁刚好在右侧圆孔外侧壁,↑:示右侧圆孔);D:A、B、C 3条CTV重合对比;E:AI+CTV勾画准确识别卵圆孔(浅黄色示AI勾画CTV1,绿色示人工勾画,↑:示双侧卵圆孔);F:AI+CTV勾画准确识别肩胛舌骨肌(紫色示AI勾画CTV2,绿色示人工勾画,↑:示双侧肩胛舌骨肌);G:AI+CTV勾画准确识别静脉角(紫色示AI勾画CTV2,该层面人工勾画已结束,↑:示双侧静脉角) 图 1 AI+CTV勾画模型在CT图像上所示颅底孔道、肩胛舌骨肌及静脉角层面勾画展示 |
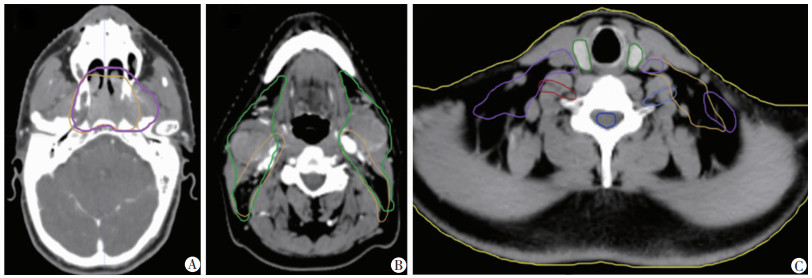
|
| A:原发灶 (浅黄色示AI+CTV勾画,紫色示人工勾画);B:淋巴结 (浅黄色示AI+CTV勾画,绿色示人工勾画);C:环状软骨下缘交界层面 (浅黄色示AI+CTV勾画CTV1,紫色示AI+CTV勾画CTV2) 图 2 AI+CTV勾画模型在CT图像上对较大原发灶、较大淋巴结患者及环状软骨交界层面的勾画展示 |
2.2 内部验证结果
利用该AI+CTV模型对本院10例患者进行AI勾画,以手动勾画为标准,内部验证该模型勾画CTV(CTV1和CTV2)的准确性。结果显示:CTV1和CTV2的中位DSC值均较高,均高于0.8,分别为0.886和0.842,且CTV1较CTV2更高。CTV1和CTV2的中位HD值均较小,分别为7.789、6.144 mm。CTV1和CTV2的中位ASSD、MSSD值均较小,分别1.683、20.52 mm和1.467、14.03 mm。CTV1和CTV2的中位RAVD值分别为16.30%和15.14%(表 1)。
| 评估参数 | CTV1 | CTV2 | t | P |
| DSC | 0.886±0.045 | 0.842±0.034 | 3.122 | 0.012 |
| HD/mm | 7.789±5.440 | 6.144±3.180 | 1.733 | 0.117 |
| ASSD/mm | 1.683±0.970 | 1.467±0.650 | 0.902 | 0.391 |
| MSSD/mm | 20.52±12.70 | 14.03±7.18 | 2.135 | 0.062 |
| RAVD(%) | 16.30±10.70 | 15.14±9.69 | 0.235 | 0.819 |
2.3 外部验证结果
利用该AI+CTV模型对三所教学医院的6例鼻咽癌进行AI勾画,以手动勾画为标准,外部验证该模型勾画CTV(CTV1和CTV2)的准确性。结果显示:CTV1勾画准确性高于CTV2。除重庆医科大学附属第二医院1例患者的CTV2-DSC值接近0.7外,6例患者CTV1和其余5例患者CTV1勾画一致性较好(DSC值均>0.7)。三所教学医院CTV1和CTV2的具体HD值、ASSD、MSSD和ABVD见表 2。
| 病例 | DSC | HD/mm | ASSD/mm | MSSD/mm | RAVD(%) | |||||||||
| CTV1 | CTV2 | CTV1 | CTV2 | CTV1 | CTV2 | CTV1 | CTV2 | CTV1 | CTV2 | |||||
| 病例1 | 0.854 | 0.720 | 5.244 | 8.000 | 1.602 | 2.450 | 17.208 | 15.297 | 9.678 | 11.443 | ||||
| 病例2 | 0.806 | 0.751 | 7.873 | 6.000 | 2.202 | 1.347 | 15.783 | 10.394 | 10.239 | 0.067 | ||||
| 病例3 | 0.853 | 0.631 | 9.000 | 13.106 | 1.918 | 4.089 | 15.613 | 23.040 | 7.704 | 14.710 | ||||
| 病例4 | 0.797 | 0.696 | 24.642 | 6.516 | 28.164 | 2.185 | 28.345 | 18.907 | 34.248 | 2.061 | ||||
| 病例5 | 0.850 | 0.729 | 6.250 | 8.880 | 1.760 | 2.490 | 15.030 | 18.590 | 6.370 | 7.280 | ||||
| 病例6 | 0.720 | 0.721 | 18.740 | 6.750 | 5.380 | 2.460 | 47.710 | 18.000 | 65.000 | 17.980 | ||||
3 讨论
本研究所训练的AI+CTV模型平均DSC值高达0.865,与最近报道的鼻咽癌CTV自动勾画准确性相当(DSC值:0.86)[19],内部(DSC均大于0.8)及外部验证(DSC值接近或超过0.7)结果均较好。先前研究报道,DSC值超过0.7即表示具有较好的重叠一致性,DSC值超过0.8则表明临床应用可行[20]。另外,研究发现该AI+CTV模型对解剖辨识度较高,能准确识别如圆孔、卵圆孔和静脉角等解剖结构,可用于分期较早(T1~2N2)且淋巴结较小(≤2 cm)患者CTV AI勾画,放疗医生只需在AI勾画基础上进行适当修改便可用于放疗计划设计,大大节省了放疗医生工作时间。此外,由于AI+CTV勾画对解剖结构的高辨识度,减少了部分低年资放疗医生因对解剖认识不足而导致的漏靶,提高了靶区勾画的准确性。
近年来,国内外关于鼻咽癌放疗靶区和危及器官AI勾画的研究越来越多[11-12, 19, 21]。MEN[21]等研究发现DDNN自动分割能准确的勾画GTVnx和CTV(DSC值分别为80.9%和82.6%), 其另一项研究[19]通过对分割技术进行改进,使得CTV的DSC值从0.82提高到0.86,同时减少了45%的工作量。LI等[12]通过对原发灶和转移淋巴结大体靶区进行AI勾画,发现AI勾画较手动勾画明显提高了效率且准确性较高,T分期越早,准确性越高[DSC值分别为77.24%(T1), 75.38%(T2), 74.13%(T3)和71.42%(T4);HD值分别为10.36 mm (T1), 11.37 mm (T2), 11.90 mm (T3)和15.72 mm (T4)];原发灶较转移淋巴结准确性更高(DSC值:74.00% vs 65.86%,HD值:12.85 mm vs 32.10 mm)。本研究内部验证CTV1和CTV2平均HD值分别为7.789 mm和6.144 mm,均小于既往研究。孙颖教授团队[11]利用AI技术在MRI影像上对1 021例鼻咽癌患者的原发灶进行自动分割,结果显示:AI辅助勾画大大提高了鼻咽癌原发灶勾画的准确性(DSC值:0.79),明显缩短了靶区勾画的时间,还减少了观察者内和观察者间的变异,提高了鼻咽癌靶区勾画的一致性。与本研究结果一致。
本研究还发现该AI+CTV模型勾画CTV1的准确性较CTV2更高,其原因可能为下颈淋巴引流区勾画时,Vb区后界和Vc区前外侧界勾画差异较大所致。大量研究发现[22-24],Vb区淋巴结的后界超过了2013版颈部淋巴结分区所划分的后界。LIN等[25]通过分析959例鼻咽癌患者10 651个淋巴结分布情况,发现13.3%的Vb区淋巴结超出Vb区后界,沿着颈横血管周围分布,并达肩胛提肌前缘。故建议将Vb水平的后界延伸到肩胛提肌的前缘,以包括横向血管。该研究还发现肩胛舌骨肌与皮肤之间未发现淋巴结,建议将Vc区前外侧界从颈阔肌缩至肩胛舌骨肌。但关于Vb区后界和Vc区前外侧界具体如何勾画,目前暂无相关指南或共识推荐,该区域的勾画有待更多的权威推荐后再优化。
该模型存在以下局限性:①该模型是利用分期较早(T1~2N2)且淋巴结较小(≤2cm)患者靶区训练而得,不适用于分期偏晚的大病灶患者,仍需继续训练针对大病灶的AI+CTV勾画模型。②该模型在环状软骨下缘即CTV1和CTV2交界层面,存在CTV1和CTV2同时出现在同一层的情况,这可能是因为环状软骨发育不对称,或者定位时患者体位原因导致双侧环状软骨下缘不在同一层面,需要进行手动修正。③该模型下颈勾画差异仍较大,急需对下颈勾画进一步规范后再优化模型。④本研究样本量较小,仅选取了30例患者进行训练,后期我们会进一步增加样本量来提升该模型的准确性。
基于小样本的AI+CTV模型在分期较早(T1~2N2)且淋巴结较小(≤2 cm)鼻咽癌患者放疗的临床应用基本可行。临床医生可以在此AI+CTV模型勾画基础上对鼻咽癌靶区进行适当修改,便可用于放疗计划设计。AI勾画的应用不仅能减少不同层级放疗单位、不同技术层次放疗医生间鼻咽癌CTV勾画的差异,提高靶区勾画准确性;还能缩短放疗医生CTV勾画时间,提高工作效率,进而实现鼻咽癌临床靶区勾画的规范化、标准化、同质化。但将来仍需进一步对鼻咽癌临床靶区勾画细节规范后再持续优化该模型。
| [1] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. |
| [2] |
ZHANG L, HUANG Y, HONG S D, et al. Gemcitabine plus cisplatin versus fluorouracil plus cisplatin in recurrent or metastatic nasopharyngeal carcinoma: a multicentre, randomised, open-label, phase 3 trial[J]. Lancet, 2016, 388(10054): 1883-1892. |
| [3] |
LANG J Y, HU C S, LU T X. Chinese expert consensus on diagnosis and treatment of nasopharyngeal carcinoma: evidence from current practice and future perspectives[J]. Cancer Manag Res, 2019, 10(11): 6365-6376. |
| [4] |
DAS I J, MOSKVIN V, JOHNSTONE P A. Analysis of treatment planning time among systems and planners for intensity-modulated radiation therapy[J]. J Am Coll Radiol, 2009, 6(7): 514-517. |
| [5] |
FENG M, DEMIROZ C, VINEBERG K A, et al. Normal tissue anatomy for oropharyngeal cancer: contouring variability and its impact on optimization[J]. Int J Radiat Oncol Biol Phys, 2012, 84(2): e245-e249. |
| [6] |
BREEN S L, PUBLICOVER J, DE S S, et al. Intraobserver and inter observer variability in GTV delineation on FDG-PET-CT images of head and neck cancers[J]. Int J Radiat Oncol Biol Phys, 2007, 68(3): 763-770. |
| [7] |
YAMAZAKI H, SHIOMI H, TSUBOKURA T, et al. Quantitative assessment of inter-observer variability in target volume delineation on stereotactic radiotherapy treatment for pituitary adenoma and meningioma near optic tract[J]. Radiat Oncol, 2011, 6: 10. |
| [8] |
VINOD S K, MIN M, JAMESON M G, et al. A review of interventions to reduce inter-observer variability in volume delineation in radiation oncology[J]. J Med Imaging Radiat Oncol, 2016, 60(3): 393-406. |
| [9] |
CARAVATTA L, MACCHIA G, MATTIUCCI G C, et al. Inter-observer variability of clinical target volume delineation in radiotherapy treatment of pancreatic cancer: a multi-institutional contouring experience[J]. Radiat Oncol, 2014, 9: 198. |
| [10] |
杜国波, 蒋利华, 郭飞, 等. ABAS软件自动勾画技术在鼻咽癌调强放疗中的应用研究[J]. 中华放射肿瘤学杂志, 2014, 23(1): 63-64. DU G B, JIANG L H, GUO F, et al. Application of ABAS software automatic sketching technology in intensity modulated radiotherapy for nasopharyngeal carcinoma[J]. Chin J Radiat Oncol, 2014, 23(1): 63-64. |
| [11] |
LIN L, DOU Q, JIN Y M, et al. Deep learning for automated contouring of primary tumor volumes by MRI for nasopharyngeal carcinoma[J]. Radiology, 2019, 291(3): 677-686. |
| [12] |
LI S H, XIAO J H, HE L, et al. The tumor target segmentation of nasopharyngeal cancer in CT images based on deep learning methods[J]. Technol Cancer Res Treat, 2019, 18: 1533033819884561. |
| [13] |
XUE X D, QIN N N, HAO X Y, et al. Sequential and iterative auto-segmentation of high-risk clinical target volume for radiotherapy of nasopharyngeal carcinoma in planning CT images[J]. Front Oncol, 2020, 23(10): 1134. |
| [14] |
YOUNG A V, WORTHAM A, WERNICK I, et al. Atlas-based segmentation improves consistency and decreases time required for contouring postoperative endometrial cancer nodal volumes[J]. Int J Radiat Oncol Biol Phys, 2011, 79(3): 943-947. |
| [15] |
GRÉGOIRE V, ANG K, BUDACH W, et al. Delineation of the neck node levels for head and neck tumors: a 2013 update. DAHANCA, EORTC, HKNPCSG, NCIC CTG, NCRI, RTOG, TROG consensus guidelines[J]. Radiother Oncol, 2014, 110(1): 172-181. |
| [16] |
LEE A W, NG W T, PAN J J, et al. International guideline for the delineation of the clinical target volumes (CTV) for nasopharyngeal carcinoma[J]. Radiother Oncol, 2018, 126(1): 25-36. |
| [17] |
罗京伟, 徐国镇, 高黎. 头颈部肿瘤放射治疗图谱[M]. (第3版). 人民卫生出版社. LUO J W, XU G Z, GAO L. Atlas of radiotherapy for head and neck tumors[M]. (3rd Edition). Beijing: People's Health Publishing House. |
| [18] |
ALLOZI R, LI X A, WHITE J, et al. Tools for consensus analysis of experts' contours for radiotherapy structure definitions[J]. Radiother Oncol, 2010, 97(3): 572-578. |
| [19] |
MEN K, CHEN X Y, ZHU J, et al. Continual improvement of nasopharyngeal carcinoma segmentation with less labeling effort[J]. Physica Medica, 2020, 80: 347-351. |
| [20] |
ZIJDENBOS A P, DAWANT B M, MARGOLIN R A, et al. Morphometric analysis of white matter lesions in MR images: method and validation[J]. IEEE Trans Med Imaging, 1994, 13(4): 716-724. |
| [21] |
MEN K, CHEN X Y, ZHANG Y, et al. Deep deconvolutional neural network for target segmentation of nasopharyngeal cancer in planning computed tomography images[J]. Front Oncol, 2017, 20(7): 315. |
| [22] |
JIANG C Y, GAO H, ZHANG L, et al. Distribution pattern and prognosis of metastatic lymph nodes in cervical posterior to level V in nasopharyngeal carcinoma patients[J]. BMC Cancer, 2020, 20(1): 667. |
| [23] |
JIANG C, GONG B, GAO H, et al. Correlation analysis of neck node levels in 960 cases of Nasopharyngeal carcinoma (NPC)[J]. Radiother Oncol, 2021, 161: 23-28. |
| [24] |
WANG X S, HU C S, YING H M, et al. Patterns of lymph node metastasis from nasopharyngeal carcinoma based on the 2013 updated consensus guidelines for neck node levels[J]. Radiother Oncol, 2015, 115(1): 41-45. |
| [25] |
LIN L, LU Y, WANG X J, et al. Delineation of neck clinical target volume specific to nasopharyngeal carcinoma based on lymph node distribution and the international consensus guidelines[J]. Int J Radiat Oncol Biol Phys, 2018, 100(4): 891-902. |




