2. 2400038 重庆,陆军军医大学(第三军医大学)基础医学院全军免疫学研究所
2. Institute of Immunology, College of Basic Medical Sciences, Army Medical University (Third Military Medical University), Chongqing, 400038, China
继人类基因组计划之后,新一轮生命医学的大科学研究计划(如人类细胞图谱计划、灵长类细胞解码计划等)聚焦在组织、器官、细胞和分子层面的精细解码,旨在进一步破译生命奥秘。人类细胞图谱等计划的快速推进,得益于单细胞测序技术的快速发展和广泛应用。单细胞转录组测序助力解码细胞表型,而单细胞表观组学测序技术则有助于进一步在表观遗传学层面解码细胞的异质性,以及在分子水平解析表观遗传学的精细调控机制,从而达到分子解码的目的。本文将主要介绍单细胞表观测序技术的进展。
1 单细胞测序技术的发展历程所谓的单细胞测序技术,其本质是将单个细胞的全部或者经过处理的核酸分子(DNA和/或RNA)通过测序方法测序(一代测序、二代测序、三代测序),进而利用生物信息学和统计学对其进行描述和分析。单细胞测序的发展基于测序技术发展而来。第一代测序技术,即Sanger测序法,是1977年由Frederick Sanger及其同事开发,通过对单一核酸分子逐个碱基标定进而达到测序目的。利用Sanger测序法,人类基因组计划于2003年得以完成,初步绘制了人类基因组序列的草图,为生命解码奠定了坚实基础。为了进一步提高测序通量和测序速度,2000年开发了二代测序技术(高通量并行测序)。二代测序技术的成熟发展和广泛应用,使得测序费用按照摩尔定律不断下降,并且起始DNA、RNA量的需求不断降低(从μg至pg水平)。第1个在单细胞中进行二代测序的尝试,是2009年TANG等[1]利用小鼠胚胎细胞绘制了单细胞水平的转录组变化,并在此基础上发展出了SMART-seq2、Drop-seq、Microwell-seq、SPLiT-seq以及目前广泛商业化应用的10×Genomics等单细胞转录组测序技术[2]。在单细胞基因组测序方向,2012年,谢晓亮团队开发出了单细胞全基因组均匀扩增的新方法——多重退火循环扩增法(MALBAC),奠定和促进了单细胞水平基因组学的发展[3]。而在单细胞表观遗传学测序方向,2015年,有研究报道了检测单细胞水平染色质可及性、组蛋白修饰等的scDNase-seq、scATAC-seq、scChIP-seq等技术[4-6]。近年来,单细胞表观组学的新方法又不断开发,解决了scDNase-seq等技术细胞通量低的问题,以及联合多个组学的多组学单细胞测序技术(图 1)。这些单细胞技术的不断开发和应用,为细胞解码等大科学计划铺平了道路。
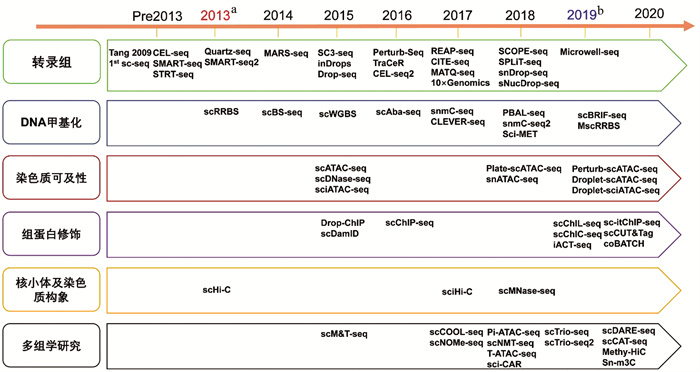
|
| a: 单细胞测序技术被Nature Methods评为2013年年度技术;b:单细胞多组学测序技术被评为2019年年度技术 图 1 单细胞测序新技术研发演进图 |
2 表观遗传学助力细胞解码
细胞的异质性很大程度取决于表观遗传学的异质性。基因的转录表达取决于基因所处的染色质状态,如转录因子的结合、顺式作用元件(增强子、启动子等)的暴露,以及三维构象、组蛋白修饰对染色质状态的维持和转录爆发的随机性[7-8]。在不同的表观调控层面,都有相应的单细胞测序技术开发。总体来说,其主要利用不同的酶处理染色质,进而捕获目标信息(图 2)。对于染色质可及性检测,利用核酸酶DNase Ⅰ或者转座酶Tn5识别切割开放染色质区域的DNA,进而利用微量DNA进行建库测序,得以获取单细胞层面的染色质可及性信息。利用核酸酶MNase切割未被核小体缠绕保护的染色体linker DNA,从而收集所有核小体缠绕的DNA片段进行测序,分析核小体的位置信息。利用组蛋白修饰特异性的抗体和Tn5、MNase等酶偶联,特异性地切断组蛋白修饰的染色质区域,进而建库测序,获取组蛋白修饰以及转录因子结合的情况。利用限制性内切酶Dpn Ⅱ对染色质切割后再原位连接,进而捕获染色质的三维结构。目前,单细胞表观测序技术的研究结果已表明,细胞间表观层面的异质性远大于转录层面的异质性,并且和转录异质性相关,但是相关调控机制尚不完全清晰[7]。不断开发新的单细胞测序技术,有望对此解析。
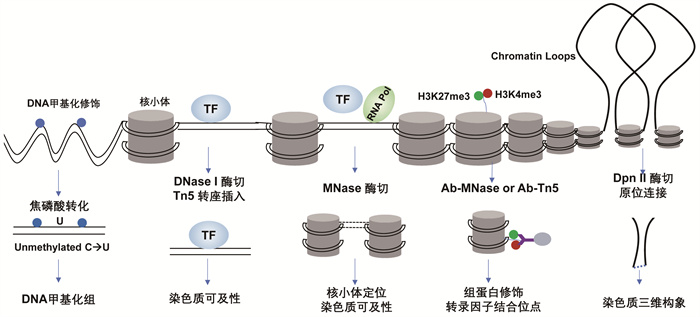
|
| 图 2 单细胞表观测序技术原理示意图 |
3 染色质可及性的细胞异质性
染色质是DNA贮存、传递遗传信息的形式,其在全基因组层面并不均一,表现为在不同的位点处于稀疏或致密状态。染色质可及性指的是核大分子能够与染色质化的DNA进行物理接触的程度,由核小体及其他阻碍接近DNA的染色质结合因子的占据和拓扑结构所决定。单细胞染色质可及性检测可通过scDNase-seq或scATAC-seq来检测。被DNase Ⅰ或Tn5酶所捕获的区域一般称为DNA酶超敏感位点(DNase Ⅰhypersensitive site, DHSs)。DHSs区多为核小体占位缺失、而被转录因子等转录相关蛋白所占据的增强子、启动子等区域。早期的scATAC-seq捕获的DHSs区域较少,而scDNase-seq可捕获约10万个的DHSs,但是随着scATAC-seq的优化,其DHSs捕获能力也随着增高。并且得益于Tn5酶在切割DHSs同时插入测序接头,scATAC-seq在文库构建便利性、增加细胞通量方面更具优势。然而,DNase-seq技术仍是研究DHSs、预测转录因子结合位点等的金标准。此外,本课题组在scDNase-seq基础上,继续开发了可提高细胞通量的iscDNase-seq技术[9]。scDNase-seq等技术的研究结果发现,染色质可及性在细胞间具有显著异质性,来自于相同细胞群体的两个单细胞也仅有约75%的DHSs相同。细胞间具有同质性的DHSs多为调控管家基因的顺式元件,而异质性的DHSs则可能为调控细胞发育异质性的重要因子。此外,不管是单细胞染色质可及性数据与bulk RNA-seq数据比较,还是来自于单细胞多组学的测序数据表明,细胞间异质性的DHSs与基因转录异质性具有显著相关性,这明确了染色质可及性异质性对基因转录异质性所发挥的重要作用。
4 核小体定位的细胞异质性核小体是由DNA缠绕组蛋白而成,连续的核小体呈串珠状排列并进一步压缩后形成染色质。核小体在基因组上的定位与排列对染色质空间高级结构、DNA复制、转录和基因表达调控起着关键作用。既往基于大量细胞检测到的核小体定位信息只能定位到DHSs区域,对于异染色质区域尚不清楚。虽然核小体在DHSs附近形成特定相位保证了染色质的开放性,但是,scDNase-seq等技术已证实细胞间具有显著的染色质可及性的异质性,意味着核小体在同一个DHS附近的定位存在着异质性。本课题组和Keji Zhao教授实验室通过构建单细胞水平核小体定位的scMNase-seq技术,在单分子水平阐述了核小体定位排布的新规律[10-11]。在沉默染色质区域,核小体定位在细胞之间表现出非常高的异质性,但是这些核小体却间距均匀地分布在基因组DNA上;在开放染色质区域,核小体定位在细胞之间几乎没有差异,但这些核小体间的间距却存在很大差异。此外,scMNase-seq分析DHSs附近的核小体间距发现其表现为双峰模式:其中一个峰的峰值与-1核小体相距190 bp(窄峰),另一个峰的峰值与-1核小体相距300 bp(宽峰),这两个峰分别对应染色质关闭和染色质开放这两种状态。并且超过80%的DHSs同时表现出两个峰,即细胞间DHSs的开放程度具有异质性。同时,宽峰的强度和染色质开放程度性正相关。这些结果为细胞间染色质的异质性研究提供了新思路。
5 组蛋白修饰的细胞异质性组成核小体的组蛋白具有丰富的修饰位点,这些修饰位点往往与不同的染色质状态密切相关。例如,组蛋白H3第4位赖氨酸甲基化修饰(H3K4me)一般位于具有转录活性的基因启动子、增强子区,而组蛋白H3第27位赖氨酸甲基化修饰(H3K27me)则位于沉默染色质区域。而同时存在H3K4me和H3K27me两种修饰的区域一般认为是待启动状态(primed state),预示着潜在的基因表达和细胞命运决定。Bradley E Bernstein实验室于2015年利用其开发的scChIP-seq技术,首次报道了单细胞水平组蛋白修饰。该技术基于微液流标记单细胞(cell barcode),之后再利用抗体去富集相应组蛋白修饰的DNA片段,所产出的单细胞reads数较低[6]。在此之后,本课题组与Keji Zhao教授实验室利用将组蛋白修饰的抗体和核酸酶MNase化学偶联,共同开发出了scChIC-seq技术[12]。而Steven Henikoff实验室利用类似的策略开发出了CUT & RUN技术[13]。后续的技术如ACT-seq、iscChIC-seq、scCUT & RUN、scCUT & TAG、CoTECH等技术则在前面基础上,通过引入cell barcode来继续提高细胞通量[14-18]。多个技术的研究结果已证实,细胞间组蛋白修饰的异质性与基因表达异质性高度正相关[7]。
6 展望不管是染色质可及性的异质性、组蛋白修饰的异质性,还是核小体排布的异质性,都会影响基因表达的异质性。这其中的机制为:当细胞处于未分化状态时,先锋转录因子结合到DNA后,通过招募相应的组蛋白修饰酶和染色质构象酶改变相应位点的染色质可及性、组蛋白修饰、核小体的排布以及染色质的三维结构,在细胞层面促使了表观遗传学异质性的产生。而细胞在接受外界分化信号后,效应转录因子结合到已处于预激活状态的染色质区域,发挥促进基因表达的作用[7]。因此,异质性的表观遗传学状态潜在指导了细胞分化命运的方向,转录因子的互作网络、表观遗传学的异质性以及转录爆发的随机性共同形成了目前转录组学层面的细胞异质性或者说细胞状态的多样性。
虽然目前多种单细胞技术已经开发,但是仍然面临以下问题:①单细胞测序数据的稀疏性和丢失问题。细致刻画细胞的异质性要求尽可能获取单细胞的所有信息,因此有必要通过新的算法或者新的建库测序策略解决此问题。②在单细胞分子层面,多组学诸如表观组学、转录组学、蛋白组学、代谢组学与细胞表型异质性的相互调控动态尚不完全清晰。解析细胞异质性的机制,需要继续开发全新的多组学技术。此外,利用单细胞技术助力细胞解码需要成熟稳定的技术平台,将会有更多商业化的单细胞试剂盒开发和推出。
基于二代测序技术的常规RNA-seq、ATAC-seq、ChIP-seq等已经成为分子生物学实验室的常规技术,随着单细胞测序技术的不断发展和成熟,单细胞测序技术将在生物学和医学实验室常规开展,助力解码器官、组织、细胞、分子的类型、状态和运行规律。
| [1] |
TANG F, BARBACIORU C, WANG Y, et al. mRNA-Seq whole-transcriptome analysis of a single cell[J]. Nat Methods, 2009, 6(5): 377-382. |
| [2] |
KHARCHENKO P V. The triumphs and limitations of computational methods for scRNA-seq[J]. Nat Methods, 2021, 18(7): 723-732. |
| [3] |
ZONG C H, LU S J, CHAPMAN A R, et al. Genome-wide detection of single-nucleotide and copy-number variations of a single human cell[J]. Science, 2012, 338(6114): 1622-1626. |
| [4] |
JIN W F, TANG Q S, WAN M M, et al. Genome-wide detection of DNase I hypersensitive sites in single cells and FFPE tissue samples[J]. Nature, 2015, 528(7580): 142-146. |
| [5] |
BUENROSTRO J D, WU B J, LITZENBURGER U M, et al. Single-cell chromatin accessibility reveals principles of regulatory variation[J]. Nature, 2015, 523(7561): 486-490. |
| [6] |
ROTEM A, RAM O, SHORESH N, et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state[J]. Nat Biotechnol, 2015, 33(11): 1165-1172. |
| [7] |
CARTER B, ZHAO K J. The epigenetic basis of cellular heterogeneity[J]. Nat Rev Genet, 2021, 22(4): 235-250. |
| [8] |
RODRIGUEZ J, REN G, DAY C R, et al. Intrinsic dynamics of a human gene reveal the basis of expression heterogeneity[J]. Cell, 2019, 176(1/2): 213-226. |
| [9] |
GAO W, KU W L, PAN L, et al. Multiplex indexing approach for the detection of DNase I hypersensitive sites in single cells[J]. Nucleic Acids Res, 2021, 49(10): e56. |
| [10] |
LAI B B, GAO W W, CUI K R, et al. Principles of nucleosome organization revealed by single-cell micrococcal nuclease sequencing[J]. Nature, 2018, 562(7726): 281-285. |
| [11] |
GAO W, LAI B, NI B, et al. Genome-wide profiling of nucleosome position and chromatin accessibility in single cells using scMNase-seq[J]. Nat Protoc, 2020, 15(1): 68-85. |
| [12] |
KU W L, NAKAMURA K, GAO W W, et al. Single-cell chromatin immunocleavage sequencing (scChIC-seq) to profile histone modification[J]. Nat Methods, 2019, 16(4): 323-325. |
| [13] |
SKENE P J, HENIKOFF S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites[J]. Elife, 2017, 6: e21856. |
| [14] |
CARTER B, KU W L, KANG J Y, et al. Mapping histone modifications in low cell number and single cells using antibody-guided chromatin tagmentation (ACT-seq)[J]. Nat Commun, 2019, 10(1): 3747. |
| [15] |
XIONG H, LUO Y, WANG Q, et al. Single-cell joint detection of chromatin occupancy and transcriptome enables higher-dimensional epigenomic reconstructions[J]. Nat Methods, 2021, 18(6): 652-660. |
| [16] |
BARTOSOVIC M, KABBE M, CASTELO-BRANCO G. Single-cell CUT&Tag profiles histone modifications and transcription factors in complex tissues[J]. Nat Biotechnol, 2021, 39(7): 825-835. |
| [17] |
HAINER S J, BOŠKOVIĆ A, MCCANNELL K N, et al. Profiling of pluripotency factors in single cells and early embryos[J]. Cell, 2019, 177(5): 1319-1329. |
| [18] |
KU W L, PAN L X, CAO Y Q, et al. Profiling single-cell histone modifications using indexing chromatin immunocleavage sequencing[J]. Genome Res, 2021. |




