2. 402460 重庆,重庆市荣昌中医院泌尿外科;
3. 402760 重庆,重庆市璧山中医院泌尿外科
2. Department of Urology, Rongchang Traditional Chinese Medicine Hospital, Chongqing, 402460;
3. Department of Urology, Bishan Traditional Chinese Medicine Hospital, Chongqing, 402760, China
膀胱癌是泌尿生殖系统常见的恶性肿瘤之一,在中国发病率呈逐年升高趋势。手术治疗是膀胱癌的主要治疗方式,但患者术后仍容易进展、复发和转移,患者5年生存率未有明显提升[1-2]。生物标志物作为最直接、快速、有效的诊断手段,其筛选与获得可在肿瘤诊断、发展、治疗以及疗效监测等多个方面发挥重要的作用[3]。因此,探究膀胱癌预后相关生物标志物将有助于预测患者的预后,指导患者的治疗。
自噬是真核细胞内发生的一种高度保守的自我降解机制,对于维持细胞的稳定性具有重要意义,并在肿瘤的发生、发展中起着重要的调控作用[4]。研究表明,饥饿诱导的自噬可以促进膀胱癌的侵袭转移并且自噬调节可以影响膀胱癌细胞对化疗药物的敏感性[5-6]。因此,探究自噬相关的生物标志物对于预测膀胱癌患者的疾病进展甚至生存预后具有重要的意义。
生物信息学可以帮助挖掘、分析肿瘤进展、预后的潜在生物标志物[7]。长链非编码RNA(lncRNA)是一类长度大于200碱基对的非蛋白编码转录体,可以通过影响染色质修饰、RNA剪切和蛋白质活性等方式在多个层面调控基因的表达进而影响膀胱癌的发生、发展、预后及化疗耐药[8]。因此,自噬相关的lncRNA在预测膀胱癌患者进展、预后中具有重要作用。本研究通过生物信息学挖掘出与自噬相关的lncRNAs,构建一个8-自噬相关lncRNA模型,并评估其在预测膀胱癌患者疾病进展和生存预后中的潜在能力。
1 材料与方法 1.1 数据获取膀胱癌患者转录组数据和临床数据均从TCGA数据库(https://cancergenome.nih.gov/)中获取。转录组数据包括19例正常膀胱样本和411例膀胱癌样本;临床数据包括402例膀胱癌患者的生存时间和生存状态。通过R软件4.0.2版本(https://www.r-project.org/)caret程序包将402例膀胱癌样本(总体集,whole set,n=402)按3 ∶1的比例随机分为训练集(training set,n=301)及验证集(validation set,n=101)。使用训练集构建模型,使用验证集和总体集验证模型。232个自噬基因从人类自噬数据库(HADb, http://www.autophagy.lu/)中获取。
1.2 构建自噬相关lncRNA预后模型自噬基因与转录组数据中lncRNA的相关性采用Pearson相关分析,满足|R2|>0.4且P < 0.001的lncRNA被认为是自噬相关的lncRNA。通过单因素Cox回归分析鉴定与膀胱癌患者总生存期(OS)相关(P < 0.05)的自噬相关lncRNA。通过危险比(HR)分析高风险相关的lncRNA(HR > 1)和保护性lncRNA(HR < 1)。通过多因素Cox回归分析对具有预后意义的自噬相关lncRNA进行进一步筛选并构建预后模型。通过Cytoscape软件3.7.1版本(https://cytoscape.org/)绘制lncRNA-mRNA共表达网络。
1.3 评估自噬相关lncRNA预后模型计算每个膀胱癌样本的风险评分(risk score,RS),并根据中位风险评分将膀胱癌患者分为高风险组和低风险组。
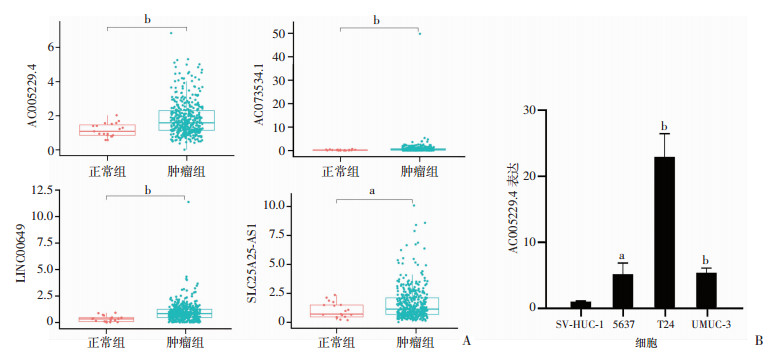
风险评分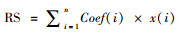 ;其中Coef(i)代表每个lncRNA通过多因素Cox回归分析得到的回归系数,x(i)代表每个lncRNA的表达数据。通过R软件survival包绘制生存曲线比较高危和低危人群之间的OS差异。通过R软件survival ROC包绘制接收者操作特征曲线(receiver operating characteristic curve,ROC)评估自噬相关lncRNA模型的灵敏度和特异度,其通过ROC曲线下方的面积(area under curve, AUC)大小展示。通过Graphpad软件8.0版本(https://www.graphpad.com/)统计每个样本风险评分在不同临床病理特征(性别、年龄、AJCC分期、TNM分期等)中的差异来评估以该模型计算的风险评分与膀胱癌进展的关系。每种病理特征根据风险评分中位数将患者分为高风险组和低风险组进行分层分析。通过单因素和多因素Cox回归分析验证该模型风险评分是否是膀胱癌患者的独立预后因素。
;其中Coef(i)代表每个lncRNA通过多因素Cox回归分析得到的回归系数,x(i)代表每个lncRNA的表达数据。通过R软件survival包绘制生存曲线比较高危和低危人群之间的OS差异。通过R软件survival ROC包绘制接收者操作特征曲线(receiver operating characteristic curve,ROC)评估自噬相关lncRNA模型的灵敏度和特异度,其通过ROC曲线下方的面积(area under curve, AUC)大小展示。通过Graphpad软件8.0版本(https://www.graphpad.com/)统计每个样本风险评分在不同临床病理特征(性别、年龄、AJCC分期、TNM分期等)中的差异来评估以该模型计算的风险评分与膀胱癌进展的关系。每种病理特征根据风险评分中位数将患者分为高风险组和低风险组进行分层分析。通过单因素和多因素Cox回归分析验证该模型风险评分是否是膀胱癌患者的独立预后因素。
通过R软件survival和rms包绘制列线图,分析临床病理特征和该模型风险评分对患者疾病进展、预后的预测效能。通过R语言foreign和timeROC包绘制校正曲线和ROC来评估列线图的准确性。
1.5 基因富集分析基因集富集分析(Gene set enrichment analysis,GSEA)用于分析高风险组和低风险组的KEGG通路富集情况。KEGG基因集(c2.cp.kegg.v7.2.symbols.gmt) 从Molecular Signatures Database中获得。在执行1 000次置换后,基于FDR < 0.25和P < 0.05的标准获得富集的通路。
1.6 细胞培养人尿路上皮永生化细胞SV-HUC-1、人膀胱癌细胞5637、人膀胱癌细胞UMUC-3均购自美国菌种保藏中心。人膀胱癌细胞T24获赠于重庆医科大学附属第一医院分子肿瘤学和表观遗传学重点实验室。所有细胞的培养条件是10%胎牛血清(fetal bovine serum, FBS)+基础培养基+1%青/链霉素。SV-HUC-1使用的基础培养基是F12-K(中乔新舟,上海),UMUC-3使用的基础培养基是DMEM(Gibco公司,美国),5637和T24使用的基础培养基是RPMI-1640(Corning公司,美国)。待细胞生长至约90%时,按1 ∶3进行传代培养。
1.7 RT-qPCR检测 1.7.1 细胞RNA提取采用FOREGENE Cell Total RNA Isolation Kit(Cat.NO.RE-03111/03112/03113)试剂盒进行RNA提取,具体方法:①将含有约106个贴壁细胞的培养皿倾斜,吸干培养基,加入500 μL Buffer CRL1进行消化、裂解。②将裂解的细胞混合液转移至DNA-Cleaning Column中,12 000 r/min离心2 min。移除DNA-Cleaning Column,保留收集管内的液体。③向上述液体中加入1.6倍体积的Buffer CRL2,轻揉混匀。④将混合液全部转移到RNA-only Column中,12 000 r/min离心1 min,弃掉收集管中的废液。⑤向纯化柱中加入500 μL Buffer RW1,12 000 r/min离心1 min,弃掉收集管中的废液。⑥向纯化柱中加入700 μL Buffer RW2,12 000 r/min离心1 min,弃掉收集管中的废液,重复2次。⑦将纯化柱放回收集管中,12 000 r/min空管离心2 min,去掉离心柱中残余的Buffer RW2。⑧将纯化柱转移至新的RNA收集管中,向纯化柱中央滴加20~50 μL已于65 ℃预热的RNase-Free dd H2O,室温放置2 min。12 000 r/min离心1 min后收集RNA溶液,然后取1 μL用紫外分光光度计(ND-1000,Nanodrop)测RNA浓度。
1.7.2 逆转录cDNA采用TaKaRa逆转录试剂盒进行,将抽提的1 000 ng的总RNA进行逆转录,逆转录体系20 μL,设置逆转录条件为:37 ℃,15 min;85 ℃,5 s,4 ℃保存。逆转录结束后将样品放置至冰上,防止cDNA降解。用紫外可见分光光度(ND-1000,Nanodrop)检测cDNA浓度。
1.7.3 RT-qPCR反应以cDNA为模板,参照SYBR® Premix ExTaqTM Ⅱ (TliRNaseH Plus)试剂盒(TaKaRa公司,日本)说明书进行荧光定量PCR操作。反应条件:95 ℃预变性30 s,95 ℃ 5 s,60 ℃ 20 s,40个循环,每组设定3个复孔。采用GAPDH作为内参基因,计算方法采用2-ΔΔCt方法。
1.8 统计学分析通过PERL语言5.30.2版本(http://www.perl.org/)和R软件4.0.2版本(https://www.r-project.org/)处理数据。通过Graphpad软件8.0版本(https://www.graphpad.com/)和R软件4.0.2版本进行图形制作和统计分析。计量资料数据以 x±s表示,均符合正态分析,组间比较采用t检验。P < 0.05表示差异具有统计学意义。
2 结果 2.1 构建8-自噬相关lncRNA预后模型首先对膀胱癌样本中的lncRNA和232个自噬基因进行person相关性分析(|R2|>0.4,P < 0.001),共鉴定出941个自噬相关lncRNA。单因素Cox回归分析显示,68个lncRNA与患者的预后相关。进一步对68个lncRNA进行多因素Cox回归分析发现,8个自噬相关lncRNA是膀胱癌患者的独立预后因素,其中7个lncRNA是保护性因素,1个lncRNA是危险因素(表 1)。通过Cytoscape软件3.7.1版本(https://cytoscape.org/)绘制lncRNA-mRNA共表达网络(图 1)。
| 基因 | Ensemble ID号 | 染色体定位 | β(cox) | HR值 | P值 |
| LINC00649 | ENSG00000237945 | chr21:33 915 534-33 984 529 | -0.355 76 | 0.478 7 | 0.000 34 |
| AC005229.4 | ENSG00000273314 | chr7:148 694 739-148 698 800 | 0.217 39 | 1.902 4 | 0.000 71 |
| USP30-AS1 | ENSG00000256262 | chr12:109 051 791-109 054 033 | -0.325 16 | 0.666 1 | 0.000 21 |
| OCIAD1-AS1 | ENSG00000248256 | chr4:48 852 007-48 860 203 | -0.274 13 | 0.542 4 | 0.003 49 |
| AC099518.2 | ENSG00000261357 | chr16:19 065 991-19 066 694 | -0.204 02 | 0.534 2 | 0.000 37 |
| SLC25A25-AS1 | ENSG00000234771 | chr9:128 106 047-128 128 124 | -0.243 79 | 0.584 0 | 0.001 22 |
| AC099343.2 | ENSG00000251878 | chr4:184 431 966-184 432 049 | -0.357 16 | 0.436 5 | 0.000 61 |
| AC073534.1 | ENSG00000268442 | chr19:24 162 193-24 163 447 | -0.585 95 | 0.480 3 | 0.001 28 |

|
| 图 1 lncRNA-mRNA共表达网络 |
2.2 8-自噬相关lncRNA模型可预测膀胱癌患者预后
在训练集(n=301)、验证集(n=101)和总体集(n=402)中,根据风险评分将患者分为高风险组和低风险组。结果发现,低风险组患者的总生存时间明显长于高风险组的患者(图 2A)。进一步通过ROC评估8-自噬相关lncRNA模型的灵敏度和特异度,在训练集中AUC值为0.803,在验证集中AUC值为0.773,在总体集中AUC值为0.759(图 2B)。表明8-自噬相关lncRNA模型可以较准确地预测患者的生存预后。

|
| A:不同群体生存曲线 低风险组患者的总生存时间明显长于高风险组的患者;B:ROC曲线评估8-自噬相关lncRNA模型的灵敏度和特异度 图 2 验证8-自噬相关lncRNA预后模型 |
2.3 8-自噬相关lncRNA模型与膀胱癌进展相关
评估模型风险评分和膀胱癌临床病理特征的相关性。模型风险评分和患者性别(P=0.239 0,图 3B)、M分期(P=0.093 4,图 3F)之间没有显著的相关性;但是模型风险评分和患者年龄(P=0.010 9,图 3A)、AJCC分期(P=0.000 019,图 3C)、T分期(P=0.000 5,图 3D)、N分期(P=0.000 1,图 3E)之间具有显著的相关性。其中,模型风险评分和M分期之间没有显著相关性的原因可能是由M期患者的样本量较少导致的。结果表明,该8-自噬相关lncRNA模型的风险评分与膀胱癌疾病进展相关。

|
| A~F:分别为模型风险评分与患者年龄、性别、AJCC分期、T分期、N分期、M分期的相关性 图 3 模型风险评分和膀胱癌患者临床病理特征的相关性 |
2.4 8-自噬相关lncRNA模型是预测膀胱癌患者生存的独立预后因素
进一步通过单因素和多因素Cox回归分析检测8-自噬相关lncRNA模型对膀胱癌患者预后的独立预测能力。结果表明,模型风险评分和患者的总生存时间显著相关(P < 0.001,图 4A、B)。ROC曲线结果表明,模型风险评分的AUC值(0.696)高于其他临床病理参数(图 4C)。以上结果表明,8-自噬相关lncRNA模型是预测膀胱癌患者预后的独立预后因素。

|
| A:单因素Cox回归分析;B:多因素Cox回归分析;C:ROC曲线 图 4 8-自噬相关lncRNA模型是预测膀胱癌患者生存的独立预后因素 |
2.5 分层分析
根据临床病理资料对膀胱癌患者进行了分层分析。在患者年龄、性别、AJCC分期、T分期、N0期和M0期中,高风险组患者的总生存时间较低风险组更短(P < 0.01,图 5)。在N1-3(P=0.171)和M1(P=0.752)这两个分层中,高风险组和低风险组患者的生存时间差异并无统计学意义(图 5),可能与该两个分层的样本量不足有关。结果表明,该8-自噬相关lncRNA模型在不同年龄、性别、AJCC分期的患者中具有较好的预测预后能力。

|
| A:年龄>65岁患者;B:年龄≤65岁患者;C:女性患者;D:男性患者;E:AJCC分期Ⅰ-Ⅱ患者;F:AJCC分期Ⅲ-Ⅳ患者;G:T1-2患者;H:T3-4患者;I:N0患者;J:N1-3患者;K:M0患者;L:M1患者 图 5 膀胱癌患者不同病理特征分层的高低风险生存分析 |
2.6 建立列线图
列线图可用于多指标联合诊断或预测疾病的发生和进展。因此,基于多因素回归分析,通过模型风险评分、患者年龄、性别、分期构建了列线图(图 6A)。进一步绘制校准曲线(图 6B、C)和ROC(图 6D)评估列线图的预测能力,3年和5年AUC值分别为0.767和0.757。
|
| A:基于多因素回归分析通过模型风险评分、患者年龄、性别、分期构建了列线图;B、C:分别为3年和5年的校准曲线 校准曲线较贴合标准曲线;D:绘制ROC曲线评估列线图的预测能力 图 6 列线图的建立、验证与评估 |
2.7 GSEA富集分析
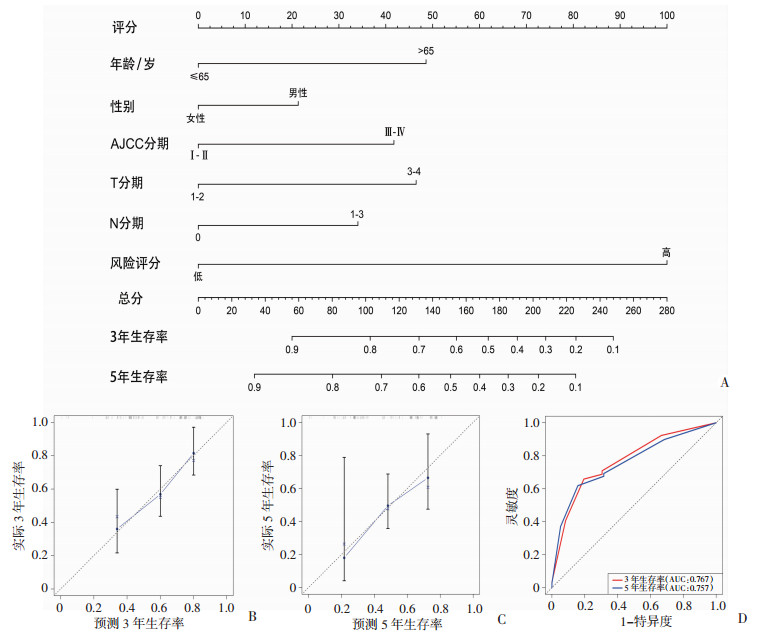
GSEA富集分析结果显示,高风险组主要富集一些自噬、肿瘤恶性生物进展的相关的TGF-β、Wnt信号通路以及一些肿瘤(结直肠癌、黑色素瘤、前列腺癌、肾癌、小细胞肺癌)相关信号通路。低风险组主要富集一些免疫(抗原提呈处理、移植物抗宿主病、肠道免疫网络、原发免疫缺陷)相关通路(图 7)。
|
| A:高风险组富集在肿瘤、结直肠癌、黑色素瘤、前列腺癌、肾癌、小细胞肺癌、TGF-β、Wnt信号通路;B:低风险组富集在抗原提呈处理、移植物抗宿主病、肠道免疫网络、原发免疫缺陷 图 7 GSEA富集分析 |
2.8 RT-qPCR对自噬相关lncRNA表达情况进行验证
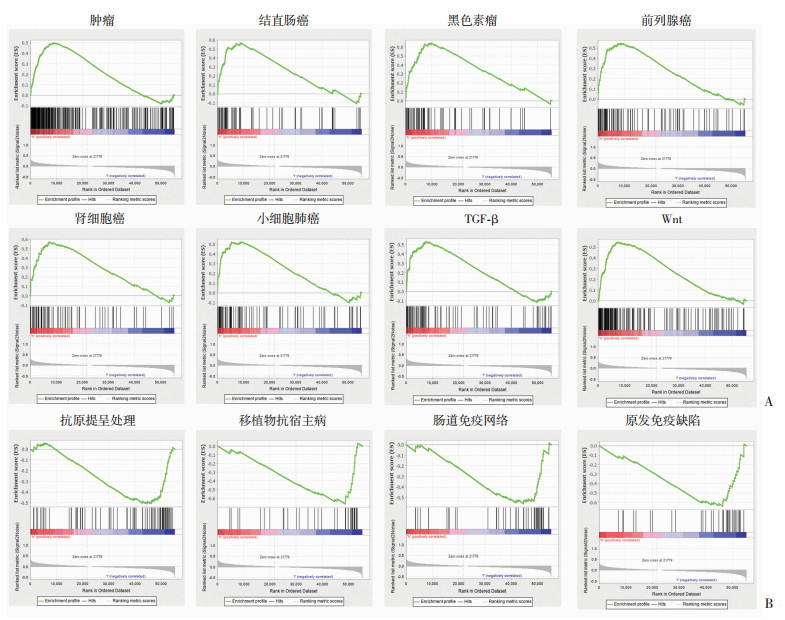
在TCGA数据库中预测8个自噬相关lncRNA的表达水平(正常样本19例,肿瘤样本411例),其中AC005229.4、AC073534.1、LINC00649、SLC25A25-AS1的差异表达具有统计学意义(图 8A)。进一步检测模型中高危lncRNA AC005229.4在正常尿路上皮细胞系SV-HUC-1和膀胱癌细胞系5637、T24、UMUC-3中的表达情况(图 8B)。相较于正常尿路上皮细胞系SV-HUC-1,AC005229.4在膀胱癌细胞系5637(P=0.013 8)、T24(P=0.000 4)、UMUC-3(P=0.000 5)中均高表达。
|
| A:TCGA数据库中预测的自噬相关lncRNA的表达 a:P < 0.05,b:P < 0.01;B:lncRNA AC005229.4在膀胱癌细胞系中的表达 a:P < 0.05,b:P < 0.01,与SV-HUC-1比较 图 8 RT-qPCR对自噬相关AC005229.4表达情况进行验证 |
3 讨论
生物标志物可以是DNA的甲基化、具有单核苷酸多态性(SNPs)的模板、蛋白质或代谢的改变及mRNA、非编码RNA的改变等,而这些变化都与机体疾病状态的发生密切相关[9]。肿瘤生物标志物在临床上主要是用于发现原发肿瘤,并且可以对一些高危人群进行筛选,且对于肿瘤患者的预后和治疗效果有着重要意义。近年来,一些膀胱癌相关肿瘤标记物才开始应用于临床,比如膀胱肿瘤抗原系列[10]、核基质蛋白22[11]、纤维蛋白降解产物[12]等。这些肿瘤标记物有助于检测出临床隐匿性膀胱癌,但是目前膀胱癌并没有高度特异性的肿瘤标志物可以预测患者的生存预后情况。因此探究膀胱癌预后相关生物标志物将具有良好的临床应用前景。
lncRNA在高等生命的基因组中占有较大的比例。相较于可编码蛋白质的序列,lncRNA有着更重要的作用[13],其可通过影响染色质修饰、RNA剪切和蛋白质活性等方式在多个层面调控基因的表达进而影响肿瘤的发生、发展、预后及化疗耐药[8]。越来越多的研究发现lncRNA可作为一种新型生物标志物在预测膀胱癌发生、发展、预后中具有重要作用。患者血清外泌体源性lncRNA LNMAT2过表达与较短的生存时间相关,外泌体源性lncRNA LNMAT2可能是膀胱癌淋巴结转移的潜在诊断生物标志物和治疗靶点[14]。正常膀胱组织来源的外泌体通过lncRNA PTENP1抑制膀胱癌的恶性进展,其可作为预测膀胱癌患者生存预后的生物标志物[15]。还有研究发现,基于TCGA数据库构建的8-lncRNA模型可有效预测膀胱癌患者的预后[16]。一个上皮间充质转化相关的14-lncRNA模型可有效预测膀胱癌患者的进展和预后[17]。一个细胞外基质相关的6-lncRNA模型可作为预测膀胱癌患者预后的新标志物[18]。随着对lncRNA在肿瘤发生、发展中作用机制的深入研究,lncRNA在膀胱癌进展预后中的诊断效能将不断提高。
细胞自噬是以胞质内出现双层膜结构包裹长寿命蛋白和细胞器的自噬体为特征的细胞“自我消化”的一系列生化过程[19]。自噬在肿瘤的发生、发展中具有重要意义。在早期阶段,自噬通过诱导线粒体重塑并去除受损和有毒性物质来抑制肿瘤进展。肿瘤进展到晚期,自噬又可通过协助肿瘤细胞应对外界刺激获得更顽强的逆境生存能力进而促进肿瘤的进展[20]。因此,自噬相关的生物标志物对预测肿瘤进展和预后具有重要的意义。WANG等[21]研究发现,一个3-自噬相关基因在膀胱癌患者预后预测中具有重要作用。在膀胱癌中自噬相关标志物的报道较少,仍具有较大的研究价值。
鉴于lncRNA和自噬在预测膀胱癌进展预后中都具有重要作用。因此,本研究利用生物信息学分析自噬相关lncRNA在膀胱癌进展预后中的预测效能。首先,对TCGA数据库的膀胱癌lncRNA表达数据和人类自噬数据库中自噬基因的表达数据进行Person相关性分析,共筛选出941个自噬相关lncRNA。通过单因素和多因素Cox回归分析发现,8个自噬相关lncRNA和膀胱癌患者的生存时间显著相关,因此基于该8个自噬相关lncRNA构建模型。在训练集、验证集、总体集中,根据模型风险评分将患者分为高风险组和低风险组。低风险组患者的总生存时间明显长于高风险组的患者,3个集合的ROC曲线AUC值均大于0.75。这提示该8-自噬相关lncRNA模型对于膀胱癌患者的预后具有较好的预测效能。列线图又称诺莫图(Nomogram图),是建立在多因素回归分析的基础上,将多个预测指标进行整合,然后采用带有刻度的线段,按照一定的比例绘制在同一平面上,从而用以多指标联合诊断或预测疾病发生或进展[22]。本研究基于患者年龄、性别、不同分期以及模型风险评分来绘制列线图。与其他传统的临床指标相比,模型的风险评分对患者预后具有更高的预测效能。通过绘制校准曲线和ROC曲线发现,该列线图的准确性较高。进一步对高风险组和低风险组患者进行GSEA通路富集分析。低风险组主要富集到一些免疫相关通路;而高风险组主要富集到一些肿瘤相关通路以及自噬调控肿瘤恶性进展相关的通路(TGF-β信号通路等)。自噬可通过TGF-β/Smad信号通路介导上皮间充质转化激活进而促进膀胱癌的侵袭转移[5]。反过来TGF-β也可上调Beclin1、ATG5、ATG7等基因诱导自噬激活[23]。因此,自噬和TGF-β可能存在这样的正反馈调控机制来影响膀胱癌的进展。
本研究也存在一定的局限性。首先,临床数据来自TCGA公共数据库,在分析过程中不可避免会产生偏倚。另外,在一些分层分析中样本量较少,可能导致结果的假阴性。最后,该模型中的lncRNA有待进行体内外基础实验以验证其对膀胱癌恶性生物学行为的影响。
总之,本研究成功构建了一个自噬相关lncRNA模型,其可准确预测膀胱癌患者的生存预后。基于该模型风险评分和临床指标构建的列线图在预测膀胱癌患者预后方面具有较好的效能。GSEA通路富集分析发现,高风险组与自噬和肿瘤通路相关,低风险组和免疫通路相关。这些结果可为未来针对不同风险群体的膀胱癌患者的潜在个体化治疗的研究提供依据。
| [1] |
ROBERTSON A G, KIM J, AL-AHMADIE H, et al. Comprehensive molecular characterization of muscle-invasive bladder cancer[J]. Cell, 2017, 171(3): 540-556.e25. |
| [2] |
LENIS A T, LEC P M, CHAMIE K, et al. Bladder cancer: a review[J]. JAMA, 2020, 324(19): 1980-1991. |
| [3] |
张敏, 周丽娜, 徐姗姗, 等. 肿瘤免疫治疗相关预测生物标志物研究进展[J]. 国际肿瘤学杂志, 2020, 47(8): 487-491. ZHANG M, ZHOU L N, XU S S, et al. Research progress in predictive biomarkers related to tumor immunotherapy[J]. J Int Oncol, 2020, 47(8): 487-491. |
| [4] |
LEVY J M M, TOWERS C G, THORBURN A. Targeting autophagy in cancer[J]. Nat Rev Cancer, 2017, 17(9): 528-542. |
| [5] |
TONG H, YIN H B, HOSSAIN M A, et al. Starvation-induced autophagy promotes the invasion and migration of human bladder cancer cells via TGF-β1/Smad3-mediated epithelial-mesenchymal transition activation[J]. J Cell Biochem, 2019, 120(4): 5118-5127. |
| [6] |
YANG X Y, YIN H B, ZHANG Y Z, et al. Hypoxia-induced autophagy promotes gemcitabine resistance in human bladder cancer cells through hypoxia-inducible factor 1α activation[J]. Int J Oncol, 2018, 53(1): 215-224. |
| [7] |
TIAN X, XU W, WANG Y, et al. Identification of tumor-infiltrating immune cells and prognostic validation of tumor-infiltrating mast cells in adrenocortical carcinoma: results from bioinformatics and real-world data[J]. Oncoimmunology, 2020, 9(1): 1784529. |
| [8] |
FLIPPOT R, BEINSE G, BOILÈVE A, et al. Long non-coding RNAs in genitourinary malignancies: a whole new world[J]. Nat Rev Urol, 2019, 16(8): 484-504. |
| [9] |
NG K, STENZL A, SHARMA A, et al. Urinary biomarkers in bladder cancer: a review of the current landscape and future directions[J]. Urol Oncol, 2021, 39(1): 41-51. |
| [10] |
CHENG M L, IYER G. Novel biomarkers in bladder cancer[J]. Urol Oncol, 2018, 36(3): 115-119. |
| [11] |
MATI Q, QAMAR S, ASHRAF S, et al. Tissue nuclear matrix protein expression 22 in various grades and stages of bladder cancer[J]. J Coll Physicians Surg Pak, 2020, 30(12): 1321-1325. |
| [12] |
LI X, SHU K P, ZHOU J, et al. Preoperative plasma fibrinogen and D-dimer as prognostic biomarkers for non-muscle-invasive bladder cancer[J]. Clin Genitourin Cancer, 2020, 18(1): 11-19.e1. |
| [13] |
ANASTASIADOU E, JACOB L S, SLACK F J. Non-coding RNA networks in cancer[J]. Nat Rev Cancer, 2018, 18(1): 5-18. |
| [14] |
CHEN C H, LUO Y M, HE W, et al. Exosomal long noncoding RNA LNMAT2 promotes lymphatic metastasis in bladder cancer[J]. J Clin Investig, 2020, 130(1): 404-421. |
| [15] |
ZHENG R, DU M L, WANG X W, et al. Exosome-transmitted long non-coding RNA PTENP1 suppresses bladder cancer progression[J]. Mol Cancer, 2018, 17(1): 143. |
| [16] |
SHEN D Y, ZHANG Y Y, ZHENG Q M, et al. A competing endogenous RNA network and an 8-lncRNA prognostic signature identify MYO16-AS1 as an oncogenic lncRNA in bladder cancer[J]. DNA Cell Biol, 2021, 40(1): 26-35. |
| [17] |
TONG H, LI T H, GAO S, et al. An epithelial-mesenchymal transition-related long noncoding RNA signature correlates with the prognosis and progression in patients with bladder cancer[J]. Biosci Rep, 2021, 41(1): BSR20203944. |
| [18] |
ZHAO D, PENG Q, WANG L, et al. Identification of a six-lncRNA signature based on a competing endogenous RNA network for predicting the risk of tumour recurrence in bladder cancer patients[J]. J Cancer, 2020, 11(1): 108-120. |
| [19] |
AMARAVADI R K, KIMMELMAN A C, DEBNATH J. Targeting autophagy in cancer: recent advances and future directions[J]. Cancer Discov, 2019, 9(9): 1167-1181. |
| [20] |
童行, 何卫阳. 自噬在肿瘤上皮-间充质转化以及侵袭转移中的作用[J]. 现代泌尿外科杂志, 2019, 24(5): 404-408. TONG H, HE W Y. Research progress on the role of autophagy in the invasion and metastasis of epithelial-mesenchymal transition[J]. J Mod Urol, 2019, 24(5): 404-408. |
| [21] |
WANG S S, CHEN G, LI S H, et al. Identification and validation of an individualized autophagy-clinical prognostic index in bladder cancer patients[J]. Onco Targets Ther, 2019, 12: 3695-3712. |
| [22] |
BALACHANDRAN V P, GONEN M, SMITH J J, et al. Nomograms in oncology: more than meets the eye[J]. Lancet Oncol, 2015, 16(4): e173-e180. |
| [23] |
KIYONO K, SUZUKI H I, MATSUYAMA H, et al. Autophagy is activated by TGF-beta and potentiates TGF-beta-mediated growth inhibition in human hepatocellular carcinoma cells[J]. Cancer Res, 2009, 69(23): 8844-8852. |




