2. 430070 湖北,华中农业大学生命科学技术学院;
3. 572000 海南,三亚市人民医院检验科
2. College of Life Science and Technology Huazhong Agricultural University, Wuhan, Hubei Province, 430070;
3. Department of Clinical Laboratory Sanya People's Hospital, Sanya, Hainan Province, 572000, China
类鼻疽伯克霍尔德菌(Burkholderia pseudomallei,B. pseudomallei,简称类鼻疽菌)是一种革兰阴性短杆菌,可以引起人兽共患疾病类鼻疽。类鼻疽作为一种热带疾病,主要流行于东南亚、澳大利亚北部以及我国海南、广东、广西、香港、台湾等地。类鼻疽临床表现多样,治疗不及时则病死率高达40%[1-2]。尽管进行了长期和适当的抗生素治疗,类鼻疽仍然保持着较高的复发率。感染类鼻疽菌后第1年复发的比例约为6%,而在10年之内达到13%[3]。复发类鼻疽是导致幸存于急性类鼻疽患者发病和死亡的重要原因。目前大部分研究聚焦于类鼻疽菌的急性感染过程,对类鼻疽的复发机制却鲜有报道。
本研究以来源于同一个患者时隔5年分离到的2株类鼻疽菌为研究对象,进行微生物学鉴定以及全基因组测序,同时检测2株类鼻疽菌的胞内存活能力和蛋白表达差异,初步分析可能与类鼻疽菌长期潜伏宿主体内有关的蛋白质,为进一步研究类鼻疽菌的持续性感染机制及防治提供有价值的参考。
1 材料与方法 1.1 菌株、细胞来源及主要试剂类鼻疽菌BP145A:2012年分离于海南省三亚市人民医院心胸外科某患者,本文称为原发株;BP145B:2017年分离于同一医院呼吸内科同一患者,本文称为复发株。菌株和人肺癌上皮细胞A549均由本实验室保存。细菌基因组DNA提取试剂盒购自北京天根公司。
1.2 全基因组提取、测序使用细菌基因组DNA提取试剂盒说明书分别提取原发株和复发株全基因组DNA,测定DNA浓度,1%琼脂糖凝胶电泳检测DNA样品有无降解,于-80 ℃储存。委托上海伯豪生物技术有限公司进行三代测序。
1.3 MLST分型表 1列出了用于扩增7个管家基因的PCR引物。PCR反应体系总体积为25 μL,PCR反应条件为95 ℃预变性5 min;95 ℃变性5 s,退火30 s(gmhD、lipA-55 ℃,ace、gltB、narK、ndh-60 ℃,lepA-62 ℃),72 ℃延伸60 s,共30个循环。将扩增片段送生工生物工程(上海)股份有限公司进行测序,获得7个管家基因的碱基序列。将7个目的片段序列上传至类鼻疽菌MLST数据库进行在线分析https://pubmlst.org/bpseudomallei/,得到每个基因对应的等位基因号。将7个等位基因号按ace-gltB-gmhD-lepA-lipA-narK-ndh顺序串联上传比对,获得菌株的ST型(Sequence type)。
| 基因 | 引物序列(3’→5’) |
| ace | 正义 GAATCGCCTTCACCATGTC |
| 反义 CCGCGCTTCTCAAAACGATA | |
| gltB | 正义 ACGCTCGCGATCGCGATGAA |
| 反义 TTCAGCAGGAGCGTCTGCTG | |
| gmhD | 正义 GCAGTTCCTGTATGCGTA |
| 反义 GAAGCACTGGTACTTGCC | |
| lepA | 正义 CATATTCGCAATTTCTCGATC |
| 反义 CACGAGCATCACGACGCCG | |
| lipA | 正义 TCCGATCAAGATCGTCCCGA |
| 反义 AGGTATTGGCCGATCGTCAG | |
| narK | 正义 CTACTCGTGCGCTGGGAT |
| 反义 GACGATGACGGCACCCAC | |
| ndh | 正义 AGTCGCGACGTTCTACAC |
| 反义 CGAGTTGCAGACGAGATAT |
1.4 同源比对进化分析
以BP145A、BP145B以及从NCBI上获得的20株类鼻疽菌菌株全基因组蛋白序列文件为输入文件,Orthofinder2进行同源比对进化分析,http://etetoolkit.org/treeview/在线网站可视化物种进化树。
1.5 SNP/Indel分析bwa软件对145A和145B进行Genomemapping,samtools和bcftools软件进行变异分析,生成结果文件。
1.6 细菌胞内生存实验使用含有10%胎牛血清的DMEM培养基(完全培养基)培养A549细胞,待细胞密度达到80%时,更换培养基,以MOI=10感染A549细胞,共培养90 min后弃去培养基,PBS洗细胞3次,加入预热的含250 μg/mL卡那霉素的完全培养基,继续孵育至相应时间点(2、4、6、8 h)后用无菌PBS清洗细胞3次,加入1 mL的0.1% Triton X-100裂解细胞,按10倍的梯度倍比稀释后,取10 μL稀释液涂布于24孔LB平板上,37 ℃培养36 h,计数菌落。
1.7 蛋白提取挑取LB琼脂平板上的类鼻疽菌单个菌落,接种到LB培养基中,振荡培养12 h后,4 ℃,4 500×g离心30 min,收集细菌沉淀和培养上清。细菌沉淀用预冷的PBS洗涤,重悬于裂解液中,超声3 min,4 ℃ 12 000×g离心10 min,收集上清即为全菌蛋白(total bacterial protein,TBP)。-80 ℃保存。
1.8 双向电泳-质谱分析取出冻存样品,加入1.75 μL IPG Buffer以及水化液至终体积350 μL,混匀,室温放置30 min,15 ℃,26 000 r/min离心1 h,取上清进行聚焦电泳,胶条平衡后进行SDS-PAGE电泳,溴酚蓝刚刚迁移出凝胶底部时终止电泳。固定30 min,脱色12 h,进行图像扫描,用保鲜膜包裹4 ℃保存备用。
将保存的凝胶置于明亮的灯光下,用剪过的吸头戳取蛋白点,放入PCR管中,更换脱色液进行脱色至无色透明,用真空离心浓缩仪抽干至胶块呈白色颗粒状,胰酶消化13.5 h后,分别加入肽段萃取液Ⅰ、Ⅱ、Ⅲ分别温育1 h,合并3次的提取液,使用真空离心浓缩仪中干燥肽段萃取至无液体残余。用2 μL 0.5%三氟乙酸溶液充分溶解管壁上的肽段(应无明显沉淀可见),用串联飞行时间质谱仪(5800MALDI-TOF/TOF)进行测试分析。
1.9 统计学分析每组实验均重复3次,结果以x±s表示,利用GraphPad Prim 7.0进行独立样本t检验,P < 0.05认为有统计学意义。
2 结果与分析 2.1 145A、145B的微生物学鉴定145A、145B均表现出典型的类鼻疽菌菌落形态,血平板上生长24 h可见光滑型菌落,48 h后变为粗糙型。出现表面褶皱,培养物伴有强烈霉臭味,革兰染色阴性,镜下可见红色短杆菌,经16s rRNA鉴定为类鼻疽菌。
2.2 145A和145B全基因组的基本特征145A的基因组全长为7 120 221 bp,其中1号染色体4 007 759 bp,2号染色体3 112 462 bp,平均GC含量为68%,共预测到5 869个编码基因、88个tRNA、12个rRNA(图 1A、B)。对其编码基因对应蛋白序列进行COG功能注释,共有5 099个蛋白获得COG功能注释(图 2Ⅰ),占全部基因的86.88%。145B的基因组全长为7 120 158 bp,1号染色体4 007 771 bp,2号染色体3 112 387 bp,平均GC含量为68%,共5 857个编码基因、88个tRNA、12个rRNA(图 1C、D)。对其编码基因对应蛋白序列进行COG功能注释,共有5 094个蛋白获得COG功能注释(图 2Ⅱ),占全部基因的86.97%。
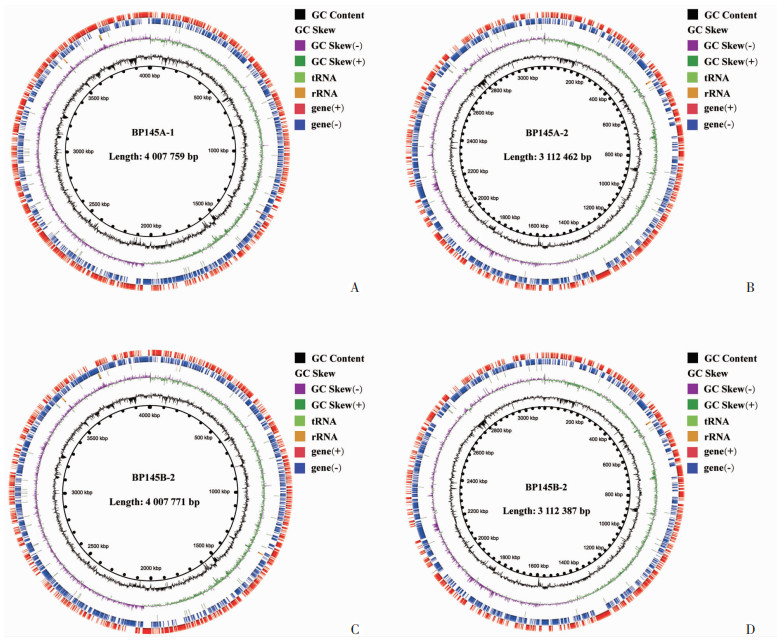
|
| A:145A 1号染色体圈图;B:145A 2号染色体圈图;C:145B 1号染色体圈图;D:145B 2号染色体圈图从内向外第1圈为基因组大小的标识;第2圈为GC含量,向外的部分表示该区域GC含量高于全基因组平均GC含量,峰值越高表示与平均GC含量差值越大,向内的部分表示该区域GC含量低于全基因组平均GC含量,峰值越高表示与平均GC含量差值越大;第3圈为GC skew值,具体算法为(G-C/G+C),在生物意义上该值为正值时正链更倾向于转录CDS,为负值时负链更倾向于转录CDS;第4圈、第5圈分别为负链和正链上的CDS 图 1 145A、145B染色体圈图 |
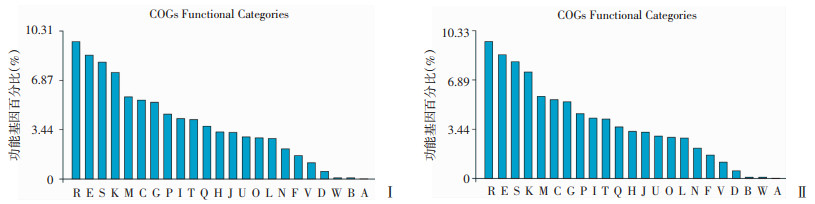
|
| Ⅰ:原发分离菌株145A COG功能注释;Ⅱ:复发分离菌株145B COG功能注释A:RNA加工和修饰;B:染色质结构和动力学;C:能量生产和转换;D:细胞周期调控、细胞分裂和染色体分配;E:氨基酸转运和代谢;F:核苷酸转运和代谢;G:碳水化合物转运和代谢;H:辅酶转运和代谢;I:脂质转运和代谢;J:翻译、核糖体结构和生物合成;K:转录;L:复制、重组和修复;M:细胞壁/细胞膜/胞外被膜生物合成;N:细胞运动;O:翻译后修饰、蛋白质转换、伴侣;P:机离子转运和代谢;Q:次级代谢产物的生成、转运和代谢;R:一般功能预测;S:功能未知;T:信号转导机制;U:胞内运输、分泌和囊泡运输;V:防御机制;W:胞外结构;Y:细胞核结构;Z:细胞骨架 图 2 145A、145B蛋白质COG聚类分析 |
2.3 MLST分型
目前PubMLST数据库中共有6 103株类鼻疽菌的MLST分型信息,其中有310株类鼻疽菌来自中国,对这310株类鼻疽菌的分析发现,分离自我国的类鼻疽菌共有89个型别,表明我国环境中类鼻疽菌的菌株多样。进一步地,根据PubMLST数据库比对7个管家基因(ace、gltB、gmhD、lepA、lipA、narK、ndh),确定145A和145B的分型均为ST58型(图 3),表明145A和145B亲缘关系较近。在PubMLST数据库中,ST58型共有47株,23株来自中国,其中BPC008、BPC025、BPC027、BPC045、BPC065、BPC079、BPC081、BPC059、BPC091、BPC096、BPC138、BPC141、BPC167、BPC104、BPC107、BPC111等等为本室提供,19株来自泰国,其余几株来自中国台湾、马来西亚、澳大利亚。
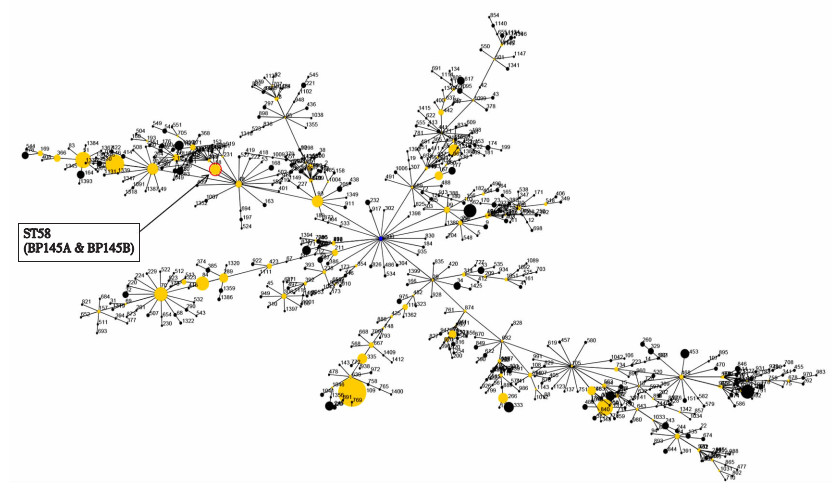
|
| 图 3 基于管家基因的MLST分型 |
2.4 同源比对进化分析
通过Orthofinder2软件构建基于全基因组蛋白序列的物种系统发育树,如图 4所示,结果表明,145A和145B位于同一分支上,提示145B可能为145A在初次感染治疗之后肺部的滞留菌株。并且与国内标准菌株BPC006以及另一国内菌株B.pseudomallei HN1亲缘关系较近,表明145A可能是本地菌株变异而来。
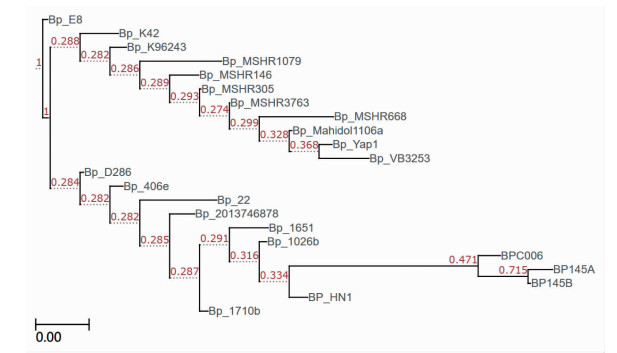
|
| 图 4 基于全基因组蛋白序列的进化树分析 |
2.5 SNP/Indel分析
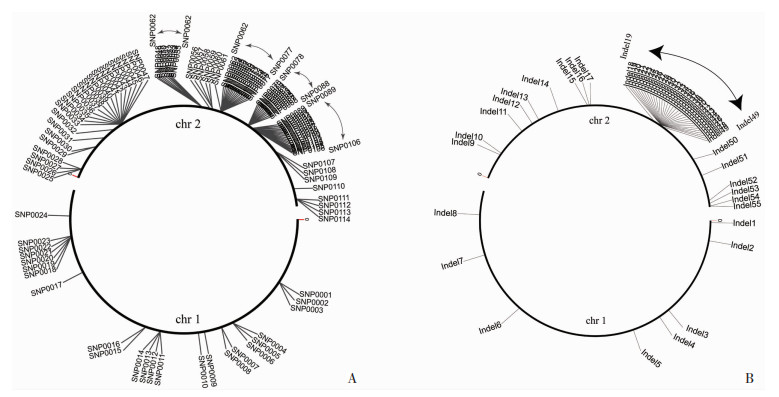
对145A、145B进行全基因组水平SNP分析,发现在145B基因组中共有114个SNP(图 5A),其中有89个SNP落入编码区域,25个SNP在非编码区域,1号染色体上只有24个SNP,大部分SNP在2号染色体上(90个)。同时共检测到55个Indel位点(图 5B),其中有31个Indel位点落入编码区域,24个Indel位于非编码区域,在1号染色体上仅有8个位点存在Indel,绝大部分Indel位于2号染色体(47)。
|
| A:145B相对于145A全基因组SNP位点;B:145B相对于145A全基因组Indel位点 图 5 145B全基因组SNP、Indel位点 |
2.6 原发株和复发株蛋白表达水平的变化
对145A和145B的蛋白表达进行分析,双向电泳结果如图 6A、图 6B所示,质谱结果显示共有68个蛋白的表达发生了显著改变(图 6C),属于20个不同的功能类别(表 2),主要集中分布在ATP binding、Oxidoreductase activity、Hydrolase activity、Catalytic activity、DNA binding、Protein secretion by the type Ⅲ secretion system、Antibiotic resistance related protein transport等7个类别,另有18个蛋白属未知功能(表 2)。
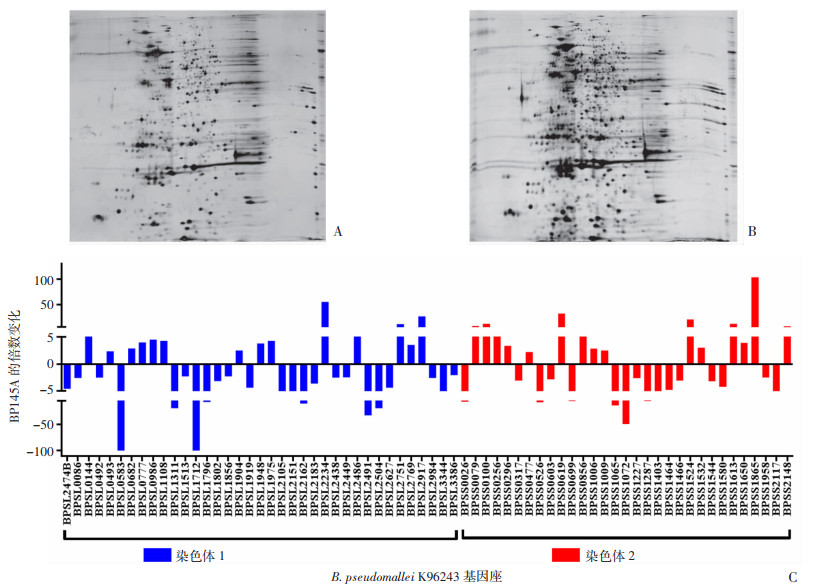
|
| A:145A双向电泳图谱;B:145B双向电泳图谱;C:145A、145B差异蛋白变化 图 6 145A、145B差异表达蛋白 |
| 类别 | 蛋白名称 | 数目 |
| ATP结合 | BPSS1865;BPSS0856;BPSS0477;BPSL2984;BPSL1856;BPSL0682 | 6 |
| 氧化还原酶活性 | BPSS1464;BPSS1466;BPSL2449 | 3 |
| 水解酶活性 | BPSL2486;BPSL1948;BPSL2504 | 3 |
| 催化活性 | BPSL2627;BPSL2438;BPSL1712;BPSL0493 | 4 |
| DNA结合 | BPSS2148;BPSS2117;BPSS0317;BPSL3344;BPSL1919;BPSL1311;BPSL0777;BPSL0583;BPSS0699 | 9 |
| Ⅲ型分泌系统分泌的蛋白质 | BPSS1613;BPSS1532;BPSS1524;BPSS1403;BPSS1544 | 5 |
| 抗生素耐药相关蛋白转运 | BPSS1287;BPSS0256;BPSL2234;BPSL2151;BPSL1975;BPSL1802 | 6 |
| 功能未知 | BPSL1108;BPSS1072;BPSL2917;BPSS1065;BPSL1904;BPSL2474B;BPSL0144;BPSL2183;BPSS1958;BPSS0026;BPSL2751;BPSL0492;BPSL0086;BPSS1227;BPSS0603;BPSL2105;BPSL1796;BPSS0079 | 18 |
| 其他 | BPSS0619;BPSS0296等 | 14 |
2.7 原发株和复发株胞内生存能力的改变
对145A和145B的胞内生存能力进行检测,结果如图 7所示,在肺上皮A549细胞中,复发株145B的胞内生存能力明显强于原发株145A。
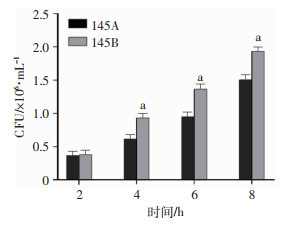
|
| P < 0.05,与145A比较 图 7 145A和145B胞内生存能力差异 |
3 讨论
本研究中,我们首先对同一个患者时隔5年分离到的2株类鼻疽菌进行了系统微生物学鉴定,表明其确为类鼻疽菌。鉴于菌株的多样性,无法准确判断导致类鼻疽复发的确切原因。为此,我们进一步通过全基因组测序、MLST分型、进化树分析、SNP/Indel分析排除了不同菌株导致的再感染,同时鉴定了复发株中主要的基因组变化,这些基因组的变化可能会增加细菌对抗生素的耐药性及其胞内生存能力。胞内生存实验表明复发株的胞内存活增殖能力明显增强,提示类鼻疽菌可能在机体长期的选择压力下微进化,使其更加适应在宿主体内的生存而实现长期滞留。
病原菌与宿主的相互作用是决定其感染和发病的前提。作为一种兼性胞内病原体,在感染过程中,类鼻疽菌面临来自宿主的多种威胁,包括营养缺乏[4]、抗生素、氧气限制[5]以及宿主自身防御机制[6]等,另一方面,类鼻疽菌在感染过程中将毒力蛋白注入宿主细胞内,干扰宿主免疫清除及代谢反应,以使其能在宿主细胞中长期存活。全面了解类鼻疽菌在宿主细胞内表达的蛋白对揭示类鼻疽的发病机理至关重要,因此我们使用双向电泳分析了从不同时期分离出的类鼻疽菌中提取的全菌蛋白,共发现了68个差异表达蛋白,这些蛋白可能是类鼻疽菌得以在宿主细胞内长期潜伏的重要原因。深入分析发现,这些差异表达蛋白涉及众多Ⅲ型分泌系统(typeⅢ secretion system,T3SS)相关蛋白。T3SS是许多革兰阴性的重要毒力因子,可将细菌分泌蛋白注入宿主细胞,干扰宿主生理过程[7]。研究表明,类鼻疽菌T3SS-3在类鼻疽菌逃逸吞噬囊泡,引起胞内持续性感染中发挥着关键作用,MUANGSOMBUT等[8]研究发现T3SS-3 bsaQ突变体表现出逃逸吞噬囊泡能力减弱,胞内生存能力降低,类似地,T3SS-3蛋白BsaZ[9]、BsaU[10]、BipC[11]、BopC[12]的表达缺失也会导致吞噬体逃逸的延迟。此外,我们发现差异蛋白中还涉及耐药相关蛋白,类鼻疽菌胞内寄生的特性使其可在宿主细胞中寄生以躲避宿主免疫系统和抗生素的攻击,同时类鼻疽菌自身具有较强的耐药性,对多种抗生素存在不同程度的耐药,使得目前在降低类鼻疽复发方面尚未取得较好的效果。现有研究显示,类鼻疽菌可通过多种途径来增强其耐药性,AUNKHAM等[13]研究发现,类鼻疽菌可以通过下调外膜孔蛋白Omp38的表达从而对头孢他啶、美罗培南及亚胺培南产生耐药;RHOLL等发现敲除penA基因后,阿莫西林、羧苄西林和氨苄西林对类鼻疽菌的MIC值降低16~42倍[14];另外,细菌可以依靠其拥有的外排泵系统将进入胞内的抗生素重新排出,减少胞内抗生素的聚积,从而产生耐药,敲除bpeEF-oprC基因后,甲氧苄啶对类鼻疽菌的最小抑菌浓度(MIC)值降低近10倍,使耐甲氧苄啶菌株转变为甲氧苄啶敏感菌株[15];而CHANTRATITA发现编码青霉素结合蛋白的BPSS1219基因缺失导致头孢他啶作用靶点的改变,是类鼻疽菌对头孢他啶耐药的主要因素[16]。而在慢性或潜伏感染状态下,细菌可能以非复制性的代谢状态(类似厌氧菌)存在于细胞内,在这种情况下,它们对常规抗生素的敏感性减弱,大大增加了细菌的胞内存活能力[17]。
综上所述,本研究通过此例复发类鼻疽的系统病原学鉴定及生物学特性研究,初步发现参与调节毒力、耐药、能量代谢等方面的蛋白质表达差异,促进类鼻疽菌在宿主内的定殖和适应,这些差异表达的蛋白质可能是类鼻疽菌建立潜伏感染的关键所在。我们拟在后续研究中围绕这些蛋白质展开进一步深入研究,以期为类鼻疽菌的持续性感染机制以及防治提供理论指导。
| [1] |
LIMMATHUROTSAKUL D, GOLDING N, DANCE D A B, et al. Predicted global distribution of Burkholderia pseudomallei and burden of melioidosis[J]. Nat Microbiol, 2016, 1(1): 1-13. |
| [2] |
WIERSINGA W J, VIRK H S, TORRES A G, et al. Limmathurotsakul D: Melioidosis[J]. Nat Rev Dis Primers, 2018, 4: 17107. |
| [3] |
PEACOCK SJ. Melioidosis[J]. Curr Opin Infect Dis, 2006, 19(5): 421-428. |
| [4] |
SCHMIDT I H E, GILDHORN C, BÖNING M A L, et al. Burkholderia pseudomallei modulates host iron homeostasis to facilitate iron availability and intracellular survival[J]. PLoS Negl Trop Dis, 2018, 12(1): e0006096. |
| [5] |
RUSTAD T R, HARRELL M I, LIAO R, et al. The enduring hypoxic response of Mycobacterium tuberculosis[J]. PLoS ONE, 2008, 3(1): e1502. |
| [6] |
ALLWOOD E M, DEVENISH R J, PRESCOTT M, et al. Strategies for intracellular survival of Burkholderia pseudomallei[J]. Front Microbiol, 2011, 2: 170. |
| [7] |
VANDER BROEK C W, STEVENS J M. TYPE Ⅲ secretion in the melioidosis pathogen Burkholderia pseudomallei[J]. Front Cell Infect Microbiol, 2017, 7: 255. |
| [8] |
MUANGSOMBUT V, SUPARAK S, PUMIRAT P, et al. Inactivation of Burkholderia pseudomallei bsaQ results in decreased invasion efficiency and delayed escape of bacteria from endocytic vesicles[J]. Arch Microbiol, 2008, 190(6): 623-631. |
| [9] |
BURTNICK M N, BRETT P J, NAIR V, et al. Burkholderia pseudomallei type Ⅲ secretion system mutants exhibit delayed vacuolar escape phenotypes in RAW 264.7 murine macrophages[J]. Infect Immun, 2008, 76(7): 2991-3000. |
| [10] |
PILATZ S, BREITBACH K, HEIN N, et al. Identification of Burkholderia pseudomallei genes required for the intracellular life cycle and in vivo virulence[J]. Infect Immun, 2006, 74(6): 3576-3586. |
| [11] |
KANG W T, VELLASAMY K M, CHUA E G, et al. Functional characterizations of effector protein BipC, a type Ⅲ secretion system protein, in Burkholderia pseudomallei pathogenesis[J]. J Infect Dis, 2015, 211(5): 827-834. |
| [12] |
SRINON V, MUANGMAN S, IMYAEM N, et al. Comparative assessment of the intracellular survival of the Burkholderia pseudomallei bopC mutant[J]. J Microbiol, 2013, 51(4): 522-526. |
| [13] |
AUNKHAM A, SCHULTE A, WINTERHALTER M, et al. Porin involvement in cephalosporin and carbapenem resistance of Burkholderia pseudomallei[J]. PLoS ONE, 2014, 9(5): e95918. |
| [14] |
RHOLL DA, PAPP-WALLACE KM, TOMARAS AP, et al. Molecular investigations of PenA-mediated beta-lactam resistance in Burkholderia pseudomallei[J]. Front Microbiol, 2011, 2: 139. |
| [15] |
PODNECKY N L, WUTHIEKANUN V, PEACOCK S J, et al. The BpeEF-OprC efflux pump is responsible for widespread trimethoprim resistance in clinical and environmental Burkholderia pseudomallei isolates[J]. Antimicrob Agents Chemother, 2013, 57(9): 4381-4386. |
| [16] |
CHANTRATITA N, RHOLL DA, SIM B, et al. Anti-microbial resistance to ceftazidime involving loss of penicillin-binding protein 3 in Burkholderia pseudomallei[J]. Proc Natl Acad Sci USA, 2011, 108(41): 17165-17170. |
| [17] |
HAMAD MA, AUSTIN CR, STEWART AL, et al. Adaptation and antibiotic tolerance of anaerobic Burkholderia pseudomallei[J]. Antimicrob Agents Chemother, 2011, 55(7): 3313-3323. |




