细胞是有机体结构和生理功能的基本单位。人体内至少有60种不同组织,100余种细胞大类,800余种细胞亚类[1]。在体外克隆形成、单细胞移植等实验中,原先认为均质的细胞群体在表型和功能上的差异逐渐显露,但由于传统群体测序方法(bulk sequencing)的局限性,导致无法从细胞群体平均值中有效提取出单个细胞的数据[2]。单细胞测序(single cell sequencing,SCS)技术凭借分子水平的高分辨率、皮克级别的低起始检测阈、无须预分选细胞等优势,在探索细胞异质性、发现细胞新类型、揭示分子调控新机制等方面展现了强大能力[3]。SCS技术不仅在2011、2013、2018年及2019年陆续被Science、Nature Methods等顶尖杂志评为年度技术,更是伴随着人类细胞图谱(human cell atlas,HCA)计划公布、人血细胞分子图谱(atlas of blood cells,ABC)研究联盟成立,来到了时代的浪尖[4-6]。
造血系统是维持生命正常运行的重要组成部分,其中造血干细胞(hematopoietic stem cell,HSC)具有自我更新和定向分化的能力, 因而被认为是所有血细胞的起源[7]。当前,对造血干细胞的研究大部分是在骨髓微环境改变或暴露于常氧水平等条件下对目标细胞进行检测,很难如实反映HSC在正常环境下的状态和功能。与此同时,通过免疫表型纯化目标细胞的方法也逐渐难以满足深入研究的需求。SCS技术的出现,可以较好地解决当前研究中所面临的矛盾,为进一步探索造血系统发育路径、谱系分化及病理改变等方面提供有力的工具。现就近年来SCS技术发展,以及在造血系统和血液病研究中的应用及进展,进行回顾与展望。
1 单细胞测序技术的概况单细胞测序技术主要分为3类,即单细胞全基因组测序、单细胞转录组测序和表观基因组测序,其分别通过检测每个细胞中DNA、RNA或者甲基化水平、组蛋白修饰、染色质结构等方面的差异,揭示细胞群体内部异质性,探索个体发育、细胞分化、病理生理发展过程等方面的基因调控网络[8-9]。单细胞测序的主要流程可以概括为四步:获取单细胞、扩增检测目标、建库及测序、数据分析和展示。其中单细胞获取和扩增技术是关键步骤,也是不同技术策略优化的主要方向。
1.1 单细胞的获取获取的单细胞样本需要达到一定的活性比例和数目要求,一是保证提取DNA或mRNA的质量,二是确保检测范围能覆盖到稀有细胞。当前常用方法有:荧光激活流式细胞分选法(flow-activated cell sorting,FACS)、激光捕获显微切割法(laser capture microdissection,LCM)、微流控技术等。
FACS是通过流式分选仪,获取特异性荧光物质标记的目标细胞,具有高通量、高精度、高特异性等优势[10]。研究人员可以通过设计染色方案,进行免疫表型鉴定、DNA含量测定、细胞亚群定性等分析,甚至可以检测到占比小于1%的稀有细胞群体。但该方式也带有FACS本身固有的技术缺陷,如荧光光谱重叠、细胞机械性损伤、样本易污染等情况。
LCM是操作人员通过显微镜,明确目标细胞位置,利用低能红外激光切取需要的区域[11]。该方法的优点是可以适用于固定样本,且保持细胞的组织形态和空间信息,缺点是细胞受到切割后完整性容易被破坏,同时人工操作也带来了样本偏好和通量较低等问题。微流控技术脱离了依据细胞本身抗体进行分离选择的传统思路,利用微流控芯片对流体的精确控制,实现单细胞离散,并将微粒与每个细胞结合,在油性试剂管道中,形成“一个细胞+一个微粒”的油包水状态,为接下来的扩增和测序创造了干扰性极小的独立空间[12]。
微流控技术拥有自动化程度高、细胞通量大、单个细胞测序成本低等巨大优势,被商用化平台广泛采用。现阶段,微流控技术对样本细胞总体数目、细胞大小均匀程度等方面仍存在较严苛的要求,是今后该技术改进的主要着力点。
1.2 单细胞扩增技术正常情况下单个细胞的DNA含量为4~6 pg,mRNA含量仅0.5 pg左右,只有扩增至纳克级别以上,才能满足深度测序的要求。为保证扩增的高效性、覆盖性以及均一性,同时减少非特异性扩增,研究人员针对单细胞的基因组、转录组和表观遗传组的不同测序需求,开发出了多种扩增技术。
全基因组扩增(whole-genome amplification,WGA)是单细胞基因组测序的主要技术。最早使用的简并寡核苷酸引物PCR (degenerate oligonucleotide primed PCR,DOP-PCR),利用含有6个随机碱基的引物进行PCR反应,实现了全基因组的扩增[13]。这类方法简单易行,成本较低,但由于存在扩增效率低、基因覆盖不全、非特异性扩增等短板,逐渐无法满足实验的需求。多重置换扩增法(multiple displacement amplification,MDA)利用Phi29 DNA聚合酶强大的延伸活性和高保真度等特点,使用6个随机碱基组成寡核苷酸引物,很好地解决了扩增效能和基因覆盖度等问题[14]。多次退火环状循环扩增(multiple annealing and looping-based amplification cycles,MALBAC)和转座子插入的线性扩增(linear amplification of transposon insertion,LIANTI)等技术的开发,有效纠正扩增过程中的序列偏移,但也带来了假阳性率高的问题[15]。因此,高均一性、高覆盖性、高效能仍然是目前技术优化的主要方向。
全转录组扩增(whole-transcriptome amplification,WTA),是将mRNA逆转录成cDNA,再利用WGA技术进行扩增,从而实现单细胞的转录组扩增目的。2009年,汤富酬研究组第一次报道了单细胞转录组的测序分析结果[16]。而后,Smart-seq技术通过识别出带有poly(A)尾的mRNA并进行逆转录,实现了全长转录本的检测,但存在高丰度转录本偏向扩增、长序列转录效能低等问题。
单细胞表观遗传测序技术主要用于探索基因组以外的遗传信息对细胞功能的影响。从经典染色体构想捕获技术发展而来的高通量染色体构象捕获技术(high-throughput chromosome conformation capture,Hi-C),可以定量检测基因组的染色质间交联[17]。随后出现了单细胞亚硫酸盐测序法(single-cell reduced-representation bifulfite sequencing,scRRBS),实现了单细胞层面上的DNA甲基化分析[18]。染色质转座酶可及性测序(assay for transposase accessible chromatin with high-throughput sequencing,ATAC-seq),通过Tn5转座酶结合开放染色质,构建了小鼠染色质可及性的细胞图谱,以及胎儿染色质可及性的人类细胞图谱,为下一步探索基因调控网络,提供更多参考信息和全新思路[19]。
2 造血系统认知的重建和完善造血系统是有机体不可或缺的重要组成部分,其可以根据机体生理需要和外界影响因素进行严格调控并达到动态平衡,从而为机体源源不断地提供各种功能不同的血细胞[20]。SCS技术的发展,促使我们从单细胞水平以更完善、更准确的方式,重新审视以往的认知体系,进而加深对造血系统复杂而又精密结构的理解。
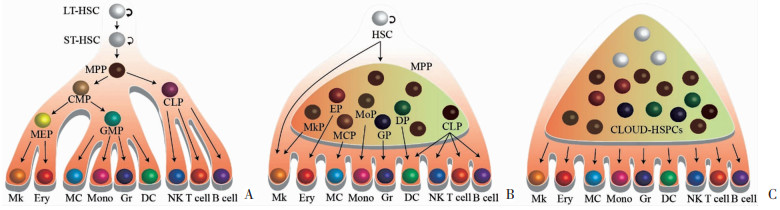
2.1 构建造血细胞图谱骨髓移植不仅可以挽救致死剂量辐照的小鼠的生命,而且在临床上,已经成功救治了无数血液系统恶性肿瘤的患者。人们将其中具备重建造血功能的细胞群体定义为HSC,并初步建立了对造血系统的认知体系。随着细胞体外集落培养和免疫标记等技术的出现,进一步加深了我们对血液细胞在发育、分化和功能方面的了解。依据细胞的免疫学特征以及展现的功能,HSC分为三群,即长期造血干细胞(long-term hematopoietic stem cell,LT-HSC)、短期造血干细胞(short-term hematopoietic cell,ST-HSC)和多潜能祖细胞(multipotent progenitor,MPP),继而向下分化出谱系限制性的造血祖细胞,最终形成各种成熟的血液细胞[21](图 1A)。这种经典“树”状结构模型的建立,对造血系统的认知产生了深刻影响。
|
| LT-HSC:长期造血干细胞;ST-HSC:短期造血干细胞;MPP:多潜能祖细胞;CMP:共同髓系祖细胞;MEP:巨核系-红系祖细胞;GMP:粒系-单核系祖细胞;CLP:共同淋巴系祖细胞;Mk: 巨核细胞;Ery: 红细胞;MC: 肥大细胞;Mono: 单核细胞;Gr: 粒细胞;DC: 树突状细胞;NK: 自然杀伤细胞;T cell: T淋巴细胞;B cell: B淋巴细胞;MkP: 巨核细胞祖细胞;EP: 红细胞祖细胞;MCP: 肥大细胞祖细胞;MoP: 单核细胞祖细胞;GP: 粒细胞祖细胞A:造血分化树状模型;B:造血分化扁平状结构模型;C:“低启动未分化造血干祖细胞连续体”模型 图 1 造血细胞分化模型 |
细胞分类应当基于功能特性、发育分化、区域分布等多个方面进行综合考量,而传统定义的免疫分型,不能明确展现出目标群体中的异质细胞。SCS技术不仅可以验证部分已知标记的细胞群体,还可以挖掘出未知的细胞类型,结合功能分析,筛选出新的细胞群体或归纳和细化已有的细胞分群。研究人员利用细胞条形码技术追踪单个细胞移植后自然状态下的发育分化情况,揭示了MPP存在着不同分化偏向的群体,这也暴露出传统流式细胞术依赖免疫表型进行细胞分类的局限[22]。2020年,程涛研究组采用单细胞转录组测序技术,建立了小鼠正常情况下造血干/祖细胞(hematopoietic stem/progenitor cell,HSPC)的单细胞转录图谱,并对比移植1周后HSPC的变化情况[23]。在高分辨率的检测水平上,HSC和MPP群体均展现出高度的异质性。根据转录组的特征,研究人员重新划分HSPC亚群,并将HSC中自我更新能力最强的细胞定义为tHSC1和tHSC2,淋系偏向分化的HSC称为tHSC3,而MPP则分为细胞干性最强的tMPP1、髓系偏向的tMPP2、巨核系-红系偏向的tMPP3和tMPP4以及淋系偏向分化的tMPP5,修改并完善了经典造血模型。随后,该课题组继续采用转录组测序技术,结合免疫表型、转录因子、长链非编码RNA(long non-coding RNA,lncRNA)等角度,检测了21例健康成年人骨髓或外周血细胞,共鉴定了32种造血细胞,覆盖了HSC到祖细胞以及各种终末分化细胞,绘制了较为完整的人血液单细胞转录组图谱[24]。
目前这些研究说明SCS技术不仅可以证实经典模型中的部分理论,如存在多向分化潜能的干/祖细胞、造血系统是分层结构等,而且对细胞发育分化的稳定性、层次分级的合理性提出了质疑,引导我们思考,如何将单细胞层面界定的新群体,映射匹配到既往定义的各个造血细胞群体。从单细胞角度看,造血细胞的分化进程是渐进推行的,自我更新和谱系分化能力的改变也是逐渐形成的,随着细胞干性降低,HSPC呈现出多能性到寡能性的转变,这解释了基于表型的细胞分群具备不明确性这一现象,契合细胞免疫标记连续的情况,也颠覆了“树”状模型里分化节点和谱系关系的传统概念。因此,经典的造血细胞分化模型需要重新进行定义和优化,才能接近细胞发育的真相。
2.2 探索造血细胞动态变化人们对造血系统的认知,随着技术进步而不断深入,造血细胞发育和分化的内在机制及影响因素成为新的关注点。其中,谱系追踪技术结合单细胞水平的测序数据,可以将细胞群体的静态信息转化为动态信息,进而推演出细胞的命运走向[25]。
既往研究通过检测干细胞移植后产生的造血细胞群体,发现淋巴系和髓系细胞的比例与供体水平产生了明显差异,且通过多次移植后,细胞构成比例的差异依旧存在,可以推断出HSC中存在不同分化偏向性的细胞群体[26]。研究人员从单细胞层面解析了免疫表型纯化的HSC,进行较为全面的图谱描绘和聚类分析,检测到具有谱系倾向性的细胞亚群,直接证明了HSC和祖细胞存在转录谱异质性,同时也发现一些谱系特异性的转录因子,如红系/巨核系分化中高表达的GATA1、粒系/单核系分化相关的PU.1等[27-28]。此外,利用SCS技术采集不同分化阶段的细胞信息数据,观察特征基因的变化,结合“伪时间”算法进行轨迹分析,可以大致推断细胞间的层级关系[29]。例如,单核细胞不仅可以来自粒系-单核系祖细胞(granulocyte-mononuclear progenitors,GMP)途径,也可以经由单核-树突状祖细胞(monocyte-dendritic progenitors,MDP)途径产生[30]。值得注意的是,巨核细胞除了通过巨核系-红系祖细胞(megakaryocyte-erythroid progenitors,MEP)产生,还可以直接来自于CD41+ HSC,这使得其分化过程在时间上较其他成熟血细胞都要提前,并且很大程度上独立于其他造血谱系[22]。
明确谱系限制的决定因素,一直是研究热点,目前主要分为两种观点,一种观点认为是造血微环境等外界因素起决定性作用,另一种则认为是在干/祖细胞阶段已经确定的内在因素占据主导。第一种观点认为,HSPC呈现一种扁平的层次结构,其中MPP是由系谱偏向的异质性细胞群体构成,可以直接分化为单/双潜能祖细胞,继而产生成熟的血细胞[31](图 1B)。第二种观点认为,细胞特征性基因表达谱提示的是分化潜能,并不是已经决定好的分化路径,因此建立了“低启动未分化造血干祖细胞连续体”(continuum of low primed undifferentiated hematopoietic stem and progenitor cell,CLOUD-HSPC)这一模型[32](图 1C)。在这种模型中,原先定义的祖细胞不应被视为一种相互独立且稳定存在的细胞类型,而是应当看成造血干细胞的“连续体”,在收到环境和机体需求的信号后,暂时聚集出现在特定位置,再向下分化成特定终末血细胞。这种观点突出干细胞的多向分化潜能,强调调控网络的重要性。研究人员通过SPRING算法、谱系特异性标记等技术,在未分化的造血祖细胞中鉴定出7个不同分支,分别对应肥大细胞、粒细胞、单核细胞、树突细胞、淋巴细胞、巨核细胞和红系祖细胞。当这些已经明确分化去向的细胞群体收到机体相应信号后,启动向下分化的程序[33]。这说明细胞分化方向在HSPC层面上就已经明确,并且一旦确定最终的谱系轨迹就不会发生变更。在这套体系内,谱系特异性的转录因子不是细胞命运的决定因素,而是作为一种辅助执行、促进分化的重要因素。
以上两种观点,均是在一定实验基础上,进行推算和分析,给出了相对合理的解释,促进了我们对造血系统的发育认知。但我们必须要清楚认识到两点:一是人为因素掺杂分析过程,对重建细胞发育轨迹和解析细胞命运的影响程度尚不清楚;二是目前技术的局限性,无法动态地观察造血细胞的连续分化过程,这可能会导致其中的一些重要细节被忽略。
3 SCS技术带给临床诊治的新希望血液疾病具有潜伏时间长短不一、临床表现各异、诊断指标繁多等特点,加上疾病进展阶段,各项指标变化较大,给临床诊断、治疗、预后判断带来了巨大挑战。SCS技术给我们提供了在单细胞层面上认识血液疾病的工具,以便重新理解血液疾病的本质,更合理地进行疾病分类分型,并可以指导临床个性化治疗方案的制定。
3.1 认识血液病发生发展的新视角利用SCS技术可以发现,白血病不是由均一的癌细胞诱发,而是呈现出多种癌细胞群体的集合。Steinmetz研究组采用Muta-Seq技术,结合谱系技术追踪细胞核以及线粒体变异情况,成功区分了正常HSC、白血病干细胞(leukemic stem cell,LSC)和白血病前干细胞间的差异,为白血病治疗提供了潜在的靶点[34]。研究人员通过SCS技术,对急性髓性细胞白血病(acute myeloid leukemia,AML)患者的骨髓样本进行了检测,根据基因表达谱差异,划分LSC亚群,并对比正常造血细胞的图谱,发现LSC亚群可以逐一对应到正常细胞的不同群体中,并且基本吻合正常分化发育的轨迹[35]。这提示我们可能在某一正常发育阶段,部分细胞出现了突变和恶性增殖,然后发育停滞,形成肿瘤干细胞。这些发现为LSC的来源以及AML的发生提供了合理解释,加深了我们对白血病发病机制和异质性的理解,正确识别癌细胞及其亚群,为制定可以发挥持续作用的LSC靶向治疗策略,以及相关白血病的靶向治疗提供了理论基础和可行途径。最近,程涛课题组完成了再生障碍性贫血的图谱,并发现DNA损伤相关的可变剪切在骨髓增生异常综合征(myelodysplastic syndrome,MDS)和再障之间出现了相同改变,提示这可能是再障恶性转化的某种机制[36]。而再障患者的CD4+和CD8+T细胞测序结果可以发现,T细胞不仅出现体细胞突变,还显著提高与HSPC的相互作用,进一步明确了免疫异常对再障的发生发展起到调控作用[36-37]。
3.2 开辟血液病治疗的新途径由于技术限制,目前血液疾病治疗手段单一且效果不稳定,无论是化疗等非靶向治疗,还是免疫治疗、基因治疗,大多不能根据细胞异质性和基因差异性制定个性化治疗策略,导致无法达到预期疗效,病情复发率高。传统化疗药物治疗过程中,疾病的进展及预后主要依赖细胞形态进行大致判断,而SCS技术可以通过基因突变、表观修饰异常等信息给予我们更为精准追踪疾病变化的能力。如地西他滨或阿扎胞苷治疗MDS时,TP53、ASXL1、RAD21等基因表达的上调或下降,可以反映化疗药物的治疗敏感性,进而指导化疗方案的调整和优化[38-39]。目前针对FLT3、IDH1/2、PDGFR、BCL-2等靶点的治疗药物已经在临床上取得了较好的疗效,但长期使用存在耐药性问题[40-41]。酪氨酸激酶抑制剂(tyrosine kinase inhibitors,TKIs),是一类有效治疗慢性粒细胞白血病(chronic granulocytic leukemia,CML)的药物,其疗效受到LSC异质性的影响[42]。针对这一问题,研究人员利用WGA技术,发现TKIs治疗不敏感的人群在LSC中存在CD45RA-的群体,结合TKIs作用特点,进一步成功找到Lin-CD34+CD38-/lowCD45RA-c-Kit-CD26+这一群体,为下一步靶向药物筛选和治疗方案的优化提供了参考[43]。造血干细胞移植(hematopoietic stem cell transplantation,HSCT)是当前唯一可以治愈急性白血病、重型再生障碍性贫血等恶性血液疾病的方案。SCS技术可以协助判断不同突变基因类型的患者进展成恶性血液病的风险大小,并且衡量能否接受HSCT治疗,以及评估预后情况,如JAK2、CBLL380P、ASXL1、IDH1/2等基因突变提示疾病恶化风险大、生存率低等情况,有进行HSCT的必要性[44]。
4 展望随着单细胞获取和扩增技术的不断迭代更新,SCS技术已经在诸多领域取得了大量突破性成果,尤其是在造血系统研究中进行了成功应用,揭示造血细胞的异质性,绘制了多个血液相关的细胞图谱,重建了发育分化的调控网络,在临床诊断和治疗中发挥了巨大作用。接下来的研究重点可能主要集中在以下几点。
4.1 技术升级与整合随着当前10X Genomics、BD Rhapsody等商用测序平台的推广运用,以及Smart-seq 2、Drop-seq和CEL-Seq(cell expression by linear amplification and sequencing)等技术的不断涌现,技术短板正被逐步消除,从而使我们尽可能接近检测目标的真实情况[45-46]。但目前SCS技术在测序过程中仍存在检测成本较高、细胞分选过程中容易发生污染、损伤等情况,应在文库构建和数据分析上进一步改进和优化,解决细胞损耗较高、检测深度不足、分析存在偏向性等实际问题,强化对单细胞的动态观察和功能检测能力,以满足深入的研究需求。
如同bulk sequencing无法在细胞群体平均值中展现细胞异质性的局限性,SCS技术也面临着获取的细胞基因组、转录组等单一维度的细胞数据后,容易出现分析片面性和主观性的困境。因此,多组学分析和空间转录组等多项技术与SCS技术联合运用是今后研究的必然趋势。多组学分析是在同一个细胞中获取两个或者更多的组学信息进行整合分析,以更加全面的角度去解析细胞,如DR-seq(DNA-mRNA sequencing)可以同时对基因组和转录组进行测序,scM & T-seq(simultaneous single-cell methylome and transcriptome sequencing)则可以同时分析DNA甲基化和RNA的信息[47-48]。而空间转录组测序技术是利用组织切片和细胞条形码,实现细胞数据的原位捕获,以弥补SCS技术无法检测基因的时间和空间特异性这一不足。目前这项技术在大脑、心脏、肿瘤的发生、发展方面的研究取得了一定成功,并在肌萎缩性侧索硬化症、黑色素瘤等疾病研究中展开了初步研究[49-50]。随着研究人员对相应技术认识和运用的深入,SCS技术向多组学联合、时空信息整合等方向进行发展,我们相信会有越来越多的细节被挖掘,因此对造血系统的认知将更加清晰、准确。
4.2 基础研究与临床诊治的突破造血系统组成复杂、高度动态,在基础研究中,细胞分类与功能研究是重中之重。目前SCS技术联合多种技术,逐步揭示造血细胞的构成,开始描绘单细胞的动态表达图谱。研究人员利用单细胞诱导移植体系,鉴别出了造血干细胞前体(pre-hematopoietic stem cell,pre-HSC),揭示HSC发育过程的分子特征,并绘制lncRNA图谱[51]。SCS技术大大拓展了观测的深度和广度,并且在细胞全局描绘和动态变化探索方面快速推进,为全面解析细胞的命运和功能指明方向。而临床诊治思路着重于特殊靶点的处理,并动态监测相关因子的变化情况,以期实现病情观察与治疗。如Dnmt3a R878H基因是AML中常见的突变体,通过SCS技术分析发现,Dnmt3a突变所致的AML与哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin, mTOR)激活有关,而mTOR抑制剂对这种AML具有较好的治疗作用,这提示了SCS技术可为疾病的治疗提供潜在治疗靶点[52]。
总体而言,SCS技术不仅对经典理论进行了单细胞尺度下的解读,进一步为临床鉴别诊断血液相关疾病提供了更为精确的指标,并为探索开发针对性更强、效果更好的治疗手段提供了有力支持,有效推动血液疾病诊治水平的精准升级。
| [1] |
HAN X, ZHOU Z, FEI L, et al. Construction of a human cell landscape at single-cell level[J]. Nature, 2020, 581(7808): 303-309. DOI:10.1038/s41586-020-2157-4 |
| [2] |
MORITA Y, EMA H, NAKAUCHI H. Heterogeneity and hierarchy within the most primitive hematopoietic stem cell compartment[J]. J Exp Med, 2010, 207(6): 1173-1182. DOI:10.1084/jem.20091318 |
| [3] |
KOKKALIARIS K D. Dissecting the spatial bone marrow microenvironment of hematopoietic stem cells[J]. Curr Opin Oncol, 2020, 32(2): 154-161. DOI:10.1097/CCO.0000000000000605 |
| [4] |
PENNISI E. Single-cell sequencing tackles basic and biomedical questions[J]. Science, 2012, 336(6084): 976-977. DOI:10.1126/science.336.6084.976 |
| [5] |
DE SOUZA N. Single-cell genetics[J]. Nat Methods, 2013, 10(9): 820. DOI:10.1038/nmeth.2626 |
| [6] |
CHI K R. Singled out for sequencing[J]. Nat Methods, 2014, 11(1): 13-17. DOI:10.1038/nmeth.2768 |
| [7] |
WATCHAM S, KUCINSKI I, GOTTGENS B. New insights into hematopoietic differentiation landscapes from single-cell RNA sequencing[J]. Blood, 2019, 133(13): 1415-1426. DOI:10.1182/blood-2018-08-835355 |
| [8] |
DEMAREE B, DELLEY C L, VASUDEVAN H N, et al. Joint profiling of DNA and proteins in single cells to dissect genotype-phenotype associations in leukemia[J]. Nat Commun, 2021, 12(1): 1583. DOI:10.1038/s41467-021-21810-3 |
| [9] |
ZHANG Y J, WANG D, PENG M, et al. Single-cell RNA sequencing in cancer research[J]. J Exp Clin Cancer Res, 2021, 40(1): 81. DOI:10.1186/s13046-021-01874-1 |
| [10] |
RINKE C, LEE J, NATH N, et al. Obtaining genomes from uncultivated environmental microorganisms using FACS-based single-cell genomics[J]. Nat Protoc, 2014, 9(5): 1038-1048. DOI:10.1038/nprot.2014.067 |
| [11] |
ELLIS P, MOORE L, SANDERS M A, et al. Reliable detection of somatic mutations in solid tissues by laser-capture microdissection and low-input DNA sequencing[J]. Nat Protoc, 2021, 16(2): 841-871. DOI:10.1038/s41596-020-00437-6 |
| [12] |
BOUNAB Y, EYER K, DIXNEUF S, et al. Dynamic single-cell phenotyping of immune cells using the microfluidic platform DropMap[J]. Nat Protoc, 2020, 15(9): 2920-2955. DOI:10.1038/s41596-020-0354-0 |
| [13] |
KISS C, KOST-ALIMOVA M, KLEIN G, et al. Optimisation of the degenerate oligonucleotide primed PCR (DOP-PCR) for capillary thermocycler[J]. Biomol Eng, 2002, 19(1): 31-34. DOI:10.1016/S1389-0344(02)00008-4 |
| [14] |
SPITS C, LE CAIGNEC C, DE RYCKE M, et al. Whole-genome multiple displacement amplification from single cells[J]. Nat Protoc, 2006, 1(4): 1965-1970. DOI:10.1038/nprot.2006.326 |
| [15] |
ZONG C, LU S, CHAPMAN A R, et al. Genome-wide detection of single-nucleotide and copy-number variations of a single human cell[J]. Science, 2012, 338(6114): 1622-1626. DOI:10.1126/science.1229164 |
| [16] |
TANG F, BARBACIORU C, WANG Y, et al. mRNA-Seq whole-transcriptome analysis of a single cell[J]. Nat Methods, 2009, 6(5): 377-382. DOI:10.1038/nmeth.1315 |
| [17] |
RAMANI V, DENG X, QIU R, et al. Massively multiplex single-cell Hi-C[J]. Nat Methods, 2017, 14(3): 263-266. DOI:10.1038/nmeth.4155 |
| [18] |
GUO H, ZHU P, WU X, et al. Single-cell methylome landscapes of mouse embryonic stem cells and early embryos analyzed using reduced representation bisulfite sequencing[J]. Genome Res, 2013, 23(12): 2126-2135. DOI:10.1101/gr.161679.113 |
| [19] |
RANZONI A M, TANGHERLONI A, BEREST I, et al. Integrative single-cell RNA-seq and ATAC-seq analysis of human developmental hematopoiesis[J]. Cell Stem Cell, 2021, 28(3): 472-487.e7. DOI:10.1016/j.stem.2020.11.015 |
| [20] |
GRANJA J M, KLEMM S, MCGINNIS L M, et al. Single-cell multiomic analysis identifies regulatory programs in mixed-phenotype acute leukemia[J]. Nat Biotechnol, 2019, 37(12): 1458-1465. DOI:10.1038/s41587-019-0332-7 |
| [21] |
EAVES C J. Hematopoietic stem cells: concepts, definitions, and the new reality[J]. Blood, 2015, 125(17): 2605-2613. DOI:10.1182/blood-2014-12-570200 |
| [22] |
RODRIGUEZ-FRATICELLI A E, WOLOCK S L, WEINREB C S, et al. Clonal analysis of lineage fate in native haematopoiesis[J]. Nature, 2018, 553(7687): 212-216. DOI:10.1038/nature25168 |
| [23] |
DONG F, HAO S, ZHANG S, et al. Differentiation of transplanted haematopoietic stem cells tracked by single-cell transcriptomic analysis[J]. Nat Cell Biol, 2020, 22(6): 630-639. DOI:10.1038/s41556-020-0512-1 |
| [24] |
XIE X W, LIU M Y, ZHANG Y W, et al. Single-cell transcriptomic landscape of human blood cells[J]. Natl Sci Rev, 2021, 8(3): nwaa180. DOI:10.1093/nsr/nwaa180 |
| [25] |
BOWLING S, SRITHARAN D, OSORIO F G, et al. An engineered CRISPR-Cas9 mouse line for simultaneous readout of lineage histories and gene expression profiles in single cells[J]. Cell, 2020, 181(6): 1410-1422.e27. DOI:10.1016/j.cell.2020.04.048 |
| [26] |
HAAS S, TRUMPP A, MILSOM M D. Causes and consequences of hematopoietic stem cell heterogeneity[J]. Cell Stem Cell, 2018, 22(5): 627-638. DOI:10.1016/j.stem.2018.04.003 |
| [27] |
STABER P B, ZHANG P, YE M, et al. Sustained PU.1 levels balance cell-cycle regulators to prevent exhaustion of adult hematopoietic stem cells[J]. Mol Cell, 2013, 49(5): 934-946. DOI:10.1016/j.molcel.2013.01.007 |
| [28] |
JUN T K, MORIGUCHI T, SUZUKI M, et al. The Gata1 5' region harbors distinct Cis-regulatory modules that direct gene activation in erythroid cells and gene inactivation in HSCs[J]. Blood, 2013, 122(20): 3450-3460. DOI:10.1182/blood-2013-01-476911 |
| [29] |
TRAPNELL C, CACCHIARELLI D, GRIMSBY J, et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells[J]. Nat Biotechnol, 2014, 32(4): 381-386. DOI:10.1038/nbt.2859 |
| [30] |
LIU Z, GU Y, CHAKAROV S, et al. Fate mapping via Ms4a3-expression history traces monocyte-derived cells[J]. Cell, 2019, 178(6): 1509-1525. e19. DOI:10.1016/j.cell.2019.08.009 |
| [31] |
BRAND M, MORRISSEY E. Single-cell fate decisions of bipotential hematopoietic progenitors[J]. Curr Opin Hematol, 2020, 27(4): 232-240. DOI:10.1097/MOH.0000000000000592 |
| [32] |
VELTEN L, HAAS S F, RAFFEL S, et al. Human haematopoietic stem cell lineage commitment is a continuous process[J]. Nat Cell Biol, 2017, 19(4): 271-281. DOI:10.1038/ncb3493 |
| [33] |
OLSSON A, VENKATASUBRAMANIAN M, CHAUDHRI V K, et al. Single-cell analysis of mixed-lineage states leading to a binary cell fate choice[J]. Nature, 2016, 537(7622): 698-702. DOI:10.1038/nature19348 |
| [34] |
VELTEN L, STORY B A, HERNÁNDEZ-MALMIERCA P, et al. Identification of leukemic and pre-leukemic stem cells by clonal tracking from single-cell transcriptomics[J]. Nat Commun, 2021, 12(1): 1366. DOI:10.1038/s41467-021-21650-1 |
| [35] |
QIN P, PANG Y, HOU W, et al. Integrated decoding hematopoiesis and leukemogenesis using single-cell sequencing and its medical implication[J]. Cell Discov, 2021, 7(1): 2. DOI:10.1038/s41421-020-00223-4 |
| [36] |
ZHU C Y, LIAN Y, WANG C C, et al. Single-cell transcriptomics dissects hematopoietic cell destruction and T cell engagement in aplastic Anemia[J]. Blood, 2021, 138(1): 23-33. DOI:10.1182/blood.2020008966 |
| [37] |
LUNDGREN S, KERÄNEN M A I, KANKAINEN M, et al. Somatic mutations in lymphocytes in patients with immune-mediated aplastic anemia[J]. Leukemia, 2021, 35(5): 1365-1379. DOI:10.1038/s41375-021-01231-3 |
| [38] |
UY G L, DUNCAVAGE E J, CHANG G S, et al. Dynamic changes in the clonal structure of MDS and AML in response to epigenetic therapy[J]. Leukemia, 2017, 31(4): 872-881. DOI:10.1038/leu.2016.282 |
| [39] |
CHEN J, KAO Y R, SUN D, et al. Myelodysplastic syndrome progression to acute myeloid leukemia at the stem cell level[J]. Nat Med, 2019, 25(1): 103-110. DOI:10.1038/s41591-018-0267-4 |
| [40] |
DAVER N, WEI A H, POLLYEA D A, et al. New directions for emerging therapies in acute myeloid leukemia: the next chapter[J]. Blood Cancer J, 2020, 10(10): 107. DOI:10.1038/s41408-020-00376-1 |
| [41] |
GEBRU M T, WANG H G. Therapeutic targeting of FLT3 and associated drug resistance in acute myeloid leukemia[J]. J Hematol Oncol, 2020, 13(1): 155. DOI:10.1186/s13045-020-00992-1 |
| [42] |
CHARAF L, MAHON F X, LAMRISSI-GARCIA I, et al. Effect of tyrosine kinase inhibitors on stemness in normal and chronic myeloid leukemia cells[J]. Leukemia, 2017, 31(1): 65-74. DOI:10.1038/leu.2016.154 |
| [43] |
WARFVINGE R, GEIRONSON L, SOMMARIN M N E, et al. Single-cell molecular analysis defines therapy response and immunophenotype of stem cell subpopulations in CML[J]. Blood, 2017, 129(17): 2384-2394. DOI:10.1182/blood-2016-07-728873 |
| [44] |
GUGLIELMELLI P, PACILLI A, ROTUNNO G, et al. Presentation and outcome of patients with 2016 WHO diagnosis of prefibrotic and overt primary myelofibrosis[J]. Blood, 2017, 129(24): 3227-3236. DOI:10.1182/blood-2017-01-761999 |
| [45] |
HASHIMSHONY T, SENDEROVICH N, AVITAL G, et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq[J]. Genome Biol, 2016, 17: 77. DOI:10.1186/s13059-016-0938-8 |
| [46] |
SAUNDERS A, MACOSKO E Z, WYSOKER A, et al. Molecular diversity and specializations among the cells of the adult mouse brain[J]. Cell, 2018, 174(4): 1015-1030.e6. DOI:10.1016/j.cell.2018.07.028 |
| [47] |
DEY S S, KESTER L, SPANJAARD B, et al. Integrated genome and transcriptome sequencing of the same cell[J]. Nat Biotechnol, 2015, 33(3): 285-289. DOI:10.1038/nbt.3129 |
| [48] |
ANGERMUELLER C, CLARK S J, LEE H J, et al. Parallel single-cell sequencing links transcriptional and epigenetic heterogeneity[J]. Nat Methods, 2016, 13(3): 229-232. DOI:10.1038/nmeth.3728 |
| [49] |
MANIATIS S, ÄIJÖ T, VICKOVIC S, et al. Spatiotemporal dynamics of molecular pathology in amyotrophic lateral sclerosis[J]. Science, 2019, 364(6435): 89-93. DOI:10.1126/science.aav9776 |
| [50] |
ZHANG P, YANG M, ZHANG Y, et al. Dissecting the single-cell transcriptome network underlying gastric premalignant lesions and early gastric cancer[J]. Cell Rep, 2019, 27(6): 1934-1947.e5. DOI:10.1016/j.celrep.2019.04.052 |
| [51] |
ZHOU J, XU J, ZHANG L, et al. Combined single-cell profiling of lncRNAs and functional screening reveals that H19 is pivotal for embryonic hematopoietic stem cell development[J]. Cell Stem Cell, 2019, 24(2): 285-298.e5. DOI:10.1016/j.stem.2018.11.023 |
| [52] |
DAI Y J, WANG Y Y, HUANG J Y, et al. Conditional knockin of Dnmt3a R878H initiates acute myeloid leukemia with mTOR pathway involvement[J]. Proc Natl Acad Sci USA, 2017, 114(20): 5237-5242. DOI:10.1073/pnas.1703476114 |




