前列腺癌在欧美国家中是老年男性发病率最高的肿瘤,也是导致癌症相关死亡的第二大原因[1]。近年来中国老年男性的前列腺癌发病率也在不断攀升,2015年前列腺癌已成为中国男性中发病率第6位的癌症[2-3]。随着相关研究进展以及基因检测技术的不断发展,大量研究表明,基因突变是肿瘤发生和进展过程中的关键分子事件[4-5];有研究表明很多基因突变与前列腺癌的发生、发展密切相关,包括TP53、PTEN、RB1、TMPRSS2-ERG融合等[6-8]。其中融合基因(包括TMPRSS2与ERG或ETV1)在前列腺癌中非常常见,在57%~80%的前列腺癌组织中检测出TMPRSS2基因与ERG基因或ETV1基因在非翻译区发生基因融合[9]。最新研究发现,约有19%的局限性前列腺癌患者携带同源重组相关基因的突变,这一比例在转移性去势抵抗性前列腺癌(metastatic castration-resistant prostate cancer, mCRPC)患者中高达23%左右[10-11]。同源重组修复基因的突变与前列腺癌的发生风险、恶性程度及预后紧密相关,并且对患者的治疗选择有重要的指导作用[12]。既往研究发现携带BRCA胚系突变患者发生前列腺癌时年龄更轻,前列腺癌Gleason评分更高,更容易发生淋巴结侵犯及远处转移,且该类患者的预后很差[13]。而最新研究发现,该类患者对于PARP抑制剂和铂类为基础的化疗敏感[14-15]。因此,美国国立综合癌症网络(The National Comprehensive Cancer Network, NCCN)前列腺癌指南建议对有阳性或可疑家族史、危险分级在高危或极高危组或确诊转移性前列腺癌以及病理类型为导管内癌的前列腺癌患者进行基因检测[16]。但是在国内,由于基因检测技术开展时间短、患者经济条件差等原因,前列腺癌基因检测未得到广泛普及,因而缺乏国人特有前列腺癌突变频谱数据。
肿瘤生长过程中伴随着凋亡和坏死等,会释放DNA片段进入血液循环系统中,也就是循环肿瘤DNA(ciculating tumor DNA, ctDNA)。ctDNA作为液态活检重要手段之一,目前在肿瘤研究领域及临床诊治过程中具有潜在应用价值。而前列腺癌在经过内分泌治疗后,原发灶多发生萎缩,疾病进展时多以骨转移灶进展为主,而骨穿刺阳性率及患者接受度均较低[17-18],在疾病进展至去势抵抗性前列腺癌(castration-resistant prostate cancer, CRPC)时很难再次获取组织行基因检测。而进行ctDNA检测仅需患者血液标本,相对创伤小,患者接受度更高,因此在前列腺癌疾病进展时具有较好的应用前景。本研究对本院46例前列腺癌患者的组织标本和/或血液ctDNA进行靶向二代测序,对与肿瘤发生、发展相关的1 021个基因的全外显子区和部分内含子区域进行突变检测,分析前列腺癌治疗前后基因突变频谱及特定突变与患者临床表型相关性。
1 资料与方法 1.1 患者选择收集2015年9月至2019年12月就诊于本院的前列腺癌患者共46例。纳入标准:①前列腺穿刺或手术标本病理确诊为前列腺癌;②具有明确前列腺癌家族史的患者,或其他肿瘤(如乳腺癌、卵巢癌、结肠癌等)家族史的患者;③前列腺组织病理类型为前列腺导管内癌(intraductal carcinoma of the prostate,IDC-P)或前列腺导管腺癌(ductal adenocarcinoma of the prostate,DAP)的患者;④确诊时发现局部进展(N1)及转移性(M1)前列腺癌的患者。排除标准:①无法获得组织及血液标本;②患者未签署知情同意参加本研究;③随访信息缺失过多;④送检样本未能达到质控要求。本研究得到陆军军医大学大坪医院伦理委员会审批(医研伦审2018第28号)。
采集全部入组患者的确诊年龄、确诊时前列腺特异性抗原(prostate-specific antigen, PSA);穿刺Gleason评分,穿刺及手术标本病理类型;转移情况;肿瘤临床分期、内分泌治疗情况及相应基因检测结果;开始内分泌治疗后睾酮及PSA变化水平。随访中确诊去势抵抗性前列腺癌诊断标准按照美国AUA指南(2018版),即:①血清睾酮 < 50 ng/dL或 < 1.7 nmol/L;②PSA升高:间隔至少3周,PSA较最低值升高>2 ng/mL,或第2次测定值较最低值升高>25%,或骨扫描发现2个及以上的新病灶或用RECIST评估软组织病灶为疾病进展。
1.2 标本获取共送检样本63份,其中前列腺组织样本31例,外周血样本32例。组织样本通过前列腺穿刺或前列腺癌根治手术获得,进行石蜡包埋后切片送检;血液样本采集自患者肘部静脉并存放于EDTA抗凝管低温保存送检。全部送检样本均达到基因测序质控要求。
1.3 基因测序基因二代测序由北京吉因加医学检验实验室有限公司完成。具体步骤如下:新鲜组织样本及石蜡包埋组织使用QIAamp DNA微型试剂盒(Qiagen)、ReliaPrep-FFPE-gDNA微型制备系统(Promega)和配套试剂Maxwell 16 FFPE Plus Lev DNA Purification Kit(货号:AS1135)进行提取,并严格按照说明书步骤进行。再使用Covaris S2超声仪(Covaris)破碎DNA产生300 bp峰值的片段,使用NEB Next Ultra DNA(Illumina)进行文库构建,生成测序文库。然后,使用定制的探针组(integrated DNA technologies,IDT)富集目标区域的文库DNA,在Illumina Hiseq 3000测序仪(Illumina,San Diego,CA,USA)上对富集的文库进行测序。
外周血采用EDTA真空管(BD Diagnostics, Franklin Lakes, NJ)收集,2 500×g离心10 min,取上清液转移至微离心管,上清液16 000×g离心10 min,去除残留细胞碎片。采用QIAamp DNA微型试剂盒从0.6~1.8 mL血浆中分离游离DNA (circulating free DNA,cfDNA)检测ctDNA。使用量子位荧光计(Invitrogen,Carlsbad,CA,USA)和双链DNA荧光定量检测试剂盒(Invitrogen)测定DNA浓度。ctDNA的大小分布采用安捷伦2100生物分析仪和DNA HS试剂盒(Agilent Technologies,Santa Clara,CA,USA)进行评估。DNA提取严格按照说明书步骤进行。
使用KAPA DNA文库制备试剂盒(KAPA Biosystems,Wilmington,MA,USA)构建DNA测序文库。捕获探针涵盖范围来自实体肿瘤中常见的1 021个基因(包含下文中除GEN1以外的17个同源重组相关基因)的编码序列或外显子,采用HiSeq3000测序系统进行DNA测序。质控标准:组织样本经HE染色后镜下评估的肿瘤细胞含量≥10%,平均测序深度≥500X;血液样本提取的DNA总量≥15 ng,平均测序深度≥4000X。
1.4 同源重组基因的筛选全部同源重组基因列表参考文献[19],具体包括ATM、ATR、BRCA1、BRCA2、BRIP1、CHEK2、FAM175A、FANCA、GEN1、MLH1、MRE11、MSH2、MSH6、NBN、PALB2、PMS2、RAD51C、RAD51D共18个相关基因。
1.5 突变基因分析胚系突变、组织突变和ctDNA突变的区分方法:组织DNA和外周血中游离DNA作为实验组(case),外周血白细胞DNA作为对照组(normal),配对的肿瘤样本和正常样本进行NGS检测,即matched tumor-normal NGS。NGS测序之后将所测得序列跟人类基因组序列(Human Genome Build 37)比较,如果在实验组和对照组中都发生了突变,为胚系变异;如果实验组有突变,对照组无突变,为体细胞变异。
胚系变异的分析遵循美国医学遗传学和基因组学学会(ACMG)指南,只报告致病突变、疑似致病突变及意义未明变异,未报告可能不会增加患癌风险的疑似良性变异和良性变异。
1.6 统计学分析采用SPSS 22.0统计学软件进行分析,以家族史、确诊时是否有转移灶、Gleason评分、PSA水平是否高于20 μg/mL为分类变量,对连续变量采用t检验,对分类变量采用卡方检验或Fisher确切检验,采用双侧检验,P < 0.05为差异具有统计学意义。
2 结果 2.1 患者临床信息46例前列腺癌患者经前列腺穿刺活检确诊为前列腺腺癌,其中1例术后病理诊断前列腺癌合并导管内癌。5例患者的一级亲属有肿瘤病史,2例患者非一级亲属有肿瘤病史,其余39例患者无相关肿瘤家族史。患者确诊平均年龄67岁(中位数65岁),确诊时中位PSA值为103 ng/mL(最小值0.48 ng/mL,最大值2 092 ng/mL)。确诊时肿瘤分期T2期患者16例,T3期患者17例,T4期患者12例(1例无随访信息)。其中出现骨转移患者21例,淋巴结转移18例,肺转移3例,同时伴有骨和肺部转移患者1例。行前列腺电切术(transurethral resection of the prostate, TURP)发现偶发癌患者8例,接受前列腺癌根治术患者10例,接受局部放疗患者6例;45例患者接受雄激素剥夺治疗(androgen deprivation therapy, ADT),根治术后接受内分泌治疗结合局部放疗患者3例,截至随访时间2020年8月1日,已进展为CRPC患者20例,因前列腺癌去世5例(表 1)。
| 分类 | HR基因突变携带者 | HR基因突变非携带者 | 合计 |
| 病例数 | 14 | 32 | 46 |
| 诊断中位年龄/岁 | 63(47~76) | 71(57~84) | 65 |
| 合并其他肿瘤 | 1 | 2 | 3 |
| 肿瘤家族史 | |||
| 直系亲属 | 4 | 1 | 5 |
| 非直系亲属 | 1 | 1 | 2 |
| 确诊时Gleason评分 | |||
| ≤7 | 0 | 6 | 6 |
| ≥8 | 13 | 26 | 39 |
| 缺失 | 1 | 0 | 1 |
| 其他病理类型 | |||
| 导管内癌 | 1 | 0 | 1 |
| 神经内分泌型癌 | 1 | 0 | 1 |
| 诊断时肿瘤分期 | |||
| T1/T2 | 4 | 12 | 16 |
| T3/T4 | 9 | 20 | 29 |
| 缺失 | 1 | 0 | 1 |
| 淋巴结转移 | 5 | 13 | 18 |
| 骨转移灶数量 | |||
| ≤2 | 4 | 9 | 13 |
| ≥3 | 4 | 4 | 8 |
| 肺转移 | 2 | 1 | 3 |
| 肺转移合并骨转移 | 1 | 0 | 1 |
| 诊断时远处转移(骨转移、肺转移等) | 11 | 14 | 25 |
| 诊断时中位PSA值/ng·mL -1 | 143 | 235 | |
| 服用阿比特龙 | 9 | 8 | 17 |
2.2 基因突变分析
在46例患者的样本检测中共检测出胚系突变5例,均为杂合突变,MEN1和SDHA同时突变1例,4例为同源重组基因胚系突变,分别为BRCA2突变2例,BRCA1突变1例,BRCA2和PALB2同时突变1例。对胚系突变的致病性分析参照ACMG指南(2015版),HR功能相关的BRCA2和PALB2基因的胚系突变是致病/疑似致病的,而MEN1、SDHA和BRCA1基因的胚系突变则尚无明确有害证据,为意义未明突变。其余疑似良性变异和良性变异,因现有证据表明这些变异很可能不会增加患癌风险,所以未进行报告。
在全部送检的63份样本中(组织DNA样本31例和血液ctDNA样本32例)共检测出362种基因的703个单核苷酸突变(SNV),涉及30个基因的34个拷贝数变化(CNV),以及4个基因的8种结构域变化(SV),见图 1A。在组织DNA样本和血液ctDNA样本中最常见检出的突变类型为错义突变,其次为框移突变和无义突变,错义突变在组织DNA和血液ctDNA中的检出比率分别占64.1%(182/284)和74.5%(301/404),见图 1B、C。涉及基因结构改变的插入、缺失和框移突变分别占组织DNA和血液ctDNA全部检出突变的28.5%(81/284)和18.6%(75/404)。其余包括启动子突变及剪切突变等分别占组织DNA和血液ctDNA全部检出突变的7.4%(21/284)和6.9%(28/404)。
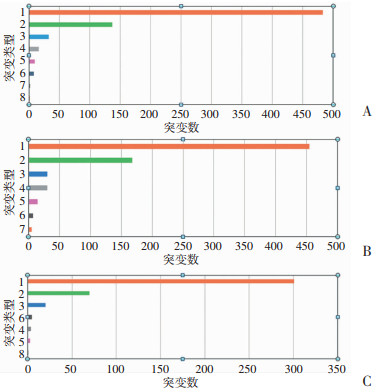
|
| 1:错义突变;2:框移突变;3:无义突变;4:编码区碱基缺失;5:启动子区突变;6:剪接突变;7:编码区碱基插入;8:编码区碱基插入/缺失A:全部63例样本检测出的突变分类;B:31例组织标本中检出的突变分类;C:32例血液ctDNA中检测出的突变分类 图 1 不同样本检出的突变类型 |
在31例患者组织DNA中检出突变率超过10%的基因为TP53(29%,9/31)、CDK12(22.6%,7/31)、FOXA1(22.6%,7/31)、JAK1(16.1%,5/31)、MLL(12.9%,4/31)、TERT(12.9%,4/31),见图 2。在32例患者血液ctDNA中检出突变率超过10%的基因分别为AR(34.3%, 11/32)、APC(25%,8/32)、CDK12(18.8%,6/32)、DNMT3a(18.8%,6/32)、TP53(18.8%,6/32)和MLL3(15.6%,5/32),见图 3。
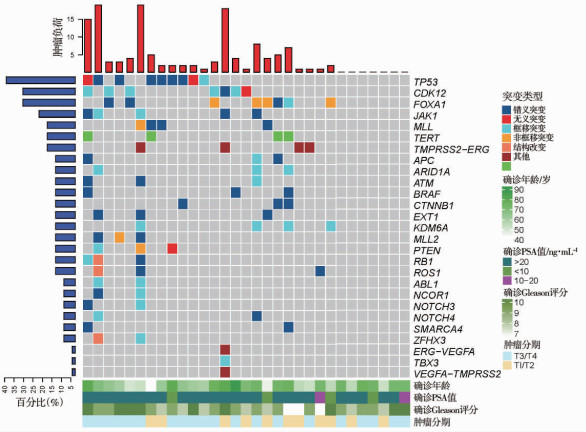
|
| 图 2 送检患者前列腺组织中检出前27位突变基因 |
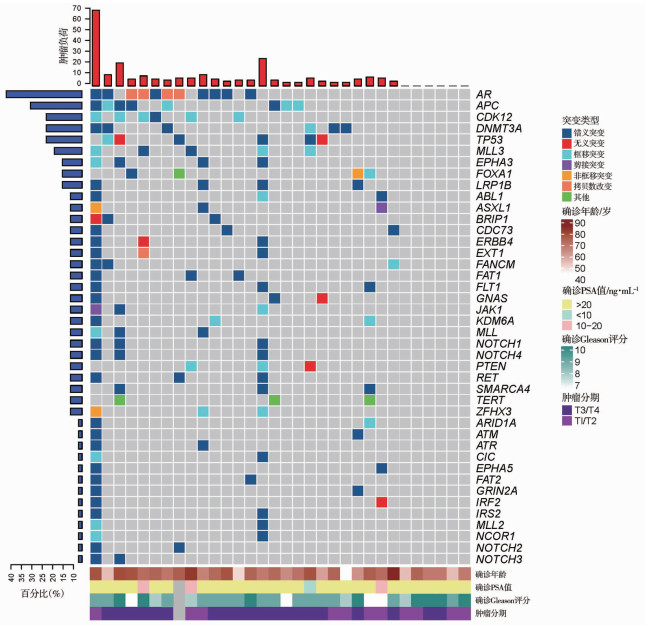
|
| 图 3 送检患者血液ctDNA中检出前42位突变基因 |
46例患者中有17例同时进行了组织标本DNA检测和血液ctDNA检测,两类样本中检出相同基因突变占总检出突变的27.9%(84/301)。其中,有9例患者是在同一时间点收取的前列腺组织标本与血液ctDNA标本;而其余8例患者的ctDNA样本是在获取前列腺组织标本诊断前列腺癌后,经过至少3个月治疗抽血获取。同一时间点收取的9例患者组织DNA和血液ctDNA检测体细胞突变结果发现,有7例患者在组织DNA和血液ctDNA中检测出相同基因突变,各例患者相同基因突变占检测出总突变率平均值为28%(14%~60%)。8例非同时送检的患者中1例患者未接受ADT治疗,7例患者均接受ADT治疗(1例治疗3个月,6例治疗1年以上)。7例ADT治疗后送检的ctDNA样本与治疗前组织DNA样本检测突变的一致率较低,总体一致率为5.6%(共检出突变基因54个,其中共同突变基因3个,各患者突变检出一致率为0%~33%),提示治疗后患者的基因突变存在明显改变。其中AR(5/54)是治疗后出现的最主要的突变,提示AR突变可能与ADT治疗抵抗(即CRPC发生)有关。
46例患者中共检测出6例存在融合基因相关的突变,4例单独在组织中检测出,1例单独在血液ctDNA样本中检测出,还有1例同时在组织DNA和血液ctDNA中检测出。这6例患者平均年龄62.2岁,确诊PSA中位数103 μg/mL,其中1例患者Gleason评分4+3分,1例Gleason评分4+4分,1例Gleason评分4+5分,2例Gleason评分5+4分。有1例分别在血液和组织中检出3种融合基因突变,分别为TMPRSS2-ERG、VEGFA-TMPRSS2、ERG-VEGFA,其确诊时PSA为127.01 μg/mL,Gleason评分5+5分,临床分期T2cN0M1b(3处骨转移灶)。
1例患者前列腺组织病理诊断为导管型和腺癌混合型前列腺癌,在前列腺肿瘤组织中检测出ATR、FBXW7、MLL2、MLL3、NTRK3、PIK3C2B、PTEN、TIPARP的突变,此患者在行前列腺电切术时诊断,并行前列腺癌根治术,1年后于膀胱颈部发生前列腺癌局部复发,之后发现全身多处骨转移。
2.3 同源重组基因突变与前列腺癌临床特点在46例患者中有14例检测出含有HR相关基因的突变,除4例胚系突变外,其余10例存在HR相关基因的体细胞突变,包括ATR、BRIP1、ATM、CHEK2、MLH1、MSH2、RAD51C、MRE11A、MSH6。通过将携带HR基因突变(无论胚系或体细胞突变)的患者与无突变的患者比较,结果表明携带HR基因突变的14例患者在诊断时的Gleason评分整体水平更高,并且确诊时往往有更多的转移灶。HR突变携带者Gleason评分≥8分的比例占100%(13/13例,1例患者无穿刺信息),高于非携带者的59.4%(19/32例),差异有统计学意义(P < 0.01);HR基因突变携带者确诊时发现转移灶的患者比例为78.5%(11/14例),高于非携带者的43.8%(14/32例),差异有统计学意义(P=0.018),且HR基因突变携带者中骨转移灶数量大于3处的患者所占比例也更高;在确诊时的肿瘤分期对比中,携带HR基因突变(无论胚系或体细胞突变)患者在确诊时的肿瘤分期为T3/T4期所占比例为69.2%(9/13例,1例患者无分期信息),高于非携带者的59.3%(19/32例),差异有统计学意义(P=0.031)。
此外,相比非胚系HR突变的患者,带有胚系HR突变的患者确诊年龄往往更低。4名胚系HR基因突变患者平均确诊年龄为51.5岁,显著小于无胚系HR突变患者的70.1岁(P < 0.01)。这提示带有胚系同源重组基因突变的前列腺癌患者往往具有更早的发病年龄。4名胚系同源重组基因突变的患者确诊Gleason评分均≥8分(100%),高于非胚系HR突变患者Gleason评分均≥8分的比例(85.3%,35/41例,1例患者无穿刺信息),差异有统计学意义(P < 0.01)。胚系突变和体细胞突变组在确诊时PSA≥20 μg/mL的比例分别为50%(2/4例)和77.7%(7/9例,1例无确诊时PSA值),差异无统计学意义。这可能与少数特殊病例(2例胚系突变患者确诊时PSA值<10 μg/mL)确诊时极低的检测值和总体样本数量较少有一定关系。
3 讨论基因突变是肿瘤发生和进展过程中的关键分子事件,在嗜铬细胞瘤、乳腺癌、卵巢癌和胰腺癌的相关研究中已经有大量报道[20-23],也有研究表明很多基因的突变与前列腺癌发生发展密切相关,例如ATM、ATR、BRCA1、BRCA2、BRIP1、CDK12、CHEK2等[24-25]。通过基因检测发现,PTEN和P53在原发前列腺癌患者中突变频率达17%和8%[26],在转移性激素抵抗性前列腺癌中有报道高达50%和40%以上[26-27];通过基因编辑小鼠,在前列腺上皮细胞中特异性敲除PTEN和P53可导致前列腺癌的发生[28]。此外,2015年Cell杂志发表的文章对150例转移性去势抵抗性前列腺癌(mCRPC)的肿瘤组织进行测序发现,mCRPC患者主要基因突变富集于AR相关通路、PI3K通路、RAF通路、WNT通路以及DNA修复通路[26]。随后,PRITCHARD等[29]对692例转移性前列腺癌进行DNA修复通路基因胚系检测,发现11.8%的患者存在DNA修复通路基因突变,BRCA2突变占5.3%,ATM突变占1.6%,CHEK2突变占1.9%,BRCA1突变占0.9%。叶定伟教授团队研究结果显示,9.8%的患者存在DNA修复通路胚系突变,其中BRCA2突变率最高(6.3%),其次为BRCA1(0.63%)和ATM(0.63%)[19, 30]。可见欧美人种与东亚人种之间前列腺癌的基因突变频谱确有一定差异。但国内尚无前列腺癌体细胞突变频谱相关报道,本研究结果显示,前列腺组织标本中最常被检出的突变基因为TP53,其次为CDK12、FOXA1与JAK1;血ctDNA标本中检测出的常见突变基因为AR、APC、CDK12、DNMT3a与TP53,融合基因占总体患者的13%(6/46)。其中携带HR缺陷相关基因体系突变患者比例为22.7%,胚系HR相关突变比例为8.8%。与文献[26, 29]结果基本一致。
DNA损伤修复过程复杂,涉及多个修复机制维持DNA稳定,包括DNA单链断裂的碱基切除修复、错配修复、同源重组修复及非同源末端连接等。其中,同源重组修复是DNA双链断裂修复最高效/保真的关键途径。BRCA1/2是同源重组修复的关键基因,BRCA1/2的突变与多种肿瘤的发生密切相关,特别是女性乳腺癌、卵巢癌及男性前列腺癌。在前列腺癌中,BRCA1和BRCA2胚系突变频率分别为0.9%和2.2%,携带BRCA1突变的个体发生前列腺癌的概率是非携带者的3.8倍;携带BRCA2突变的个体发生前列腺癌的概率是非携带者的5~7倍[31]。NYBERG等[32]研究表明,携带有BRCA2胚系突变的患者在75岁之前,前列腺癌的标准化发病率(SIR)为4.45(95%CI 2.99~6.61);而携带BRCA1胚系突变的患者前列腺癌总体SIR为2.35(95%CI 1.43~3.88)。且携带BRCA1/2的前列腺癌患者有更高的风险死于肿瘤本身。此外,携带BRCA2胚系突变患者发病年龄更早,小于65岁的前列腺癌患者中有1.2%携带有BRCA2胚系突变[33]。这与本研究发现的胚系突变携带者发病年龄较低的结果一致。提示携带BRCA1/2突变的患者其前列腺癌恶性程度更高、有更高的Gleason评分、更有可能发生淋巴结侵犯、远处转移。本研究发现携带HR基因突变的患者,无论胚系或体细胞突变,其Gleason评分及临床分期均更高。
既往研究发现胚系HR突变携带者更易因疾病进展而死亡[34-35],但本研究提示精准的治疗选择可明显改善患者生存。本研究4例HR胚系突变携带者在随访截止日期有1位进展为CRPC,从确诊到进展为CRPC的时间为12个月;其余3名携带者在接受ADT+放疗或化疗的联合疗法目前未进展为CRPC。2016年发表在European Urology杂志的文章表明:BRCA2双等位基因突变的mCRPC对铂类敏感[36]。本组1例BRCA2和PALB2胚系突变肺转移前列腺癌患者在接受ADT+铂类和多西他赛化疗后,目前肺部病灶已基本消失,PSA也在控制中,与文献[37]结论一致。另外,1例BRCA2胚系突变患者在接受ADT+前列腺及盆腔放疗后,疾病得到很好地控制,目前随访9年,现已停止内分泌治疗,TPSA一直维持在0.00左右,睾酮水平已恢复正常,该患者在随访过程中同时罹患食管鳞癌[38]。此外,美国FDA的一项Ⅱ期临床研究TOPARP-A显示49例mCRPC患者在进行DNA修复基因检测时,有16例(33%)患者存在相应突变,其中14例对奥拉帕尼治疗有效[39]。随后,TOPARP-B的研究结果进一步显示DNA修复缺陷患者对奥拉帕尼复合反应率为46.7%,其中BRCA1/2的复合反应率高达83.3%[40]。综上,携带DNA修复基因突变的患者虽然疾病恶性程度高,但是对相应的治疗,包括PARP抑制剂和铂类化疗具有更高的敏感性。
ctDNA作为近年来迅速发展的一种新检测手段,具有无创、无损,可以多次获取并实时检测疾病进展的优点,能提供比组织样品检测更多的信息。相对常规检测手段,可以在早期无创地检测出癌前病变[41]。在部分肺癌和结直肠癌中ctDNA检测已经应用于抗EGFR治疗以及确定免疫检查点抑制剂疗效的预测生物标志物,例如ctDNA检测出错配修复基因突变,或微卫星不稳定性高以及肿瘤突变负荷高的患者对免疫检查点抑制剂更敏感[42-43]。在前列腺癌中,ctDNA检测可以发现驱动性的DNA突变并预测患者生存期[44],或指导临床治疗用药(如PARP抑制剂)的选择以及预测耐药[45]。本研究有9例患者的肿瘤组织DNA标本和血液ctDNA标本是同时送检的,两种检测方法突变检出一致率约为27%;提示ctDNA检测可以部分替代传统组织检测。同时,另外7例患者的血液ctDNA标本是在接受ADT治疗后(1例治疗3个月,6例治疗1年以上)送样的,其与之前组织样本比较突变检出一致率只有7.7%,提示疾病在治疗前后其突变频谱并不完全一致,不能用手术获得的组织标本测序结果代替疾病治疗后阶段的分子特点[46-47]。ADT治疗后的突变主要包括AR、CDK12、APC,57%的患者在接受ADT治疗后出现AR基因的变异(4/7例),提示ADT治疗可能导致AR的突变,从而引起对ADT治疗抵抗发生,与2015年发表在Cell杂志上对mCRPC患者测序结果一致[27]。最后,ctDNA检测在前列腺癌疾病进展中的实时应用有重要意义。前列腺癌在经过内分泌治疗后,原发灶多发生萎缩,疾病进展时又多以骨转移为主,而骨穿刺阳性率及患者接受度均较低,因此在疾病进展时,如CRPC时很难再次进行穿刺获取组织行基因检测[17-18]。而外周血ctDNA检测作为一种无创、可重复进行的检测方式,特别适宜监测激素抵抗性前列腺癌的基因突变情况,并以此预测疾病的进展和指导选择治疗方案。
本研究对46例前列腺癌患者进行了靶向二代测序,结果表明HR基因突变患者,无论胚系或者体系,均较非携带者具有更早的发病年龄和更多的远处转移,且此类患者对以破坏肿瘤细胞DNA为靶点的治疗方案(如放疗和化疗)具有更佳的反应性。此外,同一时间点的ctDNA检测与肿瘤组织检测具有一定的一致性;治疗后的ctDNA检测与治疗前的组织DNA检测结果存在明显差异,提示需要在疾病进展阶段对患者进行动态ctDNA检测评估肿瘤分子状态;前列腺癌经过ADT治疗后可导致AR突变增加,进而引起CRPC的发生。基于以上结果,实时动态的基因检测对于前列腺癌的临床诊疗具有重要的指导意义。
利益冲突说明 所有作者均声明不存在利益冲突
| [1] |
FERLAY J, COLOMBET M, SOERJOMATARAM I, et al. Cancer incidence and mortality patterns in Europe: Estimates for 40 countries and 25 major cancers in 2018[J]. Eur J Cancer, 2018, 103: 356-387. DOI:10.1016/j.ejca.2018.07.005 |
| [2] |
TEOH J Y C, HIRAI H W, HO J M W, et al. Global incidence of prostate cancer in developing and developed countries with changing age structures[J]. PLoS ONE, 2019, 14(10): e0221775. DOI:10.1371/journal.pone.0221775 |
| [3] |
PANG C, GUAN Y Y, LI H B, et al. Urologic cancer in China[J]. Jpn J Clin Oncol, 2016, 46(6): 497-501. DOI:10.1093/jjco/hyw034 |
| [4] |
STRATTON M R, CAMPBELL P J, FUTREAL P A. The cancer genome[J]. Nature, 2009, 458(7239): 719-724. DOI:10.1038/nature07943 |
| [5] |
MA X, LIU Y, LIU Y, et al. Pan-cancer genome and transcriptome analyses of 1, 699 paediatric leukaemias and solid tumours[J]. Nature, 2018, 555(7696): 371-376. DOI:10.1038/nature25795 |
| [6] |
MARSHALL C H, FU W, WANG H, et al. Prevalence of DNA repair gene mutations in localized prostate cancer according to clinical and pathologic features: association of Gleason score and tumor stage[J]. Prostate Cancer Prostatic Dis, 2019, 22(1): 59-65. DOI:10.1038/s41391-018-0086-1 |
| [7] |
LIU J N, NEAR A, CHIARAPPA J A, et al. Clinical outcomes associated with pathogenic genomic instability mutations in prostate cancer: a retrospective analysis of US pharmacy and medical claims data[J]. J Med Econ, 2019, 22(10): 1080-1087. DOI:10.1080/13696998.2019.1649267 |
| [8] |
HAMID A A, GRAY K P, SHAW G, et al. Compound genomic alterations of TP53, PTEN, and RB1 tumor suppressors in localized and metastatic prostate cancer[J]. Eur Urol, 2019, 76(1): 89-97. DOI:10.1016/j.eururo.2018.11.045 |
| [9] |
TOMLINS S A, RHODES D R, PERNER S, et al. Recurrent fusion of TMPRSS2 and ETS transcription factor genes in prostate cancer[J]. Science, 2005, 310(5748): 644-648. DOI:10.1126/science.1117679 |
| [10] |
DALL'ERA M A, MCPHERSON J D, GAO A C, et al. Germline and somatic DNA repair gene alterations in prostate cancer[J]. Cancer, 2020, 126(13): 2980-2985. DOI:10.1002/cncr.32908 |
| [11] |
RANTAPERO T, WAHLFORS T, KÄHLER A, et al. Inherited DNA repair gene mutations in men with lethal prostate cancer[J]. Genes (Basel), 2020, 11(3): E314. DOI:10.3390/genes11030314 |
| [12] |
MARSHALL C H, ANTONARAKIS E S. Therapeutic targeting of the DNA damage response in prostate cancer[J]. Curr Opin Oncol, 2020, 32(3): 216-222. DOI:10.1097/cco.0000000000000617 |
| [13] |
CASTRO E, GOH C, OLMOS D, et al. Germline BRCA mutations are associated with higher risk of nodal involvement, distant metastasis, and poor survival outcomes in prostate cancer[J]. J Clin Oncol, 2013, 31(14): 1748-1757. DOI:10.1200/JCO.2012.43.1882 |
| [14] |
RIMAR K J, TRAN P T, MATULEWICZ R S, et al. The emerging role of homologous recombination repair and PARP inhibitors in genitourinary malignancies[J]. Cancer, 2017, 123(11): 1912-1924. DOI:10.1002/cncr.30631 |
| [15] |
ZHAO E Y, SHEN Y Q, PLEASANCE E, et al. Homologous recombination deficiency and platinum-based therapy outcomes in advanced breast cancer[J]. Clin Cancer Res, 2017, 23(24): 7521-7530. DOI:10.1158/1078-0432.CCR-17-1941 |
| [16] |
MOHLER J L, ANTONARAKIS E S, ARMSTRONG A J, et al. Prostate Cancer, Version 2.2019, NCCN Clinical Practice Guidelines in Oncology[J]. J Natl Compr Canc Netw, 2019, 17(5): 479-505. DOI:10.6004/jnccn.2019.0023 |
| [17] |
LORENTE D, OMLIN A, ZAFEIRIOU Z, et al. Castration-resistant prostate cancer tissue acquisition from bone metastases for molecular analyses[J]. Clin Genitourin Cancer, 2016, 14(6): 485-493. DOI:10.1016/j.clgc.2016.04.016 |
| [18] |
SAILER V, SCHIFFMAN M H, KOSSAI M, et al. Bone biopsy protocol for advanced prostate cancer in the era of precision medicine[J]. Cancer, 2018, 124(5): 1008-1015. DOI:10.1002/cncr.31173 |
| [19] |
WEI Y, WU J L, GU W J, et al. Germline DNA repair gene mutation landscape in Chinese prostate cancer patients[J]. Eur Urol, 2019, 76(3): 280-283. DOI:10.1016/j.eururo.2019.06.004 |
| [20] |
LITTON J K, RUGO H S, ETTL J, et al. Talazoparib in patients with advanced breast cancer and a germline BRCA mutation[J]. N Engl J Med, 2018, 379(8): 753-763. DOI:10.1056/nejmoa1802905 |
| [21] |
GOLAN T, HAMMEL P, RENI M, et al. Maintenance olaparib for germline BRCA-mutated metastatic pancreatic cancer[J]. N Engl J Med, 2019, 381(4): 317-327. DOI:10.1056/NEJMoa1903387 |
| [22] |
LEDERMANN J A, DREW Y, KRISTELEIT R S. Homologous recombination deficiency and ovarian cancer[J]. Eur J Cancer, 2016, 60: 49-58. DOI:10.1016/j.ejca.2016.03.005 |
| [23] |
WONG-BROWN M W, MELDRUM C J, CARPENTER J E, et al. Prevalence of BRCA1 and BRCA2 germline mutations in patients with triple-negative breast cancer[J]. Breast Cancer Res Treat, 2015, 150(1): 71-80. DOI:10.1007/s10549-015-3293-7 |
| [24] |
ATTARD G, PARKER C, EELES R A, et al. Prostate cancer[J]. Lancet, 2016, 387(10013): 70-82. DOI:10.1016/S0140-6736(14)61947-4 |
| [25] |
CHENG H H, SOKOLOVA A O, SCHAEFFER E M, et al. Germline and somatic mutations in prostate cancer for the clinician[J]. J Natl Compr Cancer Netw, 2019, 17(5): 515-521. DOI:10.6004/jnccn.2019.7307 |
| [26] |
CANCER GENOME ATLAS RESEARCH NETWORK. The molecular taxonomy of primary prostate cancer[J]. Cell, 2015, 163(4): 1011-1025. DOI:10.1016/j.cell.2015.10.025 |
| [27] |
ROBINSON D, VAN ALLEN E M, WU Y M, et al. Integrative clinical genomics of advanced prostate cancer[J]. Cell, 2015, 161(5): 1215-1228. DOI:10.1016/j.cell.2015.05.001 |
| [28] |
KU S Y, ROSARIO S, WANG Y Q, et al. Rb1 and Trp53 cooperate to suppress prostate cancer lineage plasticity, metastasis, and antiandrogen resistance[J]. Science, 2017, 355(6320): 78-83. DOI:10.1126/science.aah4199 |
| [29] |
PRITCHARD C C, MATEO J, WALSH M F, et al. Inherited DNA-repair gene mutations in men with metastatic prostate cancer[J]. N Engl J Med, 2016, 375(5): 443-453. DOI:10.1056/nejmoa1603144 |
| [30] |
WU J L, WEI Y, PAN J, et al. Prevalence of comprehensive DNA damage repair gene germline mutations in Chinese prostate cancer patients[J]. Int J Cancer, 2021, 148(3): 673-681. DOI:10.1002/ijc.33324 |
| [31] |
OH M, ALKHUSHAYM N, FALLATAH S, et al. The association of BRCA1 and BRCA2 mutations with prostate cancer risk, frequency, and mortality: a meta-analysis[J]. Prostate, 2019, 79(8): 880-895. DOI:10.1002/pros.23795 |
| [32] |
NYBERG T, FROST D, BARROWDALE D, et al. Prostate cancer risks for male BRCA1 and BRCA2 mutation carriers: a prospective cohort study[J]. Eur Urol, 2020, 77(1): 24-35. DOI:10.1016/j.eururo.2019.08.025 |
| [33] |
KOTE-JARAI Z, LEONGAMORNLERT D, SAUNDERS E, et al. BRCA2 is a moderate penetrance gene contributing to young-onset prostate cancer: implications for genetic testing in prostate cancer patients[J]. Br J Cancer, 2011, 105(8): 1230-1234. DOI:10.1038/bjc.2011.383 |
| [34] |
YADAV S, HART S N, HU C L, et al. Contribution of inherited DNA-repair gene mutations to hormone-sensitive and castrate-resistant metastatic prostate cancer and implications for clinical outcome[J]. JCO Precis Oncol, 2019, 3: PO.19.00067. DOI:10.1200/PO.19.00067 |
| [35] |
CARLSON A S, ACEVEDO R I, LIM D M, et al. Impact of mutations in homologous recombination repair genes on treatment outcomes for metastatic castration resistant prostate cancer[J]. PLoS ONE, 2020, 15(9): e0239686. DOI:10.1371/journal.pone.0239686 |
| [36] |
CHENG H H, PRITCHARD C C, BOYD T, et al. Biallelic inactivation of BRCA2 in platinum-sensitive metastatic castration-resistant prostate cancer[J]. Eur Urol, 2016, 69(6): 992-995. DOI:10.1016/j.eururo.2015.11.022 |
| [37] |
TANG T, WANG L N, WANG P, et al. Case report: co-existence of BRCA2 and PALB2 germline mutations in familial prostate cancer with solitary lung metastasis[J]. Front Oncol, 2020, 10: 564694. DOI:10.3389/fonc.2020.564694 |
| [38] |
KO J M, NING L, ZHAO X K, et al. BRCA2 loss-of-function germline mutations are associated with esophageal squamous cell carcinoma risk in Chinese[J]. Int J Cancer, 2020, 146(4): 1042-1051. DOI:10.1002/ijc.32619 |
| [39] |
MATEO J, CARREIRA S, SANDHU S, et al. DNA-repair defects and olaparib in metastatic prostate cancer[J]. N Engl J Med, 2015, 373(18): 1697-1708. DOI:10.1056/NEJMoa1506859 |
| [40] |
MATEO J, PORTA N, BIANCHINI D, et al. Olaparib in patients with metastatic castration-resistant prostate cancer with DNA repair gene aberrations (TOPARP-B): a multicentre, open-label, randomised, phase 2 trial[J]. Lancet Oncol, 2020, 21(1): 162-174. DOI:10.1016/S1470-2045(19)30684-9 |
| [41] |
CAMPOS-CARRILLO A, WEITZEL J N, SAHOO P, et al. Circulating tumor DNA as an early cancer detection tool[J]. Pharmacol Ther, 2020, 207: 107458. DOI:10.1016/j.pharmthera.2019.107458 |
| [42] |
OELLERICH M, SCHÜTZ E, BECK J, et al. Using circulating cell-free DNA to monitor personalized cancer therapy[J]. Crit Rev Clin Lab Sci, 2017, 54(3): 205-218. DOI:10.1080/10408363.2017.1299683 |
| [43] |
NAKAMURA Y, YOSHINO T. Clinical utility of analyzing circulating tumor DNA in patients with metastatic colorectal cancer[J]. Oncologist, 2018, 23(11): 1310-1318. DOI:10.1634/theoncologist.2017-0621 |
| [44] |
WYATT A W, ANNALA M, AGGARWAL R, et al. Concordance of circulating tumor DNA and matched metastatic tissue biopsy in prostate cancer[J]. J Natl Cancer Inst, 2017, 109(12): djx118. DOI:10.1093/jnci/djx118 |
| [45] |
GOODALL J, MATEO J, YUAN W, et al. Circulating cell-free DNA to guide prostate cancer treatment with PARP inhibition[J]. Cancer Discov, 2017, 7(9): 1006-1017. DOI:10.1158/2159-8290.CD-17-0261 |
| [46] |
VOLANTE M, TOTA D, GIORCELLI J, et al. Androgen deprivation modulates gene expression profile along prostate cancer progression[J]. Hum Pathol, 2016, 56: 81-88. DOI:10.1016/j.humpath.2016.06.004 |
| [47] |
HORNING A M, WANG Y, LIN C K, et al. Single-cell RNA-seq reveals a subpopulation of prostate cancer cells with enhanced cell-cycle-related transcription and attenuated androgen response[J]. Cancer Res, 2018, 78(4): 853-864. DOI:10.1158/0008-5472.CAN-17-1924 |




