2. 400021 重庆, 重庆市中医院检验科,重庆市中医院感染性疾病中西医结合诊治实验室
2. Department of Clinical Laboratory, Laboratory of Diagnosis and Treatment for Infectious Diseases With Integrated Traditional Chinese and Western Medicine, Chongqing Hospital of Traditional Chinese Medicine, Chongqing, 400021, China
虽然乙型肝炎病毒(hepatitis B virus,HBV)疫苗已成功应用数十年之久,但HBV感染仍然是一个重要的公共卫生问题。据统计,全球约有2.5亿感染者[1-2],每年有近100万人死于肝硬化和肝癌等相关的肝脏疾病[2]。临床的抗HBV药物主要为两大类:聚乙二醇干扰素α(PEG-IFNα)和6种核苷类似物(NAs)。干扰素治疗存在副作用大、患者耐受性差等问题,而核苷类似物因停药后易反弹,需要长期乃至终身用药,而长期用药又会导致病毒耐药[3-5]。显然,乙肝问题的最终解决,有赖于新型抗乙肝药物的开发。
抗乙肝药物的开发,应该针对乙肝病毒生命周期的各个关键环节。这些环节包括病毒入胞、病毒cccDNA的合成、病毒RNA的转录、核衣壳组装、病毒出胞等[6]。在这些环节当中,cccDNA是一个关键分子,其稳定、持续地存在,并行使其转录功能,是乙肝病毒感染得以持续且难以彻底治愈的关键[7-8]。更为重要的是,现有药物并不能直接作用于cccDNA,因此,发展能有效靶向cccDNA的治疗策略是当前亟须解决的问题。
理论上,靶向cccDNA的策略,至少包括两种,一种是降低其数量,可以通过抑制其合成或加速其降解来实现;另一种则是抑制其活性。cccDNA的主要功能是作为所有HBV RNA的转录模板,因此,抑制其活性,主要指抑制其转录活性。目前有少量具有该类效应的化合物报道,例如P300/CBP相关因子组蛋白乙酰转移酶抑制剂Curcumin[9]、Sirt1抑制剂sirtinol和Ex527[10]、JMJD3组蛋白脱甲基酶抑制剂GSK-J1[11]等。这些分子通过控制cccDNA组蛋白的乙酰化和甲基化,抑制cccDNA的转录,而目前这些分子均处于初期研究阶段,最终效果仍待观察。对cccDNA转录事件而言,前基因组RNA(pgRNA)的转录尤其重要,因为pgRNA不但是病毒DNA的复制模板,也是HBV核心蛋白和聚合酶的翻译模板。本研究构建了一种靶向HBV核心启动子转录活性的药物初筛细胞模型,利用该模型筛选了一个包含1 450种小分子的化合物库,并从中鉴定到了一种能有效抑制HBV核心启动子的转录活性,从而能抑制HBV RNA转录的化合物逆转素(Reversine),并对该化合物的生物学效应进行评价,为进一步研发新型抗乙肝药物奠定基础。
1 材料与方法 1.1 材料 1.1.1 质粒模板来源质粒pCMV-Gluc购自美国的New England Biolabs公司,质粒pEGFP-N1购自美国的Clontech公司,质粒pCH9/3091来自德国的弗莱堡大学Michael Nassal实验室[12],质粒LentiCRISPRv2、psPAX2、Pmd2.0G来自美国的麻省理工学院张峰实验室[13]。
1.1.2 质粒的构建 1.1.2.1 野生型及截短突变型核心启动子驱动的荧光素酶报告质粒野生型以质粒pCH9/3091为模板,用引物F 1070GG+R 1901GG扩增包含增强子Ⅰ、增强子Ⅱ和基本核心启动子(BCP)区域,用胶回收试剂盒回收扩增片段并将其命名为frag 1;以pCH9/3091为模板,引物F SV40+R CMV扩增载体片段并将其命名为frag 2;以质粒pCMV-Gluc为模板,用引物F Gluc+ R Gluc扩增Gluc报告基因并将其命名为frag 3,然后采用Golden-gate的方式将frag 1、2、3连接起来即得到质粒pCH9-Pcore-Gluc。
截短突变型:以pCH9-Pcore-Gluc质粒为模板,用引物F 1636GG+R CMV扩增片段frag 4、引物F 1745GG+R 1635GG扩增片段frag 5、引物F 1745GG+ R CMV扩增片段frag 6,将frag 4、5、6采用Golden-gate克隆方式连接到载体上,即得到3种截短突变型质粒:pCH9-1636-1901-Gluc、pCH9-1070-1635/1745-1901-Gluc、pCH9-1744-1901-Gluc。
1.1.2.2 Lenti-Pcore-Gluc-heo报告质粒以质粒pCH9/3091为模板,用引物F 1070GG2+R 1901GG扩增片段frag 7;以质粒pCMV-Gluc为模板,用引物F Gluc1GG+R GlucGG2扩增片段frag 8;以质粒LentiCRISPRV2为模板,分别用引物F LentiV2 flag+ R LentiV2 13773扩增片段frag 9、引物F LentiV2 13769+ R LentiV2 2607扩增片段frag 10,将frag 7、8、9、10以Golden-gate的方式连接起来构建Lenti-Pcore-Gluc质粒。为了将Lenti-Pcore-Gluc上的puromycin抗性替换为neomycin抗性,以Lenti-Pcore-Gluc为模板,引物F LentiV2 13769+R 2AGG扩增frag 11、引物F LentiV2 9947+R LentiV2 13773扩增frag 12;以pEGFP-N1质粒为模板,引物F neoGG+R neoGG扩增frag 13,将frag 11、12、13以Golden-gate方式连接起来即得到质粒Lenti-Pcore-Gluc-neo。
1.1.2.3 其他质粒Pcore-3Flag-G4S92-HBc为课题组前期构建[14],该质粒由核心启动子驱动,在HBc的N端通过G4S linker (92个氨基酸)连接3个Flag标签蛋白。同理,将该质粒上的核心启动子Pcore换成另外两种病毒来源的启动子Psv40和Pcmv即构出质粒Psv40-3Flag-G4S92-HBc和Pcmv-3Flag-G4S92-HBc。质粒HBV 1.3倍体由首都医科大学赵学森博士惠赠。实验中所涉及的引物序列见表 1。
| 引物名称 |
引物序列(5′→3′) |
| F 1070GG | GCTGACCGTCTCCCGTTGATGCCTTTGTATGCATGT |
| R 1901GG | GCTGACCGTCTCTAGACGACATGCCCCAAAGCCACCCAAGGCT |
| F SV40 | ACTCACCGTCTCTTCGTGGCCGCGACTCTAGATCAT |
| R CMV | GCTGACCGTCTCTAACGGCTATGAACTAATGACCCCGTA |
| F Gluc | GCTGACCGTCTCCGTCTGGAGTCAAAGTTCTGTTTG |
| R Gluc | ACTCACCGTCTCTACGAGTCACCACCGGCCCCCTTGAT |
| F 1636GG | GCTGACC CGTT ATTGCCCAAGGTCTTACATAAG |
| F 1745GG | GCTGACCGTCTCC CGTT GGGAGGAGATTAGGTTAAAGGT |
| R 1635GG | GCTGACCGTCTCTAACGTTTGGTGGGCGTTCACGGTGGT |
| F 1070GG2 | GCTGACCGTCTCCTGACCGTTGATGCCTTTGTATGCATGT |
| R 1901GG | GCTGACCGTCTCTAGACGACATGCCCCAAAGCCACCCAAGGCT |
| F Gluc1GG | GCTGACCGTCTCCCATGGGAGTCAAAGTTCTGTTTG |
| R GlucGG2 | ACTCACCGTCTCTAATCGTCACCACCGGCCCCCTTGAT |
| F LentiV2 flag | GCTGACCGTCTCCGATTACAAAGACGATGACGATAAG |
| R LentiV2 13773 | ACTCACCGTCTCTCCCTTAACGTGAGTTTTCGTTCCA |
| F LentiV2 13769 | GCTGACCGTCTCCAGGGATTTTGGTCATGAGATTATC |
| R LentiV2 2607 | ACTCACCGTCTCTGTCAGGTACCTTAATTAACCAAACTGG |
| F LentiV2 9947 | GCTGACCGTCTCCTGAACGCGTTAAGTCGACAATCA |
| R 2AGG | TGCGTCCGTCTCTTCCAGGATTCTCTTCGACATCT |
| F neoGG | GCTGACCGTCTCCTGGACCGATTGAACAAGATGGATTG |
| R neoGG | ACTCACCGTCTCTTTCACTCAGAAGAACTCGTCAAGAAG |
| F Actin | GTGATGGTTGGTATGGGCCA |
| R Actin | AAATCCAGTGCGACGTAGCA |
| F pgRNA | TGTTCAAGCCTCCAAGCTGT |
| R pgRNA | GACCTGCCTCGTCGTCTAAC |
| F HBVDNA | TCCTAGGACCCCTTCTCGTG |
| R HBVDNA | CTTGGCCCCCAATACCACAT |
| F NR1C2 | GGCTTCCACTACGGTGTTCA |
| R NR1C2 | TGAAGCTGGGGATGCTCTTG |
1.1.3 细胞来源
293FT及人肝癌细胞系HepG2购自美国ATCC,HepG2.2.15由重庆医科大学感染性疾病分子生物学教育部重点实验室长期保存。
1.1.4 主要仪器与试剂利用该模型进行筛选的化合物库购自美国Selleckchem公司,为一款含有1 450种小分子的生物活性化合物的定制库[名称为Cherry Pick Library(96-well)-L2000-Z286554-30uL],每种化合物的浓度为10 mmol/L,均溶解在DMSO中。主要试剂和仪器包括:X-tremeGENE HP转染试剂(Roche Diagnostics公司,美国);慢病毒浓缩试剂PEG-it Virus Precipitation Solution(5×)(SBI公司,美国);Golden-gate连接所用到的BsmBⅠ酶、Tango buffer、DTT(Thermo scientific公司,美国);ATP(New England Biolabs公司,美国);2×PrimeSTAR HS Mix(Takara公司,日本);胶回收试剂盒、基因组DNA提取试剂盒(QIAGEN公司,德国);胎牛血清(Lonsera公司,乌拉圭);胰酶、DMEM培养基、青链霉素(Gibco公司,美国);细胞培养箱HERAcell 150i(Thermo scientific公司,美国);胶原(Corning公司,美国);Lipofectamine3000 Transfection Reagent、Platinum SYBR Green qPCR、G418(Invitrogen公司,美国);TRIzol Reagent、Gaussia Luciferase Flash Assay Kit(Thermo scientific公司,美国);病毒表面抗原和E抗原酶免法检测试剂盒(科华生物工程股份有限公司,上海);逆转录试剂盒(天根生化科技有限公司,北京);Southern blotting试剂、Nylon膜(Roche Diagnostics公司,美国);酚氯仿(Amresco公司,美国);微管酶(BioLabs公司,美国);PVDF膜(Amersham Hybond公司,加拿大);Anti-Flag(1 ∶1 000,鼠抗,Sigma Aldrich公司,美国);GAPDH、β-actin小鼠单抗(1 ∶1 000,碧云天生物技术有限公司,上海);IRDye-cojugated-secondary antibodies(1 ∶10 000,Licor Biosciences公司,美国);CCK-8检测试剂盒(Bimake公司,美国);Synergy H1多孔板检测仪(Bio-Tek公司,美国);SageCreation ChampChemi凝胶成像系统(赛智创业科技有限公司,北京);Odyssey CLx system红外激光双色图像分析系统(Licor Biosciences公司,美国);UVP Hybridizer Oven分子杂交炉(UVP公司,美国)。
1.2 方法 1.2.1 细胞培养与转染实验所用到的293FT、HepG2、HepG2.2.15(HepG2细胞上构建的能够稳定产生HBV病毒颗粒的细胞系)均在37 ℃、5%CO2的环境下,于含10%胎牛血清、1%青霉素、100 μg/mL链霉素的DMEM中培养。HepG2细胞转染前的接种,需用胶原处理培养板。转染操作参照Lipo3000的转染试剂说明书进行。
1.2.2 细胞模型的构建293FT细胞以约50%密度接种到10 cm细胞培养皿,用X-tremeGENE HP转染试剂将Lenti-Pcore-Gluc-neo质粒和慢病毒包装质粒psPAX2、Pmd2.0G共转染293FT细胞,然后收取细胞培养上清,再用慢病毒浓缩试剂浓缩上清,具体操作按照产品说明书进行。将HepG2细胞以约50%密度接种到6 cm细胞培养皿,待细胞密度达到约90%时,更换为新鲜无双抗的培养基,然后用浓缩的慢病毒感染HepG2细胞,48 h后,将其消化传代至10 cm培养皿,同时用含500 μg/mL G418的培养基做筛选培养,2~3周后,取上清检测Gaussia荧光素酶的活性(以下简称Gluc活性)。
1.2.3 药物筛选利用HepG2-Pcore-Gluc细胞模型对1 450种小分子化合物进行筛选。首先,将稳定系细胞以5×104/孔种于胶原处理后的96孔板,待细胞完全贴壁后,加入不同的化合物处理(终浓度为10 μmol/L),阴性对照加入DMSO处理(DMSO终浓度不超过0.5%),48 h后取每孔上清10 μL分别加入黑边透明底的96孔板中,然后各孔再加入Gaussia荧光素酶检测试剂20 μL,用Synergy H1多孔板检测仪检测Gluc活性。每种化合物做3个复孔,通过本轮筛选,保留那些影响荧光素酶活性2倍及以上的化合物进入第2轮的剂量依赖性测试。
在第2轮剂量依赖性测试实验中,首先将第1轮筛选得到的化合物分别以1 μmol/L和10 μmol/L的终浓度处理稳定细胞系,48 h后检测Gluc活性。本轮筛选通过增加化合物的一个低浓度(1 μmol/L),在一定程度上会排除可能由于化合物的高剂量(10 μmol/L) 而导致细胞毒性,从而产生假阳性结果,然后将最终筛选得到的化合物在HepG2-Pcore-Gluc细胞模型上再做进一步的量效关系验证。
1.2.4 Western blot实验HepG2细胞接种于胶原处理过的24孔板,待细胞贴壁后分别转染Pcore-3Flag-G4S92-HBc、Pcmv-3Flag-G4S92-HBc及Psv40-3Flag-G4S92-HBc质粒,然后用不同浓度的药物(终浓度为0、1.0、5.0、10.0、20.0 μmol/L)处理细胞,48 h后用RIPA裂解液于冰上裂解细胞,然后4 ℃离心取蛋白上清,将蛋白加热变性后于10%的SDS-PAGE上进行凝胶电泳,电泳结束后将样品转到PVDF膜上,然后用5%的奶粉封闭膜1 h、一抗孵育1 h、绿色荧光二抗避光孵育1 h,最后用Odyssey CLx system红外激光双色图像分析系统曝光成像。
1.2.5 HBV core DNA的提取与Southern blot检测HepG2.2.15和HepG2细胞以5×105/孔接种于胶原处理过的12孔板,待细胞完全贴壁后加入不同浓度的药物(终浓度为0、0.5、2.5、5.0、10.0、20.0 μmol/L)处理5 d,然后用裂解液(10 mmol/L Tris-HCL、1 mmol/L EDTA、0.2% NP-40)裂解细胞,取上清加入氯化钙和微管酶,37 ℃孵育1 h后,加入EDTA、SDS,链酶蛋白酶,继续孵育1 h,然后用酚氯仿抽提,最后加入氯化钠和无水乙醇沉淀DNA。第2日,将DNA风干后溶于无酶水中,然后于1.2%的琼脂糖凝胶中进行电泳分离,再将其转移至尼龙膜。后续的DNA杂交和检测按照产品的说明书进行操作,最后用SageCreation ChampChemi凝胶成像系统进行成像。
1.2.6 细胞总RNA的提取、逆转录与荧光定量PCRHepG2.2.15和HepG2细胞接种于胶原处理过的12孔板,药物处理5 d后,加入TRIzol裂解细胞,然后加入氯仿离心,异丙醇沉淀,75%的乙醇洗涤干燥,最后加入适量无酶水至其完全溶解。根据RNA的浓度,取1 μg将其逆转录成cDNA,逆转录操作按照试剂盒说明书进行,再以cDNA为模板,利用荧光定量PCR检测cDNA的含量从而间接反映细胞内的RNA水平变化,细胞内core DNA的相对水平变化也利用荧光定量PCR检测评估,最后根据得到的CT值,利用2-△△Ct计算相对含量。荧光定量PCR所用到的引物见表 1。
1.2.7 酶联免疫吸附实验(ELISA)不同浓度的药物处理HepG2.2.15和HepG2细胞,48 h后取适量的细胞培养上清进行乙肝表面抗原和乙肝e抗原的检测,具体操作按照试剂盒说明书进行。
1.2.8 细胞活力检测HepG2细胞按照5 000/孔的密度接种于胶原处理过的96孔板,待细胞完全贴壁后,加入不同浓度的药物处理,48 h后更换成新鲜的培养基,并加入培养基体积10%的CCK-8试剂于37 ℃、5%CO2的环境中避光孵育,然后在孵育1、2、3 h后(4 h内完成检测)于多功能酶标仪上450 nm处检测各孔的光密度值D(450),并将DMSO对照孔的细胞活性设置为100%,分析其余浓度对细胞活性的影响[以细胞毒性浓度CC50(cytotoxicity concentration) 数值表示, CC50:细胞半数毒性浓度,即不同浓度药物处理后的细胞相对活性被抑制50%时所对应的浓度]。
1.3 统计学分析采用SPSS 20.00统计软件,数据以 x±s表示,对照组与用药组间比较采用两独立样本的t检验,实验重复3次。图表均使用GraphPad Prism 8绘制。检验水准: α=0.05。
2 结果 2.1 HepG2-Pcore-Gluc稳定表达细胞系的构建为了构建一种表征HBV核心启动子(Pcore)转录活性的细胞模型,设计了由Pcore驱动表达的Gaussia荧光素酶报告质粒(图 1A),然后以重组慢病毒感染HepG2细胞的方式,经抗性(neomycin)筛选得到稳定表达细胞系。为了验证HepG2-Pcore-Gluc细胞系是否构建成功,提取HepG2和HepG2-Pcore-Gluc细胞的基因组DNA,用引物F 1070GG+R Gluc做PCR扩增,结果显示:从HepG2-Pcore-Gluc细胞来源的基因组DNA,可以扩增出1 500 bp单一条带(图 1B),而阴性对照(HepG2)无此条带。取HepG2-Pcore-Gluc细胞培养上清,检测其中的Gluc活性,可检测到高于阴性对照10 000倍以上的发光信号(图 1C)。以上结果提示HepG2-Pcore-Gluc细胞系构建成功。
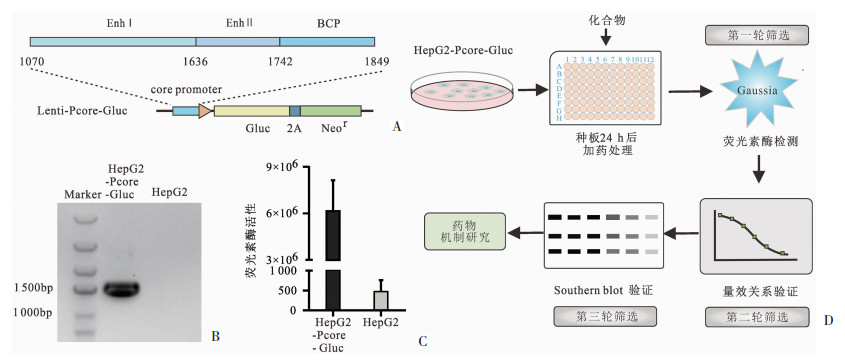
|
| A: Lenti-Pcore-Gluc质粒示意图该质粒是由HBV核心启动子(包含增强子Ⅰ、Ⅱ和基本核心启动子)驱动的Gaussia荧光素酶的表达质粒; B、C: 利用PCR扩增目的序列和检测Gluc的方式确定已成功获得由核心启动子驱动且高表达Gluc的HepG2-Pcore-Gluc稳定细胞系; D: 化合物筛选流程示意图 图 1 细胞模型的构建及化合物库的筛选流程 |
2.2 化合物的第1轮筛选
为了筛选抑制核心启动子活性的药物,采用了3轮筛选流程(图 1D)。第1轮的筛选标准是: 在10 μmol/L浓度下,能让HepG2-Pcore-Gluc细胞培养上清的Gluc活性降低2倍以上,同时显微镜下没有明显可见的细胞毒性。通过对1 450种小分子化合物进行初筛,发现有41种化合物能降低Gluc的活性2倍及以上。随后对41种化合物进行再次筛选以进一步排除假阳性,本次筛选用1 μmol/L和10 μmol/L两种浓度(图 2A~D)。经筛选后只有2种化合物Reversine和SCH58261在1 μmol/L时即表现出较好的抑制效果,在10 μmol/L时能抑制Gluc活性3倍以上,并且镜下观察细胞未发现有毒性改变(图 2A)。进一步通过细胞活力检测实验得出:Reversine在HepG2细胞中的CC50=50.53 μmol/L(图 2E),SCH58261在HepG2细胞中的CC50>100 μmol/L(图 2F),由此可见二者的活性较高且毒性较小,值得做进一步的验证。图G为Reversine和SCH58261的分子结构图。
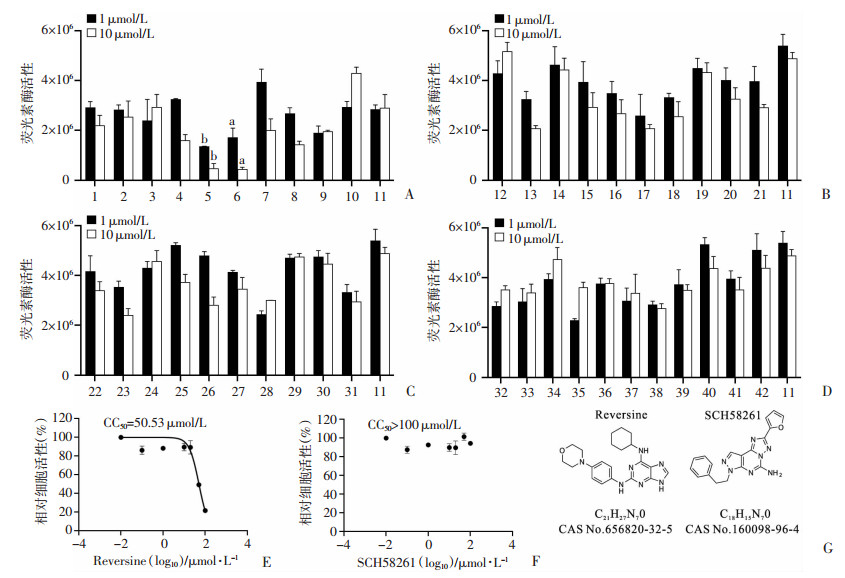
|
|
1:Aminophylline;2:Theophylline-7-acetic acid;3:Proxyphylline;4:CGS21680 HCL;5:Reversine;6:SCH58261;7:ZM241385;8:A2ARantagonist;9:Adenosine Dialdehyde;10:Adenosine;11:DMSO;12:ABT-702;13:Tubercidine;14:Turofexorate Isopropyl;15:WY-14643 (Pirinixic Acid);16:GW4064;17:SR-18292;18:GW9662;19:Tozasertib;20:Barasertib;21:MLN8054;22:ZM 447439;23:Danusertib;24:SNS-032;25:SNS-314;26:JNJ-26854165;27:ENMD-2076;28:CHIR-99021;29:Aurora A Inhibitor Ⅰ;30:PHA-680632;31:Hesperadin;32:AEE788;33:KW-2449;34:TAK-901;35:AMG-900;36:GSK1070916;37:MK-5108;38:MK-8745;39:BI-847325;40:SNS-314 Mesylate;41:CCT129202;42:CCT137690;a: P < 0.05,b: P < 0.01,与对照DMSO孔比较 A~D: 初筛得到的41种化合物在HepG2-Pcore-Gluc稳定细胞系上以低剂量(1 μmol/L)和高剂量(10 μmol/L)的终浓度进行进一步的活性测试,A图的11种化合物中化合物5(Reversine)和化合物6(SCH58261)与对照(DMSO)相比,在1 μmol/L的终浓度下能有效抑制Gluc活性, 且对细胞没有毒性; E、F: 分别为化合物Reversine、SCH58261处理48 h后HepG2细胞的活力; G: Reversine、SCH58261的分子结构图 图 2 采用荧光素酶的检测方法检测初筛得到的41种化合物的Gluc活性 |
2.3 化合物的量效关系验证
为了验证筛选出的化合物能否剂量依赖性地抑制Gluc的表达,采用两种不同的验证方式,第一种方式在HepG2-Pcore-Gluc细胞中用不同浓度药物处理细胞48 h,然后检测Gluc活性。结果发现:Reversine和SCH58261均能剂量依赖性地降低Gluc活性(图 3A、B)。第二种方式通过构建由核心启动子驱动的HBc表达质粒Pcore-3Flag-G4S92-HBc(图 3C),经转染HepG2细胞并加药处理48 h后做Western blot实验。两种验证方式的结果显示:这两种药物均能剂量依赖性地抑制核心启动子的活性。但Western blot的结果还提示:两种药物抑制由核心启动子驱动的蛋白表达效应也有可能是通过影响转录或其他环节所致(图 3D、E)。
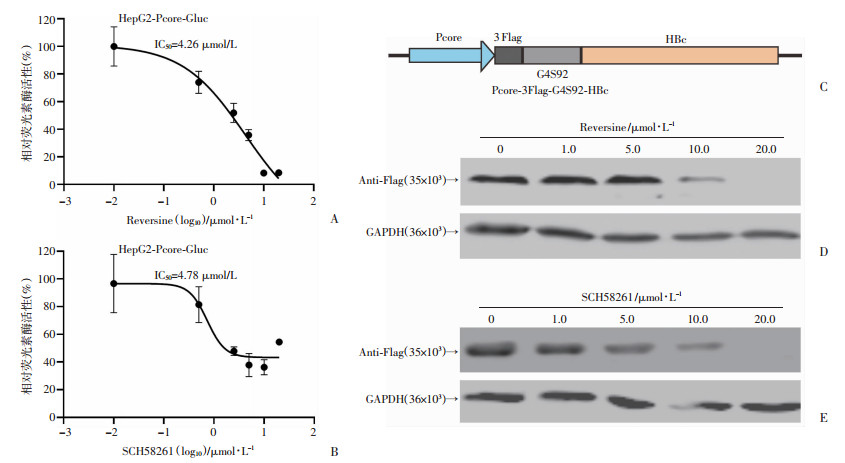
|
| A、B: 在HepG2-Pcore-Gluc细胞中,Reversine和SCH58261均能剂量依赖性地降低Gluc活性IC50为Gluc活性的半数抑制浓度,即不同浓度药物处理后的Gluc相对活性被抑制50%时所对应的浓度; C: 质粒Pcore-3Flag-G4S92-HBc的结构示意图; D、E: Reversine和SCH58261均能有效抑制由核心启动子驱动的核心蛋白的表达 图 3 Reversine和SCH58261对核心启动子活性的影响 |
2.4 化合物对HBV DNA复制水平的影响
为了进一步验证候选药物对HBV DNA复制的影响,用不同浓度的药物处理HepG2.2.15或者瞬时转染HBV 1.3倍体的HepG2细胞(药物的终浓度为0、0.5、2.5、5.0、10.0、20.0 μmol/L),5 d后提取细胞内的core DNA做Southern blot检测。如图 4A所示:在两种细胞中,Reversine均能剂量依赖性地抑制HBV DNA的复制,但SCH58261不具有此效应。在同样的HBV复制细胞系中,两种药物分别处理细胞5 d后(终浓度为0、0.1、1.0、5.0、10.0、20.0 μmol/L),利用荧光定量PCR的方式检测HBV DNA的水平变化,结果同样显示(图 4B):Reversine能够剂量依赖性地降低HBV DNA的复制水平,而SCH58261不能。在HepG2.2.15和瞬时转染HBV1.3倍体的HepG2细胞中,Reversine抑制HBV DNA的IC50分别为5.63 μmol/L和0.24 μmol/L。以上结果提示:Reversine具有良好的HBV DNA抑制活性,但SCH58261没有。
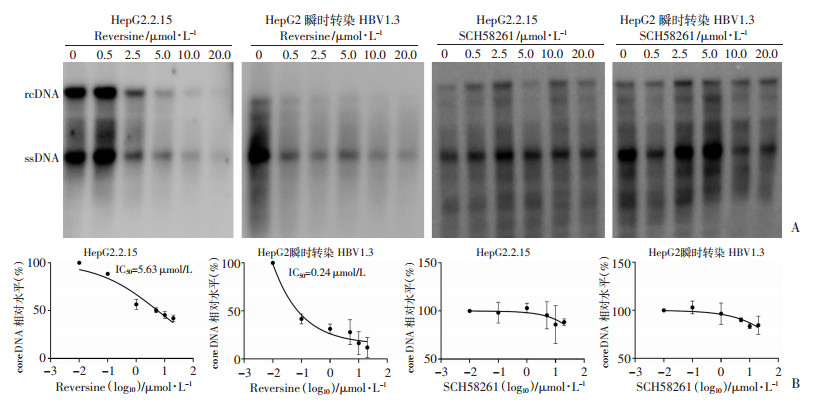
|
| A: 在HepG2.2.15和瞬时转染HBV1.3倍体的HepG2细胞中分别经药物Reversine或SCH58261处理后,Southern blot检测细胞内HBV DNA的复制水平; B: 采用荧光定量PCR进一步检测两种药物对HBV DNA复制水平的影响,其结果同Southern blot结果一致IC50为core DNA的半数抑制浓度,即不同浓度药物处理后的core DNA相对水平被抑制50%时所对应的浓度 图 4 Reversine和SCH58261对细胞内HBV DNA复制的影响 |
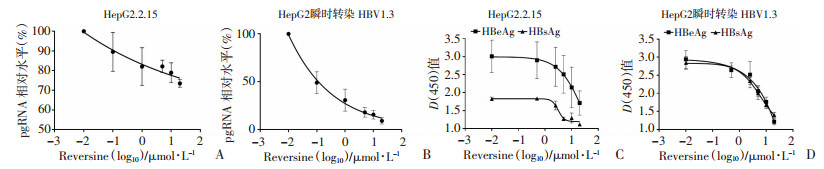
2.5 Reversine对HBV RNA转录、乙肝表面抗原和乙肝e抗原的影响
为了进一步验证Reversine的生物学效应,用不同浓度的药物(0、0.1、1.0、5.0、10.0、20.0 μmol/L)处理细胞后,提取总RNA,经逆转录后采用荧光定量PCR检测HBV前基因组RNA(pgRNA)的转录水平。结果显示:Reversine能剂量依赖性地降低HBV pgRNA的转录水平(图 5A、B)。细胞加药处理48 h后,取适量的上清做乙肝表面抗原和乙肝e抗原的检测,结果显示:Reversine也能显著降低乙肝表面抗原和乙肝e抗原的分泌水平(图 5C、D)。以上结果提示:在HepG2.2.15和瞬时转染HBV 1.3倍体的HepG2细胞中,Reversine能有效抑制HBV pgRNA的转录及乙肝表面抗原和乙肝e抗原的分泌。
|
| A、B: 在HepG2.2.15和瞬时转染HBV1.3倍体的HepG2细胞中,Reversine剂量依赖性地抑制HBV pgRNA的转录; C、D: 在同样的HBV复制细胞系中,Reversine也能有效降低乙肝表面抗原和乙肝e抗原的分泌水平 图 5 Reversine对HBV RNA转录以及乙肝表面抗原和乙肝e抗原的影响 |
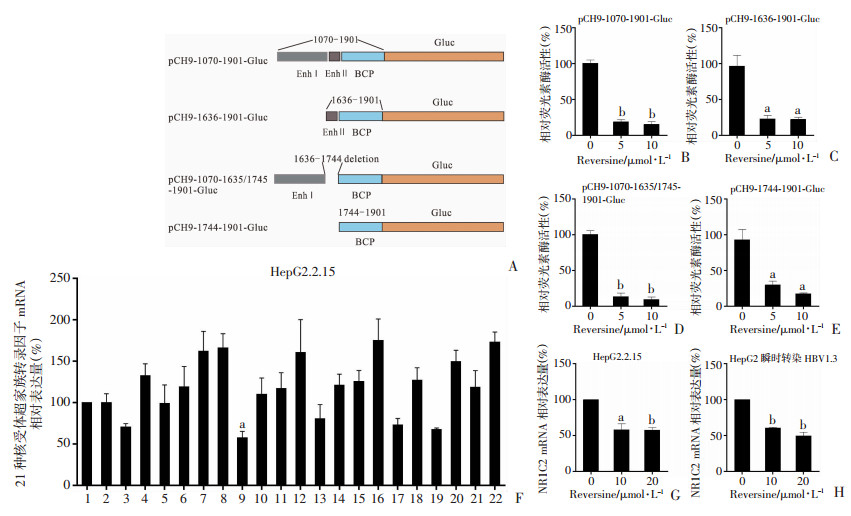
2.6 Reversine在核心启动子上的作用区域及其对核受体超家族基因转录的影响
前述实验提示Reversine能抑制由核心启动子驱动转录的pgRNA水平。为了进一步探讨Reversine的作用机制,首先对Reversine在核心启动子上的作用区域进行了研究。如图 6A所示:将4种不同启动子的荧光素酶报告质粒(分别为完整核心启动子和缺失增强子Ⅰ、或缺失增强子Ⅱ或二者均缺失的启动子)转染HepG2细胞,加药处理48 h后,检测Gluc活性(药物浓度设置为0、5、10 μmol/L)。结果显示:不论是完整的还是截短的核心启动子,Reversine均能显著降低其活性(图 6B~E)。鉴于这些报告质粒的启动子区域中均含有基本核心启动子序列(BCP),由此推测该药物可能作用在共同区域,即BCP,但具体机制有待进一步探索。
|
|
a: P < 0.05,b: P < 0.01,与对照DMSO孔(0 μmol/L)比较 A: 完整或截短突变的核心启动子的荧光素酶报告质粒示意图; B~E: 在HepG2细胞中,分别转染pCH9-1070-1901-Gluc(B)、pCH9-1636-1901-Gluc(C)、pCH9-1070-1635/1745-1901-Gluc(D)、pCH9-1744-1901-Gluc(E)4种质粒,Reversine处理48 h后,检测该化合物对野生型和截短突变型核心启动子的抑制效应; F: 在HepG2.2.15细胞中,对21种核受体超家族转录因子的筛选结果1:DMS0;2:NR1H3;3:NR1C3;4:NR5A2;5:NA1A2;6:CEBPα; 7:NR2A1;8:NR1C1;9:NR1C2;10:NR1H2;11:NR3C3;12:NR1H4;13:NR2B3;14:NR2B1;15:NR2B2; 16:NR3C2;17:NR4A2;18:NR5A1;19:NR4A1;20:NR3C1;21:NR1B3;22:NR3A2; G、H: Reversine抑制HepG2.2.15和瞬时转染HBV 1.3倍体的HepG2细胞内NR1C2的表达 图 6 Reversine抑制BCP活性且抑制NR1C2的转录 |
为了探讨Reversine对与转录相关的一些核受体超家族转录因子的影响,采用荧光定量PCR检测药物处理后(终浓度为10 μmol/L)的HepG2.2.15细胞内核受体超家族转录因子的mRNA表达(图 6F)。在21个核受体超家族转录因子中,NR1C2(PPARβ) 的mRNA表达被下调40%。对NR1C2转录因子进行进一步的验证发现(药物浓度设置为0、10、20 μmol/L):在两种HBV的复制细胞系中,NR1C2因子表达水平的下降程度随着药物浓度的增加而增加,当药物浓度为20 μmol/L时,NR1C2因子在瞬时转染HBV 1.3倍体的HepG2细胞内的表达下调了50%(图 6G、H)。以上结果提示:Reversine有可能通过抑制NR1C2 mRNA的表达来进一步影响HBV核心启动子的转录活性。
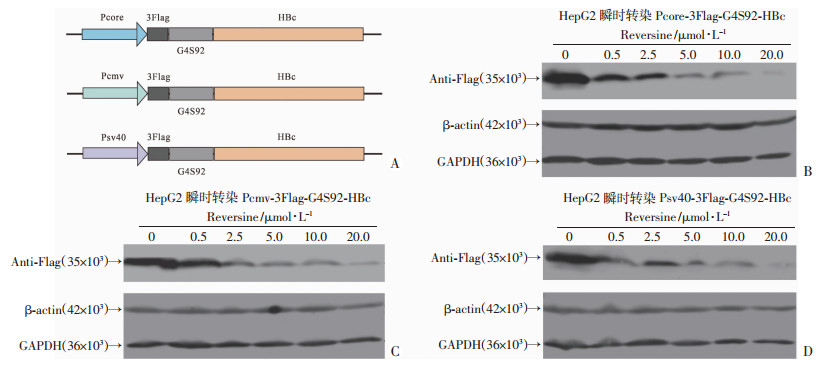
2.7 Reversine的作用特异性分析为了探索Reversine作用是否具有特异性,在HepG2细胞中分别转染Pcore-3Flag-G4S92-HBc、Pcmv-3Flag-G4S92-HBc及Psv40-3Flag-G4S92-HBc质粒,用不同浓度的Reversine(终浓度为0、0.5、2.5、5.0、10.0、20.0 μmol/L)处理细胞,48 h后做Western blot检测。结果显示(图 7):Reversine不仅对核心启动子(Pcore)具有剂量依赖性的抑制效应,对另外两种病毒启动子Pcmv和Psv40也表现出相似的抑制效应,但对两种内参基因(β-actin和GAPDH)的表达无影响。上述结果提示:Reversine的作用存在一定的启动子选择性。
|
| A: 3种病毒源性启动子(Pcore、Pcmv、Psv40)驱动的HBc蛋白表达的质粒结构示意图; B~D: 在HepG2细胞中,分别转染Pcore-3Flag-G4S92-HBc、Pcmv-3Flag-G4S92-HBc及Psv40-3Flag-G4S92-HBc质粒,经Reversine处理48 h后,Western blot检测蛋白表达 图 7 Reversine不具有HBV核心启动子的特异性 |
3 讨论
乙肝病毒的整个感染复制周期,可以看做由一系列过程组成的循环。干扰这个循环中的任一环节,理论上均可干扰病毒复制。将病毒复制周期做过程化分解,有助于我们设计的靶点相对明确和集中,同时易于实现构建高通量筛选的细胞模型。本研究将HBV核心启动子的转录这一过程单独分解出来,并用一种易于检测的方式加以表征。核心启动子是决定HBV复制肝细胞特异性的重要因素,其转录受宿主因子的严格调控[15]。该启动子在非肝源细胞中活性很低,而在肝源细胞中活性较高,这可能是由于非肝细胞中缺乏某些特异性表达的转录因子或者存在一些负性调节因子所致,而HBV的其他启动子,并不具有这种细胞特异性[16-18]。本研究采用了一种表征核心启动子活性的简便方式,即由核心启动子驱动表达的Gaussia荧光素酶,其灵敏度很高[19],且能分泌到细胞培养上清。这为检测带来巨大便利: 无需裂解细胞,而仅需取少量培养上清直接检测即可。此外,本研究构建的核心启动子驱动的Gaussia荧光素酶稳定表达细胞系,由于无需转染步骤,因而使得筛选过程更加稳定便捷,便于实现化合物的高通量筛选。当然,由于Gaussia的最终表达,不但与转录相关,也涉及转录后的诸多步骤如翻译等。因此,本细胞模型仅适合作为一种初步筛选工具,筛选所得的分子是否通过抑制转录环节起作用,还需要其他实验进一步鉴定。但HBV某些病毒成分的减少(如HBV DNA、HBsAg、HBeAg等)至少可以在很大程度上由转录被抑制来解释。
利用本筛选模型以及多轮鉴定,从1 450种化合物中筛选到两种化合物Reversine和SCH58261。后者虽然在HepG2-Pcore-Gluc细胞中表现出良好的效应,但并不能在HepG2.2.15细胞中抑制HBV DNA的复制,提示前两轮筛选的确存在假阳性情况,筛选所得化合物最终需要在HBV复制细胞系上进行确认。Reversine是一种Aurora激酶抑制剂,已经被证明是一种有效的抗肿瘤药物,主要通过诱导细胞凋亡、细胞周期阻滞和自噬来发挥抗癌作用[20-23]。在HepG2.2.15细胞和瞬时转染HBV 1.3倍体的HepG2细胞模型中,Reversine能有效抑制HBV DNA的复制。在HepG2瞬时转染模型中,其IC50=0.24 μmol/L,同时表现出较好的选择性(SI=210),好于最近KARKERI等[24]报道的SRI32007(SI=7.2)。进一步研究显示:Reversine能抑制HBV pgRNA的转录,提示其作用途径可能是影响了核心启动子的转录活性。本研究还发现:该化合物对另外两种病毒来源的启动子(Pcmv、Psv40)也具有抑制作用,但在同样的作用浓度下对细胞内β-Actin、GAPDH蛋白表达没有影响,提示该化合物的作用似乎存在一定的特异性,同时,Reversine是否对其他病毒的启动子也有抑制作用值得进一步研究。
核心启动子的截短突变体实验显示:Reversine对完整的核心启动子(含EnhⅠ、EnhⅡ和BCP)和截短的(含有EnhⅠ和BCP或EnhⅡ和BCP或只有BCP)核心启动子均具有相似的抑制效果,提示其起作用的部位应是这些表达载体共有的部分,即BCP。BCP包含多个核受体超家族转录因子的结合位点,包括HNF4、PPARα-RXRα异质二聚体,以及C/EBP等一系列转录因子,共同调控preCRNA和pgRNA的转录,对HBV的复制和致病性影响深远[15]。基于此,我们筛选了21种核受体超家族转录因子,结果显示:Reversine能将肝癌细胞内NR1C2(或PPARβ/δ,过氧化物酶体增殖物激活受体β)的mRNA水平下调50%。过氧化物酶体增殖物激活受体(PPARs)属于核激素受体超家族,它是由配体激活的转录因子组成,在调节脂肪酸摄取和氧化、脂质和碳水化合物代谢、炎症等相关过程中发挥重要作用[25]。有研究报道PPARs和RXRs形成异源二聚体,可提高Enh I/X启动子的活性,从而进一步地激活病毒核心启动子的转录[26]。因此,Reversine抑制核心启动子转录的效应是否是通过抑制PPARβ的表达来实现以及Reversine是如何下调PPARβ的表达,还有待进一步研究。有趣的是,Reversine同时也能抑制HBeAg和HBsAg的水平,提示该药物对驱动这两种抗原表达的启动子可能也有抑制作用。有报道显示HBsAg可能是导致慢性乙型肝炎患者免疫应答的原因之一[27]。因此,Reversine抑制HBsAg表达的作用,是否有助于缓解或逆转机体的免疫耐受也值得关注。
| [1] |
ARENDS J E, LIEVELD F I, AHMAD S, et al. New viral and immunological targets for hepatitis B treatment and cure: a review[J]. Infect Dis Ther, 2017, 6(4): 461-476. |
| [2] |
MARTINEZ M G, VILLERET F, TESTONI B, et al. Can we cure hepatitis B virus with novel direct-acting antivirals?[J]. Liver Int, 2020, 40(17): 27-34. |
| [3] |
TERRAULT N A, LOK A S F, MCMAHON B J, et al. Update on prevention, diagnosis, and treatment of chronic hepatitis B: AASLD 2018 hepatitis B guidance[J]. Hepatology, 2018, 67(4): 1560-1599. |
| [4] |
CHENG Z, ZHI X, SUN G, et al. Sodium selenite suppresses hepatitis B virus transcription and replication in human hepatoma cell lines[J]. J Med Virol, 2016, 88(4): 653-663. |
| [5] |
KÖSE Ș, TATLI KIȘ T, ATALAY S. Efficacy of tenofovir and entecavir in patients who relapsed after pegylated interferon therapy[J]. J Chemother, 2019, 31(4): 209-213. |
| [6] |
ANDERSON R T, LIM S G, MISHRA P, et al. Challenges, considerations, and principles to guide trials of combination therapies for chronic hepatitis B virus[J]. Gastroenterology, 2019, 156(3): 529-533. |
| [7] |
SUNG J J, WONG M L, BOWDEN S, et al. Intrahepatic hepatitis B virus covalently closed circular DNA can be a predictor of sustained response to therapy[J]. Gastroenterology, 2005, 128(7): 1890-1897. |
| [8] |
WERLE-LAPOSTOLLE B, BOWDEN S, LOCARNINI S, et al. Persistence of cccDNA during the natural history of chronic hepatitis B and decline during adefovir dipivoxil therapy[J]. Gastroenterology, 2004, 126(7): 1750-1758. |
| [9] |
WEI Z Q, ZHANG Y H, KE C Z, et al. Curcumin inhibits hepatitis B virus infection by down-regulating cccDNA-bound histone acetylation[J]. World J Gastroenterol, 2017, 23(34): 6252-6260. |
| [10] |
DENG J J, KONG K E, GAO W W, et al. Interplay between SIRT1 and hepatitis B virus X protein in the activation of viral transcription[J]. Biochim Biophys Acta Gene Regul Mech, 2017, 1860(4): 491-501. |
| [11] |
ZHANG Y, LOU C, XU Y, et al. Screening of inhibitors against histone demethylation jumonji domain-containing protein 3 by capillary electrophoresis[J]. J Chromatogr A, 2020, 1613: 460625. |
| [12] |
NASSAL M. HBV cccDNA: viral persistence reservoir and key obstacle for a cure of chronic hepatitis B[J]. Gut, 2015, 64(12): 1972-1984. |
| [13] |
SANJANA N E, SHALEM O, ZHANG F. Improved vectors and genome-wide libraries for CRISPR screening[J]. Nat Methods, 2014, 11(8): 783-784. |
| [14] |
WEI X F, GAN C Y, CUI J, et al. Identification of compounds targeting hepatitis B virus core protein dimerization through a split luciferase complementation assay[J]. Antimicrob Agents Chemother, 2018, 62(12). |
| [15] |
QUARLERI J. Core promoter: a critical region where the hepatitis B virus makes decisions[J]. World J Gastroenterol, 2014, 20(2): 425-435. |
| [16] |
KRAMVIS A, KEW M C. The core promoter of hepatitis B virus[J]. J Viral Hepat, 1999, 6(6): 415-427. |
| [17] |
LIU C J, KAO J H. Genetic variability of hepatitis B virus and response to antiviral therapy[J]. Antivir Ther, 2008, 13(5): 613-624. |
| [18] |
RAMIÈRE C, SCHOLTÈS C, DIAZ O, et al. Transactivation of the hepatitis B virus core promoter by the nuclear receptor FXRalpha[J]. J Virol, 2008, 82(21): 10832-10840. |
| [19] |
TANNOUS B A, KIM D E, FERNANDEZ J L, et al. Codon-optimized Gaussia luciferase cDNA for mammalian gene expression in culture and in vivo[J]. Mol Ther, 2005, 11(3): 435-443. |
| [20] |
BOSCO B, DEFANT A, MESSINA A, et al. Synthesis of 2, 6-diamino-substituted purine derivatives and evaluation of cell cycle arrest in breast and colorectal cancer cells[J]. Molecules, 2018, 23(8): 1996. |
| [21] |
CHENG L, WANG H, GUO K C, et al. Reversine, a substituted purine, exerts an inhibitive effect on human renal carcinoma cells via induction of cell apoptosis and polyploidy[J]. Oncotargets Ther, 2018, 11: 1025-1035. |
| [22] |
LU Y C, LEE Y R, LIAO J D, et al. Reversine induced multinucleated cells, cell apoptosis and autophagy in human non-small cell lung cancer cells[J]. PLoS One, 2016, 11(7): e0158587. |
| [23] |
PICCOLI M, GHIROLDI A, MONASKY M M, et al. Reversine: a synthetic purine with a dual activity as a cell dedifferentiating agent and a selective anticancer drug[J]. Curr Med Chem, 2020, 27(21): 3448-3462. |
| [24] |
KALKERI R, PENG J Z, HUANG C S, et al. HBV core promoter inhibition by tubulin polymerization inhibitor (SRI-32007)[J]. Adv Virol, 2020, 2020: 8844061. |
| [25] |
WAHLI W, MICHALIK L. PPARs at the crossroads of lipid signaling and inflammation[J]. Trends Endocrinol Metab, 2012, 23(7): 351-363. |
| [26] |
TURTON K L, MEIER-STEPHENSON V, BADMALIA M D, et al. Host transcription factors in hepatitis B virus RNA synthesis[J]. Viruses, 2020, 12(2): 160. |
| [27] |
GEHRING A J, PROTZER U. Targeting innate and adaptive immune responses to cure chronic HBV infection[J]. Gastroenterology, 2019, 156(2): 325-337. |




