2. 836300 新疆 阿勒泰,解放军第949医院卫勤处
2. Department of Military Health Service, No. 949 Hospital of PLA, Altay, Xinjiang Uygur Autonomous Region, 836300, China
高血压是卒中和冠状动脉疾病最主要的危险因素。仅2015年,全球共有780万人死于高血压相关疾病,1.48亿人因此丧失行动能力[1]。我国2.9亿心血管疾病患者中高血压占85%以上,而60%以上的心血管疾病死亡患者都与高血压密切相关[2]。高血压的发病机制极为复杂,其家族聚集性提示遗传因素在其中发挥了重要作用,然而目前对其遗传机制仍知之甚少。随着遗传大数据技术、平台及相关分析方法的应用,通过各类测序技术已发现多个基因可能与高血压发病相关,如肾素-血管紧张素-醛固酮系统(renin-angiotensin-aldosterone system, RAAS)中的AGT、ACE、ATIR基因[3-4],转化生长因子β(transforming growth factor β,TGFβ)通路中的SMAD、BMP2、TGFβ基因等[5-6],但遗憾的是这些位点仅能解释血压遗传能力的4%~6%。此外,由于遗传背景和环境作用的差异,不同人群中发现的高血压易感基因也不尽相同,仍有大量血压调节的分子遗传机制未被认识。
除了平原人群的高血压,高原世居人群的高血压也成为研究热点。医学高原一般指海拔2 500 m以上地区,特殊的地理环境特点如低压低氧、寒冷、干燥、大风、强紫外线辐射给生存在其中的人群造成显著的生理效应[7-8]。帕米尔高原位于中亚,是有人类居住的高海拔地区之一,而塔吉克族则世居于此。塔吉克族是我国56个民族之一,也是我国仅有的几个高海拔原住居民之一[9-10]。自“一带一路”倡议实施以来,生活在通往中西亚咽喉部位、世代守卫我国西部高原边陲的塔吉克族,其健康状况受到越来越多的关注。塔吉克族高血压患病率高,心血管健康问题亟待解决,但其遗传易感机制目前仍不清楚,与其他种族共有或种族特有的高血压相关遗传标记也未有过深入研究[11]。另一方面,塔吉克族极少与其他民族通婚,遗传环境相对隔离,长期形成的宝贵遗传资源也为增进对高血压发病机制的认识提供了新的契机。因此,本研究利用二代测序技术,筛选出与塔吉克族高血压显著关联的关键SNP位点和相关基因,以期为揭示塔吉克族高血压的遗传易感机制,进一步从高原低氧环境视角下丰富对血压调节通路的认识打下坚实基础。
1 资料与方法 1.1 研究对象通过与中国人民解放军第949医院合作,于2017年7月招募世居新疆喀什地区塔什库尔干县(平均海拔4 000 m)的塔吉克族成年人群作为研究对象。采用成组病例-对照研究设计,纳入标准为世居该地区三代以上、世代族内通婚且无血缘关系的塔吉克族成年个体,高血压组纳入标准为收缩压≥140 mmHg和(或)舒张压≥90 mmHg,并且未服用过或停止服用2周降压药的患者,对照组为血压正常个体。排除继发性高血压、慢性高原病(如高原红细胞增多症)、糖尿病、肿瘤患者以及正处于心血管疾病急性期(如冠心病急性期)和急性感染患者。
1.2 方法 1.2.1 基本情况调查研究对象均签署知情同意书,经专业人员调查其基本情况(年龄、性别、民族、生活习惯、健康状况、高原居住时间、居住海拔高度等)并做体格检查(血压、身高、体质量等)。血压测量采用标准水银血压计,取受试者静坐15 min和20 min的2次血压的平均值为一次测量值,经连续3 d测量后的平均值为最终值。本研究获陆军军医大学医学伦理委员会审查批准(2015年)。
1.2.2 血液标本采集受试者于清晨空腹时由专业医护人员统一采集5 mL静脉血于高原特殊真空抗凝采血管中,混匀后在2 h内离心处理(3 000 r/min离心10 min)分离出血细胞。采用全血基因组提取试剂盒(OMEGA公司)进行DNA提取并转移至冻存管中,放入液氮保存。
1.2.3 全外显子测序经检测合格后将DNA样品片段化处理,建库和捕获实验采用Agilent SureSelect Human All Exon V6试剂盒,之后行末端修复和3′端加A制备文库,通过QUBIT对文库进行定量,并用Agilent 2100 Bioanalyzer检测文库插入片段大小情况,最后库检合格样品在Illumina HiSeq 2500 PE150进行双端测序。外显子测序由北京博奥晶典生物技术有限公司完成。
1.2.4 生物信息学分析对测序原始数据利用fastqc进行reads和碱基质量的评估和过滤,得到高碱基质量、无接头序列污染的clean reads片段,各样本clean reads数量均达到千万数量级,基因组平均测序深度达10×以上,外显子区域捕获深度达80×以上。通过BMA-0.7.12将有效测序数据与人类参考基因组(GRCh37/hg19)进行比对,利用Picard进行排序、标记重复reads,并使用GATK (版本v3.7) Base Recalibrator重新校正已知SNP位点的碱基质量,使用DeepVariant检测群体外显子SNP,利用bcftools选取bi-allelic SNPs。利用ANNOVAR对检测出的群体外显子上SNP位点进行基因注释、氨基酸变化注释。
1.3 统计学分析通过Fisher确切概率法比较组间参考等位基因和变异等位基因频率分布差异,将群体组间等位基因频率分布差异性P值<0.05的所有SNP位点纳入统计学分析。使用metascape(https://www.metascape.org/)进行基因富集分析;应用哈代-温伯格平衡(HWE)检验分析群体资料有效性;采用Pearson χ2检验两组人员性别差异及群体显著SNP位点的5种遗传模型[共显性模型(codominant)、显性模型(dominant)、隐性模型(recessive)、乘法模型(multiple)、超显性模型(overdominant)],5种模型比较采用Bonferroni校正,显著性P值定义为0.05/5=0.01;两组人员年龄、收缩压、舒张压的比较采用非参数检验;BMI比较采用两独立样本t检验;采用非条件logistic回归检验两组人员年龄、性别因素与高血压患病的相关性并分析显著SNP位点在高血压患病中的风险度。双侧检验,P < 0.05认为差异有统计学意义。
2 结果 2.1 研究对象一般情况研究对象为塔什库尔干县62名70岁以下塔吉克族成年个体,这一群体世居海拔4 000 m高原民族聚居区,生活环境相对封闭,宗教风俗完全一致,生活习惯十分接近,均否认吸烟史(即吸烟1支/d持续1年以上)和饮酒史(即白酒≥50 g/d)。该研究群体根据是否罹患高血压分为对照组(26例)和高血压组(36例)。两组人员年龄、收缩压、舒张压不满足正态分布。对照组年龄27~68岁,中位年龄38岁,高血压组年龄19~68岁,中位年龄47.5岁,两组年龄差异有统计学意义(P < 0.05,表 1);对照组收缩压100~130 mmHg,中位数为120 mmHg,高血压组收缩压140~200 mmHg,中位数为140 mmHg;对照组舒张压60~90 mmHg,中位数为80 mmHg,高血压组舒张压80~130 mmHg,中位数为100 mmHg,两组血压差异有统计学意义(P < 0.01,表 1)。两组人员性别分布差异无统计学意义。BMI服从正态分布,组间比较差异无统计学意义,且均属于正常范围(表 1)。为进一步排除较少样本量时年龄、性别对高血压患病的影响,采用非条件logistic回归将二者纳入全变量模型进行检验,结果显示年龄(OR=1.04,95%CI=0.99~1.09,P=0.097)、性别(OR= 0.39,95%CI=0.09~1.65,P=0.199)对是否罹患高血压均无统计学意义。
| 类别 | 对照组(n=26) | 高血压组(n=36) | P |
| 年龄/岁 | 38.00(31.75, 47.00) | 47.50(35.00, 59.75) | 0.049 |
| 性别 | |||
| 男性 | 3(11.5) | 10(27.8) | 0.205 |
| 女性 | 23(88.5) | 26(72.2) | |
| BMI/kg·m-2 | 22.6±3.4 | 23.9±4.4 | 0.214 |
| 收缩压/mmHg | 120.0(117.5, 120.0) | 140.0(140.0, 150.0) | < 0.001 |
| 舒张压/mmHg | 80.0(70.0, 80.0) | 100.0(90.0, 100.0) | < 0.001 |
2.2 SNP位点注释与分析
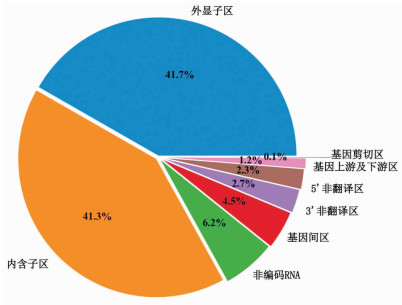
该塔吉克族群体组间等位基因频率分布有统计学差异的SNP位点共6 739个,其中位于外显子区的共2 813个,占总SNP数的41.7%,位于内含子区的共2 784个,占总SNP数的41.3%,剩余17%的SNP位点位于基因组的其他各个区域,包括基因间区、非编码RNA、5’非翻译区、3’非翻译区等(图 1)。
|
| 基因上游及下游区为上、下游1 000 bp内;基因剪切区为剪切位点上游2 bp内 图 1 差异SNP位点在基因组不同区域分布图 |
2.3 组间差异基因功能分析
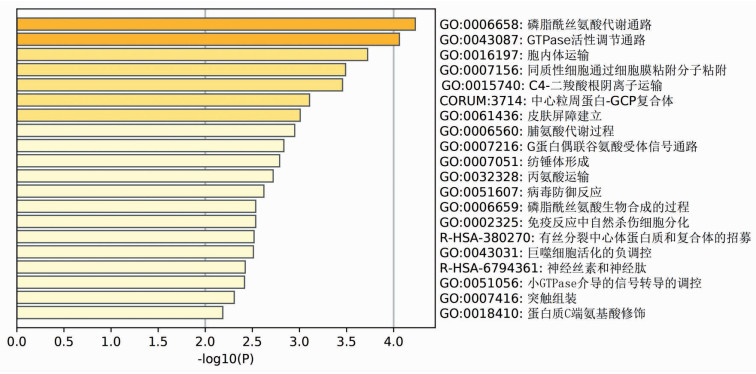
该塔吉克族群体6 739个组间差异SNP位点共定位于3 646个基因上,根据GeneCards(https://www.genecards.org/)数据库所收录的已知高血压相关基因,截止2020年6月5日共收录6 606个,以及此次发现的塔吉克族高血压相关基因,筛选出塔吉克族人群所共有的高血压已知基因,共1 178个,包括血管紧张素Ⅱ受体1型基因(angiotensin Ⅱ receptor type 1, AGTR1)、基质金属蛋白酶2基因(matrix metallopeptidase 2, MMP2)、白蛋白基因(albumin, ALB)等。此外,发现其余2 468个基因未收录于数据库,但与塔吉克族高血压发病风险相关,通过metascape在线分析网站将未收录的高血压相关基因进行富集分析(图 2),结果显示其更显著地富集在磷脂酰丝氨酸代谢通路(phosphatidylserine metabolic process)和GTPase活性调节通路(regulation of GTPase activity)。其中,富集于磷脂酰丝氨酸代谢通路的基因11个,仅占差异基因总数的0.48%,log10(P)=-4.22,而富集于GTPase活性调节通路的基因最多,共70个,占差异基因总数的3.12%,log10(P)=-4.06。提示GTPase活性调节通路更有可能参与该民族高血压的发生发展过程。
|
| 横轴表示P值的变换值-log10(P),纵轴表示最具代表性的前20个富集通路,通路富集分析使用的基因本体源包括KEGG通路、GO生物过程、Reactome基因集、Canonical通路和CORUM基因集 图 2 尚未被收录的高血压相关基因富集分析 |
2.4 显著差异基因SNP位点信息
筛选出GTPase活性调节通路中组间等位基因频率分布差异最显著的5个SNP位点进行高血压关联分析。具体信息如表 2所示。5个最显著SNP位点分别定位于该通路的5个相关基因上,其中4个位点位于基因的外显子区域,包括2个同义突变和2个非同义突变。
| 基因名 | 染色体号 | 突变位置 | 参考等位基因 | 突变等位基因 | 参考基因功能区 | 外显子功能区 | 氨基酸改变 | rs号 |
| RAB3GAP1 | 2 | 135911422 | T | C | 外显子区 | 同义突变 | p.F755F | rs17261772 |
| LRCH1 | 13 | 47297352 | G | A | 内含子区 | \\ | \\ | rs7319836 |
| ARHGEF11 | 1 | 156907115 | T | C | 外显子区 | 非同义突变 | p.S832G | rs868188 |
| SYDE2 | 1 | 85666156 | T | G | 外显子区 | 非同义突变 | p.Q175P | rs12121540 |
| RASGRF1 | 15 | 79296172 | G | A | 外显子区 | 同义突变 | p.P39P | rs1562008 |
2.5 5个SNP位点的哈代-温伯格平衡检验
对5个SNP位点在组间的频率分布进行HWE检验(表 3),5个位点在对照组均符合哈代-温伯格平衡;在高血压组中,5个位点P值分别为0.135、0.367、0.051、0.069、0.23,均大于0.05,符合哈代-温伯格平衡,表明选择的群体有很好的代表性,群体调查资料可靠,可进行后续分析。
| SNP定位 | 分组 | 基因型[例(%)] | HWE P值 | ||
| RAB3GAP1 | TT | TC | CC | ||
| (rs17261772) | 高血压组 | 2(5.6) | 7(19.4) | 27(75) | 0.135 |
| 对照组 | 4(15.4) | 13(50) | 9(34.6) | 0.845 | |
| LRCH1 | GG | GA | AA | ||
| (rs7319836) | 高血压组 | 8(22.2) | 15(41.7) | 13(36.1) | 0.367 |
| 对照组 | 2(7.7) | 5(19.2) | 19(73.1) | 0.094 | |
| ARHGEF11 | TT | TC | CC | ||
| (rs868188) | 高血压组 | 18(50) | 11(30.6) | 7(19.4) | 0.051 |
| 对照组 | 4(15.4) | 12(46.2) | 10(38.5) | 0.899 | |
| SYDE2 | TT | TG | GG | ||
| (rs12121540) | 高血压组 | 9(25) | 23(63.9) | 4(11.1) | 0.069 |
| 对照组 | 16(61.5) | 10(38.5) | 0 | 0.225 | |
| RASGRF1 | GG | GA | AA | ||
| (rs1562008) | 高血压组 | 24(66.7) | 12(33.3) | 0 | 0.23 |
| 对照组 | 12(46.2) | 8(30.7) | 6(23.1) | 0.074 | |
2.6 5个SNP位点等位基因分布和发病风险分析
表 4为各位点等位基因在组间的频率分布差异情况。在高血压组人群中,rs17261772位点的C等位基因占到了84.7%,而在正常对照组人群中仅占59.6%,组间存在统计学差异(P < 0.01)。由此可见,与参考等位基因相比,rs17261772位点C等位基因是患病的风险因子,其携带者患病风险是T等位基因携带者的3.8倍(OR=3.76,95%CI=1.61~8.77)。同理进行比较发现,rs7319836位点A等位基因则是保护性因子,其携带者患病风险仅为G等位基因携带者的28%(OR=0.28,95%CI=0.12~0.65)。rs868188位点C等位基因也是保护性因子,其携带者患病风险仅为T等位基因携带者的33%(OR=0.33,95%CI=0.16~0.70)。而rs12121540位点G等位基因则是患病的风险因子,其携带者患病风险是T等位基因携带者的3.2倍(OR= 3.17,95%CI=1.38~7.30)。rs1562008位点A等位基因则是保护性因子,其携带者患病风险仅为G等位基因携带者的32%(OR为0.32,95%CI=0.14~0.74)。
| 基因名 | SNP位点 | 高血压组 [例(%)] |
对照组 [例(%)] |
P值 | OR | 95% CI |
| RAB3GAP1 | rs17261772 | |||||
| T | 11(15.3) | 21(40.4) | 参考 | |||
| C | 61(84.7) | 31(59.6) | 0.002 | 3.76 | 1.61~8.77 | |
| LRCH1 | rs7319836 | |||||
| G | 31(43.1) | 9(17.3) | 参考 | |||
| A | 41(56.9) | 43(82.7) | 0.003 | 0.28 | 0.12~0.65 | |
| ARHGEF11 | rs868188 | |||||
| T | 47(65.3) | 20(38.5) | 参考 | |||
| C | 25(34.7) | 32(61.5) | 0.004 | 0.33 | 0.16~0.70 | |
| SYDE2 | rs12121540 | |||||
| T | 41(56.9) | 42(80.8) | 参考 | |||
| G | 31(43.1) | 10(19.2) | 0.007 | 3.17 | 1.38~7.30 | |
| RASGRF1 | rs1562008 | |||||
| G | 60(83.3) | 32(61.5) | 参考 | |||
| A | 12(16.7) | 20(38.5) | 0.007 | 0.32 | 0.14~0.74 |
2.7 5个SNP位点在不同遗传模型下的关联分析
对各位点采用五种遗传模型分析,分别计算其与高血压患病风险的关联性(表 5)。经Bonferroni校正后rs17261772、rs7319836、rs868188、rs12121540位点在隐性模型、乘法模型中显示出与患病风险相关,rs1562008位点在显性模型、乘法模型中显示出与患病风险相关。从乘法模型可看出各位点等位基因与患病风险的关联性(结果2.6已述)。从隐性模型中可看出,rs17261772位点的CC基因型携带者患病风险是CT、TT基因型携带者的5.7倍,是高血压患病的危险基因型(OR=5.67,95%CI=1.88~17.1),而rs7319836位点的AA基因型携带者其患病风险仅为AG、GG基因型携带者的21%,是高血压患病的保护性基因型(OR=0.21,95%CI=0.07~0.63),而rs868188位点的TT基因型携带者患病风险是TC、CC基因型携带者的5.5倍,是高血压患病的危险基因型(OR=5.50,95%CI=1.58~19.2),rs12121540位点的TT基因型携带者其患病风险仅为TG、GG基因型携带者的21%,是高血压患病的保护性基因型(OR=0.21,95%CI=0.07~ 0.62)。从显性模型中可看出,rs1562008位点的GG+GA基因型在组间分布具有显著统计学差异(P < 0.01),提示GG+GA基因型将增加患病风险。
| 模型 | 统计值 | RAB3GAP1 (rs17261772) |
LRCH1 (rs7319836) |
ARHGEF11 (rs868188) |
SYDE2 (rs12121540) |
RASGRF1 (rs1562008) |
| 共显性模型 | P | 0.06 | 0.07 | 0.02 | 0.03 | 0.004 |
| OR(MM/mm) | 6 | 0.17 | 6.43 | - | - | |
| 95%CI | 0.94~38.4 | 0.03~0.94 | 1.51~27.4 | - | - | |
| P | 0.005 | 0.02 | 0.03 | 0.02 | 0.77 | |
| OR(MM/Mm) | 5.57 | 0.29 | 4.91 | 0.25 | 1.33 | |
| 95%CI | 1.70~18.3 | 0.07~0.79 | 1.26~19.1 | 0.08~0.74 | 0.43~4.13 | |
| P | 1 | 1 | 0.76 | 0.56 | 0.017 | |
| OR(Mm/mm) | 1.08 | 0.75 | 1.31 | - | - | |
| 95%CI | 0.16~7.42 | 0.12~4.77 | 0.37~4.64 | - | - | |
| 显性模型 | P | 0.23 | 0.17 | 0.15 | 0.13 | 0.004 |
| OR(MM+Mm/mm) | 3.09 | 0.29 | 2.59 | - | - | |
| 95%CI | 0.52~18.3 | 0.06~1.51 | 0.83~8.12 | - | - | |
| 隐性模型 | P | 0.002 | 0.005 | 0.007 | 0.005 | 0.13 |
| OR(MM/Mm+mm) | 5.67 | 0.21 | 5.50 | 0.21 | 2.33 | |
| 95%CI | 1.88~17.1 | 0.07~0.63 | 1.58~19.2 | 0.07~0.62 | 0.83~6.58 | |
| 乘法模型 | P | 0.002 | 0.003 | 0.004 | 0.007 | 0.007 |
| OR(M/m) | 3.76 | 0.28 | 3.01 | 0.32 | 3.13 | |
| 95%CI | 1.61~8.77 | 0.12~0.65 | 1.44~6.30 | 0.14~0.72 | 1.36~7.20 | |
| 超显性模型 | P | 0.015 | 0.10 | 0.29 | 0.07 | 1 |
| OR(Mm/MM+mm) | 0.24 | 3 | 0.51 | 2.83 | 1.13 | |
| 95%CI | 0.08~0.75 | 0.92~9.75 | 0.18~1.46 | 0.99~8.03 | 0.38~3.32 | |
| M代表频率较大的等位基因,m代表频率较小的等位基因(MAF);经Bionferroni校正后显著性P值定义为0.05/5=0.01 | ||||||
2.8 5个SNP位点纯合突变与高血压患病风险的关联分析
为进一步验证5个显著差异SNP位点是否与高血压患病风险显著相关,分别在两组中找出该位点纯合突变的个体,采用非条件logistic回归将其纳入全变量模型进行检验。如表 6所示,rs17261772(RAB3GAP1)、rs7319836(LRCH1)、rs868188(ARHGEF11)位点纯合突变与高血压的患病风险显著相关,而rs12121540(SYDE2)、rs1562008(RASGRF1)位点纯合突变与高血压的患病风险无显著相关性。
| 协变量 | 纯合子型 | β | Wald χ2 | OR | 95% CI | P |
| RAB3GAP1(rs17261772) | CC | 1.88 | 5.53 | 6.53 | 1.37~31.26 | 0.019 |
| LRCH1(rs7319836) | AA | -1.92 | 6.45 | 0.15 | 0.33~0.64 | 0.011 |
| ARHGEF11(rs868188) | CC | -2.07 | 5.04 | 0.13 | 0.21~0.77 | 0.025 |
| SYDE2(rs12121540) | GG | 22.14 | 0.00 | - | - | 1 |
| RASGRF1(rs1562008) | AA | -20.38 | 0.00 | - | - | 1 |
3 讨论
世居高原人群在长期与恶劣自然环境斗争过程中发生了一系列病理生理性变化。这些适应性改变一方面促进其对氧的利用,另一方面也可能产生负面效应,如交感神经系统激活、内皮功能失衡、血容量增加等可能导致血压升高。目前对高原民族高血压的研究主要集中在世居藏族,已发现AGTR1、ACE、WNK1等基因多态性与其高血压高患病率有关[12],而对我国另一个高原世居民族——塔吉克族却鲜有关注。因此,本研究利用全外显子测序技术全面分析了塔吉克族高血压相关的差异SNP位点及所在基因,发现AGTR1等1 178个已知高血压相关基因也参与该民族高血压的发生、发展,与藏族及其他民族表现出了一致性;然而仍有2 468个差异基因尚未被收录。这些差异基因更多地富集于GTPase活性调节通路,提示该通路的基因遗传变异可能对该民族的高血压患病风险产生显著影响。
GTPase活性调节通路涉及Ras超家族的3种调节因子,即GTP酶激活蛋白(GAPs)、鸟嘌呤核苷酸交换因子(GEFs)和鸟嘌呤核苷酸解离抑制剂(GDIs)[13-15]。已有大量研究证实Ras超家族成员中Rho GTPase参与高血压、心脏病、糖尿病肾病等疾病的发生、发展进程,而其调节因子也通过影响Rho/ROCK通路的激活成为血管调节和血压控制的关键靶点[16-18]。本研究结果表明调节因子RAB3GAP1的rs17261772位点,LRCH1的rs7319836位点及ARHGEF11的rs868188位点与该民族高血压患病风险显著相关。
RAB3GAP1通过支持脂质转移以促进血管成纤维细胞内自噬体的生物形成,从而减少胞内毒性产物堆积避免细胞坏死[19-20]。本研究发现RAB3GAP1的rs17261772位点突变的C等位基因在高血压组中分布频率显著升高,尽管该位点为同义突变,但突变可能通过降低mRNA稳定性或影响翻译动力学引起蛋白水平下降,抑制血管成纤维细胞内自噬体的生物合成,增加在高原低氧暴露环境下血管损伤和局部炎症反应风险,最终导致高血压病的进展[21]。
LRCH1通过调控CDC42 GTPase阻止CD4+T细胞迁移从而调节免疫反应,而其某些位点突变促进了膝关节炎和溃疡性结肠炎中免疫细胞浸润和持续炎症反应[22]。本研究发现LRCH1的rs7319836位点突变的A等位基因在对照组中的分布频率显著升高,其可能是该高原民族患病的保护因子。该位点位于LRCH1的内含子区,可能通过影响DNA-RNA网络调控及mRNA翻译速率引起蛋白表达上升或功能激活,增强对CD4+T细胞迁移的抑制作用,减轻在高原氧化应激条件下心血管系统的免疫浸润和血管炎性损伤的进行性加重。相反,携带更多G等位基因的高血压组人群则无法有效对抗或逆转这种氧化应激进程,从而在一定程度上增加患病风险。
ARHGEF11通过激活Rho/ROCK通路抑制肌球蛋白轻链磷酸酶(myosin light chain phosphatase, MLCP)活性,从而使磷酸化肌球蛋白轻链(phospho-myosin light chain,p-MLC)无法去磷酸化,使血管平滑肌细胞中p-MLC的数量增加,引起血管持续性收缩。最近有研究显示盐敏感大鼠体内Arhgef11的缺失对高血压肾损伤有保护作用[23]。本研究发现ARHGEF11的rs868188位点突变的C等位基因在对照组中的分布频率显著升高,其可能是该高原民族患病的保护因子。该位点突变导致第832位氨基酸由甘氨酸变为丝氨酸,可能使其编码蛋白功能发生障碍,抑制血管平滑肌细胞中的RhoA GTPase的活性,在一定程度上降低p-MLC的数量,使血管持续较高的紧张状态得到缓解。
尽管本研究筛选出3个GTPase活性调节因子与高血压患病风险显著相关,但其具体的功能意义和潜在机制尚不清楚,还需进一步通过实验研究与扩大人群验证。本研究局限之处在于:①样本量较少,本研究结果可能是偶然的,但由于塔吉克族在我国境内仅5万余人,人口总数少且生活环境相对封闭,这已是目前能收集到的最大样本量,下一步我们将扩大采样范围对结果进行验证。②尽管统计学上性别在组间匹配,但两组男性例数差异较大(对照组仅3例,高血压组10例)。这是由于塔吉克族健康成年男性在气候变暖后大多离乡在外工作,因此留守的健康人群以女性为主。下一步我们将继续细化采样方案使性别匹配更加完善。③在统计学上两组年龄不匹配,尽管在纳入年龄因素后发现其并未对本研究造成影响,下一步我们在扩大样本招募时将对研究对象年龄进行更加严谨地筛选和分层。
综上,本研究利用二代测序技术筛选出与塔吉克族原发性高血压发病风险相关联的SNP位点和相关基因。在去除已知高血压相关基因后,发现该高原民族高血压发病风险与GTPase活性调节通路有关,并对其中最显著的相关基因及SNP位点进行遗传关联分析,揭示了该民族所特有的原发性高血压遗传基础,提高了对高原缺氧条件下世居人群原发性高血压遗传易感性及血压调节通路的认识。
| [1] |
EVANGELOU E, WARREN H R, MOSEN-ANSORENA D, et al. Genetic analysis of over 1 million people identifies 535 new loci associated with blood pressure traits[J]. Nat Genet, 2018, 50(10): 1412-1425. DOI:10.1038/s41588-018-0205-x |
| [2] |
胡盛寿, 高润霖, 刘力生, 等. 《中国心血管病报告2018》概要[J]. 中国循环杂志, 2019, 34(3): 209-220. HU S S, GAO R L, LIU L S, et al. Summary of the 2018 report on cardiovascular diseases in China[J]. Chin Circ J, 2019, 34(3): 209-220. DOI:10.3969/j.issn.1000-3614.2019.03.001 |
| [3] |
JI L D, LI J Y, YAO B B, et al. Are genetic polymorphisms in the renin-angiotensin-aldosterone system associated with essential hypertension? Evidence from genome-wide association studies[J]. J Hum Hypertens, 2017, 31(11): 695-698. DOI:10.1038/jhh.2017.29 |
| [4] |
FAN Z, WU G, YUE M, et al. Hypertension and hypertensive left ventricular hypertrophy are associated with ACE2 genetic polymorphism[J]. Life Sci, 2019, 225: 39-45. DOI:10.1016/j.lfs.2019.03.059 |
| [5] |
NAKAO E, ADACHI H, ENOMOTO M, et al. Elevated plasma transforming growth factor β1 levels predict the development of hypertension in normotensives: the 14-year follow-up study[J]. Am J Hypertens, 2017, 30(8): 808-814. DOI:10.1093/ajh/hpx053 |
| [6] |
FENG W, DELL'ITALIA L J, SANDERS P W. Novel paradigms of salt and hypertension[J]. J Am Soc Nephrol, 2017, 28(5): 1362-1369. DOI:10.1681/asn.2016080927 |
| [7] |
BILO G, CARAVITA S, TORLASCO C, et al. Blood pressure at high altitude: physiology and clinical implications[J]. Kardiologia Polska, 2019, 77(6): 596-603. DOI:10.33963/KP.14832 |
| [8] |
NARVAEZ-GUERRA O, HERRERA-ENRIQUEZ K, MEDINA-LEZAMA J, et al. Systemic hypertension at high altitude[J]. Hypertension, 2018, 72(3): 567-578. DOI:10.1161/hypertensionaha.118.11140 |
| [9] |
PENG M S, XU W, SONG J J, et al. Mitochondrial genomes uncover the maternal history of the Pamir populations[J]. Eur J Hum Genet, 2018, 26(1): 124-136. DOI:10.1038/s41431-017-0028-8 |
| [10] |
SULAIMAN X, XU W F, CAI L H, et al. Hematologic and spirometric characteristics of Tajik and Kyrgyz highlanders in the Pamir Mountains[J]. Am J Hum Biol, 2020, e23459. DOI:10.1002/ajhb.23459 |
| [11] |
龚亮, 阳盛洪, 高亮, 等. 1例中国塔吉克族人全基因组重测序分析及其与高原适应关联的初步研究[J]. 第三军医大学学报, 2019, 41(7): 665-672. GONG L, YANG S H, GAO L, et al. Whole genome resequencing and correlation analysis with high altitude adaptation: a preliminary study of a Tajik male in China[J]. J Third Mil Med Univ, 2019, 41(7): 665-672. DOI:10.16016/j.1000-5404.201808171 |
| [12] |
SHI R, LI J, HE J, et al. Association of with-no-lysine kinase 1 and Serine/Threonine kinase 39 gene polymorphisms and haplotypes with essential hypertension in Tibetans[J]. Environ Mol Mutagen, 2018, 59(2): 151-160. DOI:10.1002/em.22140 |
| [13] |
HODGE R G, RIDLEY A J. Regulating rho GTPases and their regulators[J]. Nat Rev Mol Cell Biol, 2016, 17(8): 496-510. DOI:10.1038/nrm.2016.67 |
| [14] |
KIM J G, ISLAM R, CHO J Y, et al. Regulation of RhoA GTPase and various transcription factors in the RhoA pathway[J]. J Cell Physiol, 2018, 233(9): 6381-6392. DOI:10.1002/jcp.26487 |
| [15] |
LAWSON C D, RIDLEY A J. Rho GTPase signaling complexes in cell migration and invasion[J]. J Cell Biol, 2018, 217(2): 447-457. DOI:10.1083/jcb.201612069 |
| [16] |
DEE R A, MANGUM K D, BAI X, et al. Druggable targets in the Rho pathway and their promise for therapeutic control of blood pressure[J]. Pharmacol Ther, 2019, 193: 121-134. DOI:10.1016/j.pharmthera.2018.09.001 |
| [17] |
SHIMOKAWA H, SUNAMURA S, SATOH K. RhoA/rho-kinase in the cardiovascular system[J]. Circ Res, 2016, 118(2): 352-366. DOI:10.1161/circresaha.115.306532 |
| [18] |
BAI X, MANGUM K D, DEE R A, et al. Blood pressure-associated polymorphism controls ARHGAP42 expression via serum response factor DNA binding[J]. J Clin Investig, 2017, 127(2): 670-680. DOI:10.1172/JCI88899 |
| [19] |
KOPARIR A, KARATAS O F, YILMAZ S S, et al. Revealing the functions of novel mutations in RAB3GAP1 in Martsolf and Warburg micro syndromes[J]. Am J Med Genet A, 2019, 179(4): 579-587. DOI:10.1002/ajmg.a.61065 |
| [20] |
GAVALI S, GUPTA M K, DASWANI B, et al. Estrogen enhances human osteoblast survival and function via promotion of autophagy[J]. Biochim Biophys Acta Mol Cell Res, 2019, 1866(9): 1498-1507. DOI:10.1016/j.bbamcr.2019.06.014 |
| [21] |
BRULE C E, GRAYHACK E J. Synonymous codons: choose wisely for expression[J]. Trends Genet, 2017, 33(4): 283-297. DOI:10.1016/j.tig.2017.02.001 |
| [22] |
WANG Y B, ZHANG H R, HE H, et al. LRCH1 suppresses migration of CD4+ T cells and refers to disease activity in ulcerative colitis[J]. Int J Med Sci, 2020, 17(5): 599-608. DOI:10.7150/ijms.39106 |
| [23] |
JOHNSON A C, WU W J, ATTIPOE E M, et al. Loss of Arhgef11 in the dahl salt-sensitive rat protects against hypertension-induced renal injury[J]. Hypertens Dallas Tex, 2020, 75(4): 1012-1024. DOI:10.1161/HYPERTENSIONAHA.119.14338 |




