2. 400038 重庆,陆军军医大学(第三军医大学)药学与检验医学系药物化学教研室
2. Department of Pharmaceutical Chemistry, Faculty of Pharmacy and Laboratory Medicine, Army Medical University (Third Military Medical University), Chongqing, 400038, China
叉头盒蛋白超家族(forkhead box super family)是一类具有特定“翼状螺旋结构”DNA结合域的转录因子,包括FOXA、FOXC、FOXM、FOXO和FOXP等几个家族[1-2],其中FOXM1(Forkhead box protein M1)在2010年被评为年度分子。研究表明,FOXM1在多种肿瘤,如乳腺癌、肺癌、肝癌、骨肉瘤、鼻咽癌、喉癌、胃癌、卵巢癌等中普遍上调[3-10],肿瘤基因表达谱分析也证实FOXM1是人类恶性肿瘤中表达上调频繁的基因之一[11]。在胃癌中,FOXM1的异常表达及通路活化与胃癌患者的表型和不良预后密切相关[10, 12]。因此,FOXM1被认为是预防和治疗人类癌症的一个潜在靶点。研究表明,盐屋霉素A通过抑制FOXM1的表达进而下调其下游靶基因Bcl-2和Mcl-1的表达以及激活caspase-3等来诱导细胞凋亡[13],硫链丝菌肽也被报道可以和FOXM1-DNA结合从而干扰下游靶点的转录[14]。但亦有研究表明盐屋霉素A还可以抑制母系胚胎亮氨酸拉链蛋白激酶(maternal embryonic leucine zipper kinase,MELK)[15],硫链丝菌肽也可以靶向Peroxiredoxin 3[16],说明它们靶向FOXM1的特异性不高,限制其临床应用。因此,寻找新型靶向FOXM1的小分子药物显得尤为重要。
近年来,合作课题组通过化学合成方法学的研究构建了许多结构新颖的分子骨架。其中,部分化合物的分子结构具有类天然产物的特征,表现出潜在的“类药性”。在前期开展的合成方法学研究中,发展了一类高选择性的不对称[3+3]环化反应,构建了一系列磺内酰胺-环己酮螺环衍生物。在对这类产物进行普遍生物活性评价的过程中,我们发现其中的代表化合物1-3-51可有效抑制胃癌细胞的增殖和侵袭迁移,进一步的机制研究发现其显著抑制FOXM1的表达,提示1-3-51可能是一个新型FOXM1小分子抑制剂。本研究为靶向FOXM1小分子抑制剂的设计提供一定的理论基础与思路。
1 材料与方法 1.1 材料人胃癌细胞株BGC823、MKN45由本实验室保存,高糖DMEM培养基、RPMI1640培养基购自美国HyClone公司,胎牛血清购自美国Gibco公司,抗人FOXM1抗体购自美国Cell Signal Technology公司,抗人TUBULIN一抗,HRP标记的山羊抗兔、山羊抗鼠二抗,CCK-8试剂盒、BCA蛋白浓度测定试剂盒、RIPA裂解液、结晶紫购自中国上海碧云天公司,多聚甲醛购自美国博士德公司,TRIzol、逆转录试剂盒和SYBR Green试剂购自日本TaKaRa公司,FOXM1过表达及其空载质粒购自长沙优宝公司,转染试剂Lipofectamine 2000和质粒小提试剂盒购自美国Invitrogen公司,PI/RNase Staining Buffer购自美国BD Pharmingen公司,PVDF膜、ECL化学发光试剂盒购自德国Millipore公司。
1.2 细胞培养人胃癌细胞BGC823、MKN45分别采用DMEM高糖培养基、RPMI1640培养基(含10%胎牛血清、100 U/mL青霉素和10 mg/L链霉素)培养,培养条件:饱和湿度、37 ℃、5%CO2恒温培养箱中培养。当细胞生长至80%~90%时,1 ∶3消化传代。
1.3 CCK-8检测细胞活性将细胞以4 000/孔接种在96孔板后,根据CCK-8说明书,在收获时将10 μL CCK-8添加到每个孔中。加入CCK-8 1 h后,在酶标仪450 nm波长处的光密度值D(450)来测定细胞活性。
1.4 提取细胞总蛋白处理后的细胞弃去培养基,用预冷的PBS洗涤2次,尽量弃去PBS,在每孔中加入适量含蛋白酶抑制剂的RIPA(强)裂解液,用细胞刮刮下细胞,转移到新的EP管中,振荡器上每隔1 min振荡30 s, 共3次,冰上裂解30 min,然后4 ℃,14 000 r/min离心15 min,最后吸取上清即为总蛋白并使用BCA法测其浓度。
1.5 提取RNA处理后的细胞弃去培养基,用预冷的PBS洗涤2次,尽量弃去PBS,在每孔中加入1 mL TRIzol,静置1 min,吹打使细胞裂解,收集细胞,振荡器上每隔1 min振荡30 s, 共3次,冰上裂解5 min;向上述裂解液中加入200 μL三氯甲烷,轻微混匀,室温静置15 min;离心:4 ℃,12 000×g,15 min;取上清于无DNA、RNA酶EP管中,加入0.5 mL异丙醇,混匀,室温放置5~10 min;离心:4 ℃,12 000 ×g,10 min;弃上清,加入1 mL 75% 酒精,离心:4 ℃,8 000×g,5 min;小心吸尽残余酒精,晾干;加入50 μL DEPC水,溶解沉淀,混匀;测浓度。
1.6 逆转录配制如下反应体系: 5×g DNA Eraser Buffer 2.0 μL,gDNA Eraser 1.0 μL,RNA 1.0 μg,RNase Free ddH2O补足至10 μL(反应条件: 42 ℃,10 min);向上述反应产物中加入如下体系:5×Prime Script Buffer 2(for Real Time)4.0 μL,Prime Script RT Enzyme Mix I 1.0 μL,RT Primer Mix 1.0 μL,RNase Free ddH2O 4.0 μL(反应条件:37 ℃、15 min,85 ℃、5 s);向反应产物cDNA中加入20 μL DEPC水,备用。
1.7 qRT-PCR检测配制如下体系:TB Green Premix Ex Taq Ⅱ 10.0 μL,cDNA 2.0 μL,上下游引物各0.8 μL,Rox 0.4 μL,DEPC水6 μL,总体积20 μL(反应条件: 95 ℃、3 min,95 ℃、15 s,58 ℃、30 s),40个循环,设置3个复孔,数值用Prism 8进行作图和统计学分析。
1.8 Western blot检测蛋白上样量为30 μg,使用10% SDS-PAGE凝胶电泳分离;湿转法转移蛋白至PVDF膜,5%脱脂奶粉封闭0.5~1 h,加入一抗4 ℃过夜(FOXM1 1 ∶1 000,TUBULIN 1 ∶10 000),二抗室温孵育1 h(山羊抗鼠1 ∶5 000,山羊抗兔1 ∶10 000),用化学发光法在化学发光仪上显影曝光。
1.9 Transwell小室实验检测细胞侵袭和迁移侵袭实验步骤:吸取2.5 mg/mL的Matergel添加到Transwell小室的上室中,在37 ℃培养箱内放置2 h。取胃癌细胞采用无血清的细胞培养液重悬细胞浓度为2×105/mL,吸取200 μL添加到上室中,然后在下室中添加550 μL的含血清的细胞培养液,放在37 ℃、5% CO2培养箱中培养24 h。PBS洗涤小室,甲醛固定,0.1%的结晶紫染色,镜下记录细胞侵袭数目即细胞穿膜数目。细胞迁移实验同侵袭实验,无Matergel包被步骤。
1.10 流式细胞术测定细胞周期处理后的细胞弃去培养基,用预冷的PBS洗涤2次,尽量弃去PBS,收集并洗涤细胞2次,用70%酒精4 ℃固定过夜,预冷的PBS洗涤细胞2次,加入300 μL PI避光染色30 min,上流式细胞仪分析细胞周期率。
1.11 裸鼠皮下移植瘤模型培养胃癌细胞BGC823,消化、计数后接种5×106个细胞于裸鼠腋窝皮下,分为2组,每组5只裸鼠,待瘤子长至合适大小时开始测量瘤体大小,并同时开始给予药物及对照处理,对照组及1-3-51组均采用腹腔注射方式,对照组注射玉米油,1-3-51组以玉米油作为溶剂,1-3-51使用剂量为50 mg/kg,采用隔天给药的方式;瘤体大小=(长×宽)2÷2,待瘤体长到合适大小后处死裸鼠并取出瘤体,称其质量并拍照。
1.12 细胞转染及处理方法6孔板每孔接种1.0×105个BGC823细胞;第2天细胞密度大约40%,然后分别转染空载质粒及过表达FOXM1质粒(oeFOXM1),首先用250 μL Opti-MEM稀释2 μg质粒DNA,轻轻吹吸3~5次混匀,室温静置5 min;然后用250 μL Opti-MEM稀释5 μL Lipofectamine 2000转染试剂,混匀,室温静置5 min;接下来转染试剂和质粒DNA稀释液混合均匀,室温静置20 min,最后将上述混合液体加入到已更换无血清无抗生素培养基的细胞中,转染6 h后更换完全培养基,同时在对照组及过表达组分别加入1-3-51 5 μmol/L,以及对照加入溶剂组,培养48 h后分别收集细胞用于后续实验。
1.13 统计学分析采用GraphPad Prism 8.0统计软件进行分析,数据以x±s表示,组间差异比较采用方差分析,检验水准α=0.05。
2 结果 2.1 小分子抑制剂1-3-51在胃癌细胞中抑制FOXM1的表达化学合成方法学构建小分子化合物1-3-51,其化学结构如图 1A所示。用其处理胃癌细胞BGC823和MKN45 48 h后的半数抑制浓度(IC50)分别为(5.429±0.225)μmol/L、(5.169±0.239)μmol/L(图 1B、C)。后续我们分别使用0、2.5、5 μmol/L 1-3-15处理上述两种胃癌细胞,采用Western blot及qRT-PCR检测1-3-51对胃癌细胞中FOXM1蛋白及其mRNA的表达情况,结果显示1-3-51呈浓度依赖的抑制FOXM1蛋白及mRNA表达(P < 0.01,图 1D)。接着使用1-3-51 5 μmol/L处理上述胃癌细胞0、24、48 h,结果显示其同样呈时间依赖的抑制FOXM1蛋白及mRNA表达(P < 0.05,图 1E)。
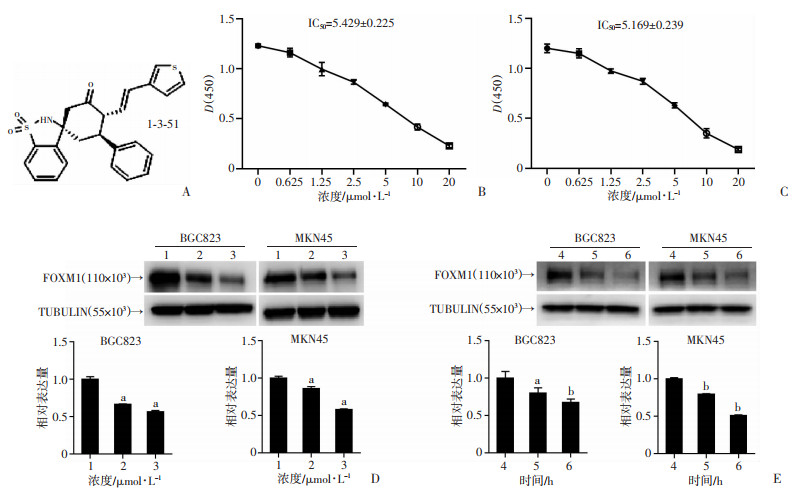
|
| 1:0 μmol/L;2:2.5 μmol/L;3:5 μmol/L;4:0 h;5:24 h;6:48 h;A:1-3-51的化学结构;B、C:CCK-8检测1-3-51处理BGC823、MKN45细胞的变化;D:不同浓度1-3-51处理BGC823、MKN45细胞后FOXM1蛋白及mRNA的表达a:P < 0.01, 与0 μmol/L比较;E:1-3-51处理BGC823、MKN45细胞后不同时间FOXM1蛋白及mRNA的表达a:P < 0.05, b:P < 0.01, 与0 h比较 图 1 1-3-51在胃癌细胞中降低FOXM1蛋白及mRNA水平 |
2.2 小分子抑制剂1-3-51在胃癌细胞中抑制FOXM1下游靶基因Cyclin D1、MMP9的表达
分别使用0、2.5、5 μmol/L处理上述2种胃癌细胞,采用qRT-PCR检测FOXM1下游靶基因Cyclin D1、MMP9的mRNA表达,结果显示1-3-51呈浓度依赖的抑制Cyclin D1、MMP9 mRNA表达(P < 0.01, 图 2A)。接着使用5 μmol/L 1-3-51处理上述胃癌细胞0、24、48 h,结果显示其同样呈时间依赖的抑制Cyclin D1、MMP9 mRNA表达(P < 0.05,图 2B)。
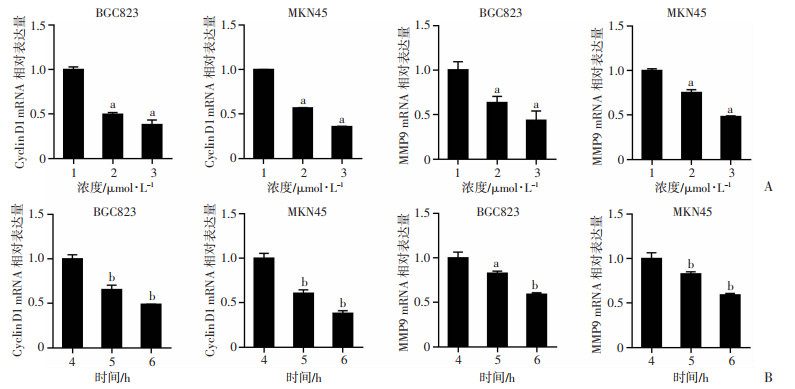
|
|
1:0 μmol/L;2:2.5 μmol/L;3:5 μmol/L;4:0 h;5:24 h;6:48 h A:不同浓度1-3-51处理BGC823、MKN45细胞后FOXM1下游靶基因Cyclin D1及MMP9 mRNA的表达a:P < 0.01, 与0 μmol/L比较;B:不同时间用1-3-51 5 μmol/L处理BGC823、MKN45细胞后FOXM1下游靶基因Cyclin D1及MMP9 mRNA的表达a:P < 0.05, b:P < 0.01, 与0 h比较 图 2 1-3-51在胃癌细胞中降低FOXM1下游靶基因Cyclin D1、MMP9的mRNA水平的表达 |
2.3 小分子抑制剂1-3-51抑制胃癌细胞增殖、迁移及侵袭
为检测小分子抑制剂1-3-51在胃癌进展中的作用,分别使用0、2.5、5 μmol/L处理上述两种胃癌细胞,通过流式细胞术检测其对胃癌细胞周期的影响,结果显示1-3-51呈浓度依赖性的使胃癌细胞阻滞在G1期(图 3A)。然后使用0、5 μmol/L处理上述两种胃癌细胞后,通过Transwell实验验证胃癌细胞的迁移和侵袭能力,结果显示1-3-51处理过的胃癌细胞穿过细胞小室膜的数量较对照组均明显减少(图 3B),提示1-3-51可以显著降低上述两种胃癌细胞的迁移及侵袭能力。以上结果说明,1-3-51可以使胃癌细胞阻滞在G1期,并且抑制胃癌细胞迁移和侵袭。

|
| a:P < 0.05,b:P < 0.01,与0 μmol/L比较; A:流式细胞术测定细胞周期变化;B:Transwell实验检测细胞迁移及侵袭 图 3 检测不同浓度1-3-51对胃癌细胞生长周期、迁移及侵袭的影响 |
2.4 1-3-51在小鼠体内抑制胃癌细胞的增殖
为进一步验证1-3-51在体内的抗胃癌效果,我们建立了异种移植裸鼠模型,在接种BGC823细胞第8天开始测量肿瘤大小,并且1-3-51组采用腹腔注射的方式,1-3-51使用剂量为50 mg/kg,隔天给药,并计算其增殖曲线,在瘤体长到合适大小后处死裸鼠取出瘤体称其质量及拍照(图 4A)。结果显示,用1-3-51处理后瘤体增殖能力显著降低,瘤体大小及质量显著下降(图 4B、C)。以上结果显示1-3-51在体内同样能够抑制胃癌的增殖。
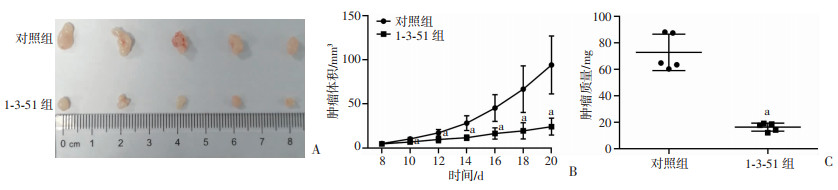
|
| a:P < 0.01,与对照组比较;A:裸鼠体内瘤体大体观察;B:肿瘤的体积变化曲线分析;C:肿瘤的质量变化 图 4 裸鼠移植瘤模型检测1-3-51对胃癌细胞增殖的影响 |
2.5 过表达FOXM1可部分逆转小分子抑制剂1-3-51抑制胃癌细胞增殖、迁移及侵袭的能力
为进一步验证小分子抑制剂1-3-51是否通过抑制FOXM1表达发挥抗胃癌效应,在用5 μmol/L 1-3-51处理胃癌细胞BGC823的同时分别转染FOXM1过表达质粒(oeFOXM1)以及空载质粒,Western blot实验证实过表达FOXM1可回复1-3-51抑制的FOXM1表达(图 5A)。随后,用同样条件处理BGC823 48 h,CCK-8和Transwell实验分别检测细胞活性、迁移和侵袭能力,结果显示回复FOXM1后细胞活性、迁移及侵袭能力也得到回复(图 5B、C)。以上结果显示1-3-51可通过抑制FOXM1从而发挥抗癌效应,过表达FOXM1可部分逆转其效果。
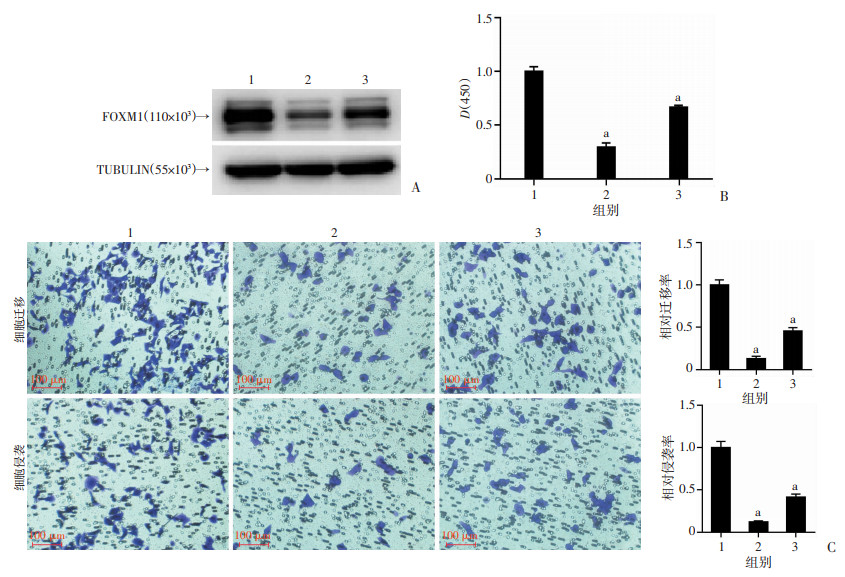
|
|
1:0 μmol/L组;2:5 μmol/L组; 3:5 μmol/L+oeFOXM1组;a:P < 0.0001,与0 μmol/L比较 A:Western blot检测BGC823细胞中1-3-51处理并过表达FOXM1(oeFOXM1)后FOXM1蛋白表达;B:CCK-8检测1-3-51处理并过表达FOXM1后BGC823增殖能力的变化;C:Transwell实验检测1-3-51处理并过表达FOXM1后BGC823迁移及侵袭变化 图 5 检测1-3-51对BGC823细胞中过表达FOXM1后细胞增殖、迁移及侵袭的变化 |
3 讨论
胃癌(gastric cancer GC)是世界上常见的消化系统恶性肿瘤之一,因其高发病率和高死亡率已成为全世界特别是发展中国家人类公共卫生的一个重大问题,严重危害人体健康和浪费社会资源。目前东亚的胃癌发生比例占全球50%左右,其中大部分病例发生在中国。据最新全球癌症统计,胃癌的发病率居世界第5位,死亡率居世界第3位,且呈上升趋势,预计到2030年,胃癌将成为全球15大主要死因之一[18]。尽管近年来胃癌的综合治疗取得一定的进展,但胃癌患者5年总体生存率仍很低,不到30%。究其原因,我国大多数胃癌患者初次诊断时已处于进展期,治疗方法有限[19],主要通过手术结合放化疗方式,且有较高的复发率和转移率[20]。因此,研究新型胃癌靶向治疗药物至关重要,对改善胃癌患者的治疗和预后有着十分重要的意义。
Forkhead家族广泛参与生命发生、发展的过程,如胚胎发生、增殖、分化、凋亡、转化、肿瘤发生、衰老和代谢稳态[21],并且在细胞增殖和细胞周期进程中具有明确的作用,与肿瘤的发生也密切相关[22],其中,FOXM1是一个典型的促增殖转录因子。目前报道FOXM1有4种异构体,分别是FOXM1A、FOXM1B、FOXM1C和FOXM1D。其中,FOXM1A的C末端插入了外显子A2使其失去转录活性[23],而基因序列中不包括外显子A2的FOXM1B、FOXM1C和FOXM1D具有转录活性[24]。研究表明,FOXM1C是一些肿瘤中过表达的转录激活因子,FOXM1D主要位于细胞质中[25],可能促进转移,FOXM1B则主要通过招募共激活因子和转录基本因子共同作用来激活相应的靶基因, 从而在细胞的多种生理和病理活动中发挥作用。近年来研究表明FOXM1B与癌症的发生、发展密切相关, 它不仅能够促进上皮间质转化(EMT)影响癌细胞的迁移、浸润和转移等过程[22, 26], 还与肿瘤耐药、化疗抵抗和患者预后不良相关[27-29]。
近年来大量有关FOXM1小分子抑制剂被发现并且应用于相关肿瘤的治疗,目前已经公认的FOXM1特异性小分子抑制剂天然噻唑类抗生素盐霉素A及与之结构相似的硫链丝菌肽等能够在多种肿瘤细胞及体外移植瘤中下调FOXM1的表达,并抑制下游基因如细胞周期蛋白D1(Cyclin D1)、Aurora B激酶(Aurora B kinase)、CDC25B、存活蛋白(survivin)等的转录活性[9, 29],但目前并未广泛应用于临床研究。有报道FOXM1可通过促进Cyclin D1、MMP9转录促进肿瘤细胞的增殖、侵袭和转移等[13-14, 16]。本实验结果显示,小分子抑制剂1-3-51可抑制FOXM1及其下游靶基因Cyclin D1、MMP9的表达,为寻找新型靶向FOXM1小分子抑制剂提供了新的方向。
随着化疗耐药现象的日趋增加,寻找新的抗肿瘤靶向小分子创新药物已成为靶向药物研究的热点。不同于传统细胞毒药物,靶向药物针对于正常细胞和肿瘤细胞之间的差异,可以达到高选择性、低毒性的治疗效果。但新型小分子靶向药物的开发,一直受到化合物来源的限制。另外,化学合成方法学的研究在源源不断地提供着大量结构新颖的复杂产物,其中不乏许多具有成药潜力的“类药性”骨架结构。但这些分子真正应用于药理研究并进一步开发成药的报道屈指可数。因此,我们对合作课题组前期开展方法学研究得到的磺内酰胺-环己酮螺环类复杂产物的生物学活性进行了普遍评价。发现其中的代表性化合物1-3-51具有较好的抗肿瘤效果,其在胃癌细胞BGC823和MKN45细胞中,1-3-51作用48 h后的IC50值分别为(5.429±0.225) μmol/L、(5.169±0.239) μmol/L,并且在有效作用浓度中可显著降低FOXM1的表达。
综上,我们发现磺内酰胺-环己酮螺环类衍生物1-3-51,可能通过FOXM1抑制人胃癌细胞BGC823、MKN45生长迁移和侵袭,从而发挥抗肿瘤的作用。这为靶向FOXM1这一靶点的新型小分子抑制剂的开发提供了一定的理论基础;也为有机合成方法学研究成果在小分子靶向药物筛选领域的应用提供了一种思路。同时,由于这类化合物结构新颖,与常规商业化合物库、已上市小分子抑制剂之间相似性低,也具有自主知识产权。但调控胃癌细胞生长侵袭和迁移、凋亡的信号通路较多,在后续的实验中我们将进一步阐明小分子抑制剂1-3-51抑制FOXM1的具体分子机制并进行结构优化,为靶向胃癌的新型小分子抑制剂提供新思路。
| [1] |
LAM E W, BROSENS J J, GOMES A R, et al. Forkhead box proteins: tuning forks for transcriptional harmony[J]. Nat Rev Cancer, 2013, 13(7): 482-495. DOI:10.1038/nrc3539 |
| [2] |
KATOH M, KATOH M. Human FOX gene family (Review)[J]. Int J Oncol, 2004, 25(5): 1495-1500. |
| [3] |
SONG X, KENSTON S S F, ZHAO J S, et al. Roles of FoxM1 in cell regulation and breast cancer targeting therapy[J]. Med Oncol, 2017, 34(3): 41. DOI:10.1007/s12032-017-0888-3 |
| [4] |
LUO W, GAO F, LI S, et al. FoxM1 promotes cell proliferation, invasion, and stem cell properties in nasopharyngeal carcinoma[J]. Front Oncol, 2018, 8: 483. DOI:10.3389/fonc.2018.00483.eCollection2018 |
| [5] |
ZHANG Y, QIAO W B, SHAN L. Expression and functional characterization of FOXM1 in non-small cell lung cancer[J]. Oncotargets Ther, 2018, 11: 3385-3393. DOI:10.2147/OTT.S162523 |
| [6] |
CHAND V, PANDEY A, KOPANJA D, et al. Opposing roles of the forkhead box factors FoxM1 and FoxA2 in liver cancer[J]. Mol Cancer Res, 2019, 17(5): 1063-1074. DOI:10.1158/1541-7786.MCR-18-0968 |
| [7] |
SHEN B, ZHOU N F, HU T, et al. LncRNA MEG3 negatively modified osteosarcoma development through regulation of miR- 361-5p and FoxM1[J]. J Cell Physiol, 2019, 234(8): 13464-13480. DOI:10.1002/jcp.28026 |
| [8] |
YAN J, HOU J, YAN Y, et al. Knockdown of FOXM1 suppresses cell growth and metastasis in human laryngeal cancer via the AKT signaling pathway[J]. Eur Rev Med Pharmacol Sci, 2020, 24(12): 6786-6793. DOI:10.26355/eurrev_202006_21667 |
| [9] |
PARASHAR D, NAIR B, GEETHADEVI A, et al. Peritoneal spread of ovarian cancer harbors therapeutic vulnerabilities regulated by FOXM1 and EGFR/ERBB2 signaling[J]. Cancer Res, 2020, 80(24): 5554-5568. DOI:10.1158/0008-5472.CAN-19-3717 |
| [10] |
XU M D, WANG Y Q, WENG W W, et al. A positive feedback loop of lncRNA-PVT1 and FOXM1 facilitates gastric cancer growth and invasion[J]. Clin Cancer Res, 2017, 23(8): 2071-2080. DOI:10.1158/1078-0432.CCR-16-0742 |
| [11] |
PILARSKY C, WENZIG M, SPECHT T, et al. Identification and validation of commonly overexpressed genes in solid tumors by comparison of microarray data[J]. Neoplasia Nov-Dec, 2004, 6(6): 744-750. DOI:10.1593/neo.04277 |
| [12] |
DIBB M, HAN N, CHOUDHURY J, et al. FOXM1 and polo-like kinase 1 are co-ordinately overexpressed in patients with gastric adenocarcinomas[J]. BMC Res Notes, 2015, 8: 676. DOI:10.1186/s13104-015-1658-y |
| [13] |
GUO X, LIU A, HUA H, et al. Siomycin a induces apoptosis in human lung adenocarcinoma A549 cells by suppressing the expression of FoxM1[J]. Nat Prod Commun, 2015, 10(9): 1603-1606. |
| [14] |
KONGSEMA M, WONGKHIEO S, KHONGKOW M, et al. Molecular mechanism of Forkhead box M1 inhibition by thiostrepton in breast cancer cells[J]. Oncol Rep, 2019, 42(3): 953-962. DOI:10.3892/or.2019.7225 |
| [15] |
NAKANO I, JOSHI K, VISNYEI K, et al. Siomycin A targets brain tumor stem cells partially through a MELK-mediated pathway[J]. Neuro Oncol, 2011, 13(6): 622-634. DOI:10.1093/neuonc/nor023 |
| [16] |
NEWICK K, CUNNIFF B, PRESTON K, et al. Peroxiredoxin 3 is a redox-dependent target of thiostrepton in malignant mesothelioma cells[J]. PLoS ONE, 2012, 7(6): e39404. DOI:10.1371/journal.pone.0039404 |
| [17] |
SIEGEL R L, MILLER K D, FUCHS H E, et al. Cancer Statistics, 2021[J]. CA Cancer J Clin, 2021, 71(1): 7-33. DOI:10.3322/caac.21654 |
| [18] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [19] |
徐惠绵, 王鑫. 我国胃癌诊治临床研究现状与展望[J]. 中华胃肠外科杂志, 2020, 23(2): 109-114. XU H M, WANG X. Current status and prospects of clinical research on diagnosis and treatment of gastric cancer in China[J]. Clin J Gastrointest Surg, 2020, 23(2): 109-114. DOI:10.3760/cma.j.issn.1671-0274.2020.02.003 |
| [20] |
CERVANTES A, RODA D, TARAZONA N, et al. Current questions for the treatment of advanced gastric cancer[J]. Cancer Treat Rev, 2013, 39(1): 60-67. DOI:10.1016/j.ctrv.2012.09.007 |
| [21] |
KALIN T V, USTIYAN V, KALINICHENKO V V. Multiple faces of FoxM1 transcription factor: lessons from transgenic mouse models[J]. Cell Cycle Georget Tex, 2011, 10(3): 396-405. DOI:10.4161/cc.10.3.14709 |
| [22] |
KIM I M, ACKERSON T, RAMAKRISHNA S, et al. The Forkhead Box m1 transcription factor stimulates the proliferation of tumor cells during development of lung cancer[J]. Cancer Res, 2006, 66(4): 2153-2161. DOI:10.1158/0008-5472.CAN-05-3003 |
| [23] |
MYATT S S, LAM E W F. The emerging roles of forkhead box (Fox) proteins in cancer[J]. Nat Rev Cancer, 2007, 7(11): 847-859. DOI:10.1038/nrc2223 |
| [24] |
LVSCHER-FIRZLAFF J M, LILISCHKIS R, LVSCHER B. Regulation of the transcription factor FOXM1c by Cyclin E/CDK2[J]. FEBS Lett, 2006, 580(7): 1716-1722. DOI:10.1016/j.febslet.2006.02.021 |
| [25] |
ZHANG X, ZHANG L, DU Y, et al. A novel FOXM1 isoform, FOXM1D, promotes epithelial-mesenchymal transition and metastasis through ROCKs activation in colorectal cancer[J]. Oncogene, 2017, 36(6): 807-819. DOI:10.1038/onc.2016.249 |
| [26] |
ZHANG J, CHEN X Y, HUANG K J, et al. Expression of FoxM1 and the EMT-associated protein E-cadherin in gastric cancer and its clinical significance[J]. Oncol Lett, 2016, 12(4): 2445-2450. DOI:10.3892/ol.2016.4917 |
| [27] |
CARR J R, PARK H J, WANG Z B, et al. FoxM1 mediates resistance to herceptin and paclitaxel[J]. Cancer Res, 2010, 70(12): 5054-5063. DOI:10.1158/0008-5472.CAN-10-0545 |
| [28] |
WANG L L, LIU J, LIU J G, et al. GLRX inhibition enhances the effects of geftinib in EGFR-TKI-resistant NSCLC cells through FoxM1 signaling pathway[J]. J Cancer Res Clin Oncol, 2019, 145(4): 861-872. DOI:10.1007/s00432-019-02845-y |
| [29] |
HU C J, WANG B, TANG B, et al. The FOXM1-induced resistance to oxaliplatin is partially mediated by its novel target gene Mcl-1 in gastric cancer cells[J]. Biochim Biophys Acta, 2015, 1849(3): 290-299. DOI:10.1016/j.bbagrm.2014.11.008 |




