2. 300211 天津,天津医科大学第二医院天津市泌尿外科研究所肿瘤免疫室
2. Department of Tumor Immunology, Tianjin Institute of Urology, Second Hospital of Tianjin Medical University, Tianjin, 300211, China
膀胱尿路上皮癌(bladderurothelial carcinoma,BUC)是膀胱癌中最常见的类型,具有高发病率、高复发率和高病死率的临床特征,严重威胁着患者的健康与生命。肿瘤标志物监测作为一种简便,无创,快捷的检测手段,广泛应用于肿瘤筛查、诊断、预后评估、疗效监测、复发预测等领域;而比较蛋白质组学研究被认为是发现肿瘤标志物的有效手段[1]。近年来同位素标记相对和绝对定量(isobaric tags for relative and absolute quantitation,iTRAQ)技术由于其高通量高定量精度的优点,被广泛应用于多种疾病的比较蛋白质组学研究中[2]。本研究采用iTRAQ技术对BUC患者和对照组的尿液标本进行比较蛋白质组学研究,应用质谱多反应监测(multiple reaction monitoring, MRM)技术对部分差异蛋白进行验证,并使用R语言进行生物信息学分析,构建BUC尿液差异蛋白数据集。通过蛋白互作网络分析进一步筛选出核心差异蛋白,用酶联免疫吸附试验(enzyme-linked immunosorbent assay,ELISA)进行验证,为BUC的诊断及治疗提供新的靶点。
1 资料与方法 1.1 标本来源收集2015年1月至2016年11月于天津医科大学第二医院泌尿外科住院病房采集BUC患者术前尿液标本78例,其中男性55人,女性23人,年龄(68.9±6.6)岁。取样前患者均未行膀胱镜检、手术、化疗、放疗等有创检查及治疗,排除了肾脏感染、炎症等慢性疾病,临床标本的使用均符合医学伦理要求。收集患者临床资料包括年龄、性别、病理诊断等。同时收集51例门诊查体健康人群尿液标本作为对照组,对照组无任何疾病征象或绝对无泌尿系统肿瘤病史及征象,其中男性34人,女性17人,年龄(60.6±11.0)岁。两组性别年龄相匹配。
用洁净容器收集受检者晨起中段尿液20 mL,1 500×g离心5 min去掉尿液沉渣, 尿液上清保存于-80 ℃冰箱。
1.2 iTRAQ技术联合质谱分析查找BUC尿液差异蛋白BUC组每5例样本等体积(20 mL)混合,获得4例BUC组混合样本T1、T2、T3和T4;对照组尿液标本中每5例样本随机等体积(20 mL)混合,获得2例对照组混合样本N1和N2。尿液蛋白提取后,用Bradford法进行蛋白定量;酶解后的肽段用StrataX柱进行除盐,真空抽干。用0.5 mol/L TEAB溶解肽段样品,并加到对应ITRAQ标签试剂中。不同样品肽段选用不同的ITRAQ标签;室温静置2 h。采用岛津LC-20AB液相系统进行肽段分离,结合色谱洗脱峰图合并样品得到20个组分,然后冷冻抽干。将抽干的肽段样品用2% 乙腈/0.1%甲酸复溶,20 000×g离心10 min后取上清,进样至岛津LC-20AD型纳升液相色谱仪中,样品首先进入trap柱富集并除盐,随后进入C18柱(7.5 mm×150 mm,3.6 μm)进行分离;分离后的肽段直接进入到ESI串联质谱仪Triple TOF 5600 (SCIEX, Framingham, MA, USA)进行分析:一级质谱扫描累积时间为250 ms,扫描质量范围为350~1 500 Da;从一级质谱中选择强度超过150 cp的前30进行二级质谱的扫描,累积时间为100 ms,扫描质量范围为350~1 500 Da。利用蛋白质鉴定软件Mascot 2.3.02对鉴定出的肽段进行搜索,对比数据库为SwissProt (Homo_sapieens, 2016-11-15),以Fold change >1.2,且P < 0.05为筛选条件,最终筛选出两组间显著性差异的蛋白。
1.3 MRM验证差异倍数排名前20的差异蛋白用于后续MRM验证。尿液样品包括对照组(N)5例,BUC组(T)10例。尿液蛋白提取后,经酶解、脱盐等处理,进样至岛津LC-20AD型纳升液相色谱仪中,样品首先进入trap柱富集并除盐,随后进入C18柱(7.5 mm×150 mm,3.6 μm)进行分离;分离后的肽段直接进入到ESI串联质谱仪QTRAP55000 (SCIEX, Framingham, MA, USA)进行分析;质谱仪的参数设置为离子源喷雾电压2 400 V,喷雾气为23。以表达差异倍数大于1.5倍为筛选条件,结合蛋白功能,选择16个目标差异蛋白,进行MRM验证;利用Skyline软件提取备选目标蛋白在质谱扫描的原始信息进行定量信息分析,利用MSstats工具包整合的线性混合模型进行目标蛋白在样品中的相对定量,统计分析后给出蛋白在比较组的比值以及校正P值(adjusted p-value),在1.5倍差异且校正P < 0.05的条件下认为是显著差异蛋白。
1.4 基于R语言的生物信息学分析应用R语言(4.0版)的ggplot2数据包构建火山图来检测蛋白标准化是否合格;应用R语言的cluster profiler数据包和enrichplot数据包等进行京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)富集分析,提取前10个生物过程及包含的蛋白;应用R语言的org.Hs.eg.db包等,进行基因本体(Gene ontology,GO)分析,对差异蛋白进行功能注释,选取具有统计学差异的前20个生物过程及其所包含的蛋白。
1.5 BUC尿液差异蛋白数据集的构建及核心差异蛋白筛选选取MRM验证的差异蛋白、KEGG富集分析及GO分析结果中具有统计学差异的生物过程包含的蛋白,共同构建BUC尿液差异蛋白数据集。应用STRINGdb对数据集中的差异蛋白进行相互作用(Protein Protein Interaction, PPI)分析,选择位于节点数高的蛋白作为核心差异蛋白,用于后续研究。
1.6 ELISA定量检测核心差异蛋白尿液样品包括对照组36例,BUC组48例。ELISA检测按照试剂盒说明进行:将倍比稀释的标准品各100 μL依次加入一排7孔板中,1孔只加样品稀释液作为对照;尿液标本1 500 r/min离心5 min去除细胞及沉渣,处理后的标本按100 μL每孔加入,每例3个复孔;37 ℃反应90 min,甩干并洗板两次;生物素抗体工作液按每孔100 μL依次加入,37 ℃反应60 min,甩干并洗板3次;将准备好的ABC工作液按每孔100 μL依次加入,37 ℃反应30 min,甩干并洗板5次;每孔依次加入的TMB显色100 μL,37 ℃避光反应10 min;用酶标仪在450 nm测定D值,通过D(450)值计算出对应的浓度。应用R语言的t.test函数进行两独立样本t检验,P < 0.05表示差异有统计学意义。
2 结果 2.1 蛋白鉴定结果在iTRAQ定量项目中共有8 828条肽段和1 949个蛋白被鉴定到,差异蛋白为101个,其中在BUC组表达上调的蛋白37个,表达下调的蛋白64个。为了全面展示被测蛋白的组间差异情况,绘制了显著差异蛋白的火山图(图 1),图中绿色点为显著上调蛋白,红色点为显著下调蛋白,蓝色点为无显著变化蛋白。与对照组相比,差异倍数排名前10的显著上调和显著下调的差异蛋白见(表 1)。针对这20个蛋白进行MRM验证分析。
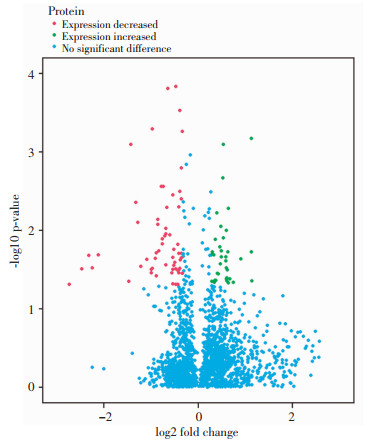
|
| 图 1 显著差异蛋白火山图 |
| UniProt编号 | 蛋白名 | 浓度变化倍数 | UniProt编号 | 蛋白名 | 浓度变化倍数 |
| P01023 | A2MG | 8.76 | O00754 | MA2B1 | 0.05 |
| P15924 | DESP | 2.2 | P01601 | KVD16 | 0.05 |
| P02649 | APOE | 2.19 | O00187 | MASP2 | 0.08 |
| P06753 | TPM3 | 2.18 | Q8IV08 | PLD3 | 0.11 |
| P06727 | APOA4 | 1.87 | P16278 | GLB1 | 0.15 |
| Q96FW1 | OTUB1 | 1.67 | P08195 | 4F2 | 0.18 |
| P17213 | BPI | 1.59 | Q9NRX4 | PHP14 | 0.20 |
| Q8TAB 3 | PCD19 | 1.56 | P48061 | SDF1 | 0.21 |
| P62263 | RS14 | 1.56 | Q5JRA6 | MIA3 | 0.23 |
| Q9QC07 | POK18 | 1.55 | P98095 | FBLN2 | 0.36 |
| BUC: 膀胱尿路上皮癌; T: BUC组; N:对照组 | |||||
2.2 MRM验证分析结果
为了验证ITRAQ的定量数据,选择表 1中的差异蛋白进行MRM验证,在iTRAQ中检测得到的20个差异蛋白中,MRM成功检测到10个,见表 2。表中分列标记有蛋白名称、比较组别、浓度变化以及统计检验的P值。这10个差异蛋白入选BUC尿液差异蛋白数据集,进行蛋白相互作用分析。
| 蛋白名 | 组间比 | 浓度变化倍数 | P |
| A2MG | T/N | 4.941 | 0.03 |
| APOE | T/N | 2.632 | 0.01 |
| APOA4 | T/N | 2.139 | 0.01 |
| FBLN2 | T/N | 0.287 | 0.00 |
| MIA3 | T/N | 0.227 | 0.00 |
| SDF1 | T/N | 0.216 | 0.02 |
| 4F2 | T/N | 0.181 | 0.02 |
| PHP14 | T/N | 0.176 | 0.00 |
| PLD3 | T/N | 0.113 | 0.00 |
| MASP2 | T/N | 0.063 | 0.02 |
| MRM: 质谱多反应监测 | |||
2.3 差异蛋白的KEGG富集分析
与iTRAQ和MRM着眼于蛋白的浓度变化不同,富集分析旨在通过发掘出现基因富集的生物过程寻找适宜的生物标志物。通过对差异蛋白进行KEGG通路分析发现了差异蛋白广泛参与的前10个生物过程,结果见图 2,其中P < 0.05的为差异蛋白显著富集的代谢通路,通路名称为:其他聚糖降解, 补体和凝血级联, 包含11个蛋白,入选BUC尿液差异蛋白数据集,留待进行蛋白相互作用分析。
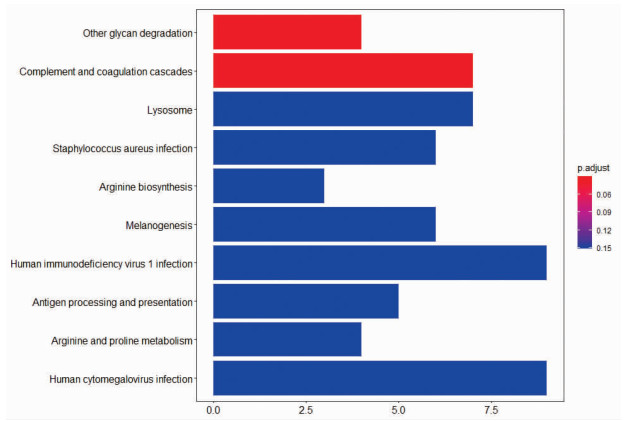
|
| 图 2 差异蛋白的KEGG富集分析(KEGG: 京都基因与基因组百科全书) |
2.4 差异蛋白的GO功能注释
经GO分析发现差异蛋白主要参与细胞黏附分子结合、羧酸结合、有机酸结合、糖胺聚糖结合、肽链内切酶激活、腺苷酸环化酶的结合、氨基多糖结合细胞因子、丝氨酸型肽链内切酶抑制剂激活通路等生物过程详见图 3。图中涉及的差异蛋白共54个,入选BUC尿液差异蛋白数据集,留待进行蛋白相互作用分析。
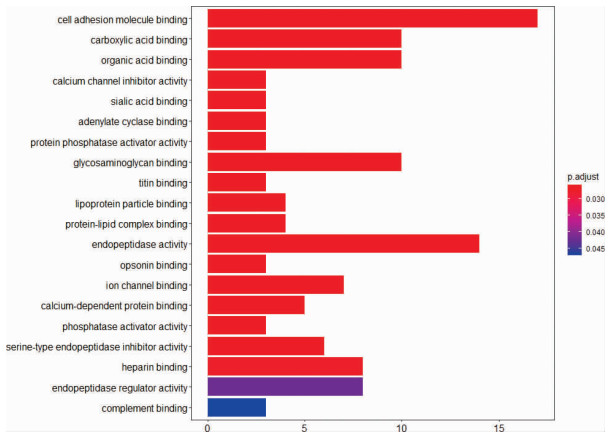
|
| 图 3 差异蛋白的GO功能注释(GO: 基因本体论) |
2.5 BUC尿液差异蛋白数据集及核心差异蛋白
选取MRM验证中出现10个差异蛋白、在KEGG富集分析结果中出现的11个差异蛋白和GO分析中出现的54个差异蛋白,共同构建由69个(6个重复蛋白)差异蛋白构成的BUC尿液差异蛋白数据集。将69个差异蛋白输入STRING数据库,进行蛋白相互作用分析。结果(图 4)所示:APOE与10个蛋白有关联,APOA4与6个蛋白有关联,以上2蛋白为与其他蛋白关联最多的蛋白,因此我们选择APOE和APOA4作为核心差异蛋白进行ELISA验证。
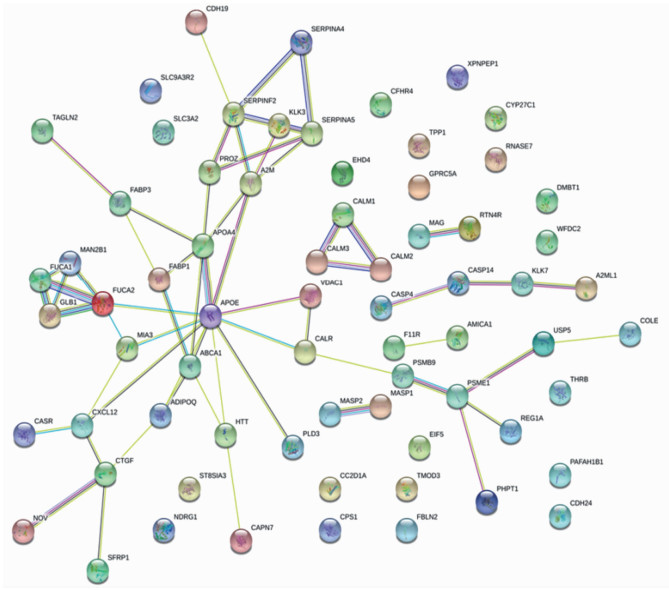
|
| 图 4 蛋白质交互作用图 |
2.6 ELISA定量检测BUC尿液核心差异蛋白
我们通过ELISA对两组样本中核心差异蛋白——APOE和APOA4的含量进行了进一步验证分析。与对照组相比,BUC组尿液中APOE和APOA4浓度均增高。经两独立样本t检验,差异有显著性(P < 0.05,表 3)。说明APOE和APOA4的浓度在BUC病患和健全人群的尿液中存在差异。
| 组别 | n | APOE(pg/mL) | APOA4 (ng/mL) |
| 对照组 | 36 | 0.30±0.10 | 3.33±1.20 |
| BUC组 | 48 | 0.55±0.12a | 7.01±1.71a |
| a:P < 0.05, 与对照组比较 | |||
3 讨论
本研究采用ITRAQ-MRM-R语言策略,构建了由69个差异蛋白构成的BUC尿液差异蛋白数据集,通过PPI分析得到APOE以及APOA4这两个核心差异蛋白,并采用ELISA验证了APOE和APOA4均在BUC组尿液中浓度显著增高。
MRM技术是一种基于已知信息或假定信息进行质谱信号采集的技术,具有灵敏度高、重现性好、准确率高和通量高等优点。对于iTRAQ获得的大量结果,用MRM进行验证,更能满足蛋白质组学的研究需求。R语言的enrichplot,org.Hs.eg.db,stringdb等相关生物信息分析包可以对iTRAQ结果进行差异蛋白富集分析、KEGG通路分析、GO分析、蛋白间相互作用分析等。ITRAQ-MRM-R语言三者结合有效的筛选出BUC尿液差异蛋白。
本研究对差异蛋白进行KEGG通路分析发现BUC差异蛋白显著富集于其他聚糖降解, 补体和凝血级联;GO分析发现差异蛋白主要参与细胞黏附分子结合、羧酸结合、有机酸结合、糖胺聚糖结合、肽链内切酶激活、腺苷酸环化酶的结合、氨基多糖结合细胞因子、丝氨酸型肽链内切酶抑制剂激活通路等生物过程。
富集分析中提示的生物通路与癌症的直接或间接关系尚不明确。但若干通路与癌症的发生、发展过程存在的潜在联系可见于在现存文献。例如LI等[3]报道在关于胆管癌生物标志物的研究中,补体与凝血过程同样出现在pathway分析的结果中。CARAYON等[4]在对甲状腺癌的研究中提示腺苷酸环化酶的结合功能与甲状腺癌分型可能存在关联。MURAMATSU等[5]指出在多种恶性肿瘤患者的血清、血浆中,都能观察到氨基多糖结合细胞因子的增加。SANDRA等[6]在关于恶性胰岛素肿瘤的生物标志物研究中发现丝氨酸型肽链内切酶抑制剂激活通路出现富集等等。
APOE基因涉及多种生物过程,其中最主要的功能是参与脂质、胆固醇的代谢和转运[7]。过往的研究发现其与阿尔兹海默症、心血管疾病、脑部炎症、精神分裂症[7-9]等多种疾患存在关联。已有研究发现胃癌、非小细胞肺癌、直肠癌等癌症,都与APOE基因表达存在关联[10-12]。APOE作为BUC的尿液差异表达蛋白被检测到的原因,可能源自其对非特异性免疫的抑制作用[13-14]。提示BUC的发生、发展过程中可能存在APOE引导的非特异性免疫抑制的发生。
APOA4编码构成淋巴细胞和血浆中乳糜微粒的主要成分[15]。现存的研究结果提示其与糖尿病肾病、阿尔兹海默症[16-17]等疾病存在关联。此外,APOA4曾同样作为BUC的生物标志物,在SOUKUP等[18]的研究中被发现,这与本次研究的成果相呼应。
受研究条件局限,本次研究的样本来源较为单一,所有标本收集自天津医科大学第二医院泌尿外科住院部。这使得本次研究的观察范围局限于居住于华北地区的东亚细亚人种的中老年住院病患。因iTRAQ检测费用较高,用于iTRAQ的样本量较小,为此我们采用混合样本的方法减小组内样本间的差异,并在后续MRM验证及ELISA验证过程中逐渐增大样本量,取得了可信的结果。本研究结果提示APOE和APOA4具有BUC诊断的潜在价值,在今后的研究中,我们将会尝试扩大样本量和样本来源,增加样本的年龄、人种、居住地等特征的异质性, 使其更具代表性;或通过综述性研究,对同类小样本研究结果进行总结归纳,得出更可靠的结论。
| [1] |
王月坤, 刘芃昊, 王裕, 等. 单细胞测序及其与未来脑恶性肿瘤的诊疗[J]. 协和医学杂志, 2020, 11(5): 606-614. WANG Y K, LIU P H, WANG Y, et al. Single-cell sequencing and its prospect in the management of brain malignant tumor[J]. Med J Peking UMCH, 2020, 11(5): 606-614. DOI:10.3969/j.issn.1674-9081.2020.05.018 |
| [2] |
WIESE S, REIDEGELD K A, MEYER H E, et al. Protein labeling by iTRAQ: a new tool for quantitative mass spectrometry in proteome research[J]. Proteomics, 2007, 7(3): 340-350. DOI:10.1002/pmic.200600422 |
| [3] |
LI H Y, LONG J Y, XIE F C, et al. Transcriptomic analysis and identification of prognostic biomarkers in cholangiocarcinoma[J]. Oncol Rep, 2019, 42(5): 1833-1842. DOI:10.3892/or.2019.7318 |
| [4] |
CARAYON P, THOMAS-MORVAN C, CASTANAS E, et al. Human thyroid cancer: membrane thyrotropin binding and adenylate cyclase activity[J]. J Clin Endocrinol Metab, 1980, 51(4): 915-920. DOI:10.1210/jcem-51-4-915 |
| [5] |
MURAMATSU T, MURAMATSU H. Glycosaminoglycan-binding cytokines as tumormarkers[J]. Proteomics, 2008, 8(16): 3350-3359. DOI:10.1002/pmic.200800042 |
| [6] |
DE SÁ S V, CORRÊA-GIANNELLA M L, MACHADO M C, et al. Serpin peptidase inhibitor clade A member 1 as a potential marker for malignancy in insulinomas[J]. Clin Cancer Res, 2007, 13(18 Pt 1): 5322-5330. DOI:10.1158/1078-0432.CCR-06-1477 |
| [7] |
YIN Y M, WANG Z. ApoE and neurodegenerative diseases in aging[J]. Adv Exp Med Biol, 2018(1086): 77-92. DOI:10.1007/978-981-13-1117-8_5 |
| [8] |
FORERO D A, LOPEZ S, GONZALEZ G Y, et al. APOE gene and neuropsychiatric disorders and endophenotypes: A comprehensive review[J]. Am J Med Genet B Neuropsychiatr Genet, 2018, 177(2): 126-142. DOI:10.1002/ajmg.b.32516 |
| [9] |
REBECK G W. The role of APOE on lipid homeostasis and inflammation in normalbrains[J]. J Lipid Res, 2017, 58(8): 1493-1499. DOI:10.1194/jlr.r075408 |
| [10] |
SAKASHITA K, TANAKA F, ZHANG X, et al. Clinical significance of ApoE expression in human gastric cancer[J]. Oncol Rep, 2008, 20(6): 1313-1319. |
| [11] |
AN H J, KOH H M, SONG D H. Apolipoprotein E is a predictive marker for assessing non-small cell lung cancer patients with lymph node metastasis[J]. Pathol Res Pract, 2019, 215(10): 152607. DOI:10.1016/j.prp.2019.152607 |
| [12] |
SLATTERY M L, SWEENEY C, MURTAUGH M, et al. Associations between apoE genotype and colon and rectal cancer[J]. Carcinogenesis, 2005, 26(8): 1422-1429. DOI:10.1093/carcin/bgi088 |
| [13] |
ZHENG P M, LUO Q, WANG W W, et al. Tumor-associated macrophages-derived exosomes promote the migration of gastric cancer cells by transfer of functional apolipoprotein E[J]. Cell Death Dis, 2018, 9(4): 434. DOI:10.1038/s41419-018-0465-5 |
| [14] |
TAVAZOIE M F, POLLACK I, TANQUECO R, et al. LXR/ApoE activation restricts innate immune suppression in cancer[J]. Cell, 2018, 172(4): 825-840. DOI:10.1016/j.cell.2017.12.026 |
| [15] |
LAMINA C, FRIEDEL S, COASSIN S, et al. A genome-wide association meta-analysis on apolipoprotein A-IV concentrations[J]. Hum Mol Genet, 2016, 25(16): 3635-3646. DOI:10.1093/hmg/ddw211 |
| [16] |
CHENG C W, CHANG CC, CHEN H W, et al. Serum ApoA4 levels predicted the progression of renal impairment in T2DM[J]. Eur J Clin Invest, 2018, 48(6): e12937. DOI:10.1111/eci.12937 |
| [17] |
LIN Q, CAO Y P, GAO J. Decreased expression of the APOA1-APOC3-APOA4 gene cluster is associated with risk of Alzheimer's disease[J]. Drug Des Devel Ther, 2015, 9: 5421-5431. DOI:10.2147/DDDT.S89279 |
| [18] |
SOUKUP V, CAPOUN O, PESL M, et al. The significance of calprotectin, CD147, APOA4 and DJ-1 in non-invasive detection of urinary bladder carcinoma[J]. Neoplasma, 2019, 66(6): 1019-1023. DOI:10.4149/neo_2019_190124N74 |




