DNA是人类遗传信息的主要载体,其中碱基是编码遗传信息的核心单位。然而DNA时刻受到内外源性致伤因子,如活性氧(reactive oxygen species, ROS)、氧化剂及紫外光照射等的攻击,而碱基是最常受到损伤的部位。常见的碱基损伤,特别是氧化性和烷化性碱基损伤就有数十种之多,例如其中最常见的8-氧鸟嘌呤(8-oxoguanine, 8-oxo-G),在人类外周血淋巴细胞中大约每106个鸟嘌呤中可以检测到2~3个8-oxo-G[1]。而在人体细胞中,由各种DNA碱基损伤脱落形成的脱嘌呤脱嘧啶位点(apurinic-apyrimidinic site/AP位点)每天产生约10 000个,远超其他类型的DNA损伤[2]。
DNA碱基损伤及AP位点的修复主要由碱基切除修复(base excision repair, BER)完成[2]。BER一般由识别DNA碱基损伤的特异性糖基化酶,例如8-氧鸟嘌呤糖基化酶(8-oxoguanine DNA glycosylase, OGG1)起始作用并形成AP位点[3-5],紧接着由共同通路的AP核酸内切酶(apurinic/apyrimidinic endonuclease1, APE1)募集下游修复酶对AP位点进行修复,并通过DNA聚合酶β(DNA polymerase beta, DNA pol β)恢复其正确的碱基配对。BER对常规的B型构象DNA双链上的碱基损伤和AP位点具有极高的修复活性,然而,我们和其他团队的前期研究发现:BER的核心限速酶APE1对多种非B型构象DNA上的AP位点修复活性具有显著差异[6]。因此,我们推测这种修复活性的差异会导致基因组里存在碱基损伤修复相对较弱的区域,导致AP位点在区域内聚集形成“热点区域”。多项前期研究发现8-oxo-G在人类基因组中同样呈现非随机分布的形式,具有一定个体差异然而又相对保守,提示DNA损伤聚集具有倾向性,被称为基因组的“疤痕”[1, 7-8]。
基因组“疤痕”现象的生物学意义直到最近才逐渐被理解,最新研究表明DNA氧化性碱基损伤可能是一种新的表观遗传学标志物。例如,8-oxo-G位置不同,其对基因表达的影响具有截然不同的作用,其中位于基因编码链上的8-oxo-G可导致基因转录终止[9],但如果位于基因上游启动子或者5′UTR区域,则很有可能促进下游基因的表达。因此,DNA碱基损伤可能代表重要的表观遗传学机制,但目前还缺乏对这些碱基损伤热点区域的全基因组全景描绘,更无法知晓其分布规律及序列特征。本研究应用染色质免疫沉淀技术(chromatin immunoprecipitation, ChIP)[10],建立可精确描述全基因组水平BER蛋白稳态结合的目的DNA片段,通过分析APE1和OGG1结合位点序列特征,初探DNA碱基损伤富集规律,为进一步阐明DNA碱基损伤的表观遗传学分子机制奠定基础。
1 材料与方法 1.1 细胞来源和培养非小细胞肺癌A549细胞株来源于本科实验室保种细胞(经STR细胞鉴定)。细胞在DMEM培养基于5%CO2的37 ℃孵箱中培养。
1.2 主要试剂及仪器包括:染色质免疫沉淀偶联测序(ChIP-seq)试剂盒(#56383, CST, 美国),APE1抗体(ab194, abcam, 美国),OGG1抗体(NB100-106, Novus Biologicals, 美国),16%甲醛(Methanol-Free 12606S, CST, 美国);超声破碎仪(非探头接触式)(Diagenode BioruptorTM UCD-200, 比利时),震荡恒温金属浴(TUS-200P, 上海一恒科学仪器有限公司)。
1.3 A549细胞ChIP实验配置含蛋白酶抑制剂的PBS溶液[每10毫升PBS中加50 μL PIC(protease inhibitor cocktail)溶液,混匀,置冰上];向含10 mL培养基的皿中加入666 μL 16%的甲醛溶液(甲醛终浓度1%),混匀,室温染色质交联10 min;然后加入1.18 mL 10×甘氨酸溶液混匀,终止交联;弃去培养基后,用冰冷PBS溶液冲洗2次;加入2 mL含蛋白酶抑制剂的PBS溶液,刮取细胞并收集在15 mL离心管中,4 ℃下1 000×g离心收集细胞沉淀;将细胞按2.0×107加入1 mL的细胞裂解液,冰上裂解10 min后,4 ℃ 5 000×g离心10 min,重复进行细胞裂解1次;加入1 mL预冷的细胞核裂解液裂解细胞核,冰上裂解10 min后,通过超声波将染色质随机切割至200~500 bp;利用APE1抗体和OGG1抗体特异性免疫沉淀反应,抗体用量:对于1份IP样本DNA含量10 μg(相当于4×106个A549细胞的DNA含量),每份DNA样本对应的阳性抗体[Histone H3(D2B12)XP® Rabbit mAb, CST, 美国]2.7 μg、阴性抗体(Normal Rabbit IgG 2729, CST, 美国)2 μg、APE1抗体(ab194,abcam,美国)4.5 μg和OGG1抗体(NB100-106, Novus, 美国)5 μg。将与之相结合的DNA片段免疫沉淀下来,解交联后纯化目的DNA。
1.4 A549细胞ChIP样品测序及生物学分析使用Qubit® DNA检测试剂盒在Qubit® 2.0中测定DNA浓度荧光计(Life Technologies, CA, USA)。将纯化的DNA用于ChIP-seq文库制备。文库由诺禾致源(中国北京)建立。随后,在Illumina平台(Illumina, CA, USA)对样品进行对端测序。高通量测序(Illumina HiSeqTM)得到的原始图像数据文件经CASAVA碱基识别(base calling)分析转化为原始测序序列(sequenced reads),进一步通过fastp(version 0.19.11)软件进行处理[11],得到清洁数据。每个IP样本测序数据量为9 G。用BWA mem(version 0.7.12)将读取数据映射到参考基因组后,使用MACS2(version 2.1.0)峰值调用软件来识别背景上IP富集的区域。ChIP效率的判断用FRiP(fraction of reads in peaks):目的蛋白结合峰中reads的个数占所有比对上的reads总数的百分比,FRiP可以简单直观地反映ChIP实验的效果,一般情况下,FRiP的值与蛋白结合峰的数目呈正比。RPM值:单个样品中1 M的reads富集到结合峰中的比例。FoldEnrich值:IP中RPM值与Input中RPM值的比值对峰进行聚类分析。使用Homer检测全新序列模体(Motif)和已知匹配模体[12]。使用ChIPseeker检索峰附近最近的基因,并注释峰的基因组区[13]。使用KOBAS软件分析KEGG(http://www.genome.jp/kegg)通路峰相关基因的统计富集性。采用PeakAnnotator检测[14]结合峰相对于转录起始点(transcription start sites, TSS)位点的分布特点。
2 结果 2.1 APE1与OGG1蛋白在全基因组范围内结合峰的分布情况由于DNA修复酶与底物结合的高度特异性,APE1结合峰可代表基因组AP位点,而OGG1结合峰可代表基因组8-oxo-G位点,因此,通过对比两者结合峰可以了解基因组中全部DNA碱基损伤和特异性氧化性DNA碱基损伤的分布。首先,通过分析APE1与OGG1在A549细胞中的全基因组结合峰的分布特点,探索DNA碱基损伤(BER修复蛋白底物)是否存在富集而非随机分布。
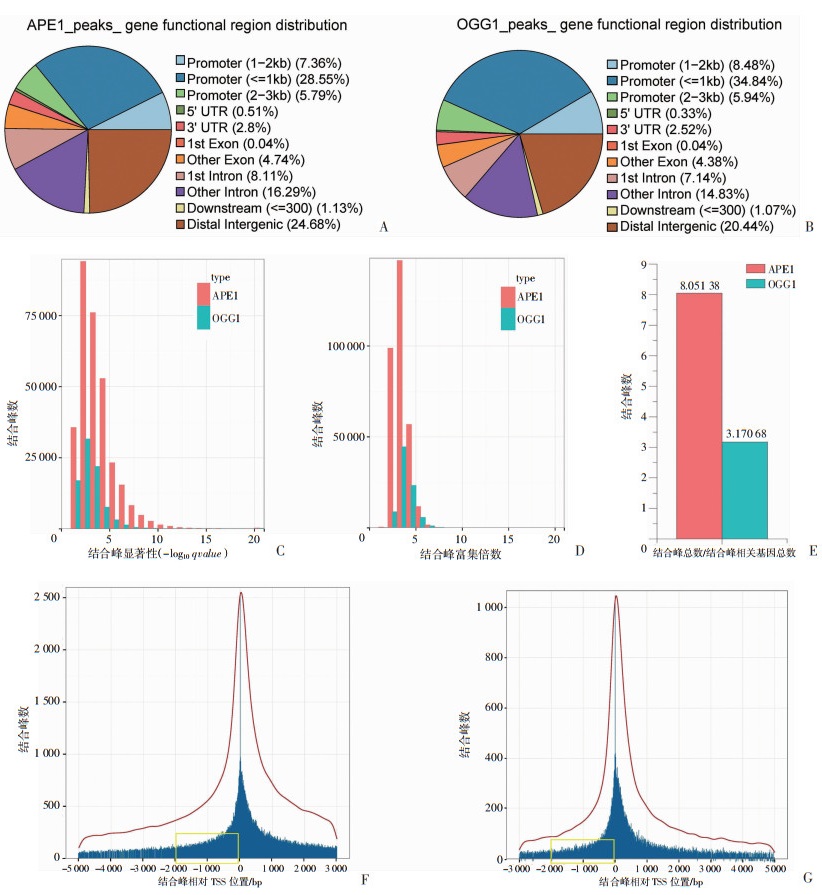
A549细胞中,APE1和OGG1结合峰主要分布在基因启动子区,所占比例分别为41.70%和49.26%,其中APE1结合在启动子区(≤1 kb、>1~2 kb、>2~3 kb)区的比例分别为7.36%、28.55%、5.79%(图 1A),OGG1结合在启动子区(≤1 kb、>1~2 kb、>2~3 kb)区的比例分别为8.48%、34.84%、5.94%(图 1B)。但仍然有24.68%的AP位点和20.44%的8-oxo-G位点分布于非基因功能区,考虑到全基因组中基因编码区结合峰所占比例很低,因此,该结果提示AP位点和8-oxo-G位点呈基因启动子区富集分布的特征,而非呈随机分布。从两个蛋白结合峰的-log10 qvalue值和Peak enrichment值来看(图 1C、D),APE1明显比OGG1在基因组上富集的显著程度和富集倍数要高,说明APE1相对于OGG1更易与基因组结合,且结合数量更多,更快速地发挥相应的调控功能。通过对比发现,APE1平均在每个基因上的结合峰数明显多于OGG1(APE1∶OGG1=8.05∶3.17)(图 1E)。APE1结合峰相对于TSS的分布主要集中在-5 000~3 000 bp,在TSS坐标0处的最大峰数为2 500,基因功能调控区TSS上游2 000 bp结合峰占比38.80%,TSS上游5 000 bp结合峰占比41.98%(图 1F)。OGG1结合峰相对于TSS的分布主要集中在-3 000~5 000 bp,TSS坐标0处最大峰数为1 000,基因功能调控区TSS上游2 000 bp结合峰占比例39.80%;TSS下游5 000 bp结合峰比例为53.88%(图 1G)。
|
| A、B:分别为APE1、OGG1蛋白结合峰基因功能区分布;C、D:分别为蛋白结合峰富集性(显著性、富集倍数)分析;E:APE1和OGG1平均每个基因上的结合峰数量;F、G:分别为APE1、OGG1蛋白结合峰相对TSS位置分析黄色框示TSS上游2 000区域 图 1 BER通路APE1与OGG1蛋白结合峰的分布特征分析 |
2.2 APE1与OGG1蛋白结合模体(motif)分析
利用Homer软件分别识别出APE1结合的motif保守序列50个,OGG1结合的motif保守序列43个。APE1和OGG1结合motif代表蛋白结合位置的序列保守性,表 1、2中分别列出5个最显著motif序列及相关参数。APE1结合的motif最佳匹配转录因子(transcription factors, TF)包括Dof2、TCP19、ZNF165等。进一步分析发现:这些转录因子多与细胞生长和发育调节、细胞周期调控、细胞因子激活相关。OGG1结合的motif序列最佳匹配转录因子包括MBNL1、CAMTA1、E2F1等,这些转录因子多与mRNA前的选择性剪接调控、转录激活剂、癌症基因转录抑制剂、细胞周期调控等相关。该结果显示APE1与OGG1结合可能与转录因子结合序列密切相关,且结合倾向性存在差异,因此,有必要对APE1与OGG1结合区域相关基因的异同进行深入分析,以进一步阐明BER参与基因表达调控的重要机制。
| APE1结合motif | 最佳匹配转录因子 | P值 | 模体序列数 | 模体序列数占APE1蛋白结合序列数比例(%) |

|
ZNF_Dof2(锌指蛋白) | 1×10-364 | 240 267 | 76.20 |

|
TCP19 | 1×10-337 | 221 399 | 70.05 |

|
ZNF165(锌指蛋白165) | 1×10-335 | 211 404 | 66.89 |

|
PLAGL1(多形性腺瘤样蛋白1) | 1×10-323 | 166 156 | 61.72 |

|
CDC5(细胞分裂周期5蛋白) | 1×10-314 | 131 062 | 49.40 |
| 最佳匹配转录因子:motif稳定结合转录因子;模体序列数(target-seq):预测motif所占所有目标序列个数 | ||||
| OGG1结合motif | 最佳匹配转录因子 | P值 | 模体序列数 | 模体序列数占APE1蛋白结合序列总数比例(%) |

|
MBNL1(盲肌样蛋白1) | 1×10-295 | 4 343 | 5.20 |

|
CAMTA1(钙调蛋白结合转录激活因子1) | 1×10-268 | 3 250 | 3.89 |

|
ZFP161(锌指蛋白161) | 1×10-259 | 3 173 | 3.80 |

|
E2F1 | 1×10-252 | 3 721 | 4.46 |

|
GT-1(三螺旋转录因子GT-1) | 1×10-224 | 3 658 | 4.38 |
| 最佳匹配转录因子:motif稳定结合转录因子;模体序列数(target-seq):预测motif所占所有目标序列个数 | ||||
2.3 APE1和OGG1结合峰与基因相关性分析
由于DNA碱基损伤存在潜在的表观遗传学调控作用,而上述的分析亦证实在非小细胞肺癌细胞中,BER蛋白更倾向于与基因的启动子区结合,也提示DNA碱基损伤可能对基因的转录具有调控作用。因此,进一步对这些BER蛋白结合的相关基因进行识别,以期发现可能受到AP和/或8-oxo-G位点调控的基因,并比较两者的交集及差异,以此推测受到DNA碱基损伤位点作为新型表观遗传学机制调控的细胞功能及信号通路。
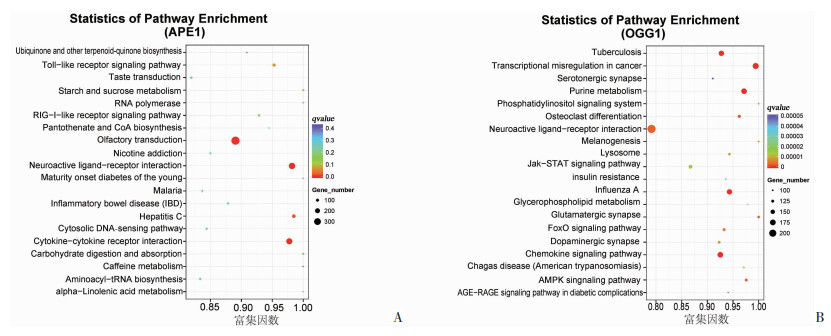
2.3.1 APE1和OGG1结合峰相关基因KEGG分析采用Kobas软件对结合峰相关基因进行KEGG通路富集分析,结果显示:APE1结合峰相关基因主要涉及物质代谢途径、生理反应、酶催化反应、细菌和病毒感染、免疫细胞因子受体等通路(图 2A);OGG1结合峰相关基因主要涉及物质代谢和细胞生理通路,同时也比对到癌症基因转录失调、细胞因子信号、破骨细胞分化、病毒感染等通路(图 2B)。
|
| 富集因数:有峰基因中位于该KEGG通路条目的基因数目与所有注释基因中位于该KEGG通路条目的基因数的比值,该比值越大,表示富集的程度越大;qvalue是做过多重假设检验校正之后的pvalue,qvalue的取值范围为[0, 1],越接近于零,表示富集越显著。qvalue的取值范围为[0, 1],从蓝至红,颜色越深,代表q值越小,富集越显著;Gene_number: 富集在该KEGG通路的基因数 图 2 APE1(A)和OGG1(B)蛋白结合峰相关基因KEGG通路分析 |
2.3.2 APE1和OGG1相同结合峰及相关基因KEGG分析
APE1与OGG1结合峰皮尔森相关系数r=0.636 3(P < 0.01,图 3A),APE1和OGG1相同结合峰54 965个(图 3B),54 965个相同峰相关基因数25 038个(图 3C),其中与肿瘤发生直接相关的基因有641个,APE1和OGG1相同峰及相关基因的KEGG通路分析(图 3D、E)主要涉及物质代谢途径、细胞生长及分化、炎症反应、病毒感染、细胞周期、疾病相关、癌症相关、病毒致癌等通路,该结果提示APE1和OGG1蛋白结合区域不仅和细胞的正常生理代谢相关,也与癌症的发生及机体炎症细胞因子分泌密切相关。

|
| A:APE1和OGG1蛋白结合峰皮尔逊相关系数;B:APE1和OGG1结合峰交叉图;C:APE1和OGG1结合峰相关基因交叉图;D:APE1和OGG1相同峰KEGG通路分析;E:APE1和OGG1相同峰相关基因KEGG通路分析 图 3 APE1和OGG1组间对比结合峰分析及相关基因KEGG分析 |
2.3.3 APE1和OGG1差异结合峰KEGG分析
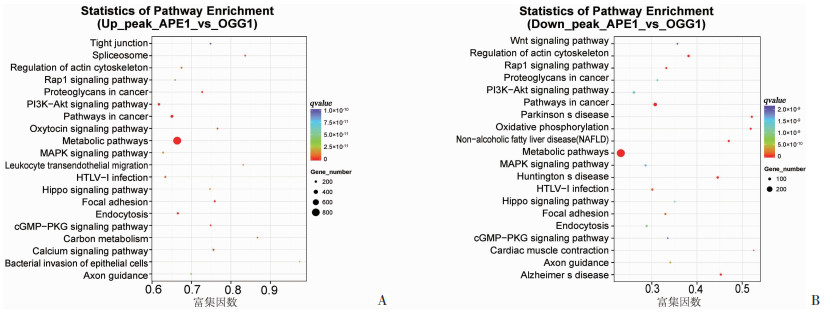
进一步分析APE1与OGG1结合峰分布的差异,通过差异结合位点定位找到差异位点相关基因,并对其差异峰相关基因KEGG富集分析。结果发现:APE1结合峰富集值高于OGG1结合峰的基因14 778个,涉及KEGG通路283条。APE1结合峰富集值低于OGG1结合峰的基因6 667个,涉及KEGG通路296条(图 4)。发现两者基因调控机制的差异主要为:APE1蛋白结合基因更倾向于细胞物质代谢途径、机体生理途径、细胞的增殖及分化通路,而OGG1蛋白结合基因则更偏向老年疾病、肿瘤发生相关通路,具体作用机制仍待进一步阐明。
|
| A:APE1结合峰富集值高于OGG1结合峰;B:APE1结合峰富集值低于OGG1结合峰 图 4 APE1和OGG1结合峰前20个显著性差异结合峰相关基因KEGG分析 |
3 讨论
本研究运用ChIP-seq技术在全基因组水平上分析了非小细胞肺癌细胞中APE1和OGG1结合的DNA序列,并建立了在全基因组水平上描绘DNA碱基损伤位点图谱的方法。本研究发现APE1和OGG1在染色体上具有广泛的结合,但在基因启动子区明显富集;APE1和OGG1结合特征模体(motif)序列是与细胞生长周期调控、细胞因子分泌调控、癌基因转录抑制调控密切相关的转录因子结合保守区域,例如APE1结合特征模体匹配的转录因子ZNF165与TGFβ1激活途径相关[15], 并且OGG1结合8-oxo-G位点可以激活NF-κB通路调节肿瘤免疫微环境[16]。因此推断:APE1和OGG1结合区域的相关基因与肿瘤的促癌基因、炎症细胞因子等通路相关。
前期研究对已知基因片段进行了APE1结合的分析,并得到了APE1可以与VEGF启动子区的AP位点结合的结果[17],但受限于技术原因,尚未从全基因组层面对BER结合位点进行分析。ChIP-seq技术目前广泛运用于分析转录因子的结合区域并预测其调控细胞功能,但很少运用于DNA碱基损伤修复蛋白的结合区域分布研究。本研究创新性地运用ChIP-seq技术,避开了直接对AP位点进行搜寻,通过与AP位点或特定碱基损伤直接结合蛋白进行ChIP的方法来搜寻DNA碱基损伤位点,通过二代测序鉴定结合位点的序列并分析APE1结合峰与OGG1结合峰代替AP和8-oxo-G位点热点区域。然而,本研究方法也存在一定局限性,近年来一些全新观点认为肿瘤全基因组存在大量染色质脆裂、基因融合和基因重排突变的存在[18],因此,用正常细胞的注释基因数据对比肿瘤细胞的DNA片段,可能存在一定程度的偏倚。
既往认为DNA碱基损伤在基因组中是随机产生的,而本研究发现在非小细胞肺癌细胞中,稳态状态下DNA碱基损伤位点是有规律的分布,分布范围主要在基因组功能区的启动子区。我们推断这种规律分布可能与基因调控相关联,但目前证据并不充分,需要进一步探索AP位点结合蛋白的调控机制。另一个重要的旁证来自于2012年在Nature发表的全基因组分析DNA“脆弱位点”的研究,发现人类染色体存在很多对DNaseI超敏的位点聚集的区域,这些区域往往存在于基因启动子区,促进下游基因转录的作用,但该研究并未详细描述这些DNaseI超敏的位点化学性质[19]。根据这些位点的酶化学性质,这些位点很可能就是8-oxo-G或者AP位点。8-oxo-G会优先在基因组中GC含量较高的序列中聚集,而这些序列多数是启动子区的潜在调控序列。经过BER机制的起始步骤移除8-oxo-G之后形成AP位点,进而促进APE1对AP位点结合,可能使下游基因持续转录激活开放[20]。APE1可以在细胞氧化还原状态改变的条件下调控包括DNA修复基因、氧化应激反应基因、缺氧相关基因及血管生成[21-23]等相关基因的表达,DNA修复与转录的耦合提供了一种在氧化应激过程中完成两项必要的细胞任务的有效机制[24-26]。本研究印证了AP/8-oxo-G位点在全基因组启动子区的主要分布形式,由此推断:APE1的DNA修复和氧化还原活性可以在调节含AP位点的启动子的转录起始中起协同作用。本研究并未明确APE1和OGG1与转录因子调控的具体机制及调控谱,仅对两个蛋白结合的序列偏好进行描述。然而,APE1与OGG1结合位点往往位于转录因子结合序列的侧翼区,以增加转录因子与增强子的结合柔性,因此,下一步工作需对热点motif的侧翼序列进行深入分析,方能揭示APE1和OGG1参与基因转录调控的真正机制。
综上所述,本研究建立了BER蛋白抗体ChIP-seq的实验方法,结果显示碱基损伤位点是有规律分布的,碱基损伤热点和启动子区域密切相关,因此推断AP/8-oxo-G位点可能是潜在的表观遗传标志。本研究建立了一个可靠的探寻方法,为今后进一步阐明肿瘤细胞DNA碱基损伤的热点分布区域特征以及碱基损伤的表观遗传重编程导致的肿瘤进展机制奠定了实验基础。
志谢 感谢本科肖何技术员对数据整理分析给予的帮助,张蕾研究实习员对实验的帮助,以及本科杨骁博士及耳鼻喉科王旭东博士对论文修改提出的意见和建议| [1] |
OHNO M, MIURA T, FURUICHI M, et al. A genome-wide distribution of 8-oxoguanine correlates with the preferred regions for recombination and single nucleotide polymorphism in the human genome[J]. Genome Res, 2006, 16(5): 567-575. DOI:10.1101/gr.4769606 |
| [2] |
LINDAHL T. DNA glycosylases, endonucleases for apurinic/apyrimidinic sites, and base excision-repair[J]. Prog Nucleic Acid Res Mol Biol, 1979, 22: 135-192. DOI:10.1016/s0079-6603(08)60800-4 |
| [3] |
MONDEN Y, ARAI T, ASANO M, et al. Human MMH(OGG1)type 1a protein is a major enzyme for repair of 8-hydroxyguanine lesions in human cells[J]. Biochem Biophys Res Commun, 1999, 258(3): 605-610. DOI:10.1006/bbrc.1999.0649 |
| [4] |
Nishimura S. Involvement of mammalian OGG1(MMH)in excision of the 8-hydroxyguanine residue in DNA[J]. Free Radic Biol Med, 2002, 32(9): 813-821. DOI:10.1016/s0891-5849(02)00778-5 |
| [5] |
KLUNGLAND A, BJELLAND S. Oxidative damage to purines in DNA: role of mammalian Ogg1[J]. DNA Repair, 2007, 6(4): 481-488. DOI:10.1016/j.dnarep.2006.10.012 |
| [6] |
LI M X, VÖLKER J, BRESLAUER K J, et al. APE1 incision activity at abasic sites in tandem repeat sequences[J]. J Mol Biol, 2014, 426(11): 2183-2198. DOI:10.1016/j.jmb.2014.03.014 |
| [7] |
HOPPE M M, SUNDAR R, TAN D S P, et al. Biomarkers for homologous recombination deficiency in cancer[J]. J Natl Cancer Inst, 2018, 110(7): 704-713. DOI:10.1093/jnci/djy085 |
| [8] |
CHAO A, LAI C H, WANG T H, et al. Genomic scar signatures associated with homologous recombination deficiency predict adverse clinical outcomes in patients with ovarian clear cell carcinoma[J]. J Mol Med, 2018, 96(6): 527-536. DOI:10.1007/s00109-018-1643-8 |
| [9] |
ALLGAYER J, KITSERA N, BARTELT S, et al. Widespread transcriptional gene inactivation initiated by a repair intermediate of 8-oxoguanine[J]. Nucleic Acids Res, 2016, 44(15): 7267-7280. DOI:10.1093/nar/gkw473 |
| [10] |
PARK P J. ChIP-seq: advantages and challenges of a maturing technology[J]. Nat Rev Genet, 2009, 10(10): 669-680. DOI:10.1038/nrg2641 |
| [11] |
CHEN S F, ZHOU Y Q, CHEN Y R, et al. Fastp: an ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34(17): i884-i890. DOI:10.1093/bioinformatics/bty560 |
| [12] |
HEINZ S, BENNER C, SPANN N, et al. Simple combinations of lineage-determining transcription factors prime Cis-regulatory elements required for macrophage and B cell identities[J]. Mol Cell, 2010, 38(4): 576-589. DOI:10.1016/j.molcel.2010.05.004 |
| [13] |
YU G C, WANG L G, HE Q Y. ChIPseeker: an R/Bioconductor package for ChIP peak annotation, comparison and visualization[J]. Bioinformatics, 2015, 31(14): 2382-2383. DOI:10.1093/bioinformatics/btv145 |
| [14] |
SALMON-DIVON M, DVINGE H, TAMMOJA K, et al. Peak Analyzer: Genome-wide annotation of chromatin binding and modification loci[J]. BMC Bioinform, 2010, 11(1): 1-12. DOI:10.1186/1471-2105-11-415 |
| [15] |
MAXFIELD K E, TAUS P J, CORCORAN K, et al. Comprehensive functional characterization of cancer-testis antigens defines obligate participation in multiple hallmarks of cancer[J]. Nat Commun, 2015, 6: 8840. DOI:10.1038/ncomms9840 |
| [16] |
PAN L, ZHU B, HAO W J, et al. Oxidized guanine base lesions function in 8-oxoguanine DNA glycosylase-1-mediated epigenetic regulation of nuclear factor κB-driven gene expression[J]. J Biol Chem, 2016, 291(49): 25553-25566. DOI:10.1074/jbc.M116.751453 |
| [17] |
PASTUKH V, ROBERTS J T, CLARK D W, et al. An oxidative DNA "damage" and repair mechanism localized in the VEGF promoter is important for hypoxia-induced VEGF mRNA expression[J]. Am J Physiol Lung Cell Mol Physiol, 2015, 309(11): L1367-L1375. DOI:10.1152/ajplung.00236.2015 |
| [18] |
ICGC/TCGA Pan-Cancer Analysis of Whole Genomes Consortium. Pan-cancer analysis of whole genomes[J]. Nature, 2020, 578(7793): 82-93. DOI:10.1038/s41586-020-1969-6 |
| [19] |
THURMAN R E, RYNES E, HUMBERT R, et al. The accessible chromatin landscape of the human genome[J]. Nature, 2012, 489(7414): 75-82. DOI:10.1038/nature11232 |
| [20] |
FLEMING A M, ZHU J, HOWPAY MANAGE S A, et al. Human NEIL3 gene expression regulated by epigenetic-like oxidative DNA modification[J]. J Am Chem Soc, 2019, 141(28): 11036-11049. DOI:10.1021/jacs.9b01847 |
| [21] |
DEGANUTO M, PITTIS M G, PINES A, et al. Altered intracellular redox status in Gaucher disease fibroblasts and impairment of adaptive response against oxidative stress[J]. J Cell Physiol, 2007, 212(1): 223-235. DOI:10.1002/jcp.21023 |
| [22] |
SCHINDL M, OBERHUBER G, PICHLBAUER E G, et al. DNA repair-redox enzyme apurinic endonuclease in cervical cancer: evaluation of redox control of HIF-1alpha and prognostic significance[J]. Int J Oncol, 2001, 19(4): 799-802. DOI:10.3892/ijo.19.4.799 |
| [23] |
LUO M H, HE H Z, KELLEY M R, et al. Redox regulation of DNA repair: implications for human health and cancer therapeutic development[J]. Antioxid Redox Signal, 2010, 12(11): 1247-1269. DOI:10.1089/ars.2009.2698 |
| [24] |
LI M X, DAI N, WANG D, et al. Distinct APE1 activities affect the regulation of VEGF transcription under hypoxic conditions[J]. Comput Struct Biotechnol J, 2019, 17: 324-332. DOI:10.1016/j.csbj.2019.02.007 |
| [25] |
LI M X, WILSON D M. Human apurinic/apyrimidinic endonuclease 1[J]. Antioxid Redox Signal, 2014, 20(4): 678-707. DOI:10.1089/ars.2013.5492 |
| [26] |
FLEMING A M, DING Y, BURROWS C J. Oxidative DNA damage is epigenetic by regulating gene transcription via base excision repair[J]. Proc Natl Acad Sci USA, 2017, 114(10): 2604-2609. DOI:10.1073/pnas.1619809114 |




