肠道病毒D68型(enterovirus D68, EV-D68)是一种小RNA病毒,原名为鼻病毒87型,后归于肠道病毒属D组,多感染儿童,最早于1962年在美国加利福尼亚地区的4名呼吸道感染患儿呼吸道标本中被分离出[1]。在被发现之后的几十年里EV-D68感染病例散发且鲜有报道,一直被认为是一种边缘性病原体而不受关注。2014年美国出现了一次EV-D68的大流行,共有1 153人被感染并多伴有严重的呼吸道症状[2],后来的许多回顾性研究表明,在同一时期欧洲也存在很高的EV-D68感染率[3],此后EV-D68才逐渐被引起重视。
目前关于EV-D68的研究多集中在流行病学,其生物学特征及其致病机理尚不完全清楚,与其他肠道病毒不同,EV-D68主要引起呼吸系统疾病,严重者可出现呼吸困难、血氧不足等情况[4]。有研究表明EV-D68的感染与哮喘发病率有密切联系[5]。此外EV-D68也会导致中枢神经系统疾病急性弛缓性麻痹、急性无力脊髓炎(acute flaccid myelitis,AFM)的发生,严重危害公众健康[6]。但目前还没有针对EV-D68的有效抗病毒药物和疫苗。本研究通过对感染EV-D68前后的宿主细胞进行转录组测序,分析验证转录组表达谱差异,寻找参与感染发生的相关基因及其信号通路的作用等,为进一步探讨其致病机制打下基础。
1 材料与方法 1.1 细胞培养与病毒感染人横纹肌瘤(rhahdomyoma, RD)细胞购于北纳生物;EV-D68选用原始Fermon毒株, 由本实验室保存。将RD细胞培养在含10%胎牛血清(Hyclone)和1%青-链霉素(碧云天)的DMEM细胞培养基(Hyclone)中。在37 ℃和5% CO2的条件下培养至细胞融合度达80%~90%时,再将培养基更换为含2%血清的培养基。然后将病毒接种到RD细胞中,感染复数(multiplicity of infection, MOI)为0.01,对照组不加病毒,每组3个生物重复,24 h后收获细胞。
1.2 RNA抽提根据说明书,使用TRIzol试剂(Invitrogen)从每组细胞中提取总RNA,去除基因组DNA,在测序之前,使用1%琼脂糖凝胶电泳、2100 Bioanalyser (Agilent)及ND-2000 (NanoDrop Technologies)检测RNA的浓度和完整性。D(260)/D(280)=1.8~2.2,D(260)/D(230)≥2.0,RIN≥7,28S:18S≥1.0,总量>2 μg为合格样本。
1.3 文库构建及测序按照说明书使用TruSeqTM RNA(Illumina, San Diego, CA)试剂盒,加入1 μg总RNA进行文库构建。首先,通过带有oligo(dT)的磁珠从总RNA中富集带有poly-A尾的mRNA,然后通过片段化缓冲液进行片段化,再使用SuperScript double-stranded cDNA synthesis kit (Invitrogen, CA)试剂盒和随机引物(Illumina)合成双链cDNA。随后根据Illumina的文库构建方案对合成的cDNA进行末端修复,并在3′末端加上一个A碱基。使用Phusion DNA聚合酶(NEB)进行PCR扩增15个PCR循环后筛选出大小为200~300 bp的条带,用TBS380定量后,使用Illumina HiSeq xten/NovaSeq 6000测序仪(2×150 bp读取长度)进行测序。
1.4 测序数据的处理与分析 1.4.1 原始数据质控及序列比对分析使用软件SeqPrep(https://github.com/jstjohn/SeqPrep)和Sickle (https://github.com/najoshi/sickle)对原始测序数据进行质控(quality control, QC),QC包括去除reads中的接头序列和没有插入片段的reads;去除3′端质量值< 20和剩余序列中质量值< 10的碱基;去除含模块碱基的reads以及舍弃去adapter及质量修剪后长度 < 30 bp的序列。质控完成后再使用HISAT2软件,将质控后的原始数据与人类参考基因组GRCh38进行比对,获得用于后续转录本组装、表达量计算等的mapped reads,比对完成后对该次转录组测序的比对结果进行质量评估。
1.4.2 表达量差异分析及GO、KEGG富集分析使用表达定量软件RSEM(http://deweylab.github.io/RSEM/)对基因的表达水平进行定量分析,并采用对基因长度和测序深度进行了均一化的TPM(Transcripts Per Million reads)作为定量指标,使得不同样本中的总表达量一致,可更直观地比较基因间表达量。使用基于负二项分布的DESeq2软件对标准化后的数据进行分析,以P<0.05且|log2FC| ≥1为标准筛选出差异表达基因(differentially expressed genes, DEGs)。
筛选完成后再对DEGs进行GO和KEGG途径富集分析。GO分析主要涵盖生物过程(biological process, BP),分子功能(molecular function, MF)和细胞成分(cellular component, CC)三个部分。KEGG可将差异表达基因按照参与的通路或行使的功能分类并统计。以P<0.05为标准进行功能富集分析。
1.5 RT-qPCR验证从测序结果中随机选取8个TPM>5的差异表达基因进行RT-qPCR验证。其中上调基因包括EGR1、c-fos、ZNF625、ARRDC3、c-jun;下调基因包括ASNS、CHAC1、ADM2。用RNA快速提取试剂盒(上海奕杉生物)分别提取对照组和EV-D68感染组细胞的总RNA,再使用反转录试剂盒(TaKaRa)将RNA反转录成cDNA。随后以此为模板根据SYBR Green PCR Master Mix(Bimake)试剂盒说明书进行RT-qPCR实验,每组3个生物重复。使用Oligo7软件根据选定基因的序列设计特异性引物并用NCBI Primer-BLAST和融解曲线对引物特异性进行评价,挑选出合格的引物进行实验(表 1)。实验采用β-actin为内参,使用2-ΔΔCt法对RT-qPCR结果进行分析,并与转录组测序结果相关基因表达量进行比较。
| 基因名称 | 引物序列(5′→ 3′) | 扩增区域 | 产物大小/bp |
| EGR1 | 上游:AAGAAAAGCCAAGCAAACCAA | 2430-3450 | 128 |
| 下游:AACGGAACAACACTCTGACAC | 2557-2537 | ||
| c-fos | 上游:CCGGGGATAGCCTCTCTTACT | 223-243 | 93 |
| 下游:CCAGGTCCGTGCAGAAGTC | 315-297 | ||
| ZNF625 | 上游:AGATTCCTAGAAGCCACCGAA | 1304-1324 | 171 |
| 下游:CCCCAGCTAAACTACTCCCAA | 1474-1454 | ||
| ARRDC3 | 上游:AGCAGCATTTTGTTACTGACT | 2266-2286 | 174 |
| 下游:ACTTTTGTGTATGTCCCGTTT | 2439-2419 | ||
| c-jun | 上游:TCCAAGTGCCGAAAAAGGAAG | 1776-1796 | 78 |
| 下游:CGAGTTCTGAGCTTTCAAGGT | 1853-1833 | ||
| ASNS | 上游:GGAAGACAGCCCCGATTTACT | 1265-1285 | 154 |
| 下游:AGCACGAACTGTTGTAATGTCA | 1418-1397 | ||
| CHAC1 | 上游:CCCAGCCATCCATAGCCCTG | 1030-1049 | 78 |
| 下游:TCCTCATGCCCTACTATCCCT | 1107-1087 | ||
| ADM2 | 上游:CTGAGCCCCATCTGAAGCC | 818-836 | 112 |
| 下游:CAGCACTGCGTGTAGACCAG | 929-910 | ||
| β-actin | 上游:CATTGCCGACAGGATGCAG | 1008-1026 | 92 |
| 下游:CGGAGTACTTGCGCTCAGGA | 1099-1080 |
1.6 统计学分析
实验数据使用GraphPad Prism 5软件进行统计学分析,计量资料用x±s表示。
2 结果 2.1 转录组测序数据统计各样本测序获得总读数(raw reads)有44 509 264~65 693 118个,经过质控后剩余的读数(clean reads)有4 316 446~64 996 826个;clean reads中Q30碱基百分比均≥91.15%,测序错误率控制在0.03%以内;所有样本碱基的质量得分均为37,且GC碱基所占比例均≥48.13%.质控完成后将clean reads与人类参考基因组GRCh38进行序列比对(表 2),比对结果显示,各样本的reads与参考基因组的比对率在91.99%~96.66%。
| 样本名称(Sample) | 总序列数(Raw reads) | 质控后读数(Clean reads) | 定位到基因组上的总序列数(Total mapped) | 多个比对位置的序列数(Multiple mapped) | 比对位置唯一的序列数(Uniquely mapped) |
| Infected1 | 44509264 | 43616446 | 40121712(91.99) | 1285393(2.95) | 38836319(89.04) |
| Infected2 | 57732532 | 57096208 | 53031046(92.88) | 1770370(3.1) | 51260676(89.78) |
| Infected3 | 56803892 | 56181770 | 52129282(92.79) | 1724017(3.07) | 50405265(89.72) |
| control1 | 55696854 | 55026242 | 52980860(96.28) | 1811150(3.29) | 51169710(92.99) |
| control2 | 62909056 | 62185334 | 59964147(96.43) | 2088945(3.36) | 57875202(93.07) |
| control3 | 65693118 | 64996826 | 62822701(96.66) | 2127168(3.27) | 60695533(93.38) |
对比完成后,将比对到基因组上的reads分布情况进行统计(表 3),定位区域共有5个,分别为编码区(CDS)、内含子(intron)、基因间区(intergenic)和5′和3′非翻译区(UTR)。在人类参考基因组中,大部分的reads通常比对到CDS区,来源于pre-mRNA的残留或由可变剪切过程中发生的内含子保留事件导致的reads则会比对到intron区,而转录自新基因或新的非编码RNA的reads则会比对到intergenic区。
| 样本名称 | 内含子区 | 3′非编码区 | 编码区 | 5′非编码区 | 基因间区 |
| Infected1 | 3280097(5.54) | 9170563(15.5) | 43201662(73.02) | 3106239(5.25) | 405048(0.68) |
| Infected2 | 4304885(5.47) | 11830199(15.04) | 57858611(73.58) | 4102487(5.22) | 537301(0.68) |
| Infected3 | 4347575(5.63) | 11643390(15.08) | 56715571(73.46) | 3956795(5.12) | 547953(0.71) |
| control1 | 4510531(5.78) | 11891302(15.23) | 57023558(73.01) | 4116075(5.27) | 557367(0.71) |
| control2 | 5117151(5.77) | 13141050(14.81) | 65146309(73.4) | 4709327(5.31) | 641421(0.72) |
| control3 | 4893303(5.23) | 13626435(14.55) | 69606105(74.34) | 4889271(5.22) | 614510(0.66) |
2.2 RT-qPCR验证
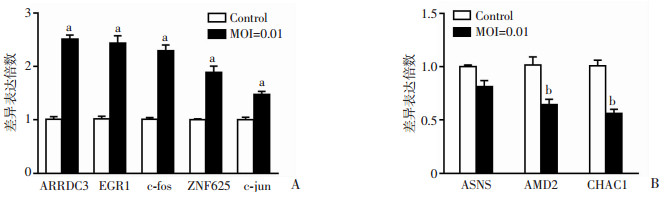
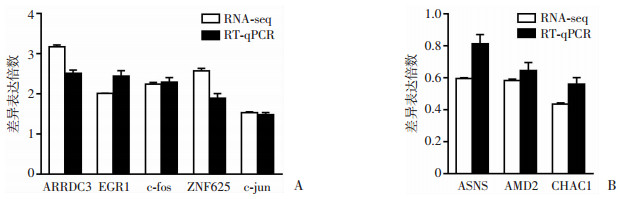
为了确定转录组测序结果的可靠性,本研究选取了8个基因进行RT-qPCR验证(EGR1、c-fos、ZNF625、ARRDC3、c-jun、ASNS、CHAC1、ADM2),并与转录组测序结果进行比较。RT-qPCR实验结果表明(图 1、2),选取的8个基因中上调或下调的差异表达倍数趋势与测序结果基本一致,说明测序结果是可信的。
|
| a:P<0.01,b:P<0.001,与control比较A:上调基因验证结果;B:下调基因验证结果 图 1 RT-qPCR验证转录组结果(n=6, x±s) |
|
| A:上调基因比较结果;B:下调基因比较结果 图 2 RT-qPCR验证结果与转录组测序结果比较 |
2.3 差异表达基因统计及GO、KEGG富集分析
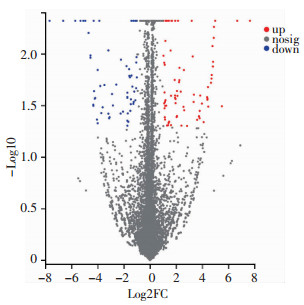
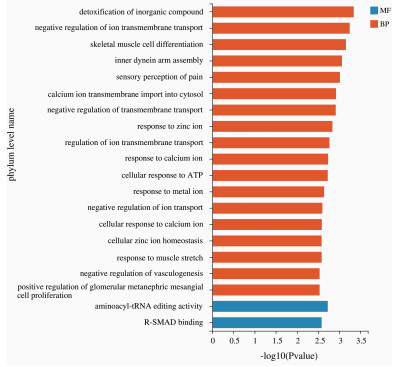
数据经过定量且标准化处理后筛选出|log2FC| ≥1的DEGs共140个,其中上调基因71个,下调基因69个(图 3)。为进一步研究这些DEGs的生物学功能,对其分别进行GO与KEGG富集分析。在GO富集分析中,显著富集到420个GO terms,其中311个涉及BP,28个涉及CC,89个涉及MF。显著富集的前20位中,18项属于BP类,其中9个与离子的调控和应答有关,其次主要富集在血管生成负调控、细胞对ATP的应答、骨骼肌分化、内动力蛋白臂组装等方面。其余2项属于MF类,分别是R-SMAD结合及氨酰基-tRNA编辑活性(图 4)。
|
| 图 3 差异表达基因火山图 |
|
| 图 4 差异基因的GO富集分析 |
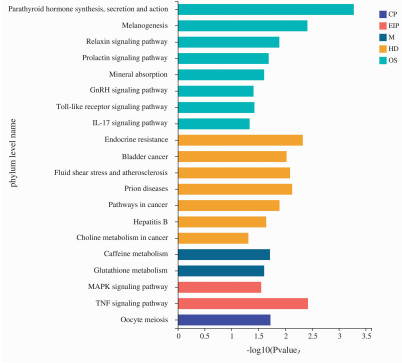
KEGG富集分析结果共富集到115条,包括代谢(metabolize, M)、遗传信息处理(genetic Information processing, GIP)、环境信息处理(environmental information processing, EIP)、细胞过程(cellular processes, CP),生物体系统(biological system, OS)、人类疾病(human diseases, HD)等6大类。其中26条显著富集,包括甲状旁腺激素的合成分泌和作用、黑色素生成、松弛素信号通路、催乳素信号通路、矿物质吸收、GnRH信号通路、Toll样受体信号通路、IL-17信号通路、内分泌抵抗、MAPK信号通路等(图 5、6)。
|
| 红色框为上调基因,黑色框为下调基因 图 5 差异基因的KEGG富集分析 |
|
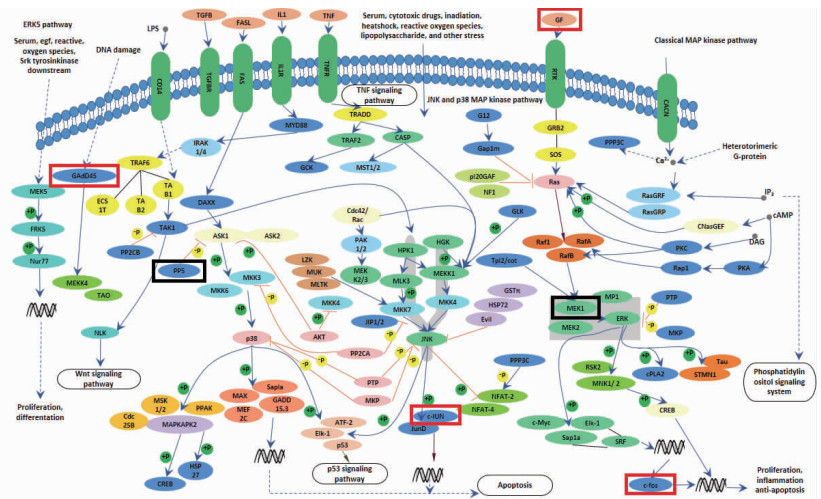
| 图 6 MAPK信号通路中差异表达的基因 |
2.4 病毒感染对宿主细胞c-fos和c-jun表达的影响
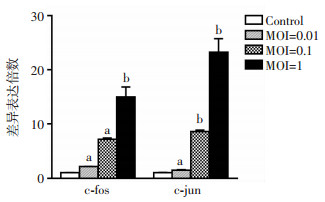
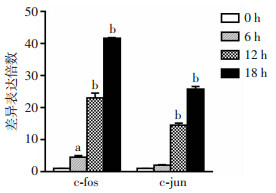
为寻找参与病毒感染的基因与信号通路,我们分别以MOI=0、MOI=0.01、MOI=0.1和MOI=1等4个病毒量梯度感染细胞24 h,再用RT-qPCR检测MAPK信号通路相关基因c-fos和c-jun mRNA水平表达变化,结果发现c-fos和c-jun的表达量随病毒量的增加而增加(图 7)。同时为了验证c-fos和c-jun基因与病毒感染时间的相关性,我们以MOI=2感染细胞,并在0、6、12、18 h收获细胞提取RNA进行RT-qPCR实验,结果表明EV-D68感染细胞后,c-fos和c-jun mRNA的表达量随时间的延长而增加(图 8)。为进一步验证这种上调趋势是否只存在于RD细胞中,我们以MOI=0、MOI=1、MOI=2的感染量作用于另一种EV-D68的易感细胞Hela细胞,检测c-fos和c-jun mRNA表达水平,得到了相似的结果(图 9),说明EV-D68感染细胞后c-fos和c-jun的调节作用在Hela细胞中同样存在。
|
| a: P<0.01, b: P<0.001, 与control比较 图 7 不同MOI处理RD细胞后c-fos和c-jun mRNA表达情况(n=3, x±s) |
|
| a: P<0.01, b: P<0.001, 与control比较 图 8 病毒感染不同时间c-fos和c-jun mRNA表达情况(n=3, x±s) |
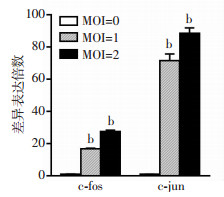
|
| b: P<0.001, 与control比较 图 9 不同MOI处理Hela细胞后c-fos和c-jun mRNA表达情况(n=3, x±s) |
3 讨论
虽然EV-D68在1962年就被发现,但直到2014年才被重视。有不少研究证明,EV-D68的流行呈季节性且两年一爆发[7-11]。在我国EV-D68没有被纳入常规检测,临床上多数为对症治疗。有研究报道在我国北京、重庆、台湾等地,人群中均有较高滴度的中和抗体存在,且抗体阳性率随年龄的增长而升高[12-14]。EV-D68易感人群为儿童,感染后轻者与感冒无异,但重者需要重症监护治疗。
哮喘是一种常见的多发性的气道慢性变态反应性炎症。越来越多的研究证明因EV-D68感染而需住院的患者中很大一部分有哮喘病史,且入院原因也是哮喘加重。WANG等[15]通过统计分析发现,2018年美国俄亥俄州哥伦布市国家儿童医院总共有277例因EV-D68感染住院的患者,其中有146名(53%)曾患有哮喘史;DREWS等[16]和MERTZ等[17]研究报告中指出住院的EV-D68感染儿童中有31%~70%有哮喘病史,而在我国台湾也有类似的报道[18];还有文献称在74.3%的哮喘发作患者的鼻咽样本中检测到EV-D68[19]。EV-D68能加重哮喘或使有哮喘病史的患者复发,其机制尚不完全清楚。
目前还未见有EV-D68感染宿主细胞后转录组表达谱差异分析的报道。RD细胞是EV-D68、EV71等新型肠道病毒感染的高效细胞[20-21],也是研究EV-D68、EV71等新型肠道病毒常用的经典细胞。在本研究中,通过对EV-D68感染前后的RD细胞进行转录组测序,筛选差异表达基因并对其进行GO和KEGG富集分析。在对测序结果的分析中,我们注意到一个特殊的基因c-fos,它在EV-D68感染后是显著上调的,通过查阅文献我们发现它与哮喘有密切联系。c-fos是
一种原癌基因,与细胞的生长、增殖活化有关,在正常人类细胞中可检测到极低水平的表达。DEMOLY等[22]在1992年第一次指出c-fos在哮喘患者支气管活检气道上皮中表达升高,在随后的研究中又发现c-fos的阳性反应程度与上皮细胞脱落程度成正比;LIU[23]等研究发现在大鼠肺中,c-fos蛋白的光密度与支气管肺泡灌洗液中P物质的浓度呈正相关,与血管活性肠肽(vasoactive intestinal peptide, VIP)的浓度呈负相关,说明c-fos蛋白表达和神经肽含量与哮喘发作有关。而c-fos在哮喘患者中表达升高不止体现在呼吸系统的组织中,也有研究表明,当哮喘模型动物被致敏,中枢系统的多个区域c-fos的表达增加[24-26]。而在我们的研究中,通过RT-qPCR证实c-fos在EV-D68感染后表达上调,且表达量随病毒MOI的增加和感染时间的延长而增加。结合测序结果和RT-qPCR验证结果我们猜想EV-D68加重哮喘的机制有可能和上调c-fos有关,在接下来的实验中,我们将进一步进行探索。
除此之外,在KEGG富集分析结果中,我们重点关注到MAPK信号通路,丝裂原活化蛋白激酶(mitogen-activated protein kinases, MAPKs)属于丝氨酸/苏氨酸蛋白激酶家族,它在真核生物中广泛存在,MAPK信号通路是细胞增殖、应激、炎症、分化、功能同步化、转化、凋亡等信号转导通路的共同交汇通路之一,与病毒感染相关的MAPK信号通路主要有3种,包括ERK、p38和JNK。MAPK信号通路的激活在肠道病毒71型、单纯疱疹病毒、柯萨奇病毒B3等都有报道[27-29]。此外,许多MAPK调节的促炎或抗病毒细胞因子(如IL-2,IL-6,IFN-β和TNF-α)的转录取决于转录因子NF-κB,c-fos和c-jun[30-32]。而在我们的结果中,富集到的MAPK通路中有6个基因有显著差异,包括c-fos、c-jun、GF、CADD45、MEK1、pp5(图 6)。除c-fos外,c-jun的表达量也随病毒MOI的增加和感染时间的延长而增加,所以我们推断,MAPK通路有可能在EV-D68感染期间发挥促炎作用和抗病毒作用,在后续实验中我们也将进一步对其进行验证。
虽然现在EV-D68逐渐引起重视,但关于其致病机制的研究却比较少,而且目前也没有预防和治疗EV-D68感染的特效药物和疫苗。本研究通过对EV-D68感染前后的宿主细胞进行转录组测序,一共发现了140个差异表达基因,并对其进行GO和KEGG富集分析,重点关注到可能参与哮喘发病机制的c-fos基因,及可能在EV-D68感染期间发挥促炎作用和抗病毒作用的MAPK信号通路,在后期实验中,我们将对以上重点关注基因的功能进行深入的探讨,为EV-D68的感染机制研究奠定实验基础。
| [1] |
SCHIEBLE J H, FOX V L, LENNETTE E H. A probable new human picornavirus associated with respiratory disease[J]. Am J Epidemiol, 1967, 85(2): 297-310. DOI:10.1093/oxfordjournals.aje.a120693 |
| [2] |
MIDGLEY C M, WATSON J T, NIX W A, et al. Severe respiratory illness associated with a nationwide outbreak of enterovirus D68 in the USA (2014): a descriptive epidemiological investigation[J]. Lancet Respir Med, 2015, 3(11): 879-887. DOI:10.1016/s2213-2600(15)00335-5 |
| [3] |
POELMAN R, SCHUFFENECKER I, VAN LEER-BUTER C, et al. European surveillance for enterovirus D68 during the emerging North-American outbreak in 2014[J]. J Clin Virol, 2015, 71: 1-9. DOI:10.1016/j.jcv.2015.07.296 |
| [4] |
OBERSTE M S, MAHER K, SCHNURR D, et al. Enterovirus 68 is associated with respiratory illness and shares biological features with both the enteroviruses and the rhinoviruses[J]. J Gen Virol, 2004, 85(9): 2577-2584. DOI:10.1099/vir.0.79925-0 |
| [5] |
RAJPUT C, HAN M Y, BENTLEY J K, et al. Enterovirus D68 infection induces IL-17-dependent neutrophilic airway inflammation and hyperresponsiveness[J]. JCI Insight, 2018, 3(16): 1-13. DOI:10.1172/jci.insight.121882 |
| [6] |
BOWERS J R, VALENTINE M, HARRISON V, et al. Genomic analyses of acute flaccid myelitis cases among a cluster in Arizona provide further evidence of enterovirus D68 role[J]. mBio, 2019, 10(1). DOI:10.1128/mbio.02262-18 |
| [7] |
MEIJER A, BENSCHOP K S, DONKER G A, et al. Continued seasonal circulation of enterovirus D68 in the Netherlands, 2011-2014[J]. Euro Surveill, 2014, 19(42). DOI:10.2807/1560-7917.es2014.19.42.20935 |
| [8] |
KRAMER R, SABATIER M, WIRTH T, et al. Molecular diversity and biennial circulation of enterovirus D68: a systematic screening study in Lyon, France, 2010 to 2016[J]. Euro Surveill, 2018, 23(37). DOI:10.2807/1560-7917.es.2018.23.37.1700711 |
| [9] |
UPRETY P, CURTIS D, ELKAN M, et al. Association of enterovirus D68 with acute flaccid myelitis, Philadelphia, Pennsylvania, USA, 2009-2018[J]. Emerg Infect Dis, 2019, 25(9): 1676-1682. DOI:10.3201/eid2509.190468 |
| [10] |
American Centers for Disease Control and Prevention: AFM investigation[EB/OL].[2020-09-13] |
| [11] |
MEYERS L, BARD J D, GALVIN B, et al. Enterovirus D68 outbreak detection through a syndromic disease epidemiology network[J]. J Clin Virol, 2020, 124: 104262. DOI:10.1016/j.jcv.2020.104262 |
| [12] |
XIANG Z, GONZALEZ R, WANG Z, et al. Coxsackievirus A21, enterovirus 68, and acute respiratory tract infection, China[J]. Emerg Infect Dis, 2012, 18(5): 821-824. DOI:10.3201/eid1805.111376 |
| [13] |
XIAO Q, REN L, ZHENG S, et al. Prevalence and molecular characterizations of enterovirus D68 among children with acute respiratory infection in China between 2012 and 2014[J]. Sci Rep, 2015, 5: 16639. DOI:10.1038/srep16639 |
| [14] |
LEE J T, SHIH W L, YEN T Y, et al. Enterovirus D68 seroepidemiology in Taiwan, a cross sectional study from 2017[J]. PLoS ONE, 2020, 15(3): e0230180. DOI:10.1371/journal.pone.0230180 |
| [15] |
WANG H Y, DIAZ A, MOYER K, et al. Molecular and clinical comparison of enterovirus D68 outbreaks among hospitalized children, Ohio, USA, 2014 and 2018[J]. Emerg Infect Dis, 2019, 25(11): 2055-2063. DOI:10.3201/eid2511.190973 |
| [16] |
DREWS S J, SIMMONDS K, USMAN H R, et al. Characterization of enterovirus activity, including that of enterovirus D68, in pediatric patients in Alberta, Canada, in 2014[J]. J Clin Microbiol, 2015, 53(3): 1042-1045. DOI:10.1128/jcm.02982-14 |
| [17] |
MERTZ D, ALAWFI A, PERNICA J M, et al. Clinical severity of pediatric respiratory illness with enterovirus D68 compared with rhinovirus or other enterovirus genotypes[J]. CMAJ, 2015, 187(17): 1279-1284. DOI:10.1503/cmaj.150619 |
| [18] |
HASEGAWA S, HIRANO R, OKAMOTO-NAKAGAWA R, et al. Enterovirus 68 infection in children with asthma attacks: virus-induced asthma in Japanese children[J]. Allergy, 2011, 66(12): 1618-1620. DOI:10.1111/j.1398-9995.2011.02725.x |
| [19] |
PAN H H, TSAI C R, TING P J, et al. Respiratory presentation of patients infected with enterovirus D68 in Taiwan[J]. Pediatr Neonatol, 2020, 61(2): 168-173. DOI:10.1016/j.pedneo.2019.09.006 |
| [20] |
WEI W, GUO H R, CHANG J L, et al. ICAM-5/telencephalin is a functional entry receptor for enterovirus D68[J]. Cell Host Microbe, 2016, 20(5): 631-641. DOI:10.1016/j.chom.2016.09.013 |
| [21] |
LIU Y, SHENG J, BAGGEN J, et al. Sialic acid-dependent cell entry of human enterovirus D68[J]. Nat Commun, 2015, 6: 8865. DOI:10.1038/ncomms9865 |
| [22] |
DEMOLY P, BASSET-SEGUIN N, CHANEZ N, et al. C-fos proto-oncogene expression in bronchial biopsies of asthmatics[J]. Am J Respir Cell Mol Biol, 1992, 7(2): 128-133. DOI:10.1165/ajrcmb/7.2.128 |
| [23] |
LIU H, YANG X, HOU W. Correlation of c-fos protein expression with neuropeptide content in the lung of bronchial asthmatic rat[J]. Int J Clin Exp Pathol, 2014, 7(12): 8657-8665. |
| [24] |
GUO R B, SUN P L, ZHAO A P, et al. Chronic asthma results in cognitive dysfunction in immaturemice[J]. Exp Neurol, 2013, 247: 209-217. DOI:10.1016/j.expneurol.2013.04.008 |
| [25] |
CHEN S D, WEN Z H, CHANG W K, et al. Acute effect of methylprednisolone on the brain in a rat model of allergic asthma[J]. Neurosci Lett, 2008, 440(2): 87-91. DOI:10.1016/j.neulet.2008.03.092 |
| [26] |
CHEN Z, LIU N N, XIAO J, et al. The amygdala via the paraventricular nucleus regulates asthma attack in rats[J]. CNS Neurosci Ther, 2020, 26(7): 730-740. DOI:10.1111/cns.13293 |
| [27] |
ZHU L, LI W H, QI G J, et al. The immune mechanism of intestinal tract Toll-like receptor in mediating EV71 virus type severe hand-foot-and-mouth disease and the MAPK pathway[J]. Exp Ther Med, 2017, 13(5): 2263-2266. DOI:10.3892/etm.2017.4245 |
| [28] |
ZHANG H, FENG H, LUO L, et al. Distinct effects of knocking down MEK1 and MEK2 on replication of Herpes simplex virus type 2[J]. Virus Res, 2010, 150(1/2): 22-27. DOI:10.1016/j.virusres.2010.02.007 |
| [29] |
SI X N, LUO H L, MORGAN A, et al. Stress-activated protein kinases are involved in coxsackievirus B3 viral progeny release[J]. J Virol, 2005, 79(22): 13875-13881. DOI:10.1128/jvi.79.22.13875-13881.2005 |
| [30] |
TUNG W H, HSIEH H L, LEE I T, et al. Enterovirus 71 modulates a COX-2/PGE2/cAMP-dependent viral replication in human neuroblastoma cells: Role of the c-Src/EGFR/p42/p44 MAPK/CREB signaling pathway[J]. J Cell Biochem, 2011, 112(2): 559-570. DOI:10.1002/jcb.22946 |
| [31] |
CEBALLOS-OLVERA I, CHÁVEZ-SALINAS S, MEDINA F, et al. JNK phosphorylation, induced during dengue virus infection, is important for viral infection and requires the presence of cholesterol[J]. Virology (Lond), 2010, 396(1): 30-36. DOI:10.1016/j.virol.2009.10.019 |
| [32] |
FEJER G, FREUDENBERG M, GREBER U F, et al. Adenovirus-triggered innate signalling pathways[J]. Eur J Microbiol Immunol, 2011, 1(4): 279-288. DOI:10.1556/eujmi.1.2011.4.3 |




