BCAR1是目前肿瘤研究的热点,其相对分子质量为130×103,是细胞外基质与胞内信号传导的桥梁分子:细胞与胞外基质接触后激活整合素→FAK激活并结合到BCAR1氨基端→磷酸化BCAR1羟基端→BCAR1与Src激酶结合→BCAR1活化→形成BCAR1/Crk复合体→Crk通路被激活[1-2]。
研究表明,BCAR1高表达与多种癌症的细胞侵袭和转移相关:在前列腺癌中,BCAR1的高表达与肿瘤早期复发密切相关[3]。在子宫内膜腺癌中,BCAR1参与肿瘤细胞的生长和转移[4]。在口腔鳞癌中,BCAR1通过调节上皮-间质转化(Epithelial mesenchymal transformation,EMT)和细胞增殖促进骨侵袭和转移[5]。我们前期研究发现:BCAR1在肺癌组织中高表达,而在癌旁组织中低表达[6-7];开展的多中心临床研究证实肺癌BCAR1-mRNA及蛋白表达水平越高,患者预后越差[7]。而且,BCAR1高表达可诱导肺腺癌细胞发生EMT促进肺腺癌细胞迁移和侵袭[8-9]。所以,BCAR1有望成为肺癌精准治疗的分子靶点[10]。
目前BCAR1研究多局限于肿瘤侵袭转移,较少聚焦细胞增殖和克隆形成。鉴于BCAR1基因促癌效应复杂,其相互作用蛋白研究较少,相关通路和网络尚不明确。本研究拟通过下调及上调BCAR1的表达及免疫共沉淀质谱分析,旨在深入探索上述问题。
1 材料与方法 1.1 材料GV371载体,Cas9载体,CV146载体,GV141载体(均来自吉凯基因),H1975、H1299、A549、293T(ATCC),胎牛血清(Gibco, 10099141),胰酶(Gibco, 25200-056),BCAR1(CST,#13846),GAPDH(CST,#2118s),MLST8(CST,#3227),Rabbit IgG(Santa Cruz,sc-2004),嘌呤霉素(Clontech,631305),MTT(Solarbio,M8180),dsDNA oligo(吉凯基因,P212-03),2×Taq Plus Master Mix(Vazyme,M0318S),TOP10感受态(吉凯基因,R0539L),BbsI(sigma,D0281-59),T7 DNA Ligase(sigma,D4041),ATP(10 μmol/L)(Takara,DP11),Yeast Extract(OXOID),Agar Powder(OXOID,A-2180),Trizol试剂盒(上海普飞),1 kp DNA ladder Marker(Fermentas,#SM0311),250 bp DNA ladder Marker(捷瑞,DL250+, 100T),In-FusionTM PCR Cloning Kit(clontech,639626),Taq polymerase(SinoBio,E001-02B),dNTP(Takara,D4030A),Primer(捷瑞生物),限制性内切酶(NEB),Plasmid抽提Kit(Promega,A1460),X-tremeGENE HP DNA Transfection Reagent(Roche,06336236001)等。
1.2 病例来源该研究方案经陆军军医大学大坪医院[TMMU-DPH]的研究伦理委员会审查和批准(2019-183),获得患者同意并签署知情同意书。收集胸外科2016年9月至2019年4月影像学诊断并经病理证实的早期肺腺癌患者54例,排除接受过治疗、患有重大基础疾病或其他恶性肿瘤患者,基线特征见表 1。获取54例蜡块癌组织,15对新鲜癌组织及远癌组织进行后续试验。
| 病例参数 | 统计量 |
| 男:女/例 | 24:30 |
| 年龄/岁 | 60.4±9.1 |
| 肿瘤大小/cm | 2.3±0.7 |
| 分期(1a:1b)/例 | 46:8 |
| 生存时间/d | 582.5±385.9 |
1.3 方法 1.3.1 western blot实验
研磨裂解组织细胞或提取细胞蛋白后BCA法测蛋白浓度,电泳,转膜,相应一抗1 :1 000稀释,孵二抗,ECL显影曝光,Image J软件(1.52a version.USA)进行蛋白条带半定量分析。
1.3.2 组织芯片制作及IHC实验步骤详见文献[8]。将前述54例肺癌样本制作组织芯片,进行BCAR1(1 :50)及MLST8(1 :50)染色。Image J软件计算染色芯片平均光密度,进行相关性分析。
1.3.3 构建人肺腺癌细胞H1975、H1299 BCAR-KO稳定株CRISPR/Cas9实验方案参见文献[11],简述如下:首先将CAS9载体(图 1A)转染H1975及H1299细胞,并通过2 μg/mL嘌呤霉素成功筛选H1975-CAS9及H1299-CAS9稳定株。依据GenBank ID(NM_001170714)的BCAR1序列设计BCAR1-sgRNA及阴性对照CON244-sgRNA干扰靶点序列(表 2),引物退火形成双链DNA,连入sgRNA载体GV371(图 1A),TOP10感受态转化,菌落PCR得到阳性克隆后测序鉴定。再将sgRNA慢病毒质粒转染H1975-CAS9、H1299-CAS9稳定细胞株,通过荧光鉴定构建的BCAR1-KO稳定细胞株后western blot验证BCAR1敲除效果。
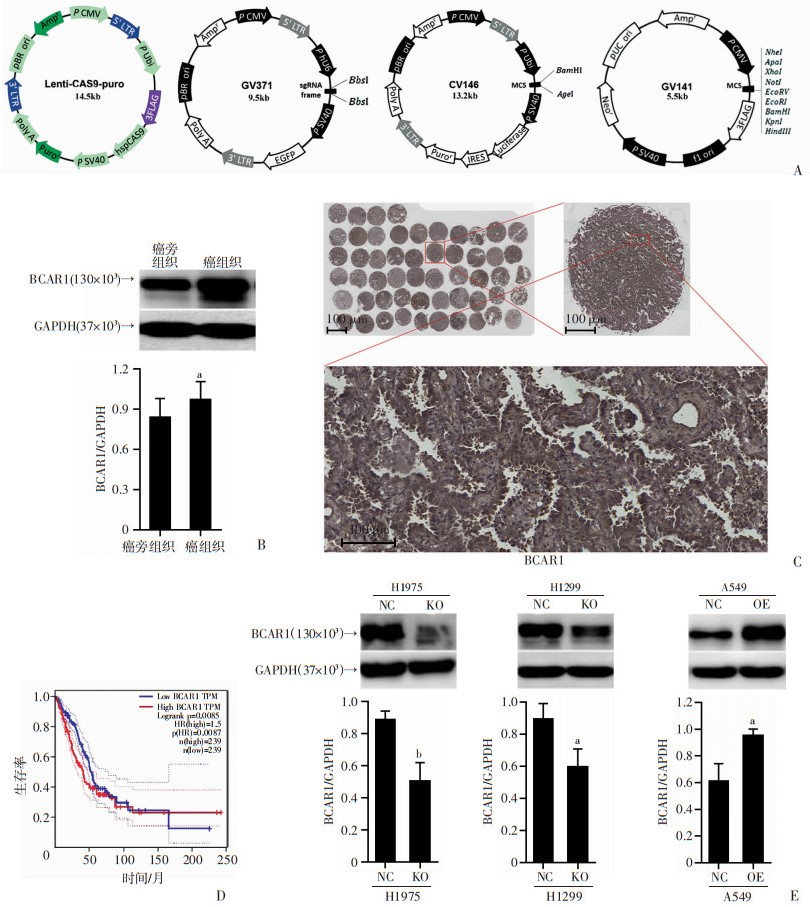
|
| A:载图谱体; B:早期肺腺癌及癌旁组织BCAR1表达,a: P < 0.05, 与癌旁组织比较;C:IHC示BCAR1表达;D:GEPIA数据库肺腺癌患者BCAR1生存分析;E:western blot验证肺腺癌细胞株BCAR1敲除与过表达,a: P < 0.05;b: P < 0.01,与NC组比较 图 1 肺腺癌患者BCAR1表达、预后分析及人肺腺癌细胞系BCAR1敲除与过表达构建 |
| 序号 | 序列5′-3′ |
| BCAR1-sgRNA-a | CACCgCAGCAACGGGTCTCGGCCAT |
| BCAR1-sgRNA-b | aaac ATGGCCGAGACCCGTTGCTGc |
1.3.4 构建人肺腺癌细胞A549 BCAR1-OE稳定株
根据GenBank ID(NM_001170714)的BCAR1基因序列设计引物如表 3,进行PCR扩增目的基因片段,PCR产物插入酶切载体CV146(图 1A)后进行转化,PCR法鉴定菌落并扩增抽提质粒。转染细胞后嘌呤霉素2 μg/mL药筛阳性细胞并扩增,western blot验证BCAR1过表达效果。
| 引物序号 | 序列 |
| BCAR1(45429-1)-P1 | AGGTCGACTCTAGAGGATCCCGCCACCATGCCTGCCAAGCCCTTCCTCTCTTCTG |
| BCAR1(45429-1)-P2 | CACAGGCTAGCTCAACCGGTTCAGGCGGCTGCCAGCTGGCCTAGGACGCGG |
1.3.5 MTT实验
0.25%胰酶消化对数生长期的细胞,制备单细胞悬液,按分组5 000/孔于96孔板接种,接种后0、24、48、72 h每孔加入20 μL MTT试剂(5 mg/mL),培养4 h,加入150 μL DMSO,震荡10 min,酶标仪490 nm测各孔吸光度值。
1.3.6 细胞平板克隆形成实验0.25%胰酶消化对数生长期的细胞,制备单细胞悬液,按分组1 000/皿(10 cm)铺板,37 ℃,5% CO2培养箱培养,直至肉眼可见明显的细胞克隆形成,4%多聚甲醛固定细胞30 min,0.25%结晶紫染色细胞15 min,晾干,拍照,Image J软件分析。
1.3.7 IP-MS实验依据BCAR1基因序列设计引物(表 4)钓取BCAR1,PCR扩增目的基因片段,产物与酶切载体GV141连接反应(图 1A),构建BCAR1过表达质粒并鉴定,质粒抽提,X-tremeGENE与过表达质粒以1 :1比例混合,转染293T细胞,24 h后换液,加入200 μg/mL G418进行药筛5 d获得293T细胞BCAR1过表达细胞株。提取细胞蛋白,BCA法测浓度,Shotgun法进行质谱分析,主要步骤如下:FASP酶解,QExactive质谱仪进行质谱分析,质谱文件转换获得数据。步骤详见文献[12],将获得的BCAR1-NC组蛋白与BCAR1-OE组蛋白进行比对分析。
| 引物序号 | 序列 |
| BCAR1(55742-1)-P1 | ACGGGCCCTCTAGACTCGAGCGCCACCATGCCTGCCAAGCCCTTCCTCTCTTC |
| BCAR1(55742-1)-P2 | AGTCACTTAAGCTTGGTACCGAGGCGGCTGCCAGCTGGCCTAGGAC |
1.3.8 生物信息学分析
TCGA数据库(https://www.cancer.gov/)查询与BCAR1表达显著正相关的基因。搜索限制条件:癌症类型:肺腺癌;位置:支气管、肺;分期:Stage ia,Stage ib;数据库选择:TCGA-LUAD;数据类型:Gene Expression Quantification;方法:RNA-Seq。选取FDR值< 10-6的BCAR1正相关基因与IP-MS结果比对分析,获取BCAR1互作蛋白。
将获得的基因输入PPI String数据库(https://string-db.org/)进行BCAR1信号通路分析,生成拥有多个节点的基因BCAR1信号通路图。
利用GEPIA在线工具(http://gepia.cancer-pku.cn/)对肺癌患者BCAR1基因进行生存分析,验证BCAR1与预后的关系;并获取的基因进行基因差异表达筛选,选取癌组织较癌旁组织显著高表达的基因研究。
1.4 统计学方法采用IBM SPSS Statistics 26.0软件进行数据分析,统计数据以x±s形式表示,计量资料两样本比较使用独立t检验,相关性分析采用两变量相关分析,生存分析采用单因素cox回归模型,P < 0.05(双侧)具有统计学意义。
2 结果 2.1 肺腺癌组织中, BCAR1高表达预后不良如图 1B,western blot示相比癌旁组织,肺腺癌组织中BCAR1显著高表达(P < 0.05);如图 1C, IHC示BCAR1在肺癌细胞质及细胞核中均有表达,BCAR1高表达患者预后差(HR=5.026,P=0.001, 95%CI: 1.948~12.963),如图 1D示,TCGA数据库生存分析亦验证了肺癌患者BCAR1高表达其预后差(HR=1.5,P=0.008 7)。
2.2 人肺腺癌细胞H1975、H1299 BCAR1-KO以及A549BCAR1-OE稳定细胞株成功构建如图 1E,在H1975细胞株及H299中,BCAR1蛋白相对表达水平KO组细胞较NC组分别降低43.43% (P < 0.01)与33.57%(P < 0.05);在A549细胞株中,BCAR1蛋白相对表达OE组较NC组增加1.55倍(P < 0.05)。
2.3 肺癌细胞增殖能力BCAR1-KO后降低,而BCAR1-OE后升高如图 2,在H1975细胞株中,在24、72 h,KO组细胞增殖较NC组均显著降低(P < 0.000 1,P < 0.000 1);在H1299细胞株中,72 h KO组细胞增殖较NC组显著降低(P < 0.000 1);在A549细胞株中,在24、48、72 h,OE组细胞增殖较NC组均显著增加(P < 0.000 1,P < 0.001,P < 0.000 1)。
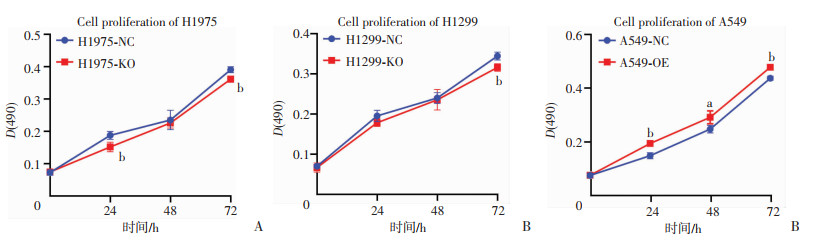
|
| A:H1975;B:H1299;C:A549,a: P < 0.01;b: P < 0.001,与NC组比较 图 2 肺腺癌细胞系BCAR1敲除或过表达对细胞增殖的影响 |
2.4 细胞克隆形成率BCAR1-KO后降低,而BCAR1-OE后升高
如图 3,在H1975细胞株中,细胞克隆形成率KO组细胞较NC组显著降低(P < 0.000 1);但在H1299细胞株中,趋势尚不明显(P>0.05);在A549细胞株中,细胞克隆形成率OE组细胞较NC组细胞显著增加(P < 0.000 1)。
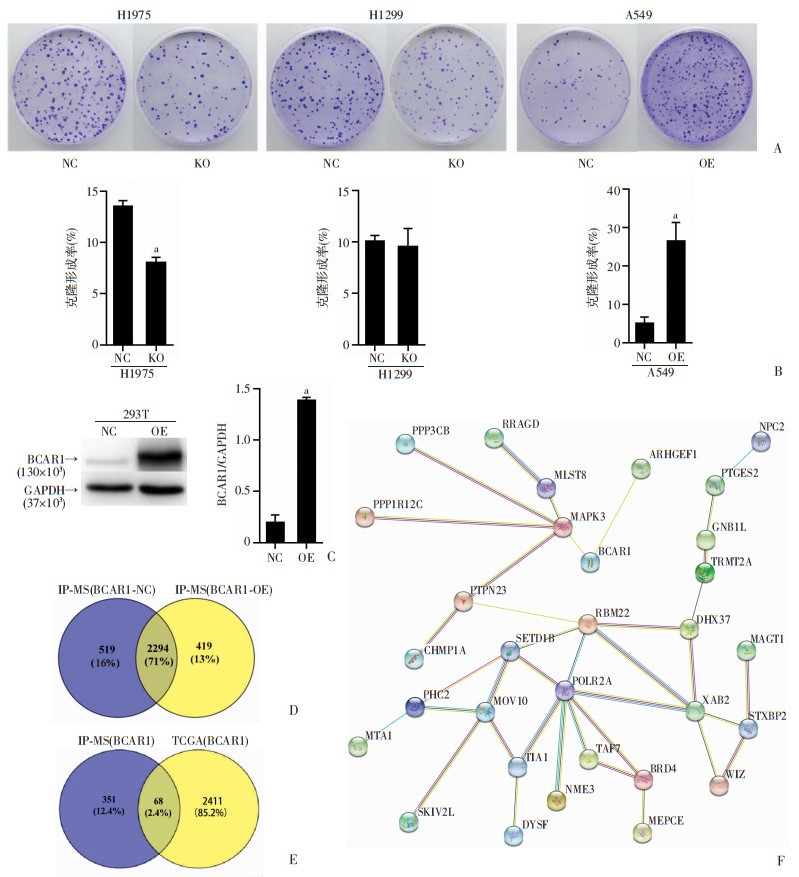
|
| A:细胞克隆形成结晶紫染色结果;B:细胞克隆形成率的比较, a: P < 0.001,与NC组比较;C:293T细胞BCAR1表达western blot验证, a: P < 0.001, 与NC组比较;D:免疫共沉淀质谱分析揭示419个潜在BCAR1作用蛋白; E:TCGA数据库揭示68个潜在BCAR1相互作用蛋白; F:PPI String数据库BCAR1互作蛋白网络分析 图 3 肺腺癌细胞系BCAR1敲除或过表达对克隆形成的影响及BCAR1的互作蛋白网络研究 |
2.5 IP-MS联合生信分析发现MSLT8为BCAR1的关键互作用蛋白
成功构建293T BCAR1过表达细胞株,BCAR1蛋白相对表达水平OE组细胞较NC组显著高表达(P < 0.001,图 3);并且IP-MS实验在293T细胞BCAR1-NC组获得2 813个蛋白(与标签蛋白互相作用),在BCAR1-OE组获得2 713个蛋白(与标签蛋白及BCAR1互相作用,表 5)。比对分析后得到419个蛋白(图 3)仅表达于BCAR1-OE组的潜在BCAR1互作蛋白通过TCGA数据库,得到2 479个与BCAR1表达显著正相关的基因比对分析后获得68个可能与BCAR1相互作用的基因(图 3)。
| 数据库 | 样品 类型 |
总肽段 数目 |
匹配到肽段的 质谱图谱总数 |
肽段数 | 蛋白 数目 |
| 人 | 293T-NC | 29 387 | 17 587 | 13 604 | 2 813 |
| 人 | 293T-OE | 28 021 | 15 823 | 12 884 | 2 713 |
通过PPI String数据库,从68个基因中获得18个拥有多节点的基因,获得信号通路图(图 3)。
GEPIA数据库的差异基因筛选显示,MSLT8,TAF7,GNB1L,PTGES2,DHX37这5个基因在癌组织中的表达显著高于癌旁正常组织(图 4A)。图 3显示MLST8与BCAR1共同连接MAPK3。而我们的前期研究证实,BCAR1是参与MAPK信号通路的衔接蛋白[8],因此以MSLT8为重点进行后续验证。
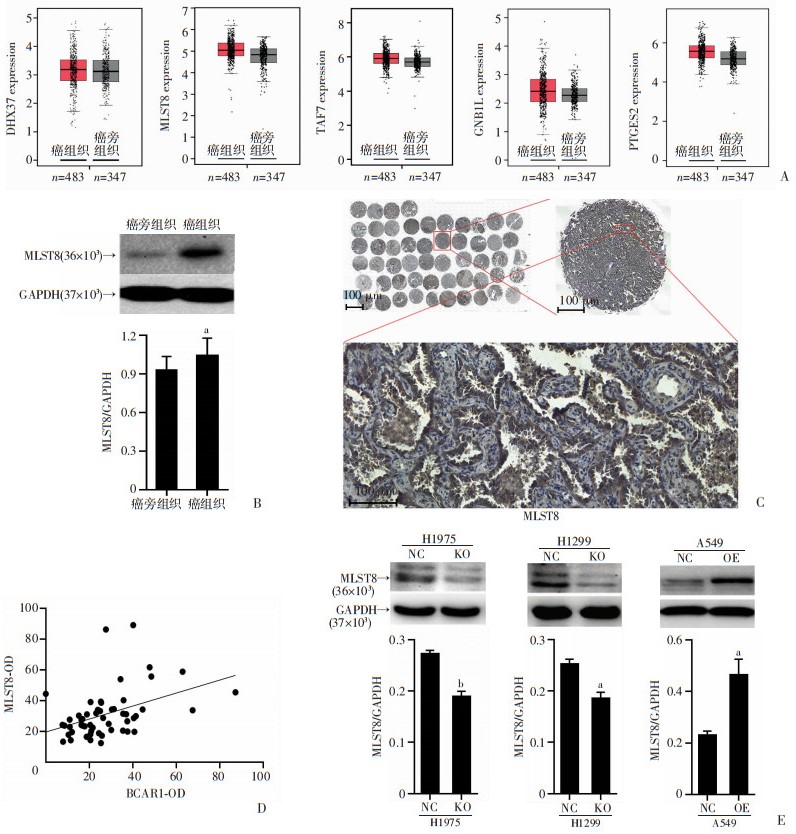
|
| A: GEPIA数据库差异基因表达筛选;B:western blot证实MLST8在肺腺癌组织显著高表达于癌旁正常组织, a: P < 0.05,与癌旁组织比较; C:IHC分析MLST8在肺腺癌组织中的表达情况; D:IHC检验BCAR1与MLST8的相关性; E:BCAR1敲除或过表达前后肺腺癌细胞株验证MLST8的表达情况,a: P < 0.01;b: P < 0.001, 与NC组比较 图 4 临床样本组织和细胞水平验证BCAR1与MLST8的表达关系 |
2.6 在肺癌细胞及组织中MLST8与BCAR1表达密切相关
如图 4B,在肺腺癌组织中,MLST8表达癌组织较癌旁组织显著高表达(P < 0.000 1);如图 4C,IHC示MLST8主要表达于细胞质;如图 4D,BCAR1与MLST8相关性分析显示BCAR1与MLST8具有显著正相关性(R=0.422 1,P=0.001 5)。
2.7 细胞水平western blot验证BCAR1与MLST8表达关系如图 4E,在H1975, H1299人肺腺癌细胞株中,BCAR1-KO后MLST8蛋白表达显著降低(P < 0.001,P < 0.01),在A549细胞株中,BCAR1-OE后MLST8蛋白表达显著增高(P < 0.01)。
3 讨论本研究发现,BCAR1在早期肺癌中亦高表达,且与预后不良密切相关,此结论与乳腺癌的研究结论类似[13]。研究揭示,BCAR1高表达与多种癌症的细胞增殖、存活密切相关[14],特别在乳腺癌中报道较多,而对肺癌细胞增殖及细胞克隆形成的影响却鲜有关注。我们研究发现,在肺癌细胞株中敲除或过表达BCAR1,细胞增殖及克隆形成能力显著降低或增高。在H1299细胞株中,虽然BCAR1敲除未影响克隆形成,考虑与BCAR1在H1299中敲除效果稍差所致,亦有可能在不同细胞背景下,BCAR1生物学功能有所差异。结合A549细胞中BCAR1过表达后,细胞增殖与克隆形成显著增加,我们认为,BCAR1在肺癌的细胞增殖、存活与克隆形成上发挥着重要的作用。
通过IP-MS结合生信分析,我们找到了几个潜在BCAR1互作蛋白,MSLT8、TAF7、GNB1L、PTGES2、DHX37。这些基因在癌组织中表达显著高于正常组织。前期研究显示,磷酸化BCAR1羟基端使得BCAR1与Src激酶结合[15-16],增强Src介导的p130cas和PI3K-AKT-mTOR信号级联反应,MMPs表达增加,促进肿瘤转移[17-18]。MLST8作为一类激酶,在基因注释上与BCAR1功能有很多相似之处。MLST8是哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin,mTOR)的结合蛋白,为激酶激活必不可少的一部分,由它们构成的复合物结构显示:MLST8可以稳定mTOR的活性位点,其表达水平上调激活mTORC1和mTORC2通路而促进肿瘤进展[19-20]。体外研究表明:PSMA与phospho-p130cas、phospho-c-Src中的FLNa结合,激活AKT/mTOR/BAD和MAPK途径,维持细胞的增殖和抗凋亡作用[21]。而我们从PPI String获得的信号通路中显示,BCAR1与MLST8紧邻着MAPK3相连接,而我们课题组前期研究证实BCAR1是参与MAPK信号通路的衔接蛋白[8],故MLST8被作为BCAR1的关键互作蛋白进行研究。
本研究通过临床与细胞水平证实,BCAR1与MLST8显著相关,MLST8随着BCAR1的变化在细胞水平出现相应的趋势变化,在BCAR1敲除后低表达,在BCAR1过表达后高表达,揭示MLST8可能为BCAR1互作的下游调节蛋白。但是,BCAR1与MLST8发生作用的具体机制仍不清楚。下一步,我们将通过免疫共沉淀或Duolink PLA实验研究BCAR1与MLST8相互关系,以及调控MLST8对于BCAR1功能的影响。总之,BCAR1及MLST8有望成为肺癌治疗的新靶点。
| [1] |
SAWADA Y, TAMADA M, DUBIN-THALER B J, et al. Force sensing by mechanical extension of the Src family kinase substrate p130Cas[J]. Cell, 2006, 127(5): 1015-1026. DOI:10.1016/j.cell.2006.09.044 |
| [2] |
GEIGER B. A role for p130Cas in mechanotransduction[J]. Cell, 2006, 127(5): 879-881. DOI:10.1016/j.cell.2006.11.020 |
| [3] |
HEUMANN A, HEINEMANN N, HUBE-MAGG C, et al. High BCAR1 expression is associated with early PSA recurrence in ERG negative prostate cancer[J]. BMC Cancer, 2018, 18(1): 37. DOI:10.1186/s12885-017-3956-3 |
| [4] |
YIN X Y, YANG X, CUI Y, et al. Expression of three proteins in endometrioid adenocarcinoma and their significance in clinical nursing, diagnosis and treatment[J]. Eur Rev Med Pharmacol Sci, 2018, 22(1): 55-61. DOI:10.26355/eurrev_201801_14100 |
| [5] |
YAGINUMA T, GAO J, NAGATA K, et al. p130Cas induces bone invasion by oral squamous cell carcinoma by regulating tumor epithelial-mesenchymal transition and cell proliferation[J]. Carcinogenesis, 2020, 41(8): 1038-1048. DOI:10.1093/carcin/bgaa007 |
| [6] |
DENG B, HUANG W, TAN Q Y, et al. Breast cancer anti- estrogen resistance protein 1 (BCAR1/p130cas) in pulmonary disease tissue and serum[J]. Mol Diagn Ther, 2011, 15(1): 31-40. DOI:10.1007/BF03257191 |
| [7] |
DENG B, SUN Z F, JASON W, et al. Increased BCAR1 predicts poor outcomes of non-small cell lung cancer in multiple- center patients[J]. Ann Surg Oncol, 2013, 20(Suppl 3): S701-S708. DOI:10.1245/s10434-013-3184-2 |
| [8] |
HUANG W, DENG B, WANG R W, et al. BCAR1 protein plays important roles in carcinogenesis and predicts poor prognosis in non-small-cell lung cancer[J]. PLoS ONE, 2012, 7(4): e36124. DOI:10.1371/journal.pone.0036124 |
| [9] |
DENG B, TAN Q Y, WANG R W, et al. P130cas is required for TGF-β1-mediated epithelial-mesenchymal transition in lung cancer[J]. Oncol Lett, 2014, 8(1): 454-460. DOI:10.3892/ol.2014.2123 |
| [10] |
GEMPERLE J, DIBUS M, KOUDELKOVA L, et al. The interaction of p130Cas with PKN3 promotes malignant growth[J]. Mol Oncol, 2019, 13(2): 264-289. DOI:10.1002/1878-0261.12401 |
| [11] |
HUDACSEK V, GYORFFY B. Genome engineering using the CRISPR-Cas9 system and applications in cancer research[J]. Magy Onkol, 2018, 62(2): 119-127. |
| [12] |
MACCARRONE G, BONFIGLIO J J, SILBERSTEIN S, et al. Characterization of a protein interactome by Co-immunoprecipitation and shotgun mass spectrometry[J]. Methods Mol Biol, 2017, 1546: 223-234. DOI:10.1007/978-1-4939-6730-8_19 |
| [13] |
GEMPERLE J, HEXNEROVÁ R, LEPŠÍK M, et al. Structural characterization of CAS SH3 domain selectivity and regulation reveals new CAS interaction partners[J]. Sci Rep, 2017, 7(1): 8057. DOI:10.1038/s41598-017-08303-4 |
| [14] |
CAMACHO LEAL M D P, COSTAMAGNA A, TASSONE B, et al. Conditional ablation of p130Cas/BCAR1 adaptor protein impairs epidermal homeostasis by altering cell adhesion and differentiation[J]. CellCommun Signal, 2018, 16(1): 73. DOI:10.1186/s12964-018-0289-z |
| [15] |
CAMACHO LEAL M D E L P, SCIORTINO M, TORNILLO G, et al. p130Cas/BCAR1 scaffold protein in tissue homeostasis and pathogenesis[J]. Gene, 2015, 562(1): 1-7. DOI:10.1016/j.gene.2015.02.027 |
| [16] |
RADULOVIĆC P, KRUŠLIN B. Immunohistochemical expression of NEDD9, E-cadherin and γ-catenin and their prognostic significance in pancreatic ductal adenocarcinoma (PDAC)[J]. Bosn J Basic Med Sci, 2018, 18(3): 246-251. DOI:10.17305/bjbms.2018.2378 |
| [17] |
TOMITA Y, DORWARD H, YOOL A J, et al. Role of aquaporin 1 signalling in cancer development and progression[J]. Int J Mol Sci, 2017, 18(2): E299. DOI:10.3390/ijms18020299 |
| [18] |
PALLARÈS V, HOYOS M, CHILLÓN M C, et al. NEDD9, an independent good prognostic factor in intermediate- risk acute myeloid leukemia patients[J]. Oncotarget, 2017, 8(44): 76003-76014. DOI:10.18632/oncotarget.18537 |
| [19] |
YU X N, ZHANG G C, SUN J L, et al. Enhanced mLST8 expression correlates with tumor progression in hepatocellular carcinoma[J]. Ann Surg Oncol, 2020, 27(5): 1546-1557. DOI:10.1245/s10434-020-08263-6 |
| [20] |
XU Y Q, HUANG Y N, WENG L H, et al. Effects of single-nucleotide polymorphisms in the mTORC1 pathway on the risk of brain metastasis in patients with non-small cell lung cancer[J]. J Cancer Res Clin Oncol, 2020, 146(1): 273-285. DOI:10.1007/s00432-019-03059-y |
| [21] |
PERICO M E, GRASSO S, BRUNELLI M, et al. Prostate-specific membrane antigen (PSMA) assembles a macromolecular complex regulating growth and survival of prostate cancer cells "in vitro" and correlating with progression "in vivo"[J]. Oncotarget, 2016, 7(45): 74189-74202. DOI:10.18632/oncotarget.12404 |






