2. 400038重庆,陆军军医大学(第三军医大学)军事预防医学系军队流行病学教研室;
3. 400037 重庆,陆军军医大学(第三军医大学)第二附属医院:胸外科,全军呼吸内科研究所,全军呼吸病研究重点实验室;
4. 400037 重庆,陆军军医大学(第三军医大学)第二附属医院:全军呼吸内科研究所,全军呼吸病研究重点实验室
2. Department of Epidemiology, Faculty of Military Preventive Medicine, Army Medical University (Third Military Medical University), Chongqing, 400038;
3. Department of Thoracic Surgery, Second Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400037, China;
4. Institute of Respiratory Diseases, Key Laboratory of Respiratory Diseases, Second Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400037, China
肺癌是全世界发病率和病死率较高的恶性肿瘤之一[1-2]。在所有肺癌中非小细胞肺癌(non-small cell lung cancer,NSCLC)的比例约为85%,其中肺腺癌是占比最高的非小细胞肺癌[3]。肺癌的5年生存率仅为15%左右[4]。肺癌属于异质性较强的一种疾病,患者的预后因素即使相近,其生存时间和生存质量也不一定相同。
随着基因组学和高通量测序的兴起,以及个性化医疗的逐渐开展,传统意义上的肺癌预后因素,如疾病的病理类型、临床分期等已经不能满足精准医疗的需求。长链非编码RNA(long non-coding RNAs,lncRNAs)是一类不参与编码蛋白质,长度超过200个核苷酸的转录产物,具有多种生物学功能[5]。目前发现的多种癌症相关的lncRNAs具有抑癌或者促癌功能,而且其特殊的组织表达特征,使其有望成为新的肿瘤生物标志物和治疗靶点。本课题组前期利用表达谱芯(Affymetrix GeneChip HumanTranscriptome Array 2.0)筛选并重点关注了在肺癌中表达下调倍数较高的lncRNA SFTA1P,但是其在肺癌中的作用和机制尚不清楚。本研究旨在通过分析非小细胞肺癌患者SFTA1P的表达差异与临床预后的关联,探索SFTA1P在肺腺癌预后预测中的应用价值,为临床决策提供新的理论依据。
1 资料与方法 1.1 临床资料和样本 1.1.1 病例的纳入以医院人群为基础,2014年11月至2015年3月从陆军军医大学第二附属医院胸外科收集病例,共计62例。肺癌的诊断和病理分型参考国际统一的标准。纳入研究对象的民族限定为汉族,排除有慢病史和有合并症的肺癌患者。本研究经过陆军军医大学第二附属医院医学伦理委员会审批(伦审研第2015030号)。
1.1.2 流行病学和临床资料收集设计统一规范的调查表,对纳入病例的人口学信息、吸烟情况、其他危险因素暴露史及家族史等进行详细的调查,并摘录临床资料信息。
1.1.3 患者组织样本的获取及保存在患者第1次接受手术中收集组织样本,且在任何放射或化疗前收集组织样本。在手术过程中切取患者一小块肺癌组织,同时切取一小块距离癌组织>5 cm的远端肺部正常组织作为对照。样本取出后,液氮冻存。
1.1.4 组织标本的病理检查切取一小块约200 mg组织,经过冰冻切片后,进行HE染色。针对组织切片,病理科医师进行镜下观察,确认肺癌的类型以及组织中肺癌细胞百分比。最终选择肺癌细胞百分比>70%的组织作为肺癌病例组,选择肺癌细胞百分比为0%的癌旁组织作为对照组。
1.1.5 公共数据库数据来源本研究预后分析数据来自癌症基因组图谱数据库(TCGA,https://tcga-data.nci.nih.gov/)的肺腺癌患者数据。共纳入了471例肺腺癌患者的临床信息,提取的临床数据包括年龄、性别、病理分期、吸烟史、放疗、分子靶向治疗和SFTA1P的表达水平。
1.2 实时荧光定量PCR按照TRIzol试剂的使用说明书,提取组织总RNA,对获得的RNA进行定量及纯度的鉴定。采用两步法逆转录RNA,第1步反应体系为5×g DNA Eraser Buffer:2 μL,gDNA Eraser:1 μL,目标RNA:1 μg,用RNase Free dH2O补足10 μL。于42 ℃反应2 min,去除RNA中可能存在的杂质DNA。第2步反应体系为第1步反应混合物:10 μL,PrimeScript RT Enzyme Mix Ⅰ:1 μL,RT Primer Mix:1 μL,5×PrimeScript Buffer 2:4 μL,RNase Free dH2O:4 μL。最终体积为20 μL,反应条件为37 ℃ 15 min,85 ℃ 15 s,目的是将RNA逆转录为cDNA。SFTA1P的引物序列上游5′-CAGCATTCCAGGTGGGCTTT-3′,下游5′-TTGTTTGGCTTACTCGTGCC-3′。β-actin为内参基因,引物序列上游5′-GCAGAGAAGCTGGTGCTCCGTA-3′,下游5′-CAGCATGTTGGACTCGATGCAG-3′。反应体系为SYBR Premix Ex Taq Ⅱ(Tli RNaseH Plus)(2X):12.5 μL,上游引物(10 μmol/L):1 μL,下游引物(10 μmol/L):1 μL,cDNA:1 μL,DEPC水:9.5 μL,体系终体积为25 μL。反应条件为第1步95 ℃ 30 s,第2步95 ℃ 5 s,60 ℃ 30 s,共40个循环。SFTA1P的表达量采用2-ΔΔCt表示,分别计算实验组、对照组的ΔCt值(ΔCt=CtSFTA1P-Ctβ-actin),而ΔΔCt=ΔCt实验组-ΔCt对照组,最后计算2-ΔΔCt得到SFTA1P的表达量。
1.3 统计学分析 1.3.1 差异检验服从正态分布的资料用x±s表示,癌组织和癌旁正常组织的统计分析用配对t检验,对于SFTA1P的表达水平与性别、年龄以及病理分期的比较采用t检验。
1.3.2 预后预测模型的构建及验证首先通过单因素Cox比例风险回归分析,将年龄、性别、吸烟史以及P<0.2的变量纳入后续的多因素Cox比例风险回归分析。通过后退法构建最终模型,基于赤池信息准则(Akaike information criterion,AIC)最小为终止条件。
在预后分析中,用X-tile软件[6]将TCGA数据库中患者的SFTA1P表达量分成高水平组和低水平组。X-tile软件能实现基于结局的界值优化,全局性的展示连续性指标分为高低表达组的各种情况。此软件不仅能提供校正的Monte Carlo P值,还能避免多重界值选择带来的问题。根据X-tile的分组,用log-rank检验比较不同SFTA1P水平的总生存期(overall survival,OS)。
预测模型进行内部验证采用Bootstrap重抽样法。一致性指数(concordance index,C-index)作为评价预测模型的区分度指标。C-index的数值范围在0.5~1.0之间,C-index=0.5代表随机结果,说明模型没有预测效果,C-index =1.0代表模型能正确地预测预后结果,说明模型预测结果与实际结果能完全一致。通过校准曲线评价模型的校准度,校准曲线是实际发生概率与预测概率的散点图。
在多因素分析时,将年龄和P<0.05的变量用于生成列线图。通过计算多因素Cox回归模型中每个变量的方差膨胀因子(variance inflation factor, VIF),用以检测纳入变量之间的多重共线性,所有变量的VIF均<2.0,因此认为在本研究多因素Cox回归模型中纳入变量之间的共线性可以忽略不计。采用R 3.6.2(http://www.r-project.org)软件进行统计分析。采用GraphPad Prism 7.0软件绘制Kaplan-Meier曲线。P<0.05为差异有统计学意义。
2 结果 2.1 临床样本中SFTA1P表达水平的变化与临床预后的关联本研究收集的62例非小细胞肺癌患者的临床基本信息见表 1,患者年龄为(56.23±8.68)岁。进一步的分析发现,SFTA1P在NSCLC组织中的表达水平与患者的各项指标(年龄、性别、NSCLC病理类型、肿瘤分期)之间无显著关联(P>0.05,表 1)。利用qRT-PCR检测收集的62对NSCLC组织以及相应的癌旁正常肺组织中SFTA1P的表达情况,结果见图 1,相比于癌旁正常肺组织(1.765±0.149),SFTA1P在肺癌组织(0.692±0.103)中表达显著下调(P<0.05)。
| 临床特征 | n | SFTA1P相对表达量 | t值 | P值 |
| 年龄/岁 | ||||
| ≥60 | 24 | 0.885±1.152 | ||
| <60 | 38 | 0.571±0.477 | 1.495 | 0.140 |
| 性别 | ||||
| 男 | 43 | 0.762±0.899 | ||
| 女 | 19 | 0.534±0.566 | 1.016 | 0.314 |
| 病理分期 | ||||
| Ⅰ期 | 31 | 0.702±0.951 | ||
| Ⅱ/Ⅲ/Ⅳ期 | 31 | 0.523±0.475 | 1.260 | 0.213 |
| 吸烟史 | ||||
| 吸烟 | 18 | 0.783±0.913 | ||
| 从未吸烟 | 42 | 0.518±0.545 | 1.170 | 0.247 |
| 缺失值 | 2 |
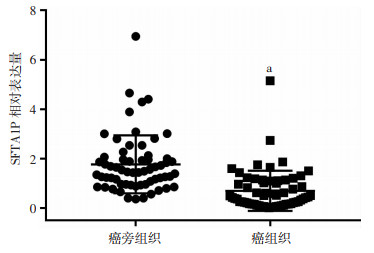
|
| a:P<0.05,与正常的癌旁组织比较 图 1 SFTA1P在肺癌组织与正常肺组织中的表达差异 |
2.2 肺腺癌预后预测模型的构建
TCGA数据集中肺腺癌患者的人口学统计特征及一般临床信息见表 2。共有471例肺腺癌患者,年龄(65.77±9.96)岁,随访时间0~6 812 d,中位随访时间448 d。
| 临床特征 | 患者(n=471) |
| 中位随访时间/d | 448(0~6 812) |
| 年龄(x±s)/岁 | 65.77±9.96 |
| 性别/n(%) | |
| 男性 | 215(45.6) |
| 女性 | 256(54.4) |
| 吸烟史 | |
| 吸烟 | 393(83.4) |
| 不吸烟 | 65(13.8) |
| 缺失值 | 13(2.8) |
| 病理分期 | |
| Ⅰ | 254(53.9) |
| Ⅱ | 112(23.8) |
| Ⅲ/Ⅳ | 104(22.1) |
| 缺失值 | 1(0.2) |
| 放疗 | |
| 是 | 49(10.4) |
| 否 | 313(66.5) |
| 缺失值 | 109(23.1) |
| 分子靶向治疗 | |
| 是 | 122(25.9) |
| 否 | 238(50.5) |
| 缺失值 | 111(23.6) |
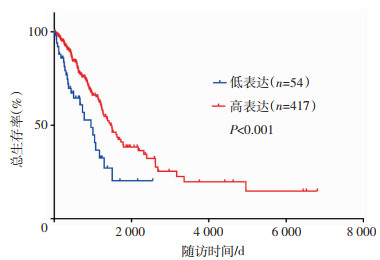
将471例肺腺癌患者的SFTA1P表达量分为高低两个组。其中,高水平组417人,低水平组54人。Kaplan- Meier曲线及log-rank检验结果表明,SFTA1P高表达水平组的OS预后较好(P<0.001),如图 2。
|
| 图 2 肺腺癌患者不同SFTA1P水平的Kaplan-Meier曲线 |
对肺腺癌患者,采用Cox比例风险回归模型对影响其OS的各变量进行单因素或者是多因素的分析,结果见表 3。Cox模型筛选出病理分期(P<0.001)、放疗(P=0.002)、分子靶向治疗(P=0.186)、SFTA1P表达水平(P=0.001)4个协变量,加上年龄、性别、吸烟史3个与肺癌密切相关的变量作为多因素Cox模型分析的协变量。校正上述协变量后,结果表明SFTA1P表达水平仍然是OS独立的预后因素(P=0.017;HR=0.535,95%CI:0.320~0.895),此外病理分期以及放疗也是患者预后的独立影响因素。最后构建的肺腺癌预后预测模型的公式为:H=0.019*A+0.954*P1+1.266*P2+0.607*R-0.625*S,其中H是相对危险度的对数值,A为年龄,P为病理分期,R为放疗,S为SFTA1P表达水平。
| 协变量 | 单因素分析 | 多因素分析 | |||
| HR(95%CI) | P值 | HR(95%CI) | P值 | ||
| 年龄 | 1.013(0.996~1.031) | 0.143 | 1.019(0.999~1.040) | 0.069 | |
| 性别 | |||||
| 女性 | 参照 | 参照 | |||
| 男性 | 1.023(0.730~1.434) | 0.894 | 0.980(0.650~1.477) | 0.924 | |
| 分期 | |||||
| Ⅰ | 参照 | 参照 | |||
| Ⅱ | 2.223(1.433~3.450) | <0.001 | 2.595(1.574~4.277) | <0.001 | |
| Ⅲ/Ⅳ | 3.983(2.673~5.937) | <0.001 | 3.574(2.119~5.939) | <0.001 | |
| 吸烟史 | |||||
| 不吸烟 | 参照 | 参照 | |||
| 吸烟 | 0.917(0.542~1.551) | 0.746 | 1.056(0.589~1.891) | 0.856 | |
| 放疗 | |||||
| 否 | 参照 | 参照 | |||
| 是 | 2.052(1.299~3.242) | 0.002 | 1.835(1.024~3.289) | 0.041 | |
| 分子靶向治疗 | |||||
| 否 | 参照 | 参照 | |||
| 是 | 1.322(0.875~1.997) | 0.186 | 0.616(0.366~1.035) | 0.067 | |
| SFTA1P表达水平 | |||||
| 低表达 | 参照 | 参照 | |||
| 高表达 | 0.471(0.308~0.720) | 0.001 | 0.535(0.320~0.895) | 0.017 | |
2.3 肺腺癌患者预后预测模型的内部验证
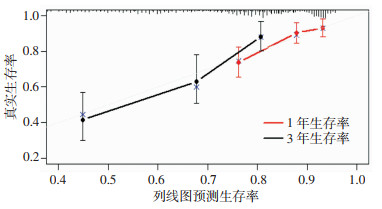
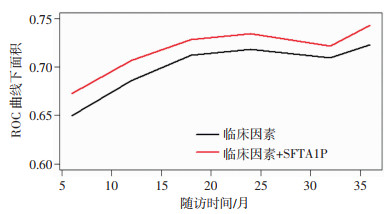
用简单Bootstrap法,对预测模型的区分度和校准度进行内部验证。区分度C-index为0.69,95%置信区间为0.63~0.75。针对1年和3年生存率所绘制的校准曲线显示了良好的预测效能(图 3)。此外,分别用临床信息和临床信息加上SFTA1P表达水平进行预后分析,比较两者的ROC曲线下面积(AUC),后者模型预测结局的效能趋于好转(图 4),可见SFTA1P表达水平增加了临床信息预测的准确度。
|
| 图 3 肺腺癌患者预后预测列线图的校准曲线 |
|
| 临床因素包括年龄、病理分期、放疗 图 4 时间依赖性AUC曲线比较 |
2.4 肺腺癌患者预后预测的列线图
为了方便临床运用,用上述预测模型构建了列线图(图 5)。在列线图中将一个样本对应指标的分值相加,得到的总分就可对应得到1年以及3年的生存率。如此次研究中某患者的年龄为65岁(5.5分)、病理分期Ⅲ期(9.65分)、进行了放疗(3.75分)、SFTA1P表达水平为低表达(5.75分),则这名患者的总分为24.65分,对应得到1年生存率>50%,3年生存率>10%。
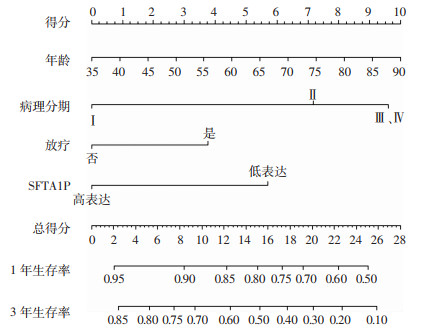
|
| 图 5 结合SFTA1P表达水平的TCGA数据集肺腺癌患者预后评估列线图 |
3 讨论
临床医师常常根据患者的年龄、病理分期、类型、肿瘤分化以及治疗措施等常规的临床指标判断肺癌的预后[7-9]。近年来,分子生物标志物作为影响肺癌的预后因素,开始逐渐被关注。LncRNA在癌症发生发展中的作用也越来越被重视,多项研究[10-12]发现其可以作为癌症预后的重要生物标志物。在癌症的不同病理分期中,有些lncRNA会呈现出特异性的表达模式,而且有些lncRNA还会在特定的组织中特异性表达,比如血浆。这些研究结果提示lncRNA可以作为预后预测的潜在分子标志物。
本研究发现了一个在肺癌组织中显著下调的lncRNA SFTA1P,SFTA1P全长693 bp,有2个外显子,位于10p14染色体区域。有关SFTA1P在肿瘤中的作用研究还较少,在MA等[13]研究中可知SFTA1P抑制了胃癌细胞的增殖、迁移和侵袭。本研究通过qRT-PCR技术检测SFTA1P的表达水平,结果显示对比癌旁的正常肺组织,SFTA1P在肺癌组织中的表达显著下调。课题组之前的研究[14]结果也显示,在细胞水平,SFTA1P能够抑制肺癌细胞的增殖、迁移和侵袭。这与ZHANG等[15]的研究结果相一致,提示SFTA1P在肺癌的发生发展中具有抑癌作用,可能是一个良性的预后因素。
临床预后预测模型[16]作为一种定量评估预后风险的工具,能够提供更加客观、准确的信息给医师以及患者,其应用越来越普遍。列线图属于临床预后预测模型的一种,能够将与患者生存相关的参数可视化,可以比较准确地计算出每个患者的生存率[16-18]。LIANG等[19]和WANG等[20]提到建立关于手术切除后的非小细胞肺癌患者以及基于临床特征和常规实验室检测的晚期非小细胞肺癌患者个体生存预测的列线图,但这些研究都只是关注患者的一般信息和临床信息,并未涉及到生物标记物。本研究经过多因素Cox分析,校正了性别、吸烟史、病例分期、临床治疗等因素后,结果表明SFTA1P高表达水平的患者总生存期较长,提示SFTA1P是肺癌独立的预后因素。因此,本研究利用临床信息和lncRNA SFTA1P表达水平构建了肺腺癌预后预测模型,模型内部验证显示具有较好的区分度和校准度。AUC比较显示SFTA1P表达水平增加了临床信息预测的准确度。同时基于构建的预测模型绘制了预测肺腺癌1年和3年生存率的列线图,以方便临床实践中运用。
本研究还存在一些不足,运用TCGA公共数据库的数据存在一些临床信息的缺失,可能会导致模型中预测因子的筛选偏倚。尽管本研究对预测模型的区分度和校准度进行了内部验证,但还缺乏独立样本的外部验证,以评价模型的可推广性。
综上,本研究发现SFTA1P在肺癌组织中表达显著下调,而且是肺腺癌患者的独立预后因素,SFTA1P表达水平与临床变量共同构建的列线图模型在一定程度上能方便预测肺腺癌患者的生存,可能对肺癌的预后预测是一个有益的补充。
| [1] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [2] |
SIEGEL R L, MILLER K D, JEMAL A. Cancer statistics, 2018[J]. CA Cancer J Clin, 2018, 68(1): 7-30. DOI:10.3322/caac.21442 |
| [3] |
ETTINGER D S, AKERLEY W, BORGHAEI H, et al. Non-small cell lung cancer, version 2.2013[J]. J Natl Compr Canc Netw, 2013, 11(6): 645-653. DOI:10.6004/jnccn.2013.0084 |
| [4] |
WENDER R, FONTHAM E T, BARRERA E Jr, et al. American Cancer Society lung cancer screening guidelines[J]. CA Cancer J Clin, 2013, 63(2): 107-117. DOI:10.3322/caac.21172 |
| [5] |
CHEN Y, LI C, PAN Y, et al. The emerging role and promise of long noncoding RNAs in lung cancer treatment[J]. Cell Physiol Biochem, 2016, 38(6): 2194-2206. DOI:10.1159/000445575 |
| [6] |
CAMP R L, DOLLED-FILHART M, RIMM D L. X-tile: a new bio-informatics tool for biomarker assessment and outcome-based cut-point optimization[J]. Clin Cancer Res, 2004, 10(21): 7252-7259. DOI:10.1158/1078-0432.CCR-04-0713 |
| [7] |
韦尉东, 傅剑华, 林鹏, 等. 非小细胞肺癌完全切除及综合治疗后预后预测模型的建立与评价[J]. 中华肿瘤杂志, 2008, 30(9): 672-675. WEI W D, FU J H, LIN P, et al. Establishment and evaluation of a prognostic prediction model for patients after complete resection of non-small cell lung cancer and multidisciplinary therapy[J]. Chin J Oncol, 2008, 30(9): 672-675. |
| [8] |
HU Z G, HU K, LI W X, et al. Prognostic factors and nomogram for cancer-specific death in non small cell lung cancer with malignant pericardial effusion[J]. PLoS ONE, 2019, 14(5): e0217007. DOI:10.1371/journal.pone.0217007 |
| [9] |
DENG J Q, REN Z P, WEN J X, et al. Construction of a nomogram predicting the overall survival of patients with distantly metastatic non-small-cell lung cancer[J]. Cancer Manag Res, 2018, 10: 6143-6156. DOI:10.2147/CMAR.S183878 |
| [10] |
XIONG Y W, ZHANG X Y, LIN Z H, et al. SFTA1P, LINC00968, GATA6-AS1, TBX5-AS1, and FEZF1-AS1 are crucial long non-coding RNAs associated with the prognosis of lung squamous cell carcinoma[J]. Oncol Lett, 2019, 18(4): 3985-3993. DOI:10.3892/ol.2019.10744 |
| [11] |
ZHANG E B, YIN D D, SUN M, et al. P53-regulated long non-coding RNA TUG1 affects cell proliferation in human non-small cell lung cancer, partly through epigenetically regulating HOXB7 expression[J]. Cell Death Dis, 2014, 5: e1243. DOI:10.1038/cddis.2014.201 |
| [12] |
SUN M, LIU X H, LU K H, et al. EZH2-mediated epigenetic suppression of long noncoding RNA SPRY4-IT1 promotes NSCLC cell proliferation and metastasis by affecting the epithelial-mesenchymal transition[J]. Cell Death Dis, 2014, 5: e1298. DOI:10.1038/cddis.2014.256 |
| [13] |
MA H W, MA T S, CHEN M, et al. The pseudogene-derived long non-coding RNA SFTA1P suppresses cell proliferation, migration, and invasion in gastric cancer[J]. Biosci Rep, 2018, 38(2): BSR20171193. DOI:10.1042/BSR20171193 |
| [14] |
周玫余, 袁帅, 向颖, 等. 长链非编码RNA SFTA1P在非小细胞肺癌组织中的表达和细胞功能研究[J]. 中华疾病控制杂志, 2020, 24(3): 341-347. ZHOU M Y, YUAN S, XIANG Y, et al. Expression and cellular function of long non-coding RNA SFTA1P in non-small cell lung cancer[J]. Chin J Dis Control Prev, 2020, 24(3): 341-347. DOI:10.16462/j.cnki.zhjbkz.2020.03.018 |
| [15] |
ZHANG H, XIONG Y Q, XIA R, et al. The pseudogene-derived long noncoding RNA SFTA1P is down-regulated and suppresses cell migration and invasion in lung adenocarcinoma[J]. Tumour Biol, 2017, 39(2): 1010428317691418. DOI:10.1177/1010428317691418 |
| [16] |
ZHOU Z R, WANG W W, LI Y, et al. In-depth mining of clinical data: the construction of clinical prediction model with R[J]. Ann Transl Med, 2019, 7(23): 796. DOI:10.21037/atm.2019.08.63 |
| [17] |
PAN H, SHI X S, XIAO D K, et al. Nomogram prediction for the survival of the patients with small cell lung cancer[J]. J Thorac Dis, 2017, 9(3): 507-518. DOI:10.21037/jtd.2017.03.121 |
| [18] |
IASONOS A, SCHRAG D, RAJ G V, et al. How to build and interpret a nomogram for cancer prognosis[J]. J Clin Oncol, 2008, 26(8): 1364-1370. DOI:10.1200/JCO.2007.12.9791 |
| [19] |
LIANG W H, ZHANG L, JIANG G N, et al. Development and validation of a nomogram for predicting survival in patients with resected non-small-cell lung cancer[J]. J Clin Oncol, 2015, 33(8): 861-869. DOI:10.1200/JCO.2014.56.6661 |
| [20] |
WANG T, LU R, LAI S, et al. Development and validation of a nomogram prognostic model for patients with advanced non-small-cell lung cancer[J]. Cancer Inform, 2019, 18: 1176935119837547. DOI:10.1177/1176935119837547 |



