2. 400038 重庆,陆军军医大学(第三军医大学)第一附属医院:老年病科;
3. 400038 重庆,陆军军医大学(第三军医大学)第一附属医院:药剂科
2. Department of Geriatrics, First Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400038, China;
3. Department of Pharmacy, First Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400038, China
碳青霉烯耐药肠杆菌科细菌感染可显著增加患者的病死率。根据我国细菌耐药监测网数据,肠杆菌科细菌对碳青霉烯类药物的耐药率逐年增高,尤其是肺炎克雷伯菌,其对碳青霉烯类耐药率已超过20%[1]。而目前针对碳青霉烯耐药肠杆菌科细菌感染的治疗仍是临床治疗的难题,2015年美国食品药品监督管理局(FDA)批准上市的头孢他啶/阿维巴坦仅对KPC或OXA-48阳性肠杆菌科细菌具有较好的疗效,而对金属酶阳性菌株的治疗效果与其他药物相比并无显著优势[2]。
对于产金属酶的菌株,目前多采取替加环素和多粘菌素为核心的联合抗菌药物治疗[3]。然而,替加环素和多粘菌素具有较高的不良反应,因此,探索其他的药物治疗方案非常重要。氨曲南是单环β-内酰胺酶类的抗生素,具有第3代头孢菌素和第4代青霉素的特点,且对导致碳青霉烯耐药的金属酶和KPC酶稳定,可能与头孢他啶/阿维巴坦联用对耐药细菌有较好的协同作用。据此,本研究拟以临床收集的碳青霉烯高耐药肺炎克雷伯菌为探究对象,研究头孢他啶/阿维巴坦与氨曲南联合对临床分离株的协同作用。
1 材料与方法 1.1 实验菌株从本院收集碳青霉烯耐药肺炎克雷伯菌,通过药敏实验筛选亚胺培南的最低抑菌浓度(MIC)值≥8 μg/mL的菌株10株。
1.2 主要试剂及仪器头孢他啶(批号:MB1334)、头孢曲松(批号:MB1333-S)、头孢吡肟(批号:MB1760-S)、庆大霉素(批号:MB1331-S)、妥布霉素(批号:MB1250-S)、阿米卡星(批号:MB1012-S)、环丙沙星(批号:MB1283-S)、左氧氟沙星(批号:MB1576-S)、氨曲南(批号:MB1025-S)、亚胺培南(批号:MB1457)、美罗培南(批号:MB1129-S)、哌拉西林(批号:MB9665-S)、他唑巴坦(批号:MB3117-S)、阿维巴坦(批号:MB2494)购于大连美仑生物技术有限公司,M-H琼脂购自北京索莱宝科技有限公司,细菌DNA提取纯化试剂盒、细菌RNA提取纯化试剂盒和2×Pfu PCR MasterMix购自北京天根生化科技有限公司,琼脂糖购自上海生工生物工程股份有限公司,逆转录试剂盒和2×SYBR Green MasterMix购自上海东洋纺生物科技有限公司,引物合成由上海生工生物工程股份有限公司完成。细菌多点接种仪购自日本Sakuma公司,PCR扩增仪、荧光定量PCR仪、电泳仪和凝胶成像系统购自美国Bio-Rad公司。
1.3 方法 1.3.1 MIC测定采用琼脂平板倍比稀释法测定细菌对抗菌药物的敏感性。细菌用生理盐水稀释比浊至0.5麦氏单位。抗菌药物用生理盐水倍比稀释成一系列浓度,取1 mL药物加入空培养皿,然后将MH琼脂培养基加入培养皿中进行混匀,制成含药琼脂平板,使抗菌药物浓度达到0.5~256.0 μg/mL。将稀释菌液点种在含药平板上,37 ℃培养18~20 h。MIC结果判读依据美国临床和实验室标准协会(CLSI,2018)和美国FDA发布的耐药折点进行。质控菌株为大肠埃希菌ATCC25922。
1.3.2 耐药基因检测参照文献[4]报道的碳青霉烯酶基因和超广谱β-内酰胺酶基因的引物序列合成引物。采用细菌基因组DNA提取试剂盒提取纯化DNA。PCR扩增体系为:2×Pfu PCR MasterMix 10 μL,正向和反向引物(10 μmol/L)各0.5 μL,DNA 1 μL,最后补足ddH2O至20 μL。PCR反应条件为:94 ℃预变性5 min,(94 ℃ 30 s;52~56 ℃ 30 s;72 ℃ 1 min)×30个循环,最后72 ℃延伸10 min。PCR反应产物在1%琼脂糖凝胶电泳上进行初步鉴定,阳性产物送上海生工生物公司进行双向测序最终鉴定。
1.3.3 碳青霉烯酶基因表达及与细菌耐药相关性细菌总RNA的提取纯化使用细菌RNA提取试剂盒进行,然后采用逆转录试剂盒合成cDNA。RT-PCR反应体系为:2×SYBR Green MasterMix 10 μL,cDNA 2 μL,正向和反向引物(10 μmol/L)各0.5 μL,ddH2O 7 μL。PCR反应条件为:94 ℃ 5 min,(94 ℃ 30 s;57 ℃ 30 s,72 ℃ 30 s)×40个循环,反应结束后,以16S rRNA基因作为内参,分析菌株的碳青霉烯酶基因表达量。此外,采用Spearman相关系数分析碳青霉烯酶基因表达量与亚胺培南耐药性之间的相关性。
1.3.4 多位点序列分型(multilocus sequence typing,MLST)参照文献[5]方法通过PCR扩增肺炎克雷伯菌的7个管家基因(gapA、infB、mdh、pgi、phoE、rpoB、tonB),确定菌株的MLST基因型。管家基因的PCR阳性产物经纯化后送至上海生工生物工程股份有限公司进行双向测序,测序结果与MLST网站(http://www.pasteur.fr/recherche/genopole/PF8/mlst/Kpneumoniae.html)上存在的等位基因序列进行比对,从而得到每株细菌相应的ST型。
1.3.5 分级抑制浓度指数(fractional inhibitory concentration index,FICI)采用棋盘法测定头孢他啶/阿维巴坦和氨曲南的协同作用[6]。按2倍梯度稀释法进行抗菌药物的稀释,然后加入96孔板中,同时加入稀释菌液,使其药物的终浓度达到1/32 MIC~2 MIC。平板放入37 ℃孵箱中培养24 h。FICI=MICA联合/MICA单独+MICB联合/MICB单独。结果判断标准:FICI≤0.5,协同作用;0.5 < FICI≤4,无关作用;FICI>4,拮抗作用[7]。
1.4 统计学分析采用SPSS17.0统计软件,计数资料以%表示,计量数据以x±s表示。碳青霉烯酶基因表达与亚胺培南耐药性之间的相关性采用Spearman相关系数分析。P < 0.05表示具有统计学意义。
2 结果 2.1 MIC测定碳青霉烯耐药肺炎克雷伯菌对抗菌药物的耐药性结果如表 1所示。菌株对替加环素的敏感性较高,而对其他实验用的抗菌药物均具有较高的耐药性。
| 抗菌药物 | MIC范围/μg·mL-1 | 耐药率[%(n)] |
| 头孢他啶 | 32~>256 | 100(10) |
| 头孢他啶/阿维巴坦 | 4/4~256/4 | 70(7) |
| 头孢曲松 | 32~>256 | 100(10) |
| 头孢吡肟 | 4~>256 | 90(9) |
| 亚胺培南 | 8~128 | 100(10) |
| 美罗培南 | 8~128 | 100(10) |
| 庆大霉素 | 4~128 | 80(8) |
| 妥布霉素 | 4~128 | 80(8) |
| 阿米卡星 | 8~>256 | 70(7) |
| 环丙沙星 | 1~64 | 90(9) |
| 左氧氟沙星 | 2~128 | 90(9) |
| 氨曲南 | 32~256 | 100(10) |
| 哌拉西林/他唑巴坦 | 128/4~>256/4 | 100(10) |
| 替加环素 | < 0.5~8.0 | 10(1) |
2.2 耐药基因分析
通过PCR扩增碳青霉烯耐药肺炎克雷伯菌的β-内酰胺酶耐药基因,结果如表 2所示。碳青霉烯酶基因扩增结果显示:有4株产NDM,2株产KPC,1株产IMP,1株同时产NDM和KPC。超广谱β-内酰胺酶耐药基因扩增结果显示:所有菌株携带CTX-M和SHV基因,6株携带TEM基因,分别有2株携带ampC和OXA基因。同时携带NDM和KPC基因的菌株(3号菌株)其亚胺培南的MIC值最大,且NDM阳性菌株对亚胺培南的耐药性大于KPC阳性菌株和IMP阳性菌株。
| 菌株号 | 亚胺培南MIC/μg·mL-1 | ST | NDM | KPC | IMP | ampC | CTX-M | SHV | TEM | OXA |
| 1 | 8 | 11 | - | + | - | - | + | + | + | - |
| 2 | 64 | 15 | + | - | - | - | + | + | - | - |
| 3 | 128 | 11 | + | + | - | + | + | + | + | + |
| 4 | 8 | 11 | - | - | - | - | + | + | + | - |
| 5 | 16 | 23 | - | - | + | - | + | + | - | - |
| 6 | 8 | 11 | - | - | - | + | + | + | - | - |
| 7 | 32 | 15 | + | - | - | - | + | + | - | - |
| 8 | 32 | 11 | - | + | - | - | + | + | + | - |
| 9 | 64 | 147 | + | - | - | - | + | + | + | + |
| 10 | 64 | 11 | + | - | - | - | + | + | + | - |
2.3 碳青霉烯酶基因表达及与细菌耐药相关性
通过RT-PCR分析菌株的碳青霉烯酶基因表达差异,结果如图 1所示。8株产碳青霉烯酶的肺炎克雷伯菌其基因表达量不同,其中NDM基因表达量较高,携带该基因的菌株对亚胺培南的耐药性也相对较高,而KPC和IMP基因表达量较低,携带该基因的菌株对亚胺培南的耐药性也相对较低。采用Spearman相关系数分析7株细菌的碳青霉烯酶基因表达和亚胺培南耐药性之间的相关性(携带两个碳青霉烯酶基因的3号菌株除外),结果显示:碳青霉烯酶基因表达与亚胺培南耐药性之间存在正相关关系(相关系数r=0.805,P=0.029)。
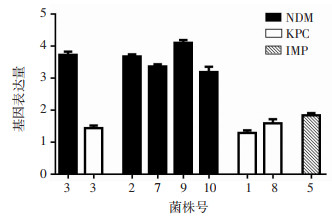
|
| 图 1 肺炎克雷伯菌的碳青霉烯酶基因表达 |
2.4 MLST分型
通过MLST分析菌株的基因多样性,结果如表 2所示。10株耐药菌株共有4个ST型,其中6株为ST11,2株为ST15,ST23和ST147各有1株。NDM阳性菌株分属于ST11、ST15和ST147 3个基因型,KPC阳性菌株均属于ST11型,IMP阳性菌株为ST23型。
2.5 FICI检测通过棋盘法测定头孢他啶/阿维巴坦与氨曲南对菌株的协同作用,结果如表 3所示。头孢他啶/阿维巴坦与氨曲南联合对7株菌具有协同作用,对3株菌表现为无关作用,无拮抗作用出现。
| 菌株号 | 单独 | 联合 | FICI | |||
| MICA | MICB | MICA | MICB | |||
| 1 | 8.0 | 32.0 | 1.0 | 8.0 | 0.375 | |
| 2 | 64.0 | 64.0 | 8.0 | 8.0 | 0.250 | |
| 3 | 256.0 | 256.0 | 64.0 | 32.0 | 0.375 | |
| 4 | 4.0 | 32.0 | 2.0 | 16.0 | 1.000 | |
| 5 | 32.0 | 64.0 | 4.0 | 8.0 | 0.250 | |
| 6 | 4.0 | 32.0 | 0.5 | 8.0 | 0.375 | |
| 7 | 128.0 | 128.0 | 64.0 | 64.0 | 1.000 | |
| 8 | 16.0 | 64.0 | 8.0 | 32.0 | 1.000 | |
| 9 | 256.0 | 128.0 | 64.0 | 32.0 | 0.500 | |
| 10 | 64.0 | 32.0 | 8.0 | 8.0 | 0.375 | |
| A药为头孢他啶/阿维巴坦,以头孢他啶MIC值计;B药为氨曲南 | ||||||
3 讨论
碳青霉烯耐药肺炎克雷伯菌药敏结果显示:菌株除对替加环素具有较高的敏感性外,对其他抗菌药物的耐药性均达到70%及以上。2012年,在多重耐药菌逐年升高的困境中在中国批准上市、被寄予厚望的替加环素对多重耐药菌具有较好的临床疗效。随着替加环素的应用增加,肺炎克雷伯菌对替加环素的耐药性也在增加,而替加环素作为抑菌剂,铜绿假单胞菌对其天然耐药,因此,替加环素多与其他药物联合使用。
实验结果发现碳青霉烯类高耐药的菌株,均为碳青霉烯酶阳性,且同时携带两种碳青霉烯酶基因的菌株和NDM阳性菌株其对碳青霉烯类药物的耐药性较高,碳青霉烯酶耐药基因表达高的菌株具有较高的耐药性,说明碳青霉烯酶特别是NDM在碳青霉烯类耐药中是首要因素。在本研究中,最主要的碳青霉烯酶基因为NDM,与研究报道相同[8-9]。NDM耐药基因最早在印度出现[10],随后在世界范围内广泛流行,目前NDM已取代KPC成为我国肠杆菌科细菌最常见的碳青霉烯酶基因[11]。ST11是国内流行的肺炎克雷伯菌基因型[12],本研究发现其在碳青霉烯耐药肺炎克雷伯菌中也占多数。本研究中NDM在肺炎克雷伯菌的多个基因型中均有分布,说明NDM耐药基因已经在肺炎克雷伯菌中广泛扩散。同时,本研究所筛选的碳青霉烯耐药菌株均携带至少2种其他β-内酰胺酶耐药基因,会导致对β-内酰胺酶类药物的高度耐药。此外,菌株对其他种类抗菌药物的高耐药提示碳青霉烯耐药菌株也应携带其他种类抗菌药物的耐药基因。
注射用头孢他啶/阿维巴坦钠已于2019年5月获得国家药品监督管理局批准,用于治疗复杂性腹腔内感染、医院获得性肺炎和呼吸机相关性肺炎,在治疗方案选择有限的成人患者中治疗对该药敏感的革兰阴性菌引起的感染。虽然头孢他啶/阿维巴坦对Ambler A类、C类和部分D类β-内酰胺酶稳定,但对产NDM肺炎克雷伯菌仍具有较高的MIC值[13]。目前国外已有临床研究表明头孢他啶/阿维巴坦和氨曲南联合对产NDM菌株可以取得较好的治疗效果[14],但两药联合在国内的临床应用研究尚未见相关报道。本研究结果显示头孢他啶/阿维巴坦联合氨曲南对大部分的碳青霉烯耐药肺炎克雷伯菌具有较好的协同作用,MIC显著降低,为头孢他啶/阿维巴坦钠在我国的临床应用提供了理论依据。但本研究细菌样本量较少,且没有进行深入的机制研究和在动物体内进行疗效评价,研究结果尚需进一步研究证实。
| [1] |
HU F P, GUO Y, YANG Y, et al. Resistance reported from China antimicrobial surveillance network (CHINET) in 2018[J]. Eur J Clin Microbiol Infect Dis, 2019, 38(12): 2275-2281. DOI:10.1007/s10096-019-03673-1 |
| [2] |
LAGACÉ-WIENS P, WALKTY A, KARLOWSKY J A. Ceftazidime-avibactam: an evidence-based review of its pharmacology and potential use in the treatment of Gram-negative bacterial infections[J]. Core Evid, 2014, 9: 13-25. DOI:10.2147/CE.S40698 |
| [3] |
FRITZENWANKER M, IMIRZALIOGLU C, HEROLD S, et al. Treatment options for Carbapenem-resistant gram-Negative infections[J]. Dtsch Arztebl Int, 2018, 115(20/21): 345-352. DOI:10.3238/arztebl.2018.0345 |
| [4] |
CHEN Z H, LI H X, FENG J, et al. NDM-1 encoded by a pNDM-BJ01-like plasmid p3SP-NDM in clinical Enterobacter aerogenes[J]. Front Microbiol, 2015, 6: 294. DOI:10.3389/fmicb.2015.00294 |
| [5] |
XIE S J, FU S M, LI M W, et al. Microbiological characteristics of carbapenem-resistant Enterobacteriaceae clinical isolates collected from county hospitals[J]. Infect Drug Resist, 2020, 13: 1163-1169. DOI:10.2147/IDR.S248147 |
| [6] |
BONAPACE C R, BOSSO J A, FRIEDRICH L V, et al. Comparison of methods of interpretation of checkerboard synergy testing[J]. Diagn Microbiol Infect Dis, 2002, 44(4): 363-366. DOI:10.1016/s0732-8893(02)00473-x |
| [7] |
ODDS F C. Synergy, antagonism, and what the chequerboard puts between them[J]. J Antimicrob Chemother, 2003, 52(1): 1. DOI:10.1093/jac/dkg301 |
| [8] |
WANG Q, WANG X J, WANG J, et al. Phenotypic and genotypic characterization of carbapenem-resistant Enterobacteriaceae: data from a longitudinal large-scale CRE study in China (2012-2016)[J]. Clin Infect Dis, 2018, 67(suppl_2): S196-S205. DOI:10.1093/cid/ciy660 |
| [9] |
TIAN D, PAN F, WANG C, et al. Resistance phenotype and clinical molecular epidemiology of carbapenem-resistant Klebsiella pneumoniae among pediatric patients in Shanghai[J]. Infect Drug Resist, 2018, 11: 1935-1943. DOI:10.2147/IDR.S175584 |
| [10] |
YONG D, TOLEMAN M A, GISKE C G, et al. Characterization of a new metallo-beta-lactamase gene, bla(NDM-1), and a novel erythromycin esterase gene carried on a unique genetic structure in Klebsiella pneumoniae sequence type 14 from India[J]. Antimicrob Agents Chemother, 2009, 53(12): 5046-5054. DOI:10.1128/AAC.00774-09 |
| [11] |
ZHOU H, ZHANG K, CHEN W, et al. Epidemiological characteristics of carbapenem-resistant Enterobacteriaceae collected from 17 hospitals in Nanjing district of China[J]. Antimicrob Resist Infect Control, 2020, 9: 15. DOI:10.1186/s13756-019-0674-4 |
| [12] |
CHEN Y, LIAO K, HUANG Y, et al. Determining the susceptibility of carbapenem resistant Klebsiella pneumoniae and Escherichia coli strains against common disinfectants at a tertiary hospital in China[J]. BMC Infect Dis, 2020, 20(1): 88. DOI:10.1186/s12879-020-4813-6 |
| [13] |
ALATOOM A, ELSAYED H, LAWLOR K, et al. Comparison of antimicrobial activity between ceftolozane-tazobactam and ceftazidime-avibactam against multidrug-resistant isolates of Escherichia coli, Klebsiella pneumoniae, and Pseudomonas aeruginosa[J]. Int J Infect Dis, 2017, 62: 39-43. DOI:10.1016/j.ijid.2017.06.007 |
| [14] |
HOBSON C A, BONACORSI S, FAHD M, et al. Successful treatment of Bacteremia due to NDM-1-producing Morganella morganii with aztreonam and ceftazidime-avibactam combination in a pediatric patient with Hematologic Malignancy[J]. Antimicrob Agents Chemother, 2019, 63(2): e02463-18. DOI:10.1128/AAC.02463-18 |



