镉被广泛应用于采矿、冶炼、电镀、合金制造以及油漆颜料生产等多个工业领域[1]。随着工农业生产的发展,环境中的镉含量也逐年上升。目前,我国镉污染土壤面积已达2×105 km2,占总耕地面积的1/6,涉及11个省市的25个地区[2]。同时,“湖南镉大米事件”“河南镉小麦事件”等重大公共卫生事件的报道进一步显示了镉污染的严峻现实及其对人民健康的巨大威胁。因此,镉的毒理机制研究已经成为我国预防医学领域研究的前沿与热点问题。
镉是对人的多种器官具有危害的一种重金属,肝脏是镉毒理作用的主要靶器官之一。现实生活中,人群镉暴露所导致的肝功能异常主要以急性暴露为主[3]。在镉的急性中毒事件中,患者会出现倦怠、恶心等症状和天冬氨酸转氨酶(AST)、丙氨酸转氨酶(ALT)等酶谱异常[4]。我们前期的实验结果显示2 mg/kg镉暴露两周会激活Drp1蛋白依赖的线粒体的分裂增多,线粒体功能障碍,ATP产生减少,从而导致SD大鼠肝脏出现明显的中性粒细胞等炎性细胞浸润、肝细胞气球样变,灶性坏死[5]。然而,镉肝细胞毒性机制较为复杂,其肝毒理作用的分子机制目前仍缺乏深入、系统的研究。因此,寻找镉急性肝脏毒性的关键基因对于确立治疗新靶点和相关药物研发至关重要。
近年来,基因芯片的运用已成为研究毒理学研究高效且必要的方法。随着数据共享时代的来临,各种数据库中拥有丰富的基因检测和分析结果,但缺少有效、精确的数据挖掘,尤其缺少相关的验证工作的开展。本研究采用生物信息技术,分析基因公共表达数据库中关于镉肝毒性的芯片数据,筛选差异表达基因及其可能的功能和信号通路,明确核心基因并结合实验室研究,以期揭示镉肝脏毒理作用的细胞靶点和分子机制。
1 材料与方法 1.1 实验动物及试剂SPF级雄性SD大鼠, 体质量250~300 g, 7~8周龄,由陆军军医大学实验动物中心提供,生产许可证号SCXK(渝) 20170002,使用许可证号SYXK(渝) 20170002。所有动物实验的开展均获得陆军军医大学实验动物伦理审查委员会批准(动物伦理证明批准号:AMUWEC20200059)。氯化镉购置于西格玛奥德里奇公司,纯度>99.99%,货号:202908。建立镉暴露大鼠模型前,以生理盐水配制氯化镉溶液。
1.2 大鼠一次性氯化镉急性暴露实验SPF级雄性大鼠16只,体质量250~300 g,适应性饲养3天,分成2组(n=8):生理盐水对照组及氯化镉暴露组。氯化镉染毒剂量为1.25 mg/kg·bw,染毒方式为一次性腹腔注射,24 h后取材进行后续实验。
1.3 血清生化指标检测25%乌拉坦麻醉大鼠,心脏取血,1 000×g离心10 min,制备血清,-80 ℃冻存。干冰运输方式送武汉赛维尔生物科技有限公司以全自动生化分析仪检测血清中谷丙转氨酶(ALT)、谷草转氨酶(AST)含量。
1.4 肝组织病理形态学观察25%乌拉坦麻醉大鼠,取肝组织0.5 cm×1 cm×1.5 cm大小,4%多聚甲醛固定,送武汉赛维尔生物科技有限公司进行肝组织常规病理切片,HE染色,应用Pannoramic MIDI病理切片扫描仪进行记录。
1.5 芯片数据获取及DEGs分析芯片数据GSE65198的基因表达谱来源于美国国立生物技术信息中心(NCBI)的公共数据库(genes expression omnibus, GEO)。该芯片检测大鼠急性Cd暴露后,肝组织基因表达改变,其中以1.25 mg/kg·bw的Cd一次性染毒,24 h取材的实验条件与我们一致。本研究应用GEO数据库在线分析工具GEO2R,提取此条件下Cd处理组与对照组数据进行差异表达基因(DEGs)分析。
1.6 GO及KEGG分析对筛选出的差异表达基因进行基因本体论(genes ontology, GO)和京都基因与基因组百科全书(kyoto encyclopendia of genes and genomes, KEGG)分析,研究DEGs可能影响的功能及信号通路。GO分析是一种广泛用于注释基因和基因产物,并且识别基因组、转录组数据的生物特征属性的重要工具[6]。KEGG是系统分析基因,关联基因功能与高阶功能信息的数据库[7]。本研究筛取差异表达上调或下调2倍以上(logFC >1或 < -1)、P < 0.05的DEGs基因,利用生物信息学分析工具Blast2GO[8]进行GO及KEGG分析,利用R语言ggplot2包进行绘图。P < 0.05设定为基因显著性富集的临界值。
1.7 蛋白-蛋白相互作用网络分析SRTING是一个检索蛋白质之间相互作用关系的在线数据库,包括蛋白质之间的直接物理相互作用及间接功能相关性,它除了包含有实验数据、从PubMed文献数据库及其他数据库摘要中挖掘的信息外,还有利用生物信息学方法预测的结果[9]。通过STRING (10.5版),以蛋白与蛋白相互作用关系置信指数>0.4构建DEGs相互作用网络,并导入软件Cytoscape(3.7.2版)[10]。利用其插件Hub Objects Analyzer (cytoHubba)[11]以Degree方法计算得出关键基因(Hub基因),进一步挖掘其可能的调控和作用机制。
1.8 实时定量荧光PCR分析以总RNA提取试剂RNAiso Plus(宝生物, 日本)溶解小于100 mg的肝组织,按说明书步骤提取样本总RNA。取等量总RNA按反转录试剂盒PrimeScript RT reagent Kit(宝生物, 日本)说明书步骤将总RNA转录为cDNA以进行后续实验。使用荧光定量检测试剂SYBR Master Mix(宝生物, 日本)及荧光定量PCR仪(CFX96,美国伯乐)进行前5位Hub基因实时定量荧光PCR分析,以明确其在镉暴露组和对照组肝组织mRNA表达差异。特异性引物序列见表 1。以GAPDH为内参,以2-ΔΔCt方法计算mRNA表达量[12]。参照对照组计算Hub基因相对表达水平。
| 基因名称 | 上游引物 | 下游引物 | 产物长度(bp) |
| Cdc20 | 5′-AGTGCTGTGGATGTGCAT-3′ | 5′-TCTGGACTCATGGTGAGACT-3′ | 182 |
| Ccnb1 | 5′-ACTTTGGCCAAATACCTCATG-3′ | 5′-TCACAGGAAGCAGAGATTCCT-3′ | 178 |
| Cdk1 | 5′-TCTTCGAGTTCCTGTCCATG-3′ | 5′-GAGGTTTCAAGTCTCTGTGAAG-3′ | 159 |
| Top2a | 5′-CAGAAGAGCATACGATATTGCT-3′ | 5′-GCTCATTGTTAAGCACACTTC-3′ | 193 |
| Ube2c | 5′-CGAATCAGACAACCTGTTC-3′ | 5′-GTGTGAGGAACTTCACTGTG-3′ | 140 |
| β-actin | 5′-CTAAGGCCAACCGTGAAAAGAT-3′ | 5′-ACCAGAGGCATACAGGGACAAC-3′ | 104 |
1.9 Western blot法检测蛋白表达
分别收集各对照组和镉暴露组肝组织提取总蛋白,BCA法测定蛋白浓度。SDS-PAGE分离,切胶、转膜、封闭。分别加入兔抗大鼠CDC20抗体(1 :1 000,Thermo Fisher Scientific, 美国)、兔抗大鼠CDK1抗体(1 :1 000,Abcam,美国)、兔抗大鼠CCNB1抗体(1 :1 000,碧云天,中国)、小鼠抗大鼠β-actin抗体(1 :5 000,Sigma,美国), 小鼠抗大鼠GAPDH抗体(1 :3 000,碧云天,中国)4 ℃孵育过夜;分别加入HRP标记的山羊抗兔IgG(1 :5 000,碧云天,中国), 山羊抗小鼠IgG(1 :5 000,碧云天,中国)室温摇床孵育2 h;洗膜,ECL显色。利用ChemiDoc XRS+系统和Image Lab软件(美国Bio-Rad)对条带进行成像和灰度值分析。
1.10 统计学分析实验数据以样本x±s表示。由SPSS 22.0统计软件进行分析,组间比较采用t检验。检验水准α=0.05。
2 结果 2.1 1.25 mg/kg·bw氯化镉急性暴露损伤大鼠肝组织通过HE染色及肝功能生化指标ALT、AST分析一次性腹腔注射1.25 mg/kg·bw氯化镉,暴露24 h后大鼠肝组织损伤情况。HE染色结果显示镉暴露组大鼠肝脏并没有明显的病理学变化,未见炎症细胞浸润,肝细胞气球样变,坏死等(图 1);与对照组相比大鼠血清中肝功能生化指标ALT、AST显著增高P < 0.01,平均值分别为ALT(U/L):对照组85.072 9,镉暴露组126.876;AST(U/L):对照组122.624,镉暴露组211.792,每组各8只大鼠。以上结果表明1.25 mg/kg·bw氯化镉急性暴露造成大鼠肝损伤。
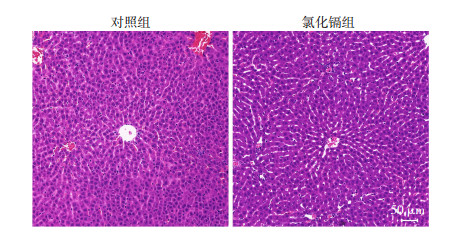
|
| 图 1 镉急性暴露对大鼠肝组织形态学影响 |
2.2 DEGs筛选结果
为分析镉暴露造成大鼠肝损伤机制,我们选择GEO公共数据库中芯片结果GSE65198,提取其中与我们相同实验条件的数据,利用在线软件GEO2R进行差异基因分析。以差异表达量上调或下调2倍以上,P < 0.05为筛选条件,其中288个基因显著上调,277个基因显著下调(图 2A)。上调及下调前25位基因热图结果显示,组内样本基因表达较为一致,组间基因表达有明显差异(图 2B)。
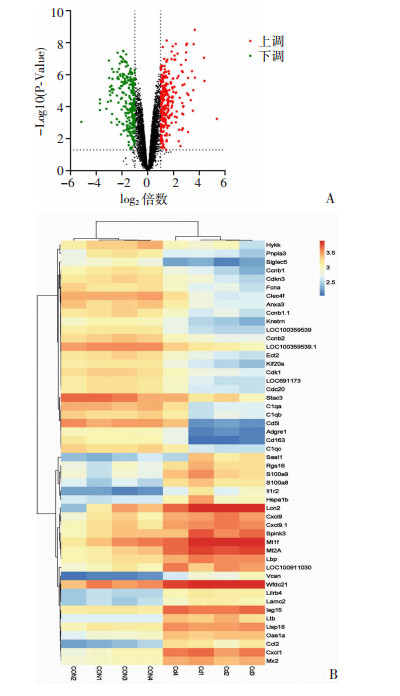
|
| 图 2 镉急性暴露肝组织中差异表达基因火山图(A)及热图分析(B) |
2.3 GO和KEGG通路富集分析显示DEGs首要参与了镉诱导的肝细胞周期阻滞
本研究采用生物信息学分析工具Blast2GO对565个差异基因进行KEGG及GO分析,展示前20位的富集类别。结果如图 3A所示,KEGG分析显示DEGs首要富集于细胞周期通路。如图 3B所示,GO分析显示DEGs涉及的生物学过程(biological process,BP)前3位为细胞分裂、有丝分裂和细胞周期,均和细胞周期密切相关,与KEGG分析结果高度吻合。因此,我们大胆推测DEGs首要参与了镉诱导的细胞周期阻滞。

|
| 图 3 差异表达基因KEGG通路富集(A)及GO分析(B) |
2.4 DEGs编码蛋白的相互作用网络分析筛选Hub基因
STRING是一个由已知和预测的蛋白质相互作用数据组成的数据库,本研究将565个显著差异表达基因导入STRING在线分析工具,获得DEGs相互作用关系,将其导入Cytoscape(3.7.2版)软件计算网络及各个节点的拓扑特性。利用Cytoscape插件cytoHubba寻找Hub基因,按照Degree分析方法排名前5位的核心(Hub)基因分别为Cdc20、Ccnb1、Cdk1、Top2a、Ube2c,表明它们可能在镉暴露损伤大鼠肝细胞中起重要作用(图 4)。
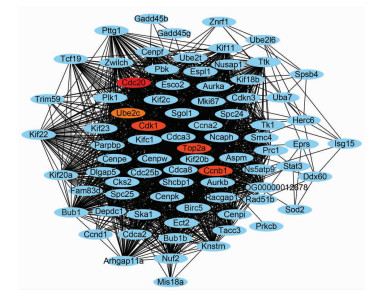
|
| 图 4 差异表达基因构建PPI网络的Hub基因分析 |
2.5 实时荧光定量PCR验证Hub基因表达
为进一步揭示镉损伤大鼠肝组织的潜在机制并验证芯片检测结果,采用实时荧光定量PCR方法检测排名前5位的Hub基因表达。如图 5所示,氯化镉急性暴露导致大鼠肝组织Cdc20、Ccnb1、Cdk1、Top2a和Ube2c mRNA表达水平显著下降,与芯片检测结果一致,进一步证实了芯片数据的有效性,并且表明它们可能是镉暴露损伤肝组织的重要靶点基因。
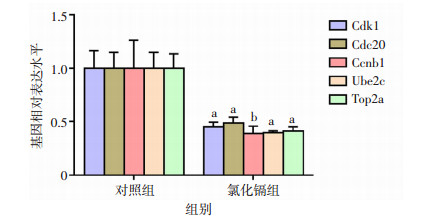
|
| a: P < 0.05,b: P < 0.01,与对照组比较 图 5 镉急性暴露对大鼠肝组织Hub基因mRNA表达影响分析(n=8, x±s) |
2.6 Western blot验证参与细胞周期的Hub基因表达
前面结果表明氯化镉急性暴露条件下大鼠肝组织差异表达基因DEGs首要影响细胞周期进程,为进一步发掘影响细胞周期通路的关键基因,本研究将细胞周期通路富集的差异基因与PPI网络分析结果排名前5位的Hub基因取交集,结果表明Cdc20、Ccnb1、Cdk1可能是氯化镉急性暴露导致细胞周期阻滞从而造成肝损伤的关键靶点(图 6A)。采用Western blot方法检测其表达水平的变化,结果表明在氯化镉暴露条件下,CDK1蛋白表达受到明显抑制(相对表达量镉处理组为对照组的66.8%,P < 0.05),CDC20及CCNB1蛋白表达没有改变(图 6B)。
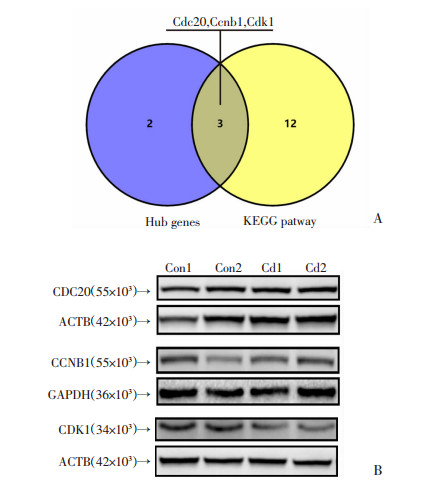
|
| A: Hub基因前5位与细胞周期通路关键基因取交集结果;B:Western blot分析核心基因蛋白表达结果 图 6 镉急性暴露对大鼠肝组织核心基因蛋白表达影响分析 |
3 讨论
肝脏是镉中毒的主要靶器官之一,目前国内外研究表明,镉肝毒性的主要机制包括改变了细胞的离子转运,竞争性的抑制细胞膜上的各种离子通道;影响了(与炎症、氧化应激和内环境稳定相关的)某些基因表达;损伤线粒体及内质网产生大量的活性氧,导致DNA损伤增加,促进凋亡及自噬性死亡[13]。尽管镉肝毒理机制研究已经有了一定进展,其肝毒理作用的机制依然缺乏系统的研究。本研究采用1.25 mg/kg·bw镉暴露SD大鼠一次建立急性镉暴露的模型,发现该暴露模式下,急性镉暴露显著诱导SD大鼠肝脏损伤,同时基于组织芯片明确Cdk1等核心基因依赖的细胞周期阻滞与镉肝毒性最为相关。本结果为镉的肝脏毒性研究提供一定的基础数据及可能的机制线索。
目前职业人群镉暴露所导致的肝脏损伤主要以急性一次性暴露为主。为了更好地分析镉的急性肝毒性机理,根据预实验结果,并参照MADEJCZYK等[14]和DE MOURA等[15]研究的镉腹腔注射剂量,本实验采用单次腹腔注射一次性给予CdCl2 1.25 mg/kg·bw的剂量建立急性Cd中毒模型。有研究表明,在镉暴露大鼠的模型中(3.9 mg/kg·bw), 肝脏可观察到的最早的形态学变化包括外周着色颗粒增加、核糖体减少、核仁凝聚及粗面内质网的膨胀。5~6 h后则可以观察到肝细胞的线粒体及内质网肿胀、细胞质内纤维性物质缠绕。在暴露10~12 h后即可出现肝细胞坏死[16]。我们用1.25 mg/kg·bw建立的镉暴露模型所用的浓度低于该文献的报道,虽然我们没有观察到文献中所提到的大鼠萎靡不振,饮食减退及相应的病理学改变等现象,却同样发现了肝损伤的血清学指标,提示进行大鼠急性镉中毒建模时镉的剂量可以选择更低的水平。
基因芯片技术是一种高通量且快速准确分析基因表达的工具,能够在不同细胞类型、不同细胞生长阶段及状态、不同条件、不同干预手段下对基因芯片做出相应的检测,并产生海量的数据,使研究者能够运用各种生物信息学方法对数据进行整合分析,从而为探索镉肝毒理作用的分子机制提供有利的线索。本研究选取数据库中与我们模型条件完全相同的芯片进行分析,共发现差异表达基因565个,288个基因显著上调,277个基因显著下调。为了进一步明确这些差异表达基因的生物学功能,应用Blast2GO在线软件对其进行了KEGG通路分析和GO富集分析。KEGG通路显示DEGs首要富集于细胞周期通路及其他相关通路,包括P53信号通路、PPAR信号通路等。GO分析显示DEGs涉及的生物学过程前3位为细胞分裂、有丝分裂和细胞周期,均和细胞周期密切相关,与KEGG分析结果高度吻合。细胞周期阻滞在镉肝脏毒性中扮演的角色已有相关文献报道。BADISA等[17]对100 μmol/L镉处理的大鼠正常肝脏细胞CRL-1439细胞24 h后进行基因芯片分析发现镉显著诱导了细胞周期相关基因的表达。ODEWUMI等[18]报道了150 μmol/L的镉暴露CRL-1439细胞24 h后明显抑制细胞周期的S期和G2/M期。基于我们分析和以往文献的报道,我们得出的结论是镉通过影响细胞周期从而引起肝损伤可能是镉肝毒理作用最重要的机制之一。
基于SRTING在线数据库,我们对镉暴露后肝脏的差异表达基因进行了蛋白互作网络分析,发现上述基因编码的蛋白产物参与了蛋白互作网络图的构建,他们之间以线连接,且部分基因作为中心节点与周围的基因关系密切。本研究通过Degree算法筛选出了网络中的5个核心基因,他们是Cdc20、Ccnb1、Cdk1、Top2a、Ube2c。同时将5个核心基因与KEGG通路中参与细胞周期的基因进行交互分析,发现Cdc20、Ccnb1、Cdk1等3个基因是参与镉诱导细胞周期阻滞的核心基因。目前这3个基因已经被广泛报道可以参与细胞周期的调控[19],且是镉诱导细胞阻滞的重要靶点[20]。有趣的是在我们的暴露模型中,镉暴露后仅诱导Cdk1蛋白表达水平降低,其他两个基因的蛋白水平并没有改变,这可能与基因的转录后调控和翻译及翻译后调控都有密切关系。此外,Top2a与细胞周期调控也存在密切的联系[21],研究表明镉可以视为一种Top2a蛋白活性抑制剂[22]。Ube2c基因是泛素化偶联酶E2基因家族成员,主要参与调控细胞的有丝分裂[23]。最近研究表明,Ube2c基因与细胞周期也密切相关。LIU等[24]报道Ube2c基因敲除会抑制黑色素瘤细胞株G2/M期。ZLLNER等[25]发现小分子化合物YK-4-279明显减少Ube2c的表达,激活Cyclin B1从而抑制尤文氏肉瘤细胞的G2/M期。目前Ube2c基因是否参与了镉诱导的肝细胞周期阻滞尚少见报道,非常值得进一步的研究分析。
本研究的局限性在于我们尚无法在动物水平直接检测肝细胞周期的改变,尤其需要在人来源的细胞株开展进一步的验证。
综上,本研究发现急性镉暴露首要通过抑制肝细胞的细胞周期从而诱导大鼠肝脏损伤(TOP1的信号通路),Cdk1等核心基因可能是镉诱导肝细胞周期阻滞的重要靶点,为进一步深入研究镉的急性肝脏毒性作用机制提供了非常有价值的信息和新的研究方向。
| [1] |
尚二萍, 许尔琪, 张红旗, 等. 中国粮食主产区耕地土壤重金属时空变化与污染源分析[J]. 环境科学, 2018, 39(10): 4670-4683. SHANG E P, XU E Q, ZHANG H Q, et al. Spatial-temporal trends and pollution source analysis for heavy metal contamination of cultivated soils in five major grain producing regions of China[J]. Chin J Environ Sci, 2018, 39(10): 4670-4683. DOI:10.13227/j.hjkx.201802139 |
| [2] |
李婧, 周艳文, 陈森, 等. 我国土壤镉污染现状、危害及其治理方法综述[J]. 安徽农学通报, 2018, 39(24): 104-107. LI J, ZHOU Y W, CHEN S, et al. Actualities, damage and management of soil cadmium pollution in China[J]. Auhui Agri Sci Bull, 2018, 39(10): 4670-4683. DOI:10.16377/j.cnki.issn1007-7731.2015.24.044 |
| [3] |
RIKANS L E, YAMANO T. Mechanisms of cadmium-mediated acute hepatotoxicity[J]. J Biochem Mol Toxicol, 2000, 14(2): 110-117. DOI:10.1002/(sici)1099-0461(2000)14:2<110::aid-jbt7>3.0.co;2-j |
| [4] |
FOWLER B A. Monitoring of human populations for early markers of cadmium toxicity: a review[J]. Toxicol Appl Pharmacol, 2009, 238(3): 294-300. DOI:10.1016/j.taap.2009.05.004 |
| [5] |
XU S, PI H, CHEN Y, et al. Cadmium induced Drp1-dependent mitochondrial fragmentation by disturbing calcium homeostasis in its hepatotoxicity[J]. Cell Death Dis, 2013, 4: e540. DOI:10.1038/cddis.2013.7 |
| [6] |
ASHBURNER M, BALL C A, BLAKE J A, et al. Gene ontology: tool for the unification of biology. The Gene Ontology Consortium[J]. Nat Genet, 2000, 25(1): 25-29. DOI:10.1038/75556 |
| [7] |
KANEHISA M, FURUMICHI M, TANABE M, et al. KEGG: new perspectives on genomes, pathways, diseases and drugs[J]. Nucleic Acids Res, 2017, 45(D1): D353-D361. DOI:10.1093/nar/gkw1092 |
| [8] |
GÖTZ S, GARCÍA-GÓMEZ J M, TEROL J, et al. High-throughput functional annotation and data mining with the Blast2GO suite[J]. Nucleic Acids Res, 2008, 36(10): 3420-3435. DOI:10.1093/nar/gkn176 |
| [9] |
SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017: quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Res, 2017, 45(D1): D362-D368. DOI:10.1093/nar/gkw937 |
| [10] |
SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003, 13(11): 2498-2504. DOI:10.1101/gr.1239303 |
| [11] |
CHIN C H, CHEN S H, WU H H, et al. CytoHubba: identifying hub objects and sub-networks from complex interactome[J]. BMC Syst Biol, 2014, 8(Suppl 4): S11. DOI:10.1186/1752-0509-8-S4-S11 |
| [12] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [13] |
ZHANG H, REYNOLDS M. Cadmium exposure in living organisms: a short review[J]. Sci Total Environ, 2019, 678: 761-767. DOI:10.1016/j.scitotenv.2019.04.395 |
| [14] |
MADEJCZYK M S, BAER C E, DENNIS W E, et al. Temporal changes in rat liver gene expression after acute cadmium and chromium exposure[J]. PLoS one, 2015, 10(5): e0127327. DOI:10.1371/journal.pone.0127327 |
| [15] |
DE MOURA C F, RIBEIRO F A, DE JESUS G P, et al. Antimutagenic and antigenotoxic potential of grape juice concentrate in blood and liver of rats exposed to cadmium[J]. Environ Sci Pollut Res Int, 2014, 21(22): 13118-13126. DOI:10.1007/s11356-014-3257-1 |
| [16] |
DUDLEY R E, SVOBODA D J, KLAASSEN C D. Time course of cadmium-induced ultrastructural changes in rat liver[J]. Toxicol Appl Pharmacol, 1984, 76(1): 150-160. DOI:10.1016/0041-008x(84)90038-3 |
| [17] |
BADISA V L, LATINWO L M, ODEWUMI C O, et al. Cytotoxicity and stress gene microarray analysis in cadmium-exposed CRL-1439 normal rat liver cells[J]. Int J Mol Med, 2008, 22(2): 213-219. |
| [18] |
ODEWUMI C O, BADISA V L, LE U T, et al. Protective effects of N-acetylcysteine against cadmium-induced damage in cultured rat normal liver cells[J]. Int J Mol Med, 2011, 27(2): 243-248. DOI:10.3892/ijmm.2010.564 |
| [19] |
NAGANO T, LUBLING Y, VÁRNAI C, et al. Cell-cycle dynamics of chromosomal organization at single-cell resolution[J]. Nature, 2017, 547(7661): 61-67. DOI:10.1038/nature23001 |
| [20] |
YU X Z, ROBINSON J F, SIDHU J S, et al. A system-based comparison of gene expression reveals alterations in oxidative stress, disruption of ubiquitin-proteasome system and altered cell cycle regulation after exposure to cadmium and methylmercury in mouse embryonic fibroblast[J]. Toxicol Sci, 2010, 114(2): 356-377. DOI:10.1093/toxsci/kfq003 |
| [21] |
LEE J H, BERGER J M. Cell cycle-dependent control and roles of DNA topoisomerase Ⅱ[J]. Genes (Basel), 2019, 10(11): E859. DOI:10.3390/genes10110859 |
| [22] |
WU X, YALOWICH J C, HASINOFF B B. Cadmium is a catalytic inhibitor of DNA topoisomerase Ⅱ[J]. J Inorg Biochem, 2011, 105(6): 833-838. DOI:10.1016/j.jinorgbio.2011.02.007 |
| [23] |
XIE C L, POWELL C, YAO M, et al. Ubiquitin-conjugating enzyme E2C: a potential cancer biomarker[J]. Int J Biochem Cell Biol, 2014, 47: 113-117. DOI:10.1016/j.biocel.2013.11.023 |
| [24] |
LIU G L, ZHAO J, PAN B Y, et al. UBE2C overexpression in melanoma and its essential role in G2/M transition[J]. J Cancer, 2019, 10(10): 2176-2184. DOI:10.7150/jca.32731 |
| [25] |
ZÖLLNER S K, SELVANATHAN S P, GRAHAM G T, et al. Inhibition of the oncogenic fusion protein EWS-FLI1 causes G2-M cell cycle arrest and enhanced vincristine sensitivity in Ewing's sarcoma[J]. Sci Signal, 2017, 10(499): eaam8429. DOI:10.1126/scisignal.aam8429 |



