2. 400038 重庆, 陆军军医大学(第三军医大学)第一附属医院骨科运动医学中心
2. Orthopaedic Sports Medicine Center, First Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400038, China
口腔鳞状细胞癌(oral squamous cell carcinoma, OSCC)是发生在口腔内主要的恶性肿瘤,通常由于烟草和酒精导致的口腔黏膜发生癌变引起[1-2]。尽管包括手术切除、放化疗和靶向治疗在内的相关治疗取得了一定的进展,但对于OSCC患者的5年生存率几乎没有明显提高[1]。目前早期诊断和预后精准评估可能是改善预后的有效措施之一。然而,由于部分患者的肿瘤异质性、遗传背景和致病因素的多样性,传统的临床分期和病理分级仍不能充分评估患者预后情况[3]。因此,迫切需要进一步提高预后相关指标的精确度,以便进行OSCC的早期诊断、风险分类和预后评估。
在细胞增殖过程中,如果miRNA、lncRNA、cicrRNA等RNA关系网络的失调以及基因表达异常等,就会导致各种疾病的发生、发展。最常见的就是由于增殖相关基因的表达异常导致各种肿瘤的发生、发展。有研究表明,OSCC的形成与细胞增殖和凋亡有密切的关系[4-6]。因此,本研究从MSigDB数据库中收集增殖相关基因,并与TCGA数据库中OSCC患者基因表达及临床数据进行匹配,筛选出与OSCC增殖相关基因,建立OSCC患者风险回归模型,以期为OSCC患者预后评估提供理论依据。
1 资料与方法 1.1 数据获取从MSigDB数据库(https://www.gsea-msigdb.org/gsea/msigdb/index.jsp)中下载了“c5.all.v7.1.symbols.gmt”数据集并筛选出1 899个增殖相关基因。随后,从TCGA数据库(https://cancergenome.nih.gov/)的头颈部鳞状细胞癌(TCGA-HNSC)数据集中,下载了原发部位位于口腔(舌、唇、颊、腭、牙龈、口咽等)的427例OSCC患者的转录组数据和临床数据。并根据文献[7]报道的分组方式,将427例OSCC患者按照7:3的比例分到训练组(299例)和验证组(128例)。
1.2 增殖相关基因差异表达分析通过训练组OSCC患者转录组数据与MSigDB数据库中筛选出的增殖相关基因比对获得OSCC患者的增殖相关基因表达矩阵,并运用Wilcoxon秩和检验对增殖相关基因在癌和癌旁组织中的表达进行差异分析。筛选出788个差异表达的增殖相关基因(筛选标准为|logFC|>2, FDR < 0.01)。
1.3 构建增殖相关基因风险回归模型使用R语言中的Survival包,将差异表达的增殖相关基因与训练组OSCC患者的随访数据进行单因素Cox分析,获得57个具有统计差异的增殖相关基因。随后为了避免模型过拟合,使用Lasso回归分析,去掉共线性基因后,确定了8个(APP、STC1、SFRP1、ABCB1、HPRT1、ZAP70、ST8SIA1、ADGRG1)具有预后评估价值的增殖相关基因,构建增殖相关基因风险回归模型。
1.4 评估增殖相关基因风险回归模型将训练组OSCC患者数据依据风险评分的中位值分为高风险组和低风险组,使用R语言中的Survival包,进行Kaplan-Meier生存分析,并使用R语言中的timeROC包绘制了ROC曲线。随后,将训练组OSCC患者的其他临床信息(性别、年龄、TNM分期)与风险评分结合,绘制危险因素评分列线图。最后,进行单因素和多因素独立预后分析,进一步评估该增殖相关基因风险回归模型的准确性。
1.5 验证增殖相关基因风险回归模型将验证组OSCC患者数据代入构建的增殖相关基因风险回归模型,计算其风险值,按照模型分组标准将验证组OSCC患者分为高风险组和低风险组,进行Kaplan-Meier生存分析以验证其预测OSCC患者预后的效果。
1.6 统计学分析使用R软件(4.0.0版本)进行统计学分析。双侧检验,检验水准α=0.05。
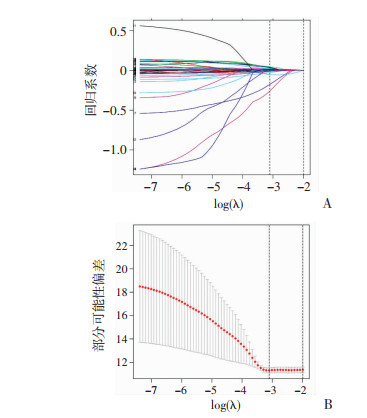
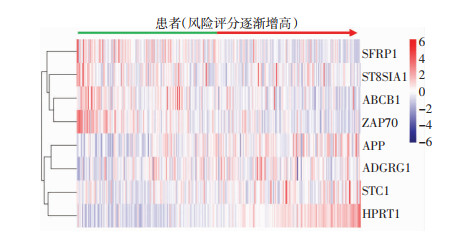
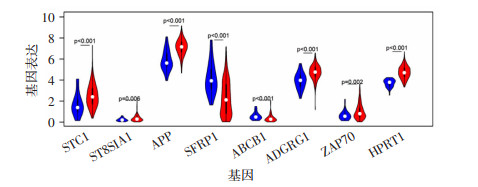
2 结果 2.1 增殖相关基因风险回归模型的建立从MSigDB和TCGA数据库获取了OSCC患者增殖相关基因,并通过差异分析、单因素Cox分析获得了57个与生存相关的基因,同时简要探究了其在染色体上的位置,发现其在7号和X染色体上分布较多(图 1)。随后使用Lasso回归分析(图 2A、B)筛选出8个具有预后评估价值的增殖相关基因:APP、STC1、SFRP1、ABCB1、HPRT1、ZAP70、ST8SIA1、ADGRG1(图 3)作为建模基因,其在OSCC患者的癌和癌旁组织中差异表达,P值均小于0.01(图 4、表 1),从而建立了增殖相关基因风险回归模型。
|
| 1~22、X、Y:染色体 图 1 生存相关差异基因的RCircos圈结果 |
|
| A:使用Lasso回归分析挑选构建模型的增殖相关基因;B:使用10倍交叉验证选择最佳的λ系数 图 2 Lasso回归模型分析 |
|
| 图 3 建模基因的热图 |
|
| 蓝色:癌旁组织;红色:癌组织;P < 0.01,OSCC患者癌旁和癌组织比较 图 4 建模基因在OSCC患者的癌旁组织中差异表达的小提琴图 |
| 建模基因 | 癌与癌旁表达量的比值(logFC) | 错误发现率(FDR) | P值 |
| STC1 | 1.208 | 1.98×10-6 | 7.63×10-7 |
| ST8SIA1 | 0.872 | 8.13×10-3 | 5.59×10-3 |
| APP | 1.125 | 1.50×10-12 | 1.99×10-13 |
| SFRP1 | -1.833 | 1.00×10-9 | 2.35×10-10 |
| ABCB1 | -0.691 | 6.95×10-6 | 2.97×10-6 |
| ADGRG1 | 0.787 | 1.77×10-9 | 4.31×10-10 |
| ZAP70 | 1.019 | 3.79×10-3 | 2.44×10-3 |
| HPRT1 | 1.160 | 9.32×10-19 | 2.35×10-20 |
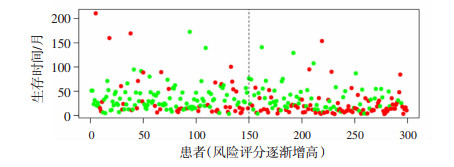
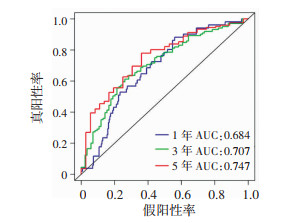
随后,训练组OSCC患者随访数据的Kaplan-Meier生存分析显示,该模型可以对OSCC患者的预后进行可靠的评估,高风险组患者的总体生存时间显著低于低风险组患者(P=1.08×10-6),其中高风险组中位生存时间为18.97个月,低风险组中位生存时间为51.12个月(图 5),且随着风险评分升高,患者风险分数增加、预后变差(图 6、7)。为了检测该模型的预测能力,按照文献[8]报道的方法,绘制了时间相关ROC曲线,结果显示,该模型具有较为满意的预测性能,其5年AUC值为0.747,95%置信区间为0.657~0.837,P=0.002(图 8)。为了便于临床实践运用,根据该模型绘制了危险因素评分列线图(图 9)。

|
| a: P < 0.01,与低风险组比较 图 5 训练组OSCC患者生存曲线 |

|
| 图 6 训练组OSCC患者风险分数分布 |
|
| 绿点:生存;红点:死亡 图 7 训练组OSCC患者增殖相关基因模型风险评分与预后的关系 |
|
| 图 8 增殖基因相关模型预测1、3、5年生存率的ROC曲线 |

|
| 图 9 OSCC患者预后相关危险因素评分列线分析 |
2.2 增殖相关基因风险回归模型的评估
使用单因素和多因素独立预后分析评估了增殖相关基因风险回归模型的准确性。单因素独立预后分析的结果表明,增殖相关基因风险回归模型风险评分能有效评估OSCC患者预后情况(P < 0.01,图 10);多因素独立预后分析的结果表明,增殖相关基因风险回归模型风险评分可作为OSCC患者的独立预后因素(P < 0.01,图 11)。

|
| 图 10 增殖相关基因风险回归模型及相关临床信息的单因素独立预后分析 |

|
| 图 11 增殖相关基因风险回归模型及相关临床信息的多因素独立预后分析 |
2.3 增殖相关基因风险回归模型的验证
将验证组OSCC患者数据代入构建的增殖相关基因风险回归模型,Kaplan-Meier生存分析显示,该模型较好地预测了OSCC患者预后,高风险组患者的总体生存时间显著低于低风险组患者(P=2.39×10-3),其中高风险组中位生存时间为36.79个月,低风险组中位生存时间为55.99个月(图 12)。

|
| a: P < 0.01, 与低风险组比较 图 12 验证组OSCC患者生存曲线 |
3 讨论
由于OSCC是一种常见的具有高发病率和高死亡率的头颈部恶性肿瘤,因此迫切需要一种功能强大且覆盖面广的生物标记物进行风险分类和预后评估[1]。虽然高通量测序技术可以在遗传水平上筛选出各种与患者生存相关的生物标志物,但测序费用高、数据量大、分析时间长。本研究使用了从MSigDB和TCGA数据库中提取的OSCC患者相关数据进行生物信息学分析,从增殖相关基因的角度出发,更加高效简便地筛选了与预后相关的预测因子,并建立了增殖相关基因风险回归模型。对比传统的临床分期和病理分级评估,多因素独立预后分析结果表明,临床分期不能作为独立预后因素,虽然病理分级可作为独立预后因素,但其风险比(HR)较小,可能无法充分评估患者的预后情况,而本研究构建的模型风险评分能较好地评估患者的预后。
在细胞生理活动过程中,细胞增殖对维持细胞生命活动起着至关重要的作用,而细胞增殖过程中相关基因的表达失调则会直接导致细胞生命活动的紊乱,最终导致各种疾病的发生、发展。因此,有关肿瘤发生、发展机制的研究离不开增殖相关基因的研究。如YANG等[9]发现增殖相关基因JMJD6的异常表达可能导致多种疾病的发生、发展,如各种肿瘤、神经性疼痛、口蹄疫、妊娠糖尿病和丙型肝炎等。YING等[10]的研究发现CCND1、NOP14和DNMT3B参与了miR-502-5p介导的膀胱癌细胞迁移和增殖的抑制。XU等[11]发现PRAME常在多种实体肿瘤中表达,例如头颈部肿瘤、乳腺癌、肾细胞癌和非小细胞肺癌等,并且参与了细胞增殖、凋亡和分化。
目前的研究表明β-淀粉样前体蛋白(APP)是一种高度保守的单一跨膜蛋白,在多种类型的肿瘤中表达增加,其与肿瘤细胞的增殖具有显著的相关性[12]。如在乳腺癌中,APP及其加工酶在癌细胞的增殖、生长、粘附和运动中起着重要作用[13]。在OSCC中,APP的表达参与OSCC的增殖和致癌作用,APP表达增加的OSCC患者生存率明显降低,APP的表达高低可以作为口腔癌变的一个潜在的标志[14]。STC1是一种分泌的糖蛋白,在乳腺癌中表达异常,并与癌细胞增殖相关,CHANG等[15]的研究发现,STC1的高表达与不良的预后相关。分泌型卷曲相关蛋白1(SFRP1)通过抑制Wnt/β-catenin通路在许多癌症中发挥抑癌作用,ZHANG等[16]的研究发现SFRP1在胃癌中低表达,它与RBMS3的共表达可作为胃癌潜在的预后指标。ABCB1即腺苷5′-三磷酸盐结合盒转运蛋白亚家族B成员1,DELOU等[17]发现在乳腺癌中,尤其是三阴乳腺癌中,ABCB1表达的缺失似乎预示了更差的预后。次黄嘌呤磷酸核糖转移酶1(HPRT1)又叫HPRT,TOWNSEND等[18]的研究发现HPRT不仅有可能成为肿瘤表征的重要生物标志物,而且还可能成为潜在的治疗手段。ZAP70是一种Syk家族的70×103酪氨酸激酶,在T细胞受体信号通路中起重要作用,FU等[19]的研究发现miR-631/ZAP70可作为研究前列腺癌细胞迁移和侵袭的新通路。ST8SIA1即ST8 α-N-乙酰神经氨酸-2, 8-唾液酸转移酶1,SHAN等[20]发现miRNA-33a和let-7e可通过调控ST8SIA1的表达抑制人结肠直肠癌的进展。黏附G蛋白偶联受体G1(ADGRG1)又叫GPR56,是各种恶性肿瘤的致癌基因,JI等[21]的研究发现GPR56通过激活PI3K/AKT信号通路,促进结直肠癌细胞增殖,并通过上皮间质转化促进转移。
由此可见本模型纳入的增殖相关基因具有文献支撑,能作为可靠的建模基因。本研究通过生物信息学分析,建立了增殖相关基因风险回归模型,并使用验证组OSCC患者数据进行验证,进一步证实了增殖相关基因风险回归模型的有效性,而多因素独立预后分析也证实该增殖相关基因风险回归模型评分可作为OSCC患者的独立预后因素。WU等[22]构建的基于独立样本T检验的数据分析模型可分析miRNA对OSCC进展的影响,但其预后预测能力偏弱。MERMOD等[23]建立的OSCC隐匿性淋巴结转移多变量预测模型,降低了过度治疗的风险。而我们根据肿瘤发生、发展机制的研究热点基因,首次构建了增殖相关基因风险回归模型,通过肿瘤发生、发展的最直观指标评估患者的预后情况。
综上,本研究从MSigDB和TCGA数据库中挖掘了OSCC增殖相关基因,建立了增殖相关基因风险回归模型,为OSCC患者预后风险评估提供了较为有效的预测模型,以期为提高OSCC患者5年生存率奠定理论基础。
| [1] |
CHI A C, DAY T A, NEVILLE B W. Oral cavity and oropharyngeal squamous cell carcinoma: an update[J]. CA Cancer J Clin, 2015, 65(5): 401-421. DOI:10.3322/caac.21293 |
| [2] |
CHATURVEDI A K, ANDERSON W F, LORTET-TIEULENT J, et al. Worldwide trends in incidence rates for oral cavity and oropharyngeal cancers[J]. J Clin Oncol, 2013, 31(36): 4550-4559. DOI:10.1200/JCO.2013.50.3870 |
| [3] |
DISSANAYAKA W L, PITIYAGE G, KUMARASIRI P V, et al. Clinical and histopathologic parameters in survival of oral squamous cell carcinoma[J]. Oral Surg Oral Med Oral Pathol Oral Radiol, 2012, 113(4): 518-525. DOI:10.1016/j.oooo.2011.11.001 |
| [4] |
GENG F X, ZHANG Y J, LU Z, et al. Fusobacterium nucleatum caused DNA damage and promoted cell proliferation by the Ku70/p53 pathway in oral cancer cells[J]. DNA Cell Biol, 2020, 39(1): 144-151. DOI:10.1089/dna.2019.5064 |
| [5] |
TIAN L, ZHANG Y, WANG Y, et al. Triptolide reduces proliferation and enhances apoptosis in drug-resistant human oral cancer cells[J]. Int J Clin Exp Pathol, 2019, 12(4): 1204-1213. |
| [6] |
LI Y Y, TAO Y W, GAO S, et al. Cancer-associated fibroblasts contribute to oral cancer cells proliferation and metastasis via exosome-mediated paracrine miR-34a-5p[J]. EBio Medicine, 2018, 36: 209-220. DOI:10.1016/j.ebiom.2018.09.006 |
| [7] |
PARK H S, PARK J S, CHUN Y J, et al. Prognostic factors and scoring model for survival in metastatic biliary tract cancer[J]. Cancer Res Treat, 2017, 49(4): 1127-1139. DOI:10.4143/crt.2016.538 |
| [8] |
HUANG R, MAO M, LU Y, et al. A novel immune-related genes prognosis biomarker for melanoma: associated with tumor microenvironment[J]. Aging (Albany NY), 2020, 12(8): 6966-6980. DOI:10.18632/aging.103054 |
| [9] |
YANG J, CHEN S, YANG Y, et al. Jumonji domain-containing protein 6 protein and its role in cancer[J]. Cell Prolif, 2020, 53(2): e12747. DOI:10.1111/cpr.12747 |
| [10] |
YING Y, LI J, XIE H, et al. CCND1, NOP14 and DNMT3B are involved in miR-502-5p-mediated inhibition of cell migration and proliferation in bladder cancer[J]. Cell Prolif, 2020, 53(2): e12751. DOI:10.1111/cpr.12751 |
| [11] |
XU Y, ZOU R, WANG J, et al. The role of the cancer testis antigen PRAME in tumorigenesis and immunotherapy in human cancer[J]. Cell Prolif, 2020, 53(3): e12770. DOI:10.1111/cpr.12770 |
| [12] |
LIM S, YOO B K, KIM H S, et al. Amyloid-β precursor protein promotes cell proliferation and motility of advanced breast cancer[J]. BMC Cancer, 2014, 14: 928. DOI:10.1186/1471-2407-14-928 |
| [13] |
DANISH RIZVI S M, HUSSAIN T, SUBAIEA G M, et al. Therapeutic targeting of amyloid precursor protein and its processing enzymes for breast cancer treatment[J]. Curr Protein Pept Sci, 2018, 19(9): 841-849. DOI:10.2174/1389203718666170828123924 |
| [14] |
KO S Y, LIN S C, CHANG K W, et al. Increased expression of amyloid precursor protein in oral squamous cell carcinoma[J]. Int J Cancer, 2004, 111(5): 727-732. DOI:10.1002/ijc.20328 |
| [15] |
CHANG A C, DOHERTY J, HUSCHTSCHA L I, et al. STC1 expression is associated with tumor growth and metastasis in breast cancer[J]. Clin Exp Metast, 2015, 32(1): 15-27. DOI:10.1007/s10585-014-9687-9 |
| [16] |
ZHANG T, WU Y, FANG Z, et al. Low expression of RBMS3 and SFRP1 are associated with poor prognosis in patients with gastric cancer[J]. Am J Cancer Res, 2016, 6(11): 2679-2689. |
| [17] |
DELOU J M A, VIGNAL G M, ÍNDIO-DO-BRASIL V, et al. Loss of constitutive expression in breast cancer associated with worse prognosis[J]. Breast Cancer(Dove Med Press), 2017, 9: 415-428. DOI:10.2147/BCTT.S131284 |
| [18] |
TOWNSEND M H, ROBISON R A, O'NEILL K L. A review of HPRT and its emerging role in cancer[J]. Med Oncol, 2018, 35(6): 89. DOI:10.1007/s12032-018-1144-1 |
| [19] |
FU D, LIU B, ZANG L E, et al. MiR-631/ZAP70: A novel axis in the migration and invasion of prostate cancer cells[J]. Biochem Biophys Res Commun, 2016, 469(3): 345-351. DOI:10.1016/j.bbrc.2015.11.093 |
| [20] |
SHAN Y, LIU Y, ZHAO L, et al. MicroRNA-33a and let-7e inhibit human colorectal cancer progression by targeting ST8SIA1[J]. Int J Biochem Cell Biol, 2017, 90: 48-58. DOI:10.1016/j.biocel.2017.07.016 |
| [21] |
JI B, FENG Y, SUN Y, et al. GPR56 promotes proliferation of colorectal cancer cells and enhances metastasis via epithelial mesenchymal transition through PI3K/AKT signaling activation[J]. Oncol Rep, 2018, 40(4): 1885-1896. DOI:10.3892/or.2018.6582 |
| [22] |
WU C, YANG M, CHEN H. Inhibition effect of miR-150 on the progression of oral squamous cell carcinoma by data analysis model based on independent sample T-test[J]. Saudi J Biol Sci, 2020, 27(2): 599-605. DOI:10.1016/j.sjbs.2019.11.022 |
| [23] |
MERMOD M, JOURDAN E F, GUPTA R, et al. Development and validation of a multivariable prediction model for the identification of occult lymph node metastasis in oral squamous cell carcinoma[J]. Head Neck, 2020. DOI:10.1002/hed.26105 |



