近年来, 基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption/ionization time-of-flight mass spectrometry, MALDI-TOF MS)技术在临床微生物中的应用越来越广泛[1-3]。随着碳青霉烯类药物在临床的大量使用, 碳青霉烯类耐药铜绿假单胞菌的出现和流行给临床抗感染治疗带来了严峻的挑战[4-5]。因此, 本研究利用MALDI-TOF MS技术建立了VIM型和SPM型金属酶铜绿假单胞菌(metallo-β-lactamase Pseudomonas aeruginosa, MBL-PA)的识别模型数据库, 并对其鉴定能力进行了性能评价, 进而明确MALDI-TOF MS在检测MBL-PA类型中的作用。
1 材料与方法 1.1 菌株收集2015—2018年重庆某三甲医院临床各个科室患者痰液、尿液、肺泡灌洗液、血液、分泌物、脓液等标本培养出的耐碳青霉烯类药铜绿假单胞菌258株, 质控菌株大肠埃希菌ATCC8739和铜绿假单胞菌ATCC27853均购于卫生部临检中心。
1.2 菌株鉴定、药敏试验及MBL-PA的表型测定临床分离的菌株使用VITEK MS进行鉴定, 利用VITEK-2 Compact进行药敏检测, 采用EDTA协同试验测定MBL-PA的表型, 具体参照文献[6]进行。
1.3 耐药基因检测铜绿假单胞菌MBL基因采用聚合酶链反应(polymerase chain reaction, PCR)方法进行测定, 挑取纯培养菌落置含200 μL蛋白酶K溶液的0.5 mL离心管中, 56 ℃温育2 h进行模板基因提取, -20 ℃冰箱保存备用。靶基因包括:IMP、VIM、SPM、GIM、SIM、NDM基因, 引物具体信息见表 1。由北京六合华大基因科技有限公司合成, 按94 ℃预变性5 min; 94 ℃变性30 s; 退火30 s; 72 ℃, 延伸60 s; 变性、退火、延伸过程设置30个循环;然后72 ℃继续延伸7 min,扩增产物经2%琼脂凝胶电泳后观察是否出现与目的基因相对分子质量相当的条带来判断结果。PCR扩增阳性的产物送重庆擎科兴业生物技术有限公司测序, 测序结果在GenBank网上进行Blast比对以确定基因型。
| 基因名称 | 引物序列(5′→3′) | 产物长度(bp) | 退火温度(℃) |
| IMP | 正义:CTACCGCAGCAGAGTCTTTG | ||
| 反义:AACCAGTTTTGCCTTACCAT | 587 | 50 | |
| VIM | 正义:GTTTGGTCGCATATCGCAAC | ||
| 反义:AATGCGCAGCACCAGGATAG | 382 | 55 | |
| SPM | 正义:CTGCTTGGATTCATGGGCGC | ||
| 反义:CCTTTTCCGCGACCTTGATC | 783 | 61 | |
| GIM | 正义:TCGACACACCTTGGTCTGAA | ||
| 反义:AACTTCCAACTTTGCCATGC | 713 | 58 | |
| SIM | 正义:TACAAGGGATTCGGCATCG | ||
| 反义:TAATGGCCTGTTCCCATGTG | 741 | 58 | |
| NDM | 正义:GGGCAGTCGCTTCCAACGGT | ||
| 反义:GTAGTGCTCAGTGTCGGCAT | 475 | 62 |
1.4 建立VIM型和SPM型MBL-PA识别模型数据库
按照质谱建库的标准, 我们前期选取临床分离的15株VIM型和15株SPM型MBL-PA的质谱光谱作为参考菌株, 每个菌株重复进行8次质谱实验。通过对VIM型和SPM型MBL-PA的质谱峰进行聚类分析, 找到二者间共有的和差异的质谱特征峰, 最后选取部分VIM型和非VIM型菌株的特征峰作为鉴定VIM型MBL-PA的识别模型数据库, 选取部分SPM型和非SPM型菌株的特征峰作为鉴定SPM型MBL-PA的识别模型数据库。
1.5 临床菌株验证新建质谱识别模型数据库为验证数据库的准确性, 我们选取临床分离的25株VIM型和20株SPM型MBL-PA作为验证菌株, 各标本1 μL上清蛋白提取液覆盖于靶板上, 均匀涂抹开, 待完全干燥后再覆盖1 μL基质液, 均匀涂抹开, 待完全干燥后于VITEK MS质谱仪上进行检测分析。
2 结果 2.1 耐碳青霉烯类药铜绿假单胞菌基本情况2015~2018年重庆某三甲医院258株耐碳青霉烯类药铜绿假单胞菌对10种抗菌药物的耐药率分别为阿米卡星(73%), 妥布霉素(71%), 哌拉西林/他唑巴坦(49%), 头孢吡肟(75%), 庆大霉素(77%), 左旋氧氟沙星(76%), 头孢他啶(76%), 环丙沙星(73%), 亚胺培南(100%), 氨曲南(64%), 见表 2。EDTA协同试验结果显示:258株菌中有106株金属酶表型初筛实验为阳性。
| 抗菌药物 | 耐药 | 中介 | 敏感 |
| 阿米卡星 | 73 | 4 | 23 |
| 妥布霉素 | 71 | 7 | 22 |
| 哌拉西林/他唑巴坦 | 49 | 12 | 39 |
| 头孢吡肟 | 75 | 4 | 21 |
| 庆大霉素 | 77 | 2 | 21 |
| 左旋氧氟沙星 | 76 | 3 | 21 |
| 头孢他啶 | 76 | 2 | 22 |
| 环丙沙星 | 73 | 5 | 22 |
| 亚胺培南 | 100 | 0 | 0 |
| 氨曲南 | 64 | 5 | 31 |
2.2 耐碳青霉烯类药铜绿假单胞菌金属酶基因检测结果
将106株金属酶表型初筛实验阳性菌株进行MBL基因扩增, 得到VIM型阳性40株, SPM型阳性35株, 未扩增出IMP、SIM、GIM和NDM阳性菌株。部分菌株PCR产物电泳及测序结果见图 1、2。
|
| M:标准; 5, 6, 7, 8, 9, 10, 11, 12, 13, 18:为VIM菌株 图 1 VIM基因阳性PCR产物电泳(A)及测序结果部分峰(B)图 |
|
| M:标准; 1, 2, 3, 4, 14, 15, 17, 21, 22, 24:为SPM菌株 图 2 SPM基因阳性PCR产物电泳(A)及测序结果部分峰(B)图 |
2.3 VIM型和SPM型MBL-PA识别模型数据库建立
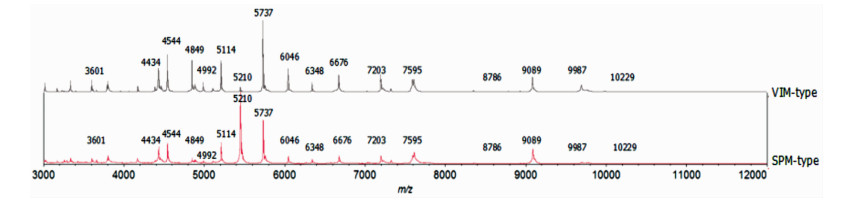
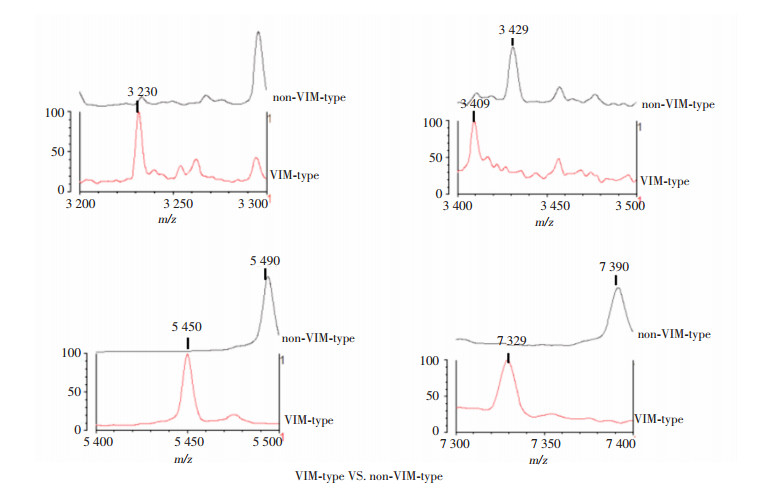
通过VITEK MS质谱仪的LaunchPad软件将VIM型和SPM型MBL-PA质谱峰进行比较分析, 得到17个高信号强度峰作为两者的共有质量峰(3 601 m/z, 4 434 m/z, 4 544 m/z, 4 849 m/z, 4 992 m/z, 5 114 m/z, 5 210 m/z, 5 737 m/z, 6 046 m/z, 6 348 m/z, 6 676 m/z, 7 203 m/z, 7 595 m/z, 8 786 m/z, 9 089 m/z, 9 987 m/z和10 229 m/z), 见图 3。为筛选VIM型和SPM型菌株之间有差异的特征峰, 再次利用LaunchPad软件分别比较VIM型菌株和非VIM型(包括SPM型和不产MBL-PA)菌株以及SPM型菌株和非SPM型(包括VIM型和不产MBL-PA)菌株的质谱特征峰, 最后选取VIM型菌株的4个特征峰(3 230 m/z, 3 409 m/z, 5 450 m/z, 7 329 m/z)和非VIM型菌株的3个特征峰(3 429 m/z, 5 490 m/z, 7 390 m/z)作为鉴定VIM型菌株的模型(图 4), 选取SPM型菌株的2个特征峰(3 663 m/z, 6264 m/z)和非SPM型菌株的2个特征峰(4 068 m/z, 4 282 m/z)作为鉴定SPM型菌株的模型(图 5)。
|
| 图 3 VIM型和SPM型MBL-PA共有特征峰 |
|
| 图 4 VIM型MBL-PA识别模型 |
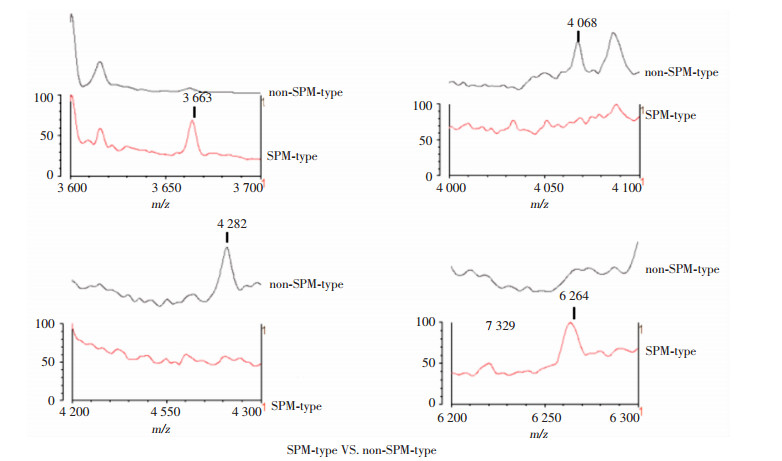
|
| 图 5 SPM型MBL-PA识别模型 |
2.4 VIM型和SPM型MBL-PA识别模型数据库的验证
新建识别模型数据库验证结果显示MALDI-TOF MS可正确鉴定23/25 VIM型菌株, 灵敏度为92.0%, 特异性为100.0%, 阳性预测值为100.0%, 阴性预测值为93.7%。对于SPM型菌株的检测灵敏度为80.0%, 特异性为100.0%, 阳性预测值均为100.0%, 阴性预测值为88.2%, 详见表 3。
| 类型 | 验证前数量 | 特征峰(m/z) | 验证后数量 | 灵敏度(%) | 特异性(%) | 阳性预测值(%) | 阴性预测值(%) |
| VIM型 | 92.0 | 100.0 | 100.0 | 93.7 | |||
| VIM菌株 | 25 | 3 230; 3 409; 5 450; 7 329 | 23 | ||||
| 非VIM菌株 | 30 | 3 429; 5 490; 7, 390 | 30 | ||||
| SPM菌株 | 80.0 | 100.0 | 100.0 | 88.2 | |||
| SPM菌株 | 20 | 3 663; 6 264 | 16 | ||||
| 非SPM菌株 | 30 | 4 068; 4 282 | 30 |
3 讨论
258株耐碳青霉烯类药铜绿假单胞菌经PCR耐药基因检测扩增出MBL基因阳性75株, 其中VIM型40株, SPM型35株, 未扩增出其他基因型阳性菌株, 这一结果与国内多家医院报道MBL以IMP基因型和VIM基因型为主的研究存在差异, 提示金属酶的基因型分布存在地区差异[7-9]。此外, 本研究结果还提示2015—2018年医院院内可能存在不同程度耐碳青霉烯类药铜绿假单胞菌交叉感染的现象, 其中部分菌株虽对碳青霉烯类耐药, 但并未检测到金属β-内酰胺酶, 因此碳青霉烯类耐药铜绿假单胞菌除产金属β-内酰胺酶以外, 还存在其他的耐药机制, 如OprD2基因缺失, 主动外排系统过度表达, 生物膜形成等[10-13]。因此, 对于这部分耐药机制尚不明确的菌株, 我们后期将对其耐药机制进行深入研究。
目前, MBL-PA的检测已引起了国外研究者的广泛关注, 而国内对MBL-PA的检测大多数还是停留在细菌分离培养后对其进行表型初筛实验的传统做法上[14-17]。传统的细菌培养耗时长, 以EDTA协同试验为代表的金属酶表型初筛实验在培养的基础上还需要18~24 h才能判定是否产金属酶, 并且金属酶表型初筛试验的结果极不稳定, 不能满足临床快速准确检测耐药表型的需求。近年来, 随着分子生物学技术的临床应用, 我们除去传统的金属酶表型初筛方法外, 还可以通过PCR的方法对MBL-PA的基因型进行分析, 但PCR检测MBL-PA基因型目前仍存在操作步骤多、易污染, 不能直接检测临床标本等缺点。因此, 目前急需开发一种MBL-PA鉴定及分型的新方法。MALDI-TOF MS作为临床细菌鉴定及其耐药性分析的一种新方法, 目前已广泛应用于病原微生物的鉴定[18-20]。并且质谱检测具有操作简单、快速、能直接检测临床标本等优点, 已引起越来越多研究者的关注[21-23]。本研究前期根据临床质谱自建库的相关指南, 再结合实验室自身需求, 通过MALDI-TOF MS对VIM型和SPM型MBL-PA的质谱蛋白特征峰进行分析, 建立区分VIM型和SPM型MBL-PA两种亚型的识别模型; 本研究通过该识别模型得到MALDI-TOF MS对25株VIM型MBL-PA的鉴定准确率为92%, 对20株SPM型MBL-PA的鉴定准确率为80%。该研究结果提示:我们前期通过自建VIM型和SPM型MBL-PA的质谱数据库, 能较好地对VIM型和SPM型MBL-PA进行区分, 具有非常重要的临床应用价值。
此外, 本研究仍存在许多不足之处, 质谱数据库验证结果中出现了4株SPM型MBL-PA被误鉴定为VIM型MBL-PA, 2株VIM型MBL-PA被误鉴定为SPM型MBL-PA。分析其原因可能是细菌蛋白提取过程出现污染, 导致质谱获取的蛋白图谱与数据库中另外一种类型识别模型图谱相似, 这种情况下会明显地干扰数据库的分析和鉴定。另外, 也可能是由于细菌基因转移与遗传背景差异等因素影响了细菌蛋白的表达, 导致部分基因型蛋白表达出现了一定的差异[24-26]。因此, 这些结果也提示我们需要进一步优化质谱识别模型, 不仅需要对4株错误鉴定的SPM型和2株错误鉴定的VIM型MBL-PA质谱峰图进行差异性对比分析, 而且还需要继续收集更多的菌株扩大建库的样本量, 以便能提高新建质谱数据库对MBL-PA分型鉴定的准确率。本研究结果不仅丰富了对质谱建库技术对鉴定MBL-PA亚型研究的认识, 而且对于今后质谱建库技术应用于其他病原菌分型的检测也具有指导和借鉴意义, 该方法具备非常广的应用前景。
| [1] |
LI J, HU W W, ZHANG F L, et al. Evaluation of matrix-assisted laser desorption/ionization time-of-flight mass spectrometry for identifying Burkholderia pseudomallei and Burkholderia thailandensis isolates[J]. Eur J Clin Microbiol Infect Dis, 2019, 38(1): 191-196. DOI:10.1007/s10096-018-3415-3 |
| [2] |
GU Y F, LI Y, ZHANG X L, et al. A new method aimed to quickly identify pathogen and drug susceptibility test based on matrix-assisted laser desorption/ionization time of flight mass spectrometry combined with flow cytometry[J]. Surg Infect (Larchmt), 2019, 20(3): 219-224. DOI:10.1089/sur.2018.145 |
| [3] |
HU Y Y, HUANG Y L, LIZOU Y Z, et al. Evaluation of Staphylococcus aureus subtyping module for methicillin-resistant Staphylococcus aureus detection based on matrix-assisted laser desorption ionization time-of-flight mass spectrometry[J]. Front Microbiol, 2019, 10: 2504. DOI:10.3389/fmicb.2019.02504 |
| [4] |
RIBEIRO Á C D S, CROZATTI M T L, SILVA AA D, et al. Pseudomonas aeruginosa in the ICU: prevalence, resistance profile, and antimicrobial consumption[J]. Rev Soc Bras Med Trop, 2019, 53: e20180498. DOI:10.1590/0037-8682-0498-2018 |
| [5] |
EL-MAHDY R, EL-KANNISHY G. Virulence factors ofcarbapenem-resistant Pseudomonas aeruginosa in hospital-acquired infections in mansoura, Egypt[J]. Infect Drug Resist, 2019, 12: 3455-3461. DOI:10.2147/IDR.S222329 |
| [6] |
李进, 胡韦维, 张峰领, 等. 重庆某三甲医院2014-2016年铜绿假单胞菌耐药表型及金属酶、外膜孔蛋白耐药基因型分析[J]. 第三军医大学学报, 2017, 39(22): 2200-2205. LI J, HU W W, ZHANG F L, et al. Drug resistance profiles and metallo-β-lactamase and outer membrane protein OprD2 genotypes of Pseudomonas aeruginosa: a 2014-2016 survey in a Grade 3 Class A general hospital in Chongqing[J]. J Third Mil Med Univ, 2017, 39(22): 2200-2205. DOI:10.16016/j.1000-5404.201706013 |
| [7] |
曾婷, 曾凌, 曹先伟, 等. 耐碳青霉烯类铜绿假单胞菌耐药基因与药敏分析[J]. 中华医院感染学杂志, 2019, 29(20): 3063-3067. ZENG T, ZENG L, CAO X W, et al. Antimicrobial susceptibility and resistance genes of carbapenem-resistant Pseudomonas aeruginosa[J]. Chin J Nosocomiol, 2019, 29(20): 3063-3067. |
| [8] |
刘先林, 欧阳琳, 谢伟, 等. 赣南地区耐碳青霉烯类药物铜绿假单胞菌耐药基因检测结果及其对药物的耐药性分析[J]. 抗感染药学, 2019, 16(9): 1512-1514. LIU X L, OUYANG L, XIE W, et al. Detection results of resistance genes of carbapenem-resistant Pseudomonas aeruginosa and analysis of drug resistance in South Jiangxi Province[J]. Anti-Infect Pharm, 2019, 16(9): 1512-1514. |
| [9] |
骆远宁. 耐碳青霉烯类铜绿假单胞菌的临床分布及其对抗菌药物的耐药性分析[J]. 临床合理用药杂志, 2019, 12(15): 93-94. LUO Y N. Clinical distribution ofcarbapenem-resistant Pseudomonas aeruginosa and its resistance to antibacterial drugs[J]. Chin J Clin Ration Drug Use, 2019, 12(15): 93-94. DOI:10.15887/j.cnki.13-1389/r.2019.15.045 |
| [10] |
许小敏, 范友芬, 张淳, 等. 烧伤患者耐碳青霉烯类铜绿假单胞菌感染影响因素分析[J]. 中华医院感染学杂志, 2019, 29(12): 1791-1795. XU X M, FAN Y F, ZHANG C, et al. Risk factors of carbapenem-resistant Pseudomonas aeruginosa infection in burn patients[J]. Chin J Nosocomiol, 2019, 29(12): 1791-1795. |
| [11] |
潘亚萍.外排泵MexAB-OprM在铜绿假单胞菌对碳青霉烯类抗生素耐药机制的研究[D].合肥: 安徽医科大学, 2017. PAN Y P. Mechanism ofMexAB-OprM efflux pump in carbapenem-resistant Pseudomonas aeruginosa[D]. Hefei: Anhui Medical University, 2017. |
| [12] |
袁莉莉, 丁百兴, 沈震, 等. 耐碳青霉烯类铜绿假单胞菌的耐药性及分子流行病学研究[J]. 中国感染与化疗杂志, 2017, 17(3): 289-292. YUAN L L, DING B X, SHEN Z, et al. Antibiotic resistance and molecular epidemiology of carbapenem-resistant Pseudomonas aeruginosa[J]. Chin J Infect Chemother, 2017, 17(3): 289-292. DOI:10.16718/j.1009-7708.2017.03.012 |
| [13] |
张智琪.氟喹诺酮药物诱导铜绿假单胞菌生物膜及耐药机制研究[D].太原: 山西医科大学, 2018. ZHANG Z Q. Mechanism of resistance and biofilm of Pseudomonas aeruginosa induced by fluoroquinolones in vitro[D]. Taiyuan: Shanxi Medical University, 2018. |
| [14] |
SONNABEND M S, KLEIN K, BEIER, et al. Identification of drug resistance determinants in a clinical isolate of Pseudomonas aeruginosa by high-density transposon mutagenesis[J]. Antimicrob Agents Chemother, 2020, 64(3): e01771-e01719. DOI:10.1128/AAC.01771-19 |
| [15] |
徐雁峰, 王慧敏, 张冬. 铜绿假单胞菌对碳青霉烯类抗生素耐药机制研究[J]. 包头医学院学报, 2019, 35(9): 108-112. XU Y F, WANG H M, ZHANG D. Study on the resistance mechanism of Pseudomonas aeruginosa tocarbapenem antibiotics[J]. J Baotou Med Coll, 2019, 35(9): 108-112. DOI:10.16833/j.cnki.jbmc.2019.09.048 |
| [16] |
KHALILI Y, YEKANI M, GOLI H R, et al. Characterization of carbapenem-resistant but cephalosporin-susceptible Pseudomonas aeruginosa[J]. Acta Microbiol Immunol Hung, 2019, 66(4): 529-540. DOI:10.1556/030.66.2019.036 |
| [17] |
PETROVA A, FEODOROVA Y, MITEVA-KATRANDZH-IEVA T, et al. First detected OXA-50 carbapenem-resistant clinical isolates Pseudomonas aeruginosa from Bulgaria and interplay between the expression of main efflux pumps, OprD and intrinsic AmpC[J]. J Med Microbiol, 2019, 68(12): 1723-1731. DOI:10.1099/jmm.0.001106 |
| [18] |
ALCOLEA-MEDINA A, FERNANDEZ M T C, MONTIEL N, et al. An improved simple method for the identification of Mycobacteria by MALDI-TOF MS (Matrix-Assisted Laser Desorption-Ionization mass spectrometry)[J]. Sci Rep, 2019, 9(1): 20216. DOI:10.1038/s41598-019-56604-7 |
| [19] |
PENG Y, ZHANG Q, XU C, et al. MALDI-TOF MS for the rapid identification and drug susceptibility testing of filamentous fungi[J]. Exp Ther Med, 2019, 18(6): 4865-4873. DOI:10.3892/etm.2019.8118 |
| [20] |
SAMARANAYAKE W A M P, DEMPSEY S, HOWARD-JONES A R, et al. Rapid direct identification of positive paediatric blood cultures by MALDI-TOF MS technology and its clinical impact in the paediatric hospital setting[J]. BMC Res Notes, 2020, 13(1): 12. DOI:10.1186/s13104-019-4861-4 |
| [21] |
PIAMSOMBOON P, JARESITTHIKUNCHAI J, HUNG T Q, et al. Identification of bacterial pathogens in cultured fish with a custom peptide database constructed by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF MS)[J]. BMC Vet Res, 2020, 16(1): 52. DOI:10.1186/s12917-020-2274-1 |
| [22] |
QUÉRO L, COURAULT P, CELLIÈRE B, et al. Application of MALDI-TOF MS to species complex differentiation and strain typing of food related fungi: Case studies with Aspergillus section Flavi species and Penicillium roqueforti isolates[J]. Food Microbiol, 2020, 86: 103311. DOI:10.1016/j.fm.2019.103311 |
| [23] |
LUO LL, LIU W J, LI B, et al. Evaluation of matrix-assisted laser desorption ionization-time of flight mass spectrometry for identification of Mycobacterium abscessus subspecies according to whole-genome sequencing[J]. J Clin Microbiol, 2016, 54(12): 2982-2989. DOI:10.1128/JCM.01151-16 |
| [24] |
YOO G Y, KIM E, KANG H, et al. Mass spectrometric investigation of concentration-dependent effect of curcumin and oxidative stress on intracellular glutathione levels[J]. Anal Bioanal Chem, 2020, 412(12): 2873-2880. DOI:10.1007/s00216-020-02524-9 |
| [25] |
SÁNCHEZ-JUANES F, TEIXEIRA-MARTÍN V, GONZÁLEZ-BUITRAGO J M, et al. Identification of species and subspecies of lactic acid bacteria present in Spanish cheeses type "torta" by MALDI-TOF MS and pheS gene analyses[J]. Microorganisms, 2020, 8(2): E301. DOI:10.3390/microorganisms8020301 |
| [26] |
GOU JJ, LIU N, GUO L H, et al. Carbapenem-resistant Enterobacter hormaechei ST1103 with IMP-26 carbapenemase and ESBL gene blaSHV-178[J]. Infect Drug Resist, 2020, 13: 597-605. DOI:10.2147/IDR.S232514 |



