2. 100700 北京,解放军总医院第七医学中心全军皮肤损伤修复研究所;
3. 100120 北京,军委政治工作部原机关门诊部;
4. 150001 哈尔滨,93199 部队门诊部
2. PLA Institute of skin Damage and Repair, The Seventh Medical Center of Chinese PLA General Hospital, Beijing, 100700;
3. Clinic of Former Organization of Political Work Department of the Military Commission, Beijing, 100120;
4. Clinic of 93199 Troop of PLA, Harbin, Heilongjiang Province, 150001, China
播散性毛孢子菌病是一种可累及包括皮肤、泌尿道、肺、肝脏、脾脏、中枢神经系统、心脏等器官的复杂真菌感染[1]。如诊治不及时,其病死率非常高,特别是中性粒细胞减少的癌症患者、接受侵袭性治疗以及广谱抗生素治疗的危重患者,病死率为30%~80%不等[2-3]。而阿萨希毛孢子菌(Trichosporon asahii,T. asahii)是引起播散性毛孢子菌病的最主要致病菌,近年来该菌的感染也呈明显上升的趋势。DE等[2]回顾分析了1994~2015年间537例侵袭性毛孢子菌感染的报道,其中由T. asahii引起的感染数量从1994~2004年间的25.8%,迅速增长到2005~2015年间的74.2%。GUO等[4]对2009~2016年间中国多个中心收集并经DNA准确鉴定的133株毛孢子菌分析发现,其中81.2%(108株)是T. asahii感染。以氟康唑为代表的三唑类抗真菌药物是临床上治疗毛孢子菌感染的最常用药物,而且对大部分T. asahii感染有效,但近年来,有关该菌对氟康唑等三唑类耐药的报道不断增多[2-4]。考虑到现有的对T. asahii具有抗真菌活性的药物数量有限,对三唑类药物耐药的发展可能严重影响侵袭性感染患者的病死率。但目前该菌对唑类药物耐药的机制不清,因此,开展唑类药物耐药机制研究,对寻找新的药物作用靶点、开发新型抗真菌药、降低患者病死率均有重要意义。
1 材料与方法 1.1 实验菌株从我中心保存的T. asahii临床分离株中挑取了3株,编号分别为JM4、JM5、JM6,其中JM4分离至国内首例播散性毛孢子菌病患者面部皮肤,JM5和JM6分别分离至皮肤感染和泌尿系感染者的皮肤和尿液。
1.2 药物试剂和仪器药物:氟康唑干粉购于美国Sigma公司。试剂:RPMI-1640干粉、二甲基亚砜(DMSO)、3-N-吗啉基丙磺酸干粉购于美国Sigma公司;酵母提取物购于英国Oxoid公司;PBS磷酸盐缓冲液干粉购于美国Hyclone公司;Trizol RNA提取试剂盒购于美国Invitrogen公司;真菌DNA提取试剂盒购于美国Omega公司;DNaseI购于美国Thermo公司;KAPA链RNA-Seq试剂盒、KAPA DNA样品制备试剂盒购于美国Kapa公司。仪器:HiSeq 3000第二代基因测序仪,美国Illumina公司。
1.3 方法 1.3.1 菌株复苏与药敏实验从-80 ℃冰箱中取出实验用菌株,融化后挑取少量菌落接种至马铃薯葡萄糖琼脂培养基(PDA)上培养48 h,再取单菌落转种培养48 h。制备菌悬液并进行菌落计数,参照CLSI指南中推荐的M27-A3标准化方案[5]对3株菌株进行氟康唑药敏实验,测定其MIC值。
1.3.2 氟康唑体外诱导参照T. asahii氟康唑体外诱导方法[6],对3株菌进行体外诱导耐药培养。氟康唑浓度从0.25 μg/mL开始,依次倍增,直至氟康唑浓度≥128 μg/mL,再无药传代30次,传代结束后进行氟康唑药敏实验,测定其MIC值。
1.3.3 RNA提取与转录组测序将敏感菌株JM4及其体外诱导培养获得的耐药菌株(JM9)的单菌落分别接种于PDA固体培养基中,37 ℃培养48 h。用Trizol RNA提取试剂盒分别提取2株菌的总RNA,然后用DNase I去除潜在的基因组DNA污染。在定量和质量控制后,用KAPA链RNA-Seq试剂盒,从这些RNA样品中构建RNA-Seq文库。以上过程的具体步骤均参照试剂盒说明书进行。然后遵循标准Illumina样品制备方案并使用HiSeq 3000二代基因测序仪进行高通量测序。
1.3.4 DNA提取与基因组测序将敏感菌株JM4、JM5、JM6及其体外诱导耐药菌株JM9、JM10、JM11的单菌落分别接种于PDA固体培养基中,37 ℃培养至48 h。参照试剂盒使用说明书,用真菌DNA提取试剂盒分别提取6株菌的DNA,定量化后用KAPA DNA样品制备试剂盒,构建双末端的DNA文库。然后遵循标准Illumina样品制备方案并使用HiSeq 3000二代基因测序仪进行高通量测序。
1.3.5 生物信息学分析对T. asahii转录组进行高通量测序后,首先去除了低质量的序列和接头序列,然后使用TopHat软件(版本号v2)将转录组测序读段与T. asahii标准株(CBS 2479)基因组进行比对。通过公式FPKM(每百万测序读段中每Kb碱基长度上比对上的测序读段的数目)评估基因的表达水平。通过R语言包中edgeR包计算差异表达的基因(DEG)。标准如下:如果某基因的FDR值< 0.001且倍数变化> 1.3(上调)或 < 0.7(下调),则认为这些基因是差异表达基因。使用ABySS软件拼接敏感菌株与耐药菌株的基因组序列,通过BLASTN软件鉴定出ERG11的基因序列。
2 结果 2.1 药敏与氟康唑体外诱导诱导前3株菌JM4、JM5、JM6的氟康唑MIC值分别为2 μg/mL、1 μg/mL、2 μg/mL,氟康唑体外诱导后及无药传代30代后其MIC值均≥64 μg/mL,意味着将3株氟康唑敏感菌株诱导成为稳定的氟康唑耐药菌株,并分别命名为JM9、JM10、JM11(表 1)。
| 菌株编号 | 来源 | 诱导前MIC值 | 诱导后MIC值 | 传代后MIC值(编号) |
| JM4 | 皮肤 | 2 μg/mL | ≥128 μg/mL | ≥64 μg/mL(JM9) |
| JM5 | 皮肤 | 1 μg/mL | ≥128 μg/mL | ≥64 μg/mL(JM10) |
| JM6 | 尿液 | 2 μg/mL | ≥64 μg/mL | ≥64 μg/mL(JM11) |
2.2 RNA提取与转录组测序质量
成功提取了敏感菌株JM4及其诱导耐药菌株JM9的总RNA,均未发生降解,可用于建立文库及高通量测序。对转录组测序进行2个生物学重复,表 2是转录组测序数据统计表,总共获得4Gb高质量数据量,两株菌的转录组测序数据饱和图如图 1所示,表明测序数据量可以满足后续对表达量差异变化基因的筛选。
| 样本 | 原始序列数 | 原始数据量/bp | 高质量序列数 | 高质量数据量/bp |
| 敏感株重复1 | 12633260 | 1212792960 | 12095840 | 1037129254 |
| 敏感株重复2 | 12633260 | 1212792960 | 12095840 | 931176844 |
| 耐药株重复1 | 10726491 | 1029743136 | 10332991 | 952714950 |
| 耐药株重复2 | 10726491 | 1029743136 | 10332991 | 867364305 |
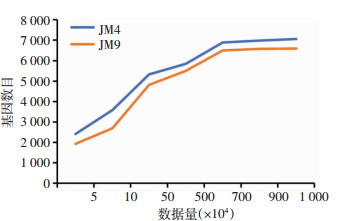
|
| (JM4为敏感菌株,JM9为耐药菌株) 图 1 转录组数据饱和图 |
2.3 差异表达基因筛选
根据相关生物信息学软件分析、比对,计算得到表达量信息和差异表达情况,其中,1 405个基因表达上调,2 112个基因表达下调。初步确定了73个和耐药相关的基因,其中38个为上调表达的基因。主要包括麦角固醇生物合成途径上的相关基因,如ERG11、ERG3等,其中氟康唑药物作用靶酶基因ERG11在耐药菌株中的表达量是敏感菌株的3.7倍(表 3);药物外排转运有关的基因,如ATP结合盒(ABC)、主要易化子超家族(MFS)转运蛋白基因等(表 4);以及抗氧化基因等,如硫氧还蛋白过氧化物酶(TPx)基因在耐药菌株中的表达量是敏感菌株的40倍。
| 基因编号 | 耐药均值 | 敏感均值 | 差异倍数 | 注释 | 基因 |
| A1Q1_00523 | 650.032 | 55.945 1 | 11.6 | C-8 sterol isomerase | ERG2 |
| A1Q1_00651 | 2 158.87 | 1 071.2 | 2.0 | C-4 methyl sterol oxidase | ERG25 |
| A1Q1_00690 | 430.775 | 505.762 | -1.2 | C-22 sterol desaturase | ERG5 |
| A1Q1_00880 | 199.086 | 78.789 | 2.5 | farnesyl-diphosphate farnesyltransferase | ERG9 |
| A1Q1_01564 | 50.593 | 56.022 7 | -1.1 | hydroxymethylglutaryl-CoA reductase (NADPH) | HMG |
| A1Q1_01672 | 1 670.04 | 778.04 | 2.1 | squalene monooxygenase | ERG1 |
| A1Q1_01819 | 292.278 | 64.035 3 | 4.6 | C-3 sterol dehydrogenase (C-4 sterol decarboxylase) | ERG26 |
| A1Q1_02098 | 1 217.17 | 329.852 | 3.7 | lanosterol 14-alpha-demethylase | ERG11 |
| A1Q1_03320 | 319.9 | 145.559 | 2.2 | C-5 sterol desaturase | ERG3 |
| A1Q1_05989 | 1 008.21 | 171.551 | 5.9 | acetyl-CoA C-acetyltransferase | ERG10 |
| A1Q1_06312 | 193.116 | 95.859 2 | 2.0 | C-14 sterol reductase | ERG24 |
| A1Q1_06727 | 1 067.68 | 182.765 | 5.8 | sterol 24-C-methyltransferase | ERG6 |
| A1Q1_07715 | 357.714 | 58.171 1 | 6.1 | diphosphomevalonate decarboxylase | ERG19 |
| A1Q1_07921 | 114.98 | 62.960 3 | 1.8 | lanosterol synthase | ERG7 |
| 基因编号 | 耐药/敏感 | 注释 |
| A1Q1_07193 | 2.9 | Homolog of MFS transporter. Multidrug resistance efflux pump. |
| A1Q1_07942 | 8.8 | ABC protein |
| A1Q1_04656 | 1.2 | efflux proteinEncT |
| A1Q1_07398 | 45.1 | Homolog of MFS transporter transporter, tetracycline efflux pump. |
| A1Q1_08012 | 2.3 | Homolog of MFS transporter transporter. Multidrug resistance efflux pump. |
| A1Q1_08156 | 19.3 | Homolog of MFS transportertransporter, tetracycline efflux pump. |
| A1Q1_08276 | 42.0 | Homolog of MFS transporter, ACS family, pantothenate transporter or FEN2 |
| A1Q1_00730 | 38.0 | aflatoxin efflux pump AFLT |
| A1Q1_04359 | 11.2 | MFS alpha-glucoside transporter |
| A1Q1_06164 | 1.8 | xenobiotic-transporting ATPase |
| A1Q1_01754 | 2.2 | ER togolgi family transport-related protein |
| A1Q1_00017 | 10.0 | phosphatidylinositol transporter |
| A1Q1_00031 | 3.2 | amino acid transporter |
| A1Q1_00867 | 9.6 | metal ion transporter |
| A1Q1_01225 | 2.1 | flavin-adenine dinucleotide transporter |
| A1Q1_01481 | 1.9 | inner membrane citrate transporter Ctp1p |
| A1Q1_01626 | 5.8 | phospholipid transporter |
| A1Q1_02511 | 2.8 | transporter |
| A1Q1_04142 | 1.4 | organic acid transporter |
| A1Q1_04426 | 3.2 | electron transporter transferring electrons within CoQH2-cytochrome creductase complex |
| A1Q1_05965 | 3.0 | dicarboxylic acid transporter |
| A1Q1_06363 | 2.2 | S-adenosylmethionine transporter |
| A1Q1_02271 | 1.8 | hexose transport-related protein |
| A1Q1_03522 | 2.3 | electron transporter |
| A1Q1_04374 | 15.4 | inorganic phosphate transporter |
| A1Q1_06083 | 38.0 | ammonium transporter |
2.4 ERG11全基因序列比较
为了明确DNA水平突变特别是ERG11基因是否与氟康唑耐药相关,对3株敏感菌株及其耐药菌株分别进行基因组测序,数据量均约3G,覆盖在100X以上,数据量足以满足后续基因组拼接和突变检测分析。通过拼接和比对分析,确定了6株菌ERG11基因的全基因序列,发现6株菌的ERG11基因在DNA水平上序列没有发生突变,3株敏感菌株与3株诱导耐药菌株的ERG11基因序列完全一致。
3 讨论不同于白念珠菌等真菌,目前有关T. asahii唑类药物耐药的分子机制研究较少,而大量的研究报道表明氟康唑(FLC)等三唑类药物的临床耐药现象及严重后果正日益凸显。BASU等[7]在3例败血症患者体内分离出两性霉素B(AMB)和FLC双重耐药的T. asahii分离株,这对患者来说是致命的。VASHISHTHA等[8]在8例新生儿败血症患者中均分离到了FLC耐药的T. asahii,其中6例在接受抗真菌治疗后死亡。根据流行病学界值(ECV),FLC的ECV值为8 μg/mL,T. asahii的MIC大于该值即定义为非野生型菌株,表示已经出现了耐药[3]。PADOVAN等[9]据此回顾分析了19项有关T. asahii对三唑类药物敏感性的研究,其中15项均至少发现了1株FLC耐药的T. asahii。根据GUO等[4]的研究数据分析,中国临床分离的T. asahii菌株有25%为FLC耐药菌株。而我们[10]也在国内首例播散性毛孢子菌病患者治疗15年后的面部皮损中分离到FLC耐药的T. asahii。而通过体外诱导获得单药耐药菌株是真菌耐药机制研究的常用方法,可与体内耐药菌研究互补。本研究中通过体外诱导获得的3株菌的MIC值均≥64 μg/mL,是理想的体外诱导耐药的研究模型。
麦角固醇是真菌细胞膜发挥多种细胞功能所不可或缺的成分,是由其前体物质羊毛固醇在14-α-脱甲基酶等一系列酶的催化下合成的。而氟康唑等唑类药物以羊毛固醇14-α-脱甲基酶为作用靶酶,通过抑制该酶的催化活性,阻碍麦角固醇的合成而达到抗真菌的作用。ERG11基因是羊毛固醇14-α-脱甲基酶的编码基因,该基因的突变以及上调表达均与唑类耐药有关[11]。GOŁABEK等[12]对120株白念珠菌的ERG11基因的表达变化进行了研究,发现60株耐药菌株的平均表达量显著高于60株敏感菌株的平均表达量。我们的研究也发现ERG11基因在耐药菌株中的表达量是敏感菌株的3.7倍,表明在T. asahii中ERG11基因的上调表达与FLC耐药机制相关。而ERG11基因的表达上调可能与其调控基因的突变有关,其中最主要的是编码锌簇转录调节因子的Upc2p基因。但我们在其上游启动子区序列中未发现Upc2p的同源基因。我们因此推测,ERG11基因的上调表达可能是T. asahii对氟康唑的一种应激反应,而非由基因表达调控改变所致。
除了表达量的变化,ERG11基因突变也是唑类耐药的主要原因之一。对白念珠菌ERG11基因的突变研究发现,ERG11基因突变大多数位于三个热点区域中,这些编码区域的突变,可以导致氨基酸的取代,进而使14-α-脱甲基酶活性区域的三维结构发生改变,导致唑类药物与酶的结合下降,引起耐药[13]。我们在体外诱导的3株FLC耐药菌株中均未发现ERG11基因突变,这与笔者之前在体内分离到的T. asahii氟康唑耐药菌株中发现多个突变位点的结果并不一致[10]。KUSHIMA等[14]从菌血症患者体内分离到6株对唑类(氟康唑、咪康唑、伊曲康唑、伏立康唑)具有交叉耐药的临床菌株,发现ERG11基因在所有分离株中均存在点突变(G448A),这一突变导致了甘氨酸替代,而这与他们之前在体外诱导耐药菌株中发现的一个突变位点并不同。他们认为,持续的唑类暴露诱导突变,从而产生对这类抗真菌药物的耐药。此外,在另外一项针对T. asahii临床标准株和环境株的氟康唑体外诱导耐药研究中也未在临床株中发现ERG11基因的突变,仅在环境株中发现了一个突变位点[15]。而在白念珠菌等其他真菌中也发现了仅有ERG11基因的表达上调,而无ERG11基因突变的情况[16]。因此,我们认为这种体内与体外、临床与环境、菌株与菌株之间的突变差异性,也正说明了真菌耐药机制的复杂性以及我们目前对耐药机制研究所知的局限性。
唑类耐药性的产生除了与ERG11基因改变有关外,通常还与药物外排泵的激活以及抗氧化应激等有关。目前已知的外排泵蛋白家族基因主要包括两大类:ATP结合盒(ABC)转运蛋白基因和主要易化子超家族(MFS)转运蛋白基因。我们的研究也同样发现了在FLC耐药的T. asahii菌株中ABC、MFS等外排泵基因的表达上调,而且与念珠菌的FLC耐药菌株中外排泵基因的高表达有着极高的相似性[17]。此外,除了外排泵基因,抗氧化基因也参与了耐药机制的形成,表现为硫氧还蛋白过氧化物酶(TPx)基因表达上调了40倍。另一研究也发现氧化还原过程和跨膜运输相关的基因在T. asahii的FLC暴露时发生了变化[15]。JIA等[18]研究发现氟康唑可以诱导对唑类和两性霉素B交叉耐药的热带念珠菌产生活性氧(ROS)。而ROS的产生超过抗氧化酶系统的清除能力时,可引起蛋白质、脂类等生物大分子的损伤,白念珠菌等通过表达超氧化物歧化酶和过氧化氢酶,以及谷氨酰胺多辛和硫氧还蛋白等来降低ROS的损伤,导致药物的敏感性下降。我们研究发现的TPx是细胞内重要的氧化还原调节分子之一,可以帮助真菌细胞清除ROS,从而减少ROS的聚积,降低细胞的损伤。这也说明抵抗氧化应激的抗氧化反应可能也是引起T. asahii耐药的机制之一,这对于未来寻找新的抗真菌靶点及新药开发研究都有着重要的意义和启发。但真菌通过抗氧化应激的防御反应来产生对唑类药物耐药的机制并不完全明确,目前仍有争议,如MARTINS等[19]发现过氧化氢酶活性的抑制反而增加了啤酒酵母对唑类药物的耐药性,而不是我们所理解的当抗氧化防御被削弱时,真菌的耐药性降低。
总之,我们的研究虽然表明ERG11基因、外排转运相关基因及抗氧化基因等表达上调均可能与T. asahii氟康唑耐药机制有关,但对于其耐药机制我们了解的仍太少,特别是对于抗氧化机制的研究。
| [1] |
MILAN E P, SILVA-ROCHA W P, DE ALMEIDA J J S, et al. Trichosporon inkin meningitis in Northeast Brazil: first case report and review of the literature[J]. BMC Infect Dis, 2018, 18(1): 470. DOI:10.1186/s12879-018-3363-7 |
| [2] |
DE ALMEIDA JÚNIOR J N, HENNEQUIN C. Invasive Trichosporon infection: a systematic review on a Re-emerging fungal pathogen[J]. Front Microbiol, 2016, 7: 1629. DOI:10.3389/fmicb.2016.01629 |
| [3] |
FRANCISCO E C, DE ALMEIDA JUNIOR J N, DE QUEIROZ TELLES F, et al. Species distribution and antifungal susceptibility of 358 Trichosporon clinical isolates collected in 24 medical centres[J]. Clin Microbiol Infect, 2019, 25(7): 909. DOI:10.1016/j.cmi.2019.03.026 |
| [4] |
GUO L N, YU S Y, HSUEH P R, et al. Invasive infections due to Trichosporon: species distribution, genotyping, and antifungal susceptibilities from a multicenter study in China[J]. J Clin Microbiol, 2019, 57(2): e01505-e01518. DOI:10.1128/JCM.01505-18 |
| [5] |
National Committee for Clinical Laboratory Standards. Reference method for broth dilution antifungal susceptibility testing of yeasts[M]. Nattional Committee for Clinical Laboratory Standards, Wayne, PA; 2002.
|
| [6] |
KUSHIMA H, TOKIMATSU I, ISHⅡ H, et al. Cloning of thelanosterol 14-α-demethylase (ERG11) gene in Trichosporon asahii: a possible association between G453R amino acid substitution and azole resistance in T. asahii[J]. FEMS Yeast Res, 2012, 12(6): 662-667. DOI:10.1111/j.1567-1364.2012.00816.x |
| [7] |
BASU S, TILAK R, KUMAR A. Multidrug-resistant Trichosporon: an unusual fungal Sepsis in preterm neonates[J]. Pathog Glob Health, 2015, 109(4): 202-206. DOI:10.1179/2047773215Y.0000000019 |
| [8] |
VASHISHTHA V M, MITTAL A, GARG A. A fatal outbreak of Trichosporon asahii sepsis in a neonatal intensive care unit[J]. Indian Pediatr, 2012, 49(9): 745-747. DOI:10.1007/s13312-012-0137-y |
| [9] |
PADOVAN A C B, ROCHA W P D S, TOTI A C M, et al. Exploring the resistance mechanisms in Trichosporon asahii: Triazoles as the last defense for invasive trichosporonosis[J]. Fungal Genet Biol, 2019, 133: 103267. DOI:10.1016/j.fgb.2019.103267 |
| [10] |
XIA Z K, YU H Y, WANG C M, et al. Genomic andtranscriptome identification of fluconazole-resistant genes for Trichosporon asahii[J]. Med Mycol, 2020, 58(3): 393-400. DOI:10.1093/mmy/myz088 |
| [11] |
FLOWERS S A, COLÓN B, WHALEY S G, et al. Contribution of clinically derived mutations in ERG11 to azole resistance in Candida albicans[J]. Antimicrob Agents Chemother, 2015, 59(1): 450-460. DOI:10.1128/AAC.03470-14 |
| [12] |
GOŁABEK K, STRZELCZYK J K, OWCZAREK A, et al. Selected mechanisms of molecular resistance of Candida albicans to azole drugs[J]. Acta Biochim Pol, 2015, 62(2): 247-251. DOI:10.18388/abp.2014_940 |
| [13] |
FENG L J, WAN Z, WANG X H, et al. Relationship between antifungal resistance of fluconazole resistant Candida albicans and mutations in ERG11 gene[J]. Chin Med J, 2010, 123(5): 544-548. |
| [14] |
KUSHIMA H, TOKIMATSU I, ISHⅡ H, et al. A new amino acid substitution at G150S inlanosterol 14-α demethylase (Erg11 protein) in multi-azole-resistant Trichosporon asahii[J]. Med Mycol J, 2017, 58(1): E23-E28. DOI:10.3314/mmj.16-00027 |
| [15] |
LI H T, WANG C M, CHEN Y, et al. Integratedtranscriptomic analysis of Trichosporon Asahii uncovers the core genes and pathways of fluconazole resistance[J]. Sci Rep, 2017, 7(1): 17847. DOI:10.1038/s41598-017-18072-9 |
| [16] |
ALIZADEH F, KHODAVANDI A, ZALAKIAN S. Quantitation ofergosterol content and gene expression profile of ERG11 gene in fluconazole-resistant Candida albicans[J]. Curr Med Mycol, 2017, 3(1): 13-19. DOI:10.29252/cmm.3.1.13 |
| [17] |
SOUZA A C, FUCHS B B, PINHATI H M, et al. Candida parapsilosis resistance to fluconazole: molecular mechanisms and in vivo impact in infected Galleria mellonella larvae[J]. Antimicrob Agents Chemother, 2015, 59(10): 6581-6587. DOI:10.1128/AAC.01177-15 |
| [18] |
JIA C, ZHANG J, ZHUGE Y Z, et al. Synergistic effects of geldanamycin with fluconazole are associated with reactive oxygen species in Candida tropicalis resistant to azoles and amphotericin B[J]. Free Radic Res, 2019, 53(6): 618-628. DOI:10.1080/10715762.2019.1610563 |
| [19] |
MARTINS D, NGUYEN D, ENGLISH A M. Ctt1 catalase activity potentiates antifungal azoles in the emerging opportunistic pathogen Saccharomyces cerevisiae[J]. Sci Rep, 2019, 9(1): 9185. DOI:10.1038/s41598-019-45070-w |



