何首乌为蓼科植物何首乌(Polygonum multiflorum thunb)的块根,是一味养生抗衰老中药[1],具有抗氧化[2]、细胞保护特性[3]、乌发[4]、治疗与衰老相关的神经退行性疾病[5]以及改善与衰老有关的疾病等作用。何首乌可通过调节和增强机体非特异性及特异性免疫功能,消除衰老动物体内自由基达到抗衰老作用[6],但其具体机制还未明了,尤其是对衰老细胞表观遗传学、基因的表达以及一些物质代谢的作用靶点还不清楚。
在衰老过程中,表观遗传学变化包括甲基化(如组蛋白H4K20、H3K4高度甲基化,H3K9、H3K27低度甲基化)、H4K16乙酰化以及异染色质蛋白1-α(HP1α)减少等[7]。而DNA甲基化是调节基因表达的关键表观遗传修饰之一,可作为衰老的标志,但迄今为止尚无证据表明特定DNA甲基化模式的改变可以延长寿命[7-8]。脑老化作为衰老的主要特征之一,表现为广泛的行为缺陷,包括焦虑和认知功能受损,并可影响全身的老化[9]。相对于其他组织,大脑显示出较高水平的甲基化,特别是在基因组的重复序列。DNA甲基化、组蛋白修饰、染色质构象改变等表观遗传学修饰均可调控基因表达并参与衰老及许多疾病的发生、发展。
研究表明,在阿尔茨海默病的早期,Synpo作为神经元间的功能性突触蛋白减少[10];Mapkapk5在细胞生长、营养饥饿反应、程序性细胞死亡、衰老和运动过程中扮演着多种角色[11],且有证据证明Mapkapk5可以磷酸化多种底物,包括Foxo1、Foxoa3和Rheb,但Mapkapk5的生物学作用远未完全了解[12-14];作为T细胞特异性转录因子的Tcf7及RAS家族中的Rab15参与了机体的免疫调节过程[15-16]。在衰老过程相关研究中,这些基因的作用尚不明确。因此,本研究应用简化甲基化测序(reduced representation bisulfite sequencing,RRBS)和RNA-seq测序方法,观察何首乌对D-半乳糖致衰老C57BL/6小鼠脑组织DNA甲基化和转录的影响,探讨何首乌延缓衰老的分子机制。
1 材料与方法 1.1 试剂何首乌:用75%乙醇提取,减压回收乙醇,减压浓缩至100 g生药/100 mL(遵义医科大学附属医院中药房,批号:160801);D-半乳糖购自美国Sigma公司;SOD、MDA和GSH-Px检测试剂盒购自中国南京建成生物工程研究所;RNAiso Plus、PrimeScriptTMRT试剂盒及TB GreenTM Premix Ex TaqTMⅡ试剂盒均购自日本TaKaRa公司。
1.2 动物分组及给药C57BL/6雄性小鼠购自第三军医大学医学实验动物中心,许可证号[SCXK(辽)015-0001],体质量(20±2)g,饲养于普通环境,自由饮食,适应性喂养1周。按处理方式不同将50只小鼠分为5组:正常对照组(CC)、衰老组(MC)、何首乌提取液高剂量组(MH)、中剂量组(MM)以及低剂量组(ML)。除CC组外,其余4组用D-半乳糖(800 mg· mL-1· kg-1)连续腹腔注射60 d以建立亚急性衰老模型,而CC组腹腔注射等体积生理盐水;衰老模型建立好后,参照LEE等[17]和吕锦芳等[18]的研究及前期药物浓度实验摸索确定用药浓度,MH组、MM组和ML组分别按1、0.6、0.3 g·mL-1·kg-1 PME每天灌胃,CC组和MC组灌胃等量生理盐水,连续灌胃60 d。造模结束,小鼠腹腔注射4%水合氯醛(每100 g体质量1 mL)。待小鼠麻醉后,摘除眼球取血,在4 ℃下3 500 r/min离心10 min,取上清于-20 ℃待用;断头处死小鼠,取出脑组织,并标记组别及日期,于-80 ℃冰箱保存。
1.3 血清样本测定取每组离心所得血清样本,根据试剂盒说明,配置检测GSH-Px活性、SOD活性及MDA含量所需的试剂溶液,通过化学比色分析并计算酶活性和MDA含量。
1.4 脑组织DNA的提取及测序根据建模结果,选取CC组、MC组及MM组小鼠脑组织进行甲基化测序,每组选取3只小鼠的脑组织,按TaKaRa MiniBEST Universal Genomic DNA Extraction Kit操作说明提取脑组织基因组DNA,用微量核酸蛋白分析仪对DNA进行定量检测。合格的DNA样本送至深圳华大基因科技服务有限公司进行建库及RRBS测序。流程如下:将质量检测合格的基因组DNA片段用限制性内切酶MspⅠ进行处理,对酶切后的DNA片段进行末端修复、3′端加A碱基,并连接甲基化接头,切胶选择40~200 bp的酶切片段,采用ZYMO EZ DNA Methylation-Gold kit进行亚硫酸盐(Bisulfite)处理;聚合酶链式反应(PCR)扩增文库,合格的文库通过高通量测序平台Illumina Hiseq X-ten进行上机测序得到数据。
1.5 脑组织RNA的提取及测序根据建模结果,选取CC组、MC组及MM组小鼠脑组织进行转录组测序,每组选取5只冻存的小鼠脑组织,称取30 mg,按照RNAiso Plus操作步骤提取RNA,用微量核酸蛋白分析仪检测D(260)/D(280),结果在1.8~2.0之间,最后用1%琼脂糖凝胶电泳检测RNA质量,均符合转录组测序要求。将合格的总RNA送至深圳华大基因科技服务有限公司建立DNA文库,且合格的文库通过高通量测序平台BGISEQ-500进行上机测序得到转录组测序数据。
1.6 差异基因启动子区DMR相关基因功能聚类分析筛选出启动子区甲基化发生差异变化的基因,并用GO功能聚类及KEGG通路富集分析。
1.7 基因表达分析用RNAiso Plus试剂盒提取脑组织的总RNA,核酸蛋白仪测总RNA的浓度及纯度。将1 μg RNA用逆转录试剂盒Pri-meScriptTMRT逆转为cDNA,用TB GreenTM Premix Ex TaqTMⅡ试剂盒进行荧光定量PCR,总反应体系为20 μL,反应程序为95 ℃,30 s;GOTO(95 ℃,5 s;60 ℃,30 s);95 ℃,10 s;60 ℃,5 s;95 ℃,50 s;40个循环。以GAPDH为内参,通过2-ΔΔCt法计算目的基因的相对表达量。目的基因的引物设计和合成均由生工生物(上海)有限公司完成,引物序列见表 1。
| 基因 | 引物序列(5′→ 3′) | 产物长度/bp |
| Gapdh | 上游:TGTGTCCGTCGTGGATCTGA 下游:CCTGCTTCACCACCTTCTTGA |
161 |
| Tcf7 | 上游:GCAGCATCCGCAGCCTCAAC 下游:GCCTGTGAACTCCTTGCTTCTGG |
152 |
| Rab15 | 上游:AGGAGTACGGCATGGACTTCTACG 下游:CTCCTCCAGCTCGGCCAGTG |
164 |
| Synpo | 上游:GTGGCACCAGCAGAACAAGAGG 下游:GCAGGCTTGGCTAGTGTGAGATG |
95 |
| Mapkapk5 | 上游:TGATGATGGATAAGGCGGTGGTTG 下游:GCAGCTTCCTCTTCCTGAGAATGG |
141 |
1.8 统计学分析
采用SPSS 25.0统计软件进行分析,计量资料以x±s表示,两组间数据比较采用t检验,多组间数据比较采用单因素方差分析。检验水准:α=0.05。
2 结果 2.1 PME对衰老小鼠血清抗氧化指标的影响通过对血清中氧化酶的检测,衰老组(MC)SOD、GSH-Px的酶活性较正常对照组(CC)显著降低(P < 0.05),MDA的含量较CC组显著升高(P < 0.05);灌胃PME后,何首乌提取液中、高剂量组的SOD、GSH-Px酶活性较MC组显著升高(P < 0.05),中、高剂量组的MDA含量较MC组显著降低(P < 0.05,表 2),且中剂量组(MM)作用效果相对明显。因此,选取正常对照组、衰老组及何首乌提取液中剂量组进行甲基化和转录组测序以及后续实验。
| 组别 | SOD/U·mL-1 | MDA/nmol·mL-1 | GSH-Px/U·mL-1 |
| 正常对照组 | 85.68±4.79 | 1.947±0.765 | 431.77±24.94 |
| 衰老组 | 71.95±3.12a | 4.132±0.926a | 384.30±31.46a |
| 何首乌提取液 | |||
| 高剂量组 | 79.20±4.14b | 2.358±0.722b | 436.33±33.66b |
| 中剂量组 | 82.37±3.22b | 2.252±1.350b | 424.94±19.40b |
| 低剂量组 | 68.09±7.70 | 3.351±1.032 | 414.68±53.79 |
| a:P < 0.05,与正常对照组比较;b:P < 0.05,与衰老组比较 | |||
2.2 RRBS测序的质控及PME作用后脑组织基因组整体甲基化水平的变化
DNA甲基化测序数据显示,经过去污染、去测序接头和去低质量碱基比例过高的reads后,在Bisulfite处理时,非甲基化胞嘧啶的转化效率均在99%以上,捕获效率均在83%以上,即比对到目标区域的base数占所有比对上参考基因组的base总数的比例在83%以上。对3组样本的启动子区和CpG岛甲基化C碱基中CG、CHG与CHH分布比例进行分析,正常对照组(CC)、衰老组(MC)和何首乌提取液中剂量组(MM)中甲基化C碱基以CG类型为主,在启动子区域和CpG岛CG碱基分别占全部甲基化C碱基类型(CG、CHG、CHH)的85%和95%以上。对所有甲基化C碱基的平均甲基化水平进行分析,MC组基因启动子区域的所有甲基化C碱基及甲基化CG类型的平均甲基化水平均显著高于CC组和MM组(P < 0.05);3组CpG岛的所有甲基化C碱基的平均甲基化水平差异无统计学意义(P>0.05,图 1)。
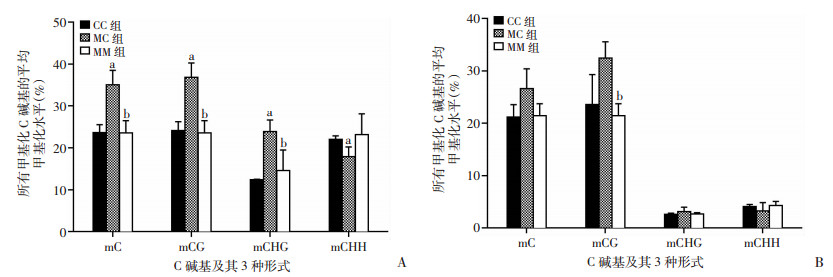
|
| A:启动子区;B:CpG岛 a:P < 0.05,与CC组比较;b:P < 0.05,与MC组比较 图 1 PME作用于衰老小鼠后脑组织基因组平均甲基化水平分析 |
2.3 RNA-seq测序数据的质控报告
转录组测序结果显示,去除原始数据中的低质量、接头污染以及未知碱基N含量过高的reads,得到高质量reads,测得每个样品平均产出21.53M数据。
2.4 PME作用后脑组织基因组DMR及DMR位于启动子区的相关基因分析和鉴定进一步分析启动子区域有5个以上CpG覆盖的基因甲基化情况,最终获得启动子区C位点的平均甲基化水平和基因区C位点的甲基化水平。用CG甲基化水平的差异来寻找DMR,从而确定在每条染色体上有差异的区域长度和有差异的基因。与正常对照组比较,衰老组有164个DMR,平均长度为15 750 bp;与衰老组比较,MM组有140个DMR,平均长度为13 672 bp。根据启动子区域发生甲基化差异的基因,正常对照组与衰老组相比有178个,而衰老组与MM组相比有154个,在3组中有52个DMR位于启动子区的相关基因,包括与衰老密切相关的4个基因:Rab15、Tcf7、Synpo和Mapkapk5。
2.5 DNA甲基化与基因表达之间的重叠变化为了找到何首乌延缓衰老作用的分子靶点,在CC组与MC组及MC组与MM组之间,对启动子区域发生甲基化差异及相应发生基因表达差异的基因进行了分析,发现27个基因在两者之间均发生了变化,其中Cmip、Deaf1、Arhgef2、Tmem2、Rab15、Tcf7和Erbb3与基因表达呈现负性相关, Igf2r、Adgrl4、Slc15a1、Dnase1l2、Smad3、Synpo和Mapkapk5等13个基因与其表达呈正相关,结果见表 3。
| 基因名 | 差异甲基化 | 基因表达 | ||||||
| Log2差异甲基化倍数 (CC/MC) |
Log2差异甲基化倍数 (MC/MM) |
P值 | Log2差异表达倍数 (CC/MC) |
Log2差异表达倍数 (MC/MM) |
校正P值 (CC/MC) |
校正P值 (MC/MM) |
||
| Cmip | 6.161 | -6.118 | < 0.05 | -0.382 | 0.137 | < 0.05 | < 0.05 | |
| Deaf1 | 1.216 | 0.521 | < 0.05 | -0.226 | -0.120 | < 0.05 | < 0.05 | |
| Rab15 | 1.049 | -0.895 | < 0.05 | -0.158 | 0.320 | < 0.05 | < 0.05 | |
| Tcf7 | 1.049 | -0.646 | < 0.05 | -0.231 | 0.316 | < 0.05 | < 0.05 | |
| Arhgef2 | 0.214 | -0.191 | < 0.05 | -0.100 | 0.272 | < 0.05 | < 0.05 | |
| Tmem2 | -0.326 | 0.327 | < 0.05 | 0.133 | -0.319 | < 0.05 | < 0.05 | |
| Erbb3 | -1.110 | 1.191 | < 0.05 | 0.148 | -0.325 | < 0.05 | < 0.05 | |
| Igf2r | 1.390 | -0.836 | < 0.05 | 0.152 | -0.092 | < 0.05 | < 0.05 | |
| Adgrl4 | 0.822 | -0.631 | < 0.05 | 0.478 | -0.356 | < 0.05 | < 0.05 | |
| Slc15a1 | -0.203 | 0.252 | < 0.05 | -1.080 | 0.459 | < 0.05 | < 0.05 | |
| Dnase1l2 | -0.357 | 0.294 | < 0.05 | -1.457 | 1.086 | < 0.05 | < 0.05 | |
| Smad3 | -0.454 | 0.301 | < 0.05 | -0.195 | 0.204 | < 0.05 | < 0.05 | |
| Nnat | -0.223 | 0.329 | < 0.05 | -1.014 | 0.160 | < 0.05 | < 0.05 | |
| Catsperg1 | -0.259 | 0.330 | < 0.05 | -0.821 | 0.950 | < 0.05 | < 0.05 | |
| Synpo | -0.654 | 0.922 | < 0.05 | -0.572 | 0.366 | < 0.05 | < 0.05 | |
| Mapkapk5 | -1.570 | 1.394 | < 0.05 | -0.251 | 0.289 | < 0.05 | < 0.05 | |
| 9-Sep | -1.140 | 1.423 | < 0.05 | -0.335 | 0.076 | < 0.05 | < 0.05 | |
| Nfatc2 | -0.388 | 1.494 | < 0.05 | -0.342 | 0.547 | < 0.05 | < 0.05 | |
| P3h3 | -0.504 | 1.514 | < 0.05 | -0.801 | 0.984 | < 0.05 | < 0.05 | |
| Nub1 | -2.226 | 2.198 | < 0.05 | -0.181 | 0.129 | < 0.05 | < 0.05 | |
2.6 基因启动子区DMR相关基因功能注释、聚类及通路分析
为了评估启动子区发生甲基化变化的差异甲基化基因是否富集于特定的生物过程或功能,用GO分析对启动子DMR相关的基因进行功能分类。CC组与MC组之间以及MC组与MM组之间,启动子DMR相关的基因都富含了24种细胞成分类别,注释基因也都参与了14个分子功能和9个生物过程(图 2)。在生物过程中,CC组与MC组和MC组与MM组出现了不同组分,在CC组与MC组比较中,有排毒和参与化学突触传递的突触前过程;MC组与MM组比较中,除细胞杀伤和色素沉着功能,其余组分均相同。就CC组与MC组之间以及MC组与MM组之间,对启动子区域发生甲基化差异基因进行KEGG富集通路分析,参与多条生物通路, 如肿瘤通路、急性髓性白血病、黏着力和B细胞受体信号通路等,见表 4。其中Mapkapk5和Tcf7分别参与了与衰老密切相关的MAPK信号通路和Wnt信号通路。
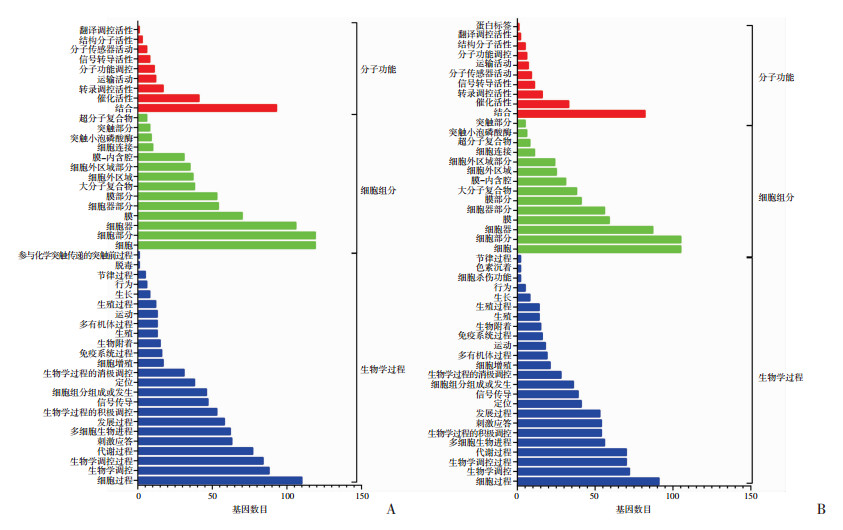
|
| A:正常对照组(CC)与衰老组(MC)的启动子区DMR相关基因的GO功能分类图; B:衰老组(MC)与何首乌提取液中剂量组(MM)的启动子区DMR相关基因的GO功能分类图 图 2 PME作用于衰老小鼠后脑组织基因启动子区DMR相关基因GO分析 |
| 通路ID | 通路名称 | P值 |
| 正常对照组与衰老组比较 | ||
| ko00514 | 其他类型的O-聚糖生物合成 | 0.00 271 593 |
| ko05221 | 急性髓细胞白血病 | 0.02 175 338 |
| ko04672 | 肠免疫网络产生IgA | 0.04 122 130 |
| ko04512 | ECM-受体相互作用 | 0.04 125 208 |
| ko04550 | 调节干细胞多能性的信号通路 | 0.04 857 240 |
| 衰老组与何首乌提取液中剂量组比较 | ||
| ko05200 | 癌症通路 | 0.00 006 475 |
| ko05221 | 急性髓细胞白血病 | 0.00 017 332 |
| ko04510 | 粘着斑 | 0.00 170 871 |
| ko04662 | B细胞受体信号通路 | 0.00 217 413 |
| ko04012 | ErbB信号通路 | 0.00 428 466 |
| ko05161 | 乙型肝炎 | 0.00 576 464 |
| ko04550 | 调节干细胞多能性的信号通路 | 0.00 703 979 |
| ko05321 | 炎性肠病 | 0.00 721 948 |
| ko04024 | cAMP信号通路 | 0.00 770 444 |
| ko05416 | 病毒性心肌炎 | 0.00 857 148 |
| ko04921 | 催产素信号通路 | 0.00 958 345 |
| ko05202 | 癌症中的转录失调 | 0.01 001 671 |
| ko04520 | 黏着连接 | 0.01 220 692 |
| ko04664 | FcεRI信号通路 | 0.01 292 794 |
| ko04370 | VEGF信号通路 | 0.01 383 705 |
| ko04650 | 自然杀伤细胞介导的细胞毒性 | 0.01 742 397 |
| ko04950 | 青年成熟期发病型糖尿病 | 0.01 806 356 |
| ko05130 | 致病性大肠杆菌感染 | 0.01 891 861 |
| ko04974 | 蛋白质消化和吸收 | 0.01 900 314 |
| ko05212 | 胰腺癌 | 0.02 426 306 |
| ko04010 | MAPK信号通路 | 0.02 453 871 |
| ko05216 | 甲状腺癌 | 0.02 490 214 |
| ko05210 | 结直肠癌 | 0.02 490 214 |
| ko04670 | 白细胞经内皮迁移 | 0.02 565 680 |
| ko05220 | 慢性髓细胞白血病 | 0.02 687 362 |
| ko05412 | 致心律失常性右室心肌病 | 0.02 962 858 |
| ko04672 | 肠免疫网络产生IgA | 0.03 226 757 |
| ko05142 | 美洲锥虫病 | 0.03 327 437 |
| ko04310 | Wnt信号通路 | 0.03 368 032 |
| ko05205 | 癌症中的蛋白多糖 | 0.03 709 201 |
| ko05032 | 吗啡成瘾 | 0.03 714 303 |
| ko04066 | HIF-1信号通路 | 0.03 714 303 |
| ko0492 | 醛固酮的合成和分泌 | 0.03 794 331 |
| ko04660 | T细胞受体信号通路 | 0.03 957 026 |
| ko05215 | 前列腺癌 | 0.04 039 688 |
| ko05206 | 癌症中的微小RNA | 0.04 116 418 |
| ko05131 | 志贺氏菌病 | 0.04 292 905 |
| ko04810 | 肌动蛋白细胞骨架的调控 | 0.04 910 963 |
| ko04015 | Rap1信号通路 | 0.04 965 487 |
2.7 实时荧光定量PCR结果
结果显示,经PME作用后,衰老小鼠脑组织中Synpo、Mapkapk5、Tcf7及Rab15的表达均上调,差异有统计学意义(P < 0.05,图 3),且与RNA-seq测序结果一致。
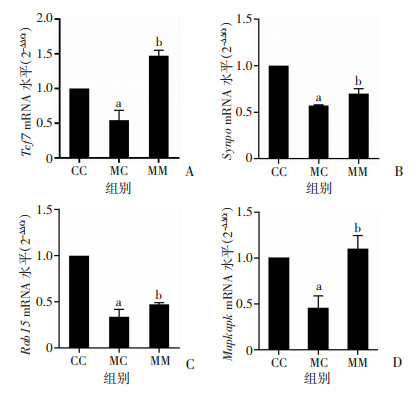
|
| A:Tcf7;B:Synpo;C:Rab15;D:Mapkapk5 a:P < 0.01, 与CC组比较;b:P < 0.05,与MC组比较 图 3 PME对衰老小鼠脑组织中4个基因mRNA表达的影响 |
3 讨论
衰老是一个极其复杂的过程,包括机体的各方面机能下降以及对环境的适应性降低等,如机体的免疫能力下降、神经退行性病变、抗氧化力减弱及与衰老相关的脏器病变等。机体可通过调节免疫能力、抑制神经变性和抗氧化等达到抗衰老目的。何首乌作为抗衰老药物,可以抗炎、抗氧化[19],预防和治疗衰老相关的骨质流失[20]和减轻神经退行性疾病,如阿尔茨海默症和帕金森病[21]等,但具体抗衰老分子途径不明了。为探寻何首乌延缓衰老的作用机制,本研究将何首乌作用后的衰老小鼠脑组织进行RRBS和RNA-seq测序。
在衰老机制研究中,常采用D-半乳糖建立小鼠亚急性衰老模型模拟人体衰老[22]。此模型建立机制是过量D-半乳糖在机体不能被代谢,会在细胞内聚集产生大量ROS,使机体氧化损伤[23],致使机体逐渐衰老。常用MDA、SOD和GSH-Px等指标验证衰老模型建立是否成功,本实验结果与以往实验结果一致[24],用D-半乳糖建立的衰老小鼠模型,血清中MDA含量升高,SOD和GSH-Px酶活性降低。经PME作用于衰老小鼠后,血清中抗氧化酶活性明显提高,MDA含量下降,尤以中剂量组效果相对显著。这些结果表明,本实验衰老模型建立成功,且何首乌具有延缓衰老作用。
小鼠脑组织DNA甲基化及转录组测序结果显示,3组启动子区超过85%的CG位点都被甲基化。D-半乳糖致小鼠衰老前后以及经PME作用前后,小鼠脑组织中基因启动子区甲基化水平及转录水平均发生差异变化的有20个。由于启动子相关的CpG岛甲基化水平通常与基因表达呈负相关,但某些基因也会出现相反结果[25],故对启动子甲基化变化与转录水平变化相同或相反基因均进行研究。结果显示,启动子甲基化与转录水平变化趋势相同的13个基因中的Synpo和Mapkapk5基因,以及变化趋势相反的7个基因中的Tcf7和Rab15基因,均可能与何首乌延缓衰老作用相关。
最新研究表明,在神经元中Bag3与Synpo相互协同调节自噬[26],Synpo在阿尔茨海默症患者中的表达显著下调[27],突触功能障碍与自噬的调节密切相关,可以通过调节自噬功能来治疗与年龄相关的神经系统疾病[28]。本研究在D-半乳糖致衰老的小鼠脑组织中发现Synpo低甲基化,基因表达下调,经PME作用后甲基化水平和基因表达水平均升高。由此,我们推测何首乌可能通过调节Synpo基因表达来调控自噬系统以此达到延缓衰老的目的。
Mapkapk5亦称PRAK,属于p38 MAPK信号转导途径的关键成分,p38调节/活化激酶(PRAK)被细胞应激激活。但是,PRAK及其下游交互作用蛋白的功能尚未明确。文献[29]报道PRAK与DJ-1相互作用可以防止氧化应激导致的细胞死亡。本研究发现,衰老小鼠经PME作用后,脑组织中Mapkapk5甲基化水平和转录水平均提高,结果提示:何首乌发挥延缓衰老作用,可能是通过调节Mapkapk5基因,防止因氧化应激导致的细胞死亡。
Tcf7作为调节T细胞分化的关键因子之一,在衰老过程中其表达量降低与DNA高甲基化相关[30],即在抗衰老过程中Tcf7表达量增加与DNA低甲基化相关。此外,Tcf7与衰老有关的骨质疏松密切相关,与视黄觉受体相关的Rorβ缺失可通过增加Tcf7的表达和Wnt信号反应来增强骨形成,从而防止骨质疏松[31]。衰老可降低人体的整体免疫能力,尽管所有免疫细胞受到影响,但T细胞所受的影响最大[32],且衰老的免疫系统失去抵御感染的能力[33]。树突状细胞(DC)对于病毒感染会发起保护性T细胞免疫,而Rab15可赋予CD亚群中的CD141+DC抗感染能力,当Rab15表达水平提高可提高抗感染能力[34]。本研究发现,经PME作用后衰老小鼠脑组织中Tcf7和Rab15两者低甲基化,基因表达上调,且通过分析DMR位于启动子区基因的富集通路发现,Tcf7参与了Wnt信号通路。表明何首乌可能是通过调控Tcf7和Rab15的表观遗传来影响其表达,从而提高机体免疫力以达到延缓衰老作用,且通过调节Tcf7的表达来防止衰老相关的骨质疏松。
综上,本研究利用DNA甲基化及转录组测序技术,分析比较启动子甲基化水平及转录水平均发生变化的基因,初步确定何首乌可能通过调节Synpo、Mapkapk5、Tcf7和Rab15的启动子区域甲基化变化来影响其表达。推测何首乌可能是通过调节自噬系统以防止神经退行性病变、抗氧化及调控或增强机体免疫功能等,影响基因的表观遗传进而调节其基因的表达,达到延缓衰老的作用。为后续何首乌延缓衰老的具体机制研究提供了方向。
| [1] |
CHEN T, YANG Y J, LI Y K, et al. Chronic administration tetrahydroxystilbene glucoside promotes hippocampal memory and synaptic plasticity and activates ERKs, CaMKⅡ and SIRT1/miR-134 in vivo[J]. J Ethnopharmacol, 2016, 190: 74-82. DOI:10.1016/j.jep.2016.06.012 |
| [2] |
LV L, GU X H, TANG J, et al. Antioxidant activity of stilbene glycoside from Polygonum multiflorum Thunb in vivo[J]. Food Chem, 2007, 104(4): 1678-1681. DOI:10.1016/j.foodchem.2007.03.022 |
| [3] |
STEELE M L, TRUONG J, GOVINDARAGHAVAN S, et al. Cytoprotective properties of traditional Chinese medicinal herbal extracts in hydrogen peroxide challenged human U373 astroglia cells[J]. Neurochem Int, 2013, 62(5): 522-529. DOI:10.1016/j.neuint.2012.08.018 |
| [4] |
HAN M N, LU J M, ZHANG G Y, et al. Mechanistic studies on the use of Polygonum multiflorum for the treatment of hair graying[J]. Biomed Res Int, 2015, 2015(2): 647-655. DOI:10.1155/2015/651048 |
| [5] |
CHAN Y C, WANG M F, CHANG H C. Polygonum multiflorum extracts improve cognitive performance in senescence accelerated mice[J]. Am J Chin Med, 2003, 31(2): 171-179. DOI:10.1142/S0192415X03000862 |
| [6] |
LEE S Y, AHN S M, WANG Z Y, et al. Neuroprotective effects of 2, 3, 5, 4'-tetrahydoxystilbene-2-O-β-D-glucoside from Polygonum multiflorum against glutamate-induced oxidative toxicity in HT22 cells[J]. J Ethnopharmacol, 2017, 195: 64-70. DOI:10.1016/j.jep.2016.12.001 |
| [7] |
LÓPEZ-OTÍN C, BLASCO M A, PARTRIDGE L, et al. The hallmarks of aging[J]. Cell, 2013, 153(6): 1194-1217. DOI:10.1016/j.cell.2013.05.039 |
| [8] |
JUNG M, PFEIFER G P. Aging and DNA methylation[J]. BMC Biol, 2015, 13(1): 7-14. DOI:10.1186/s12915-015-0118-4 |
| [9] |
SADIGH-ETEGHAD S, MAJDI A, MCCANN S K, et al. D-galactose-induced brain ageing model: A systematic review and meta-analysis on cognitive outcomes and oxidative stress indices[J]. PLoS ONE, 2017, 12(8): e0184122. DOI:10.1371/journal.pone.0184122 |
| [10] |
GOETZL E J, KAPOGIANNIS D, SCHWARTZ J B, et al. Decreased synaptic proteins in neuronal exosomes of frontotemporal dementia and Alzheimer's disease[J]. FASEB J, 2016, 30(12): 4141-4148. DOI:10.1096/fj.201600816R |
| [11] |
DWYER S F, GELMAN I H. Cross-phosphorylation and interaction between src/FAK and MAPKAP5/PRAK in early focal adhesions controls cell motility[J]. J Cancer Biol Res, 2014, 2(1): 1045. |
| [12] |
CHOW K T, TIMBLIN G A, MCWHIRTER S M, et al. MK5 activates Rag transcription via Foxo1 in developing B cells[J]. J Exp Med, 2013, 210(8): 1621-1634. DOI:10.1084/jem.20130498 |
| [13] |
KOSTENKO S. Physiological roles of mitogen-activated-protein-kinase-activated p38-regulated/activated protein kinase[J]. World J Biol Chem, 2011, 2(5): 73-89. DOI:10.4331/wjbc.v2.i5.73 |
| [14] |
ZHENG M, WANG Y H, WU X N, et al. Inactivation of Rheb by PRAK-mediated phosphorylation is essential for energy-depletion-induced suppression of mTORC1[J]. Nat Cell Biol, 2011, 13(3): 263-272. DOI:10.1038/ncb2168 |
| [15] |
UTZSCHNEIDER D T, DELPOUX A, WIELAND D, et al. Active maintenance of T cell memory in acute and chronic viral infection depends on continuous expression of FOXO1[J]. Cell Rep, 2018, 22(13): 3454-3467. DOI:10.1016/j.celrep.2018.03.020 |
| [16] |
WONG P, IWASAKI A. RAB15 empowers dendritic cells to drive antiviral immunity[J]. Sci Immunol, 2017, 2(13): eaan6448. DOI:10.1126/sciimmunol.aan6448 |
| [17] |
LEE S V, CHOI K H, CHOI Y W, et al. Hexane extracts of Polygonum multiflorum improve tissue and functional outcome following focal cerebral ischemia in mice[J]. Mol Med Rep, 2014, 9(4): 1415-1421. DOI:10.3892/mmr.2014.1943 |
| [18] |
吕锦芳, 宁康健, 丁小燕, 等. 何首乌粗提物对小鼠部分抗氧化抗衰老指标的影响[J]. 畜牧与兽医, 2012, 44(7): 58-60. LYU J F, NING K J, DING X Y, et al. Effects of crude extract of polygonum multiflorum on partial antioxidant and anti-aging index in mice[J]. Animal Husb Vet Med, 2012, 44(7): 58-60. |
| [19] |
KIM S J, HWANG Y H, MUN S K, et al. Protective effects of 2, 3, 5, 4'-tetrahydroxystilbene-2-O-β-d-glucoside on ovariectomy induced osteoporosis mouse model[J]. Int J Mol Sci, 2018, 19(9): E2554. DOI:10.3390/ijms19092554 |
| [20] |
ZHOU M R, LI J, WU J K, et al. Preventive effects of Polygonum multiflorum on glucocorticoid-induced osteoporosis in rats[J]. Exp Ther Med, 2017, 14(3): 2445-2460. DOI:10.3892/etm.2017.4802 |
| [21] |
YU Y, LANG X Y, LI X X, et al. 2, 3, 5, 4'-Tetrahydroxystilbene-2-O-β-d-glucoside attenuates MPP+/MPTP-induced neurotoxicity in vitro and in vivo by restoring the BDNF-TrkB and FGF2-Akt signaling axis and inhibition of apoptosis[J]. Food Funct, 2019, 10(9): 6009-6019. DOI:10.1039/c9fo01309a |
| [22] |
ZHANG Y, LIU B, CHEN X, et al. Naringenin ameliorates behavioral dysfunction and neurological deficits in a d-galactose-induced aging mouse model through activation of PI3K/akt/Nrf2 pathway[J]. Rejuv Res, 2017, 20(6): 462-472. DOI:10.1089/rej.2017.1960 |
| [23] |
FENG Y, YU Y H, WANG S T, et al. Chlorogenic acid protects D-galactose-induced liver and kidney injury via antioxidation and anti-inflammation effects in mice[J]. Pharm Biol, 2016, 54(6): 1027-1034. DOI:10.3109/13880209.2015.1093510 |
| [24] |
GAO J Q, YU Z P, JING S, et al. Protective effect of Anwulignan against D-galactose-induced hepatic injury through activating p38 MAPK-Nrf2-HO-1 pathway in mice[J]. Clin Interv Aging, 2018, 13: 1859-1869. DOI:10.2147/CIA.S173838 |
| [25] |
GUTIERREZ-ARCELUS M, LAPPALAINEN T, MONTGOMERY S B, et al. Correction: Passive and active DNA methylation and the interplay with genetic variation in gene regulation[J]. eLife, 2013, 2: e01045. DOI:10.7554/elife.01045 |
| [26] |
JI C Y, TANG M P, ZEIDLER C, et al. BAG3 and SYNPO (synaptopodin) facilitate phospho-MAPT/Tau degradation via autophagy in neuronal processes[J]. Autophagy, 2019, 15(7): 1199-1213. DOI:10.1080/15548627.2019.1580096 |
| [27] |
DATTA A, CHAI Y L, TAN J M, et al. An iTRAQ-based proteomic analysis reveals dysregulation of neocortical synaptopodin in Lewy body dementias[J]. Mol Brain, 2017, 10(1): 36. DOI:10.1186/s13041-017-0316-9 |
| [28] |
BOURDENX M, DEHAY B. Autophagy and brain: the case of neurodegenerative diseases[J]. Med Sci (Paris), 2017, 33(3): 268-274. DOI:10.1051/medsci/20173303013 |
| [29] |
TANG J, LIU J H, LI X, et al. PRAK interacts with DJ-1 and prevents oxidative stress-induced cell death[J]. Oxid Med Cell Longev, 2014, 2014: 735618. DOI:10.1155/2014/735618 |
| [30] |
TSEREL L, KOLDE R, LIMBACH M, et al. Age-related profiling of DNA methylation in CD8+ T cells reveals changes in immune response and transcriptional regulator genes[J]. Sci Rep, 2015, 5: 13107. DOI:10.1038/srep13107 |
| [31] |
FARR J N, WEIVODA M M, NICKS K M, et al. Osteoprotection through the deletion of the transcription factor rorβ in mice[J]. J Bone Miner Res, 2018, 33(4): 720-731. DOI:10.1002/jbmr.3351 |
| [32] |
TSEREL L, KOLDE R, LIMBACH M, et al. Age-related profiling of DNA methylation in CD8+ T cells reveals changes in immune response and transcriptional regulator genes[J]. Sci Rep, 2015, 5: 13107. DOI:10.1038/srep13107 |
| [33] |
WEYAND C M, GORONZY J J. Aging of the immune system. mechanisms and therapeutic targets[J]. Ann Am Thorac Soc, 2016, 13(Suppl 5): S422-S428. DOI:10.1513/AnnalsATS.201602-095AW |
| [34] |
SILVIN A, YU C N, LAHAYE X, et al. Constitutive resistance to viral infection in human CD141+ dendritic cells[J]. Sci Immunol, 2017, 2(13): eaai8071. DOI:10.1126/sciimmunol.aai8071 |



