2. 400038 重庆,陆军军医大学(第三军医大学)军事预防医学系:军事环境卫生学教研室;
3. 400038 重庆,陆军军医大学(第三军医大学)军事预防医学系:毒理学研究所
2. Department of Environment Health, Faculty of Military Preventive Medicine, Army Medical University (Third Military Medical University), Chongqing, 400038, China;
3. Institute of Toxicology, Faculty of Military Preventive Medicine, Army Medical University (Third Military Medical University), Chongqing, 400038, China
结直肠癌为全球最常见的消化系统恶性肿瘤也是世界第三大最常见的癌症,占所有癌症发病的9%以上[1]。2015年我国结直肠癌患者为37.63万例,死亡人数为19.1万人,结直肠癌在预防和治疗问题上面临着巨大的挑战[2]。因此,寻找有助于CRC预防、治疗和预后的生物标志物十分迫切。
细胞在正常代谢过程中因各种内、外因素如内源性活性氧等均可能导致DNA损伤,若细胞累积的DNA损伤得不到正确修复,则可能引起基因突变、癌基因激活、抑癌基因失活及染色体不稳定等,并引发细胞衰老、凋亡、代谢等调控异常,最终可能导致包括CRC在内的肿瘤等各种疾病。
大量研究表明个体DNA损伤修复能力与CRC发病风险有关,而在DNA损伤修复相关基因与CRC风险的研究目前主要集中于DNA错配修复基因和DNA聚合酶基因,其他典型的DNA修复途径与CRC的关联性尚未明确[3]。同源重组修复(homologous recombination repair,HRR)作为核心的DNA损伤修复方式之一,是维持基因组完整性确保遗传信息高保真传递的一种DNA修复机制。其修复能力受损的个体通常与癌症的风险增加有关[4-7]。
但是,HRR通路基因的遗传变异是否会影响相关基因功能从而导致个体间HRR能力差异,以及这些变异与CRC的发病风险是否存在关联等,目前尚缺乏研究,特别是在亚洲人群中HRR通路基因遗传变异与CRC的研究资料极少。本研究对筛选到的HRR通路中17个关键基因的单核苷酸多态性(single nucleotide polymorphisms,SNP)与结直肠癌的风险关联进行了分析,以探寻HRR基因的遗传变异情况对CRC风险易感性的影响。
1 资料与方法 1.1 研究对象于2001年1月至2004年6月在陆军军医大学3所附属医院收集的经病理诊断的新发结直肠癌病例478例,并以性别、居住地为匹配纳入同期非肿瘤住院患者为对照,详细纳入排除标准说明见文献[8]。病例纳入标准:①年龄:30~80岁;②性别:不限;③发病时间:初次诊断后6个月以内且未接受医学治疗的新发病例;④居住地及居住时间:重庆地区患者,在研究区域内连续居住>15年,或累计居住>30年且在外持续居住时间≤5年;⑤诊断:组织病理学确诊。病例排除标准:①复发结直肠癌患者;②家族性腺瘤性息肉病变;③遗传性非息肉病性结直肠癌患者;④患其他肿瘤或有其他肿瘤史;⑤患严重消化性疾病2年以上;⑥患有重度高血压、严重心脑血管疾病、肝硬化、脂肪肝、糖尿病、代谢综合征等相关疾病。对照纳入标准:①以性别、年龄(±3岁)为匹配因素;②同时期住院患者;③居住地及居住时间标准同病例纳入标准。对照排除标准:①肿瘤患者或有既往肿瘤史者;②患严重消化性疾病2年以上;③患有重度高血压、严重心脑血管疾病、肝硬化、脂肪肝、糖尿病、代谢综合征等相关疾病。所有参与人员均通过书面同意,同时采集5 mL外周静脉血液进行DNA样本取样。经排除DNA质量不合格样品后,最终纳入413例病例和1 671例对照,其中,男性病例/对照分别为230/890例,女性病例/对照分别为183/781例,病例和对照间性别分布差异无统计学意义(P>0.05)。
1.2 研究方法 1.2.1 通路候选基因的选择以关键词Homologous Recombination与double-strand break repair via homologous recombination分别检索KEGG数据库[9]与Amigo数据库,选择同时在两个数据库且均包含进入HRR通路的基因。在此基础上,以最近2年文献报道对同源重组通路中可能对结直肠癌发病风险有影响的候选基因进行筛查补充,共纳入17个关键基因。
1.2.2 DNA提取使用Wizard Genomic DNA Purification Kit(Promega)试剂盒按说明从外周血中提取基因组DNA,Nanodrop测量DNA原液浓度后统一稀释为50 ng/μL共50 μL的DNA样本。取10 μL至96孔板中密封并储存在-80 ℃。
1.2.3 SNP基因分型由上海晶能生物科技有限公司使用Illumina微芯片(Illumina,San Diego,California,USA)进行基因分型。
1.2.4 质量控制使用PLINK软件对测序样本与数据进行质量控制:剔除基因分型失败率大于5%、MAF≤1%以及对照组Hardy-Weinberg不平衡的SNP位点。
1.3 统计学分析数据分析采用Plink 3.0和SAS 9.1(SAS Institute,Cary,NC)进行,以Logistic回归模型计算SNP基因型与CRC发病风险的关联,以性别校正,以经bonferroni校正后P值(Pbonferroni)反映统计学差异,检验水准:α=0.05。
2 结果 2.1 HRR通路中筛选到的基因信息通过KEGG与Amigo数据库分析与同源重组修复功能有关的基因,筛选到同时在两个数据库均包含进入HRR通路的15个基因;同时根据最近2年文献报道对同源重组通路中可能对结直肠癌发病风险有影响的候选基因进行筛查补充,发现BARD1[10]和BRIP1[11]基因在HRR中也发挥着重要作用,且新近的研究提示可使乳腺癌、卵巢癌等肿瘤表型增加3倍或以上,但尚无对CRC影响的相关研究。因此,在纳入此2个基因后本次研究共纳入17个HRR通路的关键基因(图 1)。
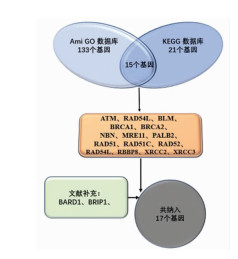
|
| 图 1 HRR通路基因筛选结果 |
2.2 SNPs等位基因与CRC发病风险的关联分析
在17个候选基因及其上下游各50 kb的区域内,有效分型的TagSNPs共2 207个,Logistic关联分析结果显示有16个位点与CRC显著相关(P < 0.05),见表 1。其中10个位点位于候选基因的远端位置,6个位点位于基因上下游2 kb区域,位于基因功能区域有2个SNPs(rs11226,P=6.67×10-6;rs4987114,P=7.37×10-3)。但经bonferroni校正后,在16个显著关联的位点中,仅位于RAD52基因3’-UTR的rs11226位点(OR=1.42,Pbonferroni=1.07×10-4)和CRTC3-AS1基因内含子区rs75893366位点(OR=0.43,Pbonferroni =2.91×10-2)与CRC的关联仍具有显著性,其余均不显著。
| SNPa | 基因区域 | 等位基因 | 病例 (n=413) |
对照 (n=1 671) |
OR(95% CI) | P值 | Pbonferroni |
| rs11226 | RAD52 3’-UTR | A/G | 438/386 | 1482/1 856 | 1.42 (1.22-1.66) | 6.67×10-6 | 1.07×10-4 |
| rs75893366 | CRCT3-AS1 | G/A | 15/811 | 136/3 194 | 0.43 (0.25-0.74) | 1.82×10-3 | 2.91×10-2 |
| rs4987114 | ATM 3’-UTR | G/A | 53/771 | 137/3 105 | 1.56 (1.12-2.16) | 7.37×10-3 | 1.18×10-1 |
| rs76629177 | C11orf65 intronic | G/A | 62/762 | 172/3 160 | 1.50 (1.11-2.02) | 8.44×10-3 | 1.35×10-1 |
| rs8066911 | UNC45B intronic | A/C | 175/649 | 855/2 479 | 0.78 (0.65-0.94) | 8.69×10-3 | 1.39×10-1 |
| rs191789336 | BLM missense | A/G | 10/814 | 15/3 319 | 2.72 (1.22-6.07) | 1.11×10-2 | 1.78×10-1 |
| rs3816754 | RAD51D intronic | A/C | 142/676 | 704/2 630 | 0.78 (0.64-0.96) | 1.68×10-2 | 2.69×10-1 |
| rs8069929 | 1.2kb 5’ of UNC45B | A/C | 119/703 | 388/2 950 | 1.20 (1.03-1.61) | 2.48×10-2 | 3.97×10-1 |
| rs10932562 | 16kb 5’ of AC107218.3 | G/A | 209/613 | 726/2 606 | 1.22 (1.03-1.46) | 2.51×10-2 | 4.02×10-1 |
| rs78416118 | 5.8kb 3’ of RND2 | A/G | 22/802 | 52/3 282 | 1.73 (1.05-2.87) | 2.53×10-2 | 4.05×10-1 |
| rs2277869 | WNK1 intronic | G/A | 52/774 | 286/3 048 | 0.72 (0.53-0.97) | 3.16×10-2 | 5.06×10-1 |
| rs13245470 | 3.1kb 5’ of SNORA26 | G/A | 57/765 | 169/3 163 | 1.40 (1.02-1.90) | 3.50×10-2 | 5.60×10-1 |
| rs11651601 | BRIP1 intronic | C/A | 109/711 | 542/2 796 | 0.79 (0.63-0.99) | 3.76×10-2 | 6.02×10-1 |
| rs7579026 | AC107218.3 | A/G | 195/625 | 682/2 640 | 1.21 (1.01-1.45) | 4.13×10-2 | 6.61×10-1 |
| rs2310928 | 17kb 3’ of SNORA26 | A/G | 162/664 | 762/2 576 | 0.82 (0.68-1.00) | 4.46×10-2 | 7.14×10-1 |
| rs1799944 | BRCA2 missense | G/A | 69/751 | 359/2 979 | 0.76 (0.58-1.00) | 4.65×10-2 | 7.44×10-1 |
| a:rs11226、rs75893366、rs4987114、rs76629177、rs8066911、rs191789336、rs3816754、rs8069929、rs10932562、rs78416118、rs2277869、rs13245470、rs11651601、rs7579026、rs55794205、rs2310928与rs1799944分别有3、6、51、6、5、5、8、4、7、5、2、7、5、13、13、8、5个样品基因分型失败 | |||||||
经性别分层后,在16个位点中有8个位点在男性中的关联具有显著性(表 2);有4个在女性中具有显著性(表 3)。但仅rs11226位点经bonferroni校正后在男性(OR=1.42,Pbonferroni=2.54×10-3)和女性(OR=1.41,Pbonferroni=4.69×10-2)中均具有显著性。
| SNPa | 位置信息 | 等位基因 | 病例(n=230) | 对照(n=890) | OR (95% CI) | P值 | Pbonferroni |
| rs11226 | RAD52 3’-UTR | A/G | 198/154 | 560/688 | 1.42 (1.16~1.75) | 1.59×10-4 | 2.54×10-3 |
| rs13245470 | 3.1kb 5’ of SNORA26 | G/A | 40/420 | 78/1702 | 2.07 (1.40~3.09) | 2.22×10-4 | 3.55×10-3 |
| rs4987114 | ATM 3’-UTR | G/A | 32/428 | 68/1 660 | 1.83 (1.18~2.82) | 5.82×10-2 | 9.31×10-2 |
| rs76629177 | C11orf65 intronic | G/A | 36/424 | 89/1 685 | 1.61 (1.08~2.40) | 1.95×10-2 | 3.12×10-1 |
| rs8066911 | UNC45B intronic | A/C | 100/360 | 483/1 295 | 0.74 (0.58~0.95) | 1.81×10-2 | 2.90×10-1 |
| rs191789336 | BLM missense | A/G | 5/453 | 4/1 772 | 4.89 (1.31~18.28) | 9.05×10-3 | 1.45×10-1 |
| rs8069929 | 1.2kb 5’ of UNC45B | A/C | 75/385 | 206/1 574 | 1.49 (1.12~1.98) | 6.31×10-2 | 1.00×10-0 |
| rs2277869 | WNK1 intronic | G/A | 22/438 | 158/1 620 | 0.52 (0.33~0.81) | 3.92×10-3 | 6.27×10-2 |
| a:rs11226、rs13245470、rs4987114、rs76629177、rs8066911、rs191789336、rs8069929、rs2277869与rs13245470分别有3、7、51、6、5、5、4、2个样品基因分型失败 | |||||||
| SNPa | 位置信息 | 等位基因 | 病例 (n=183) |
对照 (n=781) |
OR(95% CI) | P值 | Pbonferroni |
| rs11226 | RAD52 3’-UT R | A/C | 187/179 | 662/896 | 1.41 (1.12~1.78) | 2.93×10-3 | 4.69×10-2 |
| rs75893366 | CRCT3-AS1 | G/A | 7/359 | 77/1 479 | 0.37 (0.17~0.82) | 1.06×10-2 | 1.70×10-1 |
| rs10932562 | AC107218.3 intronic | A/G | 98/264 | 344/1 208 | 1.30 (1.00~1.69) | 4.61×10-2 | 7.38×10-1 |
| rs1799944 | BRCA2 missense | G/A | 23/337 | 182/1 376 | 0.52 (0.33~0.81) | 3.40×10-3 | 5.44×10-2 |
| a:rs11226、rs75893366、rs10932562与rs1799944分别有3、6、7、5个样品基因分型失败 | |||||||
2.3 SNPs等位基因型与CRC发病风险的关联分析
对16个显著性位点进行性别分层后分析其在显隐性遗传模型中的效应,在显性遗传模型中男性基因型与CRC的易感性在性别分层前后共有3个位点(表 4),其中在候选基因上下游2 kb区域RAD52 3’-UTR的rs11226 AG与AA基因(OR=1.42,95%CI=1.01~ 1.99)与ATM 3’-UTR的rs4987114 GG和GA基因型(OR=1.89,95%CI=1.20~2.97)可增加CRC的患病风险,但经bonferroni校正后仅rs11226位点的基因型与CRC关联仍显著。而女性在分层前后仅有rs11226 AG与AA基因型(OR=1.57,95%CI=1.10~2.27)具有风险效应(表 5)。分析结果提示rs11226在经过Logistic回归模型调整性别协变量后与CRC发病风险密切关联,可增加CRC的患病风险性。
| SNPa | 病例[n=230,例(%)] | 对照[n=890,例(%)] | OR(95% CI) | P值 | Pbonferroni |
| rs11226 | 1.59×10-4 | 2.54×10-3 | |||
| GG | 54 (23.58) | 271 (30.45) | 1 | - | - |
| GA | 99 (43.23) | 418 (46.97) | 1.19 (0.83~1.71) | - | - |
| AA | 76 (33.39) | 201 (22.58) | 1.89 (1.28~2.81) | - | - |
| AA+GA | 175 (76.42) | 619 (69.55) | 1.42 (1.01~1.99) | - | - |
| rs8069929 | 6.31×10-2 | 1.00×10-0 | |||
| CC | 164 (71.31) | 695 (78.09) | 1.00 | - | - |
| AC | 57 (24.78) | 184 (20.67) | 1.31 (0.93~1.85) | - | - |
| AA | 9 (3.91) | 11 (1.24) | 3.47 (1.41~8.50) | - | - |
| AA+AC | 66 (28.70) | 195 (21.91) | 1.43 (1.03~1.99) | - | - |
| rs4987114 | 5.82×10-2 | 9.31×10-2 | |||
| AA | 199 (86.51) | 798 (92.36) | 1.00 | - | - |
| GA | 30 (13.04) | 64 (7.41) | 60.12(14.26~253.82) | - | - |
| GG | 1 (0.45) | 2 (0.23) | 2.01 (0.18~22.22) | - | - |
| GG+GA | 31 (13.48) | 66 (7.64) | 1.89 (1.20~2.97) | - | - |
| a:在男性中rs11226和rs4987114位点分别有1、26个样品基因分型失败 | |||||
| SNPa | 病例[n=183,例(%)] | 对照[n=781,例(%)] | OR (95% CI) | P值 | Pbonferroni |
| rs11226 | 2.93×10-3 | 4.69×10-2 | |||
| GG | 45 (24.59) | 264 (33.89) | 1.00 | - | - |
| AA | 49 (26.78) | 147 (18.87) | 1.96 (1.24~3.07) | - | - |
| AG | 89 (48.64) | 368 (47.24) | 0.29 (0.09~2.10) | - | - |
| AA+GA | 138 (45.41) | 515 (66.11) | 1.57 (1.10~2.27) | - | - |
| rs75893366 | 1.06×10-2 | 1.70×10-1 | |||
| AA | 176 (96.17) | 704 (90.49) | 1.00 | - | - |
| GA | 7 (3.83) | 71 (9.13) | 0.39 (0.18~0.87) | - | - |
| GG | 0 (0.00) | 3 (0.38) | - | - | - |
| GG+GA | 7 (3.83) | 74 (9.51) | 0.38 (0.17~0.84) | - | - |
| a:在女性中rs11226与rs75893366分别有2、3个样品基因型分型失败 | |||||
3 讨论
近年来越来越多的证据表明,HRR通路紊乱是多种类型癌症的基础,其复杂的过程涉及多种基因,在多种肿瘤中发挥并扮演重要的角色[12],HRR能力强弱可能是体细胞突变及相应的多种类型肿瘤的主要诱因。如BRCA1/2与乳腺癌,卵巢癌;RAD54与非霍奇金淋巴瘤[13];RECQL4与骨肉瘤和皮肤癌[14];ATM、NBS1、BLM和WRN与淋巴瘤和白血病[15]等。但是,HRR通路基因的遗传多态性是否与CRC的发病风险存在关联,目前尚未明确。因此,本研究采用人类基因组芯片分型技术探讨中国西南地区人群在同源重组修复通路中发挥主要作用的17个基因的遗传多态性与结直肠发病风险之间的关联。
本研究基于生物信息学和文献筛查共筛选候选基因17个,在2 207个分型位点中共发现16个SNP与CRC显著关联,其中在候选基因上下游2 kb内存在6个SNP位点,位于候选基因的远端位置有10个位点,经bonferroni校正后,在男性等位基因频率关联分析中仅rs11226(P=2.54×10-3)和rs13245470(P=3.55×10-3)两位点的关联显著,在女性中仅rs11226(P=4.69×10-2)位点仍关联显著;而在SNP位点的基因型与CRC关联的性别分层分析中,仅RAD52基因3’-UTR区域内的rs11226位点的基因型与CRC仍存在显著关联。
RAD52在HRR通路中发挥着重要的作用,在哺乳动物细胞中作为DNA链交换和同源重组期间的关键蛋白,以寡聚体形式存在,负责修复双链DNA断裂。主要参与结合单链DNA(ssDNA),促进ssDNA退火,与复制蛋白A相互作用,刺激RAD51介导的同源DNA配对[16]。RAD52还可在BRCA2缺陷引起的细胞周期检查点紊乱中起到补偿BRCA2丧失功能的作用[17]。RAD52的异常与肝癌[18]、乳腺癌等息息相关,其多态性可作为宫颈癌的铂耐药性和预后的分子标记[19]。
在RAD52启动子区和3′非翻译区已鉴定出多个单核苷酸多态性(SNPs)。JIANG等[20]研究显示位于3′-UTR的rs7963551位点的A>C多态性可通过miRNA-mRNA结合作用,降低miRNA let-7与3′-UTR的结合亲和力,从而提高RAD52基因的转录,降低乳腺癌发病风险。ZHANG等[21]在中国人群中发现RAD52与远处CRC转移之间存在显著相关性,rs7963551 GT与GG基因型增加CRC淋巴转移的风险,在3′-UTR区域rs1051669、rs10774474、rs11571378、rs6489769和rs7963551常见的T-T-T-T-T,C-T-T-T-T和C-T-A-C-T单倍型约使CRC的风险增加两倍。NACCARATI等[22]以修复DNA双键断裂的HRR通路为背景,探索HRR通路基因3′-UTR的基因多态性与癌症的易感性和预后情况,结果表明位于RAD52 3′-UTR的rs11226、rs1051669和rs11571475位点的多态性与结直肠癌风险相关,rs1051669的A等位基因可使直肠癌的风险增加1.7倍,而rs11571475杂合子TC基因型的携带者可降低患结肠癌的风险,其中大肠癌患者在隐性遗传模型中携带rs11226 TT基因型会表现出更好的生存率。3′-UTR区域的变体多态性可调节基因表达并对肿瘤的发展风险产生影响,rs11226多态性位于3′非翻译区,具备调节RAD52基因的表达能力。本研究结果显示RAD52 3′-UTR rs11226是结果中最为显著的位点(P=6.67×10-6),其A等位基因为风险因素(OR=1.42,95%CI=1.22~1.66),在性别分层前后的显隐性遗传模型中均表现出统一的风险效应(OR>1)。基因对疾病的复杂性状影响方面在很大程度上是由数量性状的微效多基因决定的,SNP位点可能通过影响基因表达量对这些数量性状产生轻微的作用。因此,我们推测rs11226多态性位点的改变可能影响到miRNA的结合能力从而影响RAD52基因表达量的变化,最终增加了患CRC的风险。但是,本研究仍然存在不足之处。由于缺失了较多数量对照样本的年龄等资料信息,本研究在分析中未能对年龄等混杂因素进行校正,导致未能有效控制可能由年龄导致的关联分析结果偏倚。但考虑到SNP属于遗传背景变异并不受年龄因素影响,研究中SNP位点与CRC风险关联的结果仍可能因未能校正年龄对CRC的影响而导致最终的关联水平与真实关联之间可能存在差异。因此,本研究结果及各位点与CRC易感性之间的真实关联还需要更多研究进一步验证。
综上所述,我们有理由相信同源重组修复通路中基因的遗传多态性与CRC易感性之间存在显著关联性,RAD52基因功能区域的rs11226位点存在作为CRC新遗传分子标记的可能性,但其遗传多态性是否可能影响RAD52基因的功能从而导致个体间CRC患病易感性的差异,本研究尚未对其进行相关的功能验证。同时,因为人群的差异以及样本数量较少,本研究结果仍需在更大的人群样本中进行重复研究加以验证。
| [1] |
SIEGEL R L, MILLER K D, JEMAL A. Cancer statistics, 2017[J]. CA: Cancer J Clin, 2017, 67(1): 7-30. DOI:10.3322/caac.21387 |
| [2] |
张玥, 石菊芳, 黄慧瑶, 等. 中国人群结直肠癌疾病负担分析[J]. 中华流行病学杂志, 2015, 36(7): 709-714. ZHANG Y, SHI J F, HUANG H Y, et al. Burden of colorectal cancer in China[J]. Chin J Epidemiol, 2015, 36(7): 709-714. DOI:10.3760/cma.j.issn.0254-6450.2015.07.010 |
| [3] |
ALDUBAYAN S H, GIANNAKIS M, MOORE N D, et al. Inherited DNA-repair defects in colorectal cancer[J]. Am J Hum Genet, 2018, 102(3): 401-414. DOI:10.1016/j.ajhg.2018.01.018 |
| [4] |
MIKI Y, SWENSEN J, SHATTUCK-EIDENS D, et al. A strong candidate for the breast and ovarian cancer susceptibility gene BRCA1[J]. Science, 1994, 266(5182): 66-71. DOI:10.1126/science.7545954 |
| [5] |
WOOSTER R, BIGNELL G, LANCASTER J, et al. Identification of the breast cancer susceptibility gene BRCA2[J]. Nature, 1995, 378(6559): 789-792. DOI:10.1038/378789a0 |
| [6] |
ROY R, CHUN J, POWELL S N. BRCA1 and BRCA2: different roles in a common pathway of genome protection[J]. Nat Rev Cancer, 2012, 12(1): 68-78. DOI:10.1038/nrc3181 |
| [7] |
ORTHWEIN A, NOORDERMEER S M, WILSON M D, et al. A mechanism for the suppression of homologous recombination in G1 cells[J]. Nature, 2015, 528(7582): 422-426. DOI:10.1038/nature16142 |
| [8] |
YANG H, ZHOU Y H, ZHOU Z Y, et al. A novel polymorphism rs1329149 of CYP2E1 and a known polymorphism rs671 of ALDH2 of alcohol metabolizing enzymes are associated with colorectal cancer in a Southwestern Chinese population[J]. Cancer Epidemiol Biomarkers Prev, 2009, 18(9): 2522-2527. DOI:10.1158/1055-9965.EPI-09-0398 |
| [9] |
KANEHISA M, SATO Y, KAWASHIMA M, et al. KEGG as a reference resource for gene and protein annotation[J]. Nucleic Acids Res, 2016, 44(D1): D457-D462. DOI:10.1093/nar/gkv1070 |
| [10] |
BILLING D, HORIGUCHI M, WU-BAER F, et al. The BRCT domains of the BRCA1 and BARD1 tumor suppressors differentially regulate homology-directed repair and stalled fork protection[J]. Mol Cell, 2018, 72(1): 127-139.e8. DOI:10.1016/j.molcel.2018.08.016 |
| [11] |
RAFNAR T, GUDBJARTSSOND F, SULEM P, et al. Mutations in BRIP1 confer high risk of ovarian cancer[J]. Nat Genet, 2011, 43(11): 1104-1107. DOI:10.1038/ng.955 |
| [12] |
SMILENOVL B. Tumor development: haploinsufficiency and local network assembly[J]. Cancer Lett, 2006, 240(1): 17-28. DOI:10.1016/j.canlet.2005.08.015 |
| [13] |
WONGA K, ORMONDE P A, PERO R, et al. Characterization of a carboxy-terminal BRCA1 interacting protein[J]. Oncogene, 1998, 17(18): 2279-2285. DOI:10.1038/sj.onc.1202150 |
| [14] |
HIRAMOTO T, NAKANISHI T, SUMIYOSHI T, et al. Mutations of a novel human RAD54 homologue, RAD54B, in primary cancer[J]. Oncogene, 1999, 18(22): 3422-3426. DOI:10.1038/sj.onc.1202691 |
| [15] |
HELLEDAY T. Homologous recombination in cancer development, treatment and development of drug resistance[J]. Carcinogenesis, 2010, 31(6): 955-960. DOI:10.1093/carcin/bgq064 |
| [16] |
LOKB H, POWELL S N. Molecular pathways: understanding the role of Rad52 in homologous recombination for therapeutic advancement[J]. Clin Cancer Res, 2012, 18(23): 6400-6406. DOI:10.1158/1078-0432.CCR-11-3150 |
| [17] |
MAHAJAN S, RAINA K, VERMA S, et al. Human RAD52 protein regulates homologous recombination and checkpoint function in BRCA2 deficient cells[J]. Int J Biochem Cell Biol, 2019, 107: 128-139. DOI:10.1016/j.biocel.2018.12.013 |
| [18] |
LI Z Q, GUO Y, ZHOU L Q, et al. Association of a functional RAD52 genetic variant locating in a miRNA binding site with risk of HBV-related hepatocellular carcinoma[J]. Mol Carcinog, 2015, 54(9): 853-858. DOI:10.1002/mc.22156 |
| [19] |
SHIT Y, YANG G, TU X Y, et al. RAD52 variants predict platinum resistance and prognosis of cervical cancer[J]. PLoS ONE, 2012, 7(11): e50461. DOI:10.1371/journal.pone.0050461 |
| [20] |
JIANG Y, QIN Z Z, HU Z B, et al. Genetic variation in a hsa-let-7 binding site in RAD52 is associated with breast cancer susceptibility[J]. Carcinogenesis, 2013, 34(3): 689-693. DOI:10.1093/carcin/bgs373 |
| [21] |
ZHANG L Y, ZHANG Y J, TANG C H, et al. RAD52 gene polymorphisms are associated with risk of colorectal cancer in a Chinese Han population[J]. Medicine (Baltimore), 2017, 96(49): e8994. DOI:10.1097/MD.0000000000008994 |
| [22] |
NACCARATI A, ROSA F, VYMETALKOVA V, et al. Double-strand break repair and colorectal cancer: gene variants within 3' UTRs and microRNAs binding as modulators of cancer risk and clinical outcome[J]. Oncotarget, 2016, 7(17): 23156-23169. DOI:10.18632/oncotarget.6804 |



