2. 400038 重庆,陆军军医大学(第三军医大学)第一附属医院感染管理科
2. Department of Hospital Infection Management, First Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400038, China
鲍曼不动杆菌(Acinetobacter baumannii)为非发酵革兰阴性杆菌,广泛存在于自然界的水、土壤等潮湿环境中,常寄居于人或动物体表皮肤、泌尿生殖道、消化道和呼吸道。研究表明,鲍曼不动杆菌在医院的床单位、医疗仪器等物表分布广泛且长期存活,极易造成老年患者、危重患者及机体免疫力低下患者的感染,是引起严重院内感染最常见的条件致病菌[1]。目前,鲍曼不动杆菌对临床常用抗菌药物的耐药性严重,碳青霉烯类、β-内酰胺类抗生素复合制剂等抗菌药物是临床治疗鲍曼不动杆菌严重感染的有效手段,但随着近年来该类抗菌药物在临床的大量及不合理使用,造成了鲍曼不动杆菌的耐药菌,特别是多重耐药或“泛耐药”越来越多,可供选择的抗菌药物越来越少,给临床抗感染治疗带来了严峻挑战[2]。
噬菌体(bacteriophage)是一类能感染细菌等微生物的病毒。它必须依靠宿主细胞才能进行复制和繁殖,具有寄生性和严格的宿主特异性。按照噬菌体与宿主菌的关系分为裂解性噬菌体和溶源性噬菌体,鉴于裂解性噬菌体的溶菌作用而被认为是细菌的天然杀手[3]。随着细菌耐药性问题日益严重,新抗生素研发的速度远低于耐药菌产生的速度[4]。作为可替代抗生素成为新型抗菌剂的噬菌体,重新引起了国内外研究者的重视。目前其针对多耐药菌感染性疾病治疗的有效性研究报道较多[5],特别是生物工程噬菌体的出现,克服了许多传统噬菌体疗法的缺陷,使靶向给药或逆转耐药菌的耐药性成为可能[6]。然而,噬菌体广泛存在于自然界中,常伴随着细菌而存在,理论上每种细菌均有其对应的噬菌体,所有噬菌体种类超过108种[7]。因此,筛选某种细菌的特异性噬菌体尤其是具有较强溶菌作用的裂解性噬菌体的难度较大,需要获得大量的噬菌体,并对其进行生物学特性研究和基因组分析[8]。为此,本研究在医院污水中分离获得1株鲍曼不动杆菌的裂解性噬菌体SWH-Ab-3,并对其生物学特性和基因组进行初步研究,为噬菌体用于多重耐药鲍曼不动杆菌所致院内感染的预防控制以及感染治疗提供基础。
1 材料与方法 1.1 主要试剂和仪器琼脂粉、酵母提取物、蛋白胨购自英国OXOID公司;氯化钠购自重庆川东化工集团公司;噬菌体基因组DNA提取试剂盒(Code No. AB114)购自北京艾比根生物技术有限公司。ThermoFisher Micro 21R离心机为赛默飞世尔科技(中国)有限公司产品;电热恒温培养箱为上海跃进医疗器械有限公司产品;JEM-1400 Plus透射电子显微镜为日本精工(JEOL)公司产品;HiSeq2000高通量测序仪为美国Illumina公司产品。
1.2 菌株和培养基42株鲍曼不动杆菌、2株大肠杆菌、2株金黄色葡萄球菌、2株铜绿假单胞菌及2株肺炎克雷伯菌均由陆军军医大学(第三军医大学)第一附属医院检验科微生物室分离鉴定,本实验室保存。所有菌株均采用Vitek 2 Compact -32全自动细菌鉴定系统(法国生物梅里埃公司)进行生化性质鉴定。自配LB液体培养基:氯化钠10.0 g,蛋白胨10.0 g,酵母提取物5.0 g;半固体培养基在LB培养基中加入琼脂粉7.0 g,固体培养基在LB培养基中加入琼脂粉15.0 g,加水至1 L,pH 7.4,高压灭菌后分装备用。污水样品采自本院污水站,加消毒剂前采集。
1.3 噬菌体分离与鉴定 1.3.1 宿主菌制备将-80 ℃保存的42株鲍曼不动杆菌、2株大肠杆菌、2株金黄色葡萄球菌、2株铜绿假单胞菌及2株肺炎克雷伯菌用三线法接种于固体LB平板上,37 ℃过夜培养,复苏菌株。次日,挑取细菌单个菌落分别接种于2 mL液体LB培养基中,于37 ℃、200 r/min培养至光密度值D(600)为0.6左右,制备宿主菌菌悬液,置于4 ℃保存备用。
1.3.2 噬菌体富集与分离按照罗娟等[9]的方法,从医院污水中富集和分离鲍曼不动杆菌裂解性噬菌体,并观察噬菌斑的形成及特点。
1.3.3 噬菌体纯化与保存从LB双层琼脂平板上挑取单个噬菌斑,参照罗娟等[9]的方法纯化噬菌体3~5次,至噬斑的形状和大小均一。纯化的噬菌体与50%甘油等体积混匀后放于-20 ℃和-80 ℃冻存、备用。
1.3.4 噬菌体形态观察参照文献[9]报道的方法观察噬菌体的形态特征。
1.4 噬菌体生物学特性研究 1.4.1 噬菌体裂解谱测定采用邢少贞等[10]建立的点板试验测定噬菌体的裂解谱。受试菌为鲍曼不动杆菌42株、大肠杆菌2株、金黄色葡萄球菌2株、铜绿假单胞菌2株和肺炎克雷伯菌2株。若平板上点加噬菌体后的菌苔出现规则、透明空斑即为裂菌试验阳性。
1.4.2 噬菌体最佳感染复数(multiplicity of infection,MOI)测定及一步生长曲线绘制参照文献[10]报道的方法进行。噬菌体与宿主菌的比值(MOI)分别为0.001、0.01、0.1、1、10、100,培养后得到噬菌体滴度最高的MOI即为最佳感染复数。再按照最佳MOI的比例加入噬菌体和宿主菌共培养,分别在0时刻和每间隔10 min取样,测定样品噬菌体滴度,并绘制噬菌体的一步生长曲线。
1.4.3 噬菌体的热稳定性将0.7 mL液体LB培养基加入无菌EP管中,分别在30、40、50、60、70、80 ℃的水浴中孵育30 min,每个温度点分别设置3个平行管。待试管内外温度稳定后,加入0.3 mL噬菌体(24×109 PFU/mL),分别在30、40、50、60、70、80 ℃的水浴中再孵育30 min后,取出并立即置于冰浴中冷却。取孵育液10 μL梯度稀释后,采用双层琼脂平板法检测噬菌体的滴度,并绘制噬菌体的热稳定性曲线。
1.4.4 噬菌体对pH敏感性用浓盐酸(1 mol/L)、氢氧化钠溶液(1 mol/L)分别将液体LB培养基调至pH值为2.0、3.0、4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0,高压灭菌后备用。取不同pH的液体LB培养基2 mL加至10 mL无菌玻璃试管中,分别加入100 μL噬菌体(109 PFU/mL),37 ℃恒温摇床中孵育4 h,每个pH值分别设置3个平行实验管。取孵育液10 μL做梯度稀释后,采用双层琼脂平板法检测噬菌体的滴度,并绘制噬菌体的酸碱敏感性曲线。
1.5 噬菌体全基因组测序及序列分析用λ噬菌体基因组DNA提取试剂盒(Cat:AB114)抽提噬菌体SWH-Ab-3基因组DNA,序列测定由北京博奥晶典生物技术有限公司完成。主要步骤如下:检测DNA样品的浓度和纯度,用检测合格的样品构建测序文库和cluster制备,采用第2代高通量测序技术Illumina Hiseq2000测序。所获原始测序数据,经预处理去除接头、引物及低质量数据后,通过Spades软件进行噬菌体基因组序列组装,获得噬菌体SWH-Ab-3最好的DNA序列拼接结果。
噬菌体基因组信息分析采用Glimmer(http://www.ncbi.nlm.nih.gov/genomes/MICROBES/glimmer_3.cgi)软件预测噬菌体基因组的开放阅读框(open reading frame, ORF),同时使用BLAST在线工具(http://blast.ncbi.nlm.nih.gov/)进行碱基序列相似性比对及编码基因的注释。再通过tRNAscan-SE(http://lowelab.ucsc.edu/tRNAscan-SE/)确定基因组上是否存在tRNA基因。最后,将基因组注释序列提交NCBI数据库,获取序列登录号。
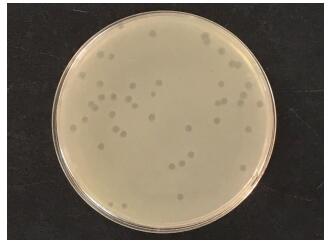
2 结果 2.1 噬菌体生物学特性研究 2.1.1 噬菌体的分离及其噬菌斑特征以临床分离的鲍曼不动杆菌为宿主菌,经双层琼脂平板法筛选和纯化,从医院污水中分离到1株裂解性鲍曼不动杆菌噬菌体,命名为噬菌体SWH-Ab-3。在双层琼脂平板上可见噬菌体SWH-Ab-3能形成形态规则的噬菌斑,直径0.5~1.0 mm,圆形透明、边缘清晰,呈现出裂解性噬菌体的噬菌斑特征(图 1)。
|
| 图 1 噬菌体SWH-Ab-3的噬菌斑形态特征 |
2.1.2 噬菌体SWH-Ab-3的形态
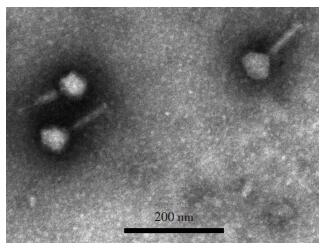
纯化的噬菌体SWH-Ab-3经磷钨酸负染后,透射电子显微镜观察的形态如图 2所示。噬菌体SWH-Ab-3的头部呈正多面体对称结构,头部直径约为70 nm,尾长约为80 nm。根据噬菌体分类方法,把该噬菌体初步归于肌尾噬菌体科(Myoviridae)。
|
| 图 2 电镜观察噬菌体SWH-Ab-3的形态 |
2.1.3 噬菌体SWH-Ab-3的裂菌谱
噬菌体SWH-Ab-3对包括鲍曼不动杆菌、大肠杆菌、金黄色葡萄球菌、铜绿假单胞菌及肺炎克雷伯菌在内的50株临床分离菌株的裂解作用见表 1。结果显示,噬菌体SWH-Ab-3可裂解6株鲍曼不动杆菌临床分离菌株,裂解率为14.28%(6/42),但对大肠杆菌、金黄色葡萄球菌、铜绿假单胞菌及肺炎克雷伯菌均无裂解能力。
| 菌株 | 敏感性 | 菌株 | 敏感性 | 菌株 | 敏感性 |
| A. baumannii 01 | + | A. baumannii 18 | - | A. baumannii 35 | - |
| A. baumannii 02 | - | A. baumannii 19 | - | A. baumannii 36 | - |
| A. baumannii 03 | - | A. baumannii 20 | + | A. baumannii 40 | - |
| A. baumannii 04 | + | A. baumannii 21 | - | A. baumannii 46 | - |
| A. baumannii 05 | - | A. baumannii 22 | + | A. baumannii 48 | + |
| A. baumannii 06 | - | A. baumannii 23 | - | A. baumannii 53 | - |
| A. baumannii 07 | - | A. baumannii 24 | - | A. baumannii 61 | - |
| A. baumannii 08 | - | A. baumannii 25 | - | A. baumannii 150 | + |
| A. baumannii 09 | - | A. baumannii 26 | - | E. coli 01 | - |
| A. baumannii 10 | - | A. baumannii 27 | - | E. coli 02 | - |
| A. baumannii 11 | - | A. baumannii 28 | - | S. aureus 01 | - |
| A. baumannii 12 | - | A. baumannii 29 | - | S. aureus 02 | - |
| A. baumannii 13 | - | A. baumannii 30 | - | P. aeruginosa 01 | - |
| A. baumannii 14 | - | A. baumannii 31 | - | P. aeruginosa 02 | - |
| A. baumannii 16 | - | A. baumannii 33 | - | K. pneumoniae 01 | - |
| A. baumannii 17 | - | A. baumannii 34 | - | K. pneumoniae 02 | - |
| +:裂解;-:不裂解 | |||||
2.1.4 噬菌体SWH-Ab-3的最佳感染复数
分别按照不同比例将鲍曼不动杆菌宿主菌(A. baumannii 01)与噬菌体SWH-Ab-3混合,培养4 h后测定噬菌体滴度。结果显示,当MOI=0.1时,培养后上清液中噬菌体的滴度最高(表 2),表明噬菌体SWH-Ab-3的最佳感染复数为0.1。
| 管号 | 噬菌体数/PFU | 细菌数/CFU | MOI | 4 h噬菌体滴度/PFU·mL-1 |
| 1 | 108 | 106 | 100.00 | 2.55×109 |
| 2 | 108 | 107 | 10.00 | 5.60×109 |
| 3 | 108 | 108 | 1.00 | 2.89×1010 |
| 4 | 108 | 109 | 0.10 | 8.40×1010 |
| 5 | 107 | 109 | 0.01 | 6.75×1010 |
| 6 | 106 | 109 | 0.001 | 7.30×1010 |
2.1.5 噬菌体SWH-Ab-3的一步生长曲线
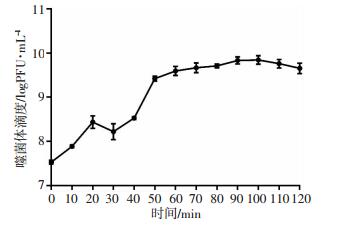
噬菌体和宿主菌按照最佳MOI的比例共培养后,以噬菌体滴度为纵坐标,培养时间为横坐标,绘制噬菌体的一步生长曲线(图 3),以评估噬菌体的裂解能力。从图中可以看出,SWH-Ab-3感染宿主菌40 min内,噬菌体的数量没有明显增加,该时间段为噬菌体的潜伏期;感染后40~90 min内噬菌体的数量急剧增加,之后趋于稳定,表明其爆发期为50 min。根据裂解量计算公式:裂解量=爆发末期噬菌体滴度/感染初期宿主菌浓度,即得出噬菌体SWH-Ab-3的裂解量约为31.4 PFU/cell,表明该噬菌体的增殖能力较强。
|
| 图 3 噬菌体SWH-Ab-3的一步生长曲线 |
2.1.6 噬菌体SWH-Ab-3的热稳定性
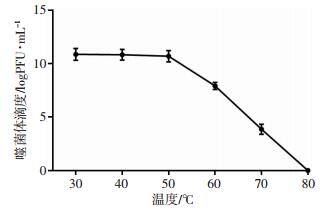
以不同温度孵育后噬菌体的残留滴度为纵坐标,温度为横坐标绘制噬菌体的热稳定性曲线(图 4)。噬菌体SWH-Ab-3在30~50 ℃孵育30 min后,其活性滴度基本不变,当温度高于60 ℃,噬菌体残留滴度开始下降,70 ℃时滴度急剧下降,当孵育温度达到80 ℃时,噬菌体全部失活。说明该噬菌体对热处理有一定抗性,但不耐高温。
|
| 图 4 噬菌体SWH-Ab-3的热稳定性测定 |
2.1.7 噬菌体SWH-Ab-3的pH稳定性
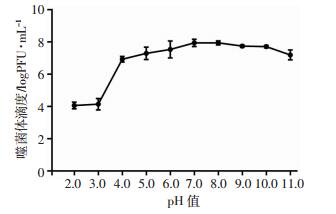
以不同pH条件下孵育后噬菌体的残留滴度为纵坐标,pH值为横坐标绘制噬菌体的酸碱稳定性曲线(图 5)。噬菌体SWH-Ab-3经不同pH处理4 h后,随着pH的降低,pH在4.0~5.0范围内噬菌体活性开始下降,随后噬菌体的活性呈快速下降趋势;pH在6.0~8.0范围内对噬菌体的活性影响不大,且当pH为7.0时噬菌体的活性最高;随着pH值的增高,噬菌体的活性小幅下降,但仍保留较高活性。结果表明该噬菌体对酸碱环境有一定的抗性,其耐碱性更强。
|
| 图 5 噬菌体SWH-Ab-3的pH稳定性测定 |
2.2 噬菌体基因组学研究 2.2.1 噬菌体SWH-Ab-3的全基因组概况
经测序数据分析,噬菌体SWH-Ab-3基因组长度为41 730 bp,G+C%含量为39.38%。将噬菌体SWH-Ab-3全基因组核酸序列提交GenBank,获取的登录号为MG599035。通过tRNAscan-SE预测软件分析,发现噬菌体SWH-Ab-3的基因组中无tRNA存在。经BLASTn比对分析发现,噬菌体SWH-Ab-3与鲍曼不动杆菌噬菌体Abp1(42 185 bp,GenBank登录号JX658790)和vB_AbaP_B3(40 598 bp,GenBank登录号MF033348)的比对覆盖率分别为96%和94%。尽管它们之间存在较高的相似性,但Abp1和vB_AbaP_B3的基因组长度与SWH-Ab-3不同,另一方面从噬菌体形态看,Abp1和vB_AbaP_B3均属于短尾噬菌体科,而SWH-Ab-3属于肌尾噬菌体科。由此可见,噬菌体SWH-Ab-3是一株新分离的鲍曼不动杆菌裂解性噬菌体。
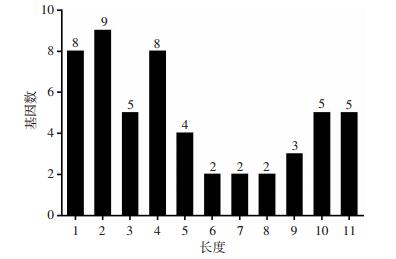
2.2.2 噬菌体SWH-Ab-3的功能性ORF分析噬菌体全基因组通过Glimmer软件在线初步预测,有53个推定的开放阅读框(ORF)。将噬菌体SWH-Ab-3的ORF长度进行统计分析,从图 6中可以看到,推定的功能基因长度具有较大差异,其长度在100~200、>200~300、>400~500 bp的功能基因数较多,占47.17%(25/53)。但有5个推定功能基因的长度超过2 000 bp,有可能是噬菌体结构蛋白的编码基因。总之,噬菌体SWH-Ab-3的功能基因呈现多样性,可编码较为复杂的噬菌体的结构蛋白。运用NCBI的序列比对软件BLASTp进行噬菌体功能基因的ORF注释,结果见表 3,其中20个ORF为假想蛋白(hypothetical protein),33个ORF的功能已知。ORF02(噬菌体裂解酶)和ORF01(穿孔蛋白)是噬菌体的裂解效应相关基因,ORF02是一种溶菌酶,可导致细胞壁破裂而使细菌溶解。ORF01编码的蛋白质能使细菌细胞膜形成空洞,有助于噬菌体基因组核酸注入宿主细菌,同时该蛋白还具有连接噬菌体头部与尾部的功能。结构蛋白ORF45是噬菌体主要衣壳蛋白,与包装蛋白邻近。ORF47-ORF49编码噬菌体尾部相关蛋白,具有收缩和运动功能,该基因簇在噬菌体感染、吸附宿主菌时发挥重要作用。此外,噬菌体DNA复制及调控相关基因主要包括:ORF30(DNA聚合酶)、ORF22(DNA引物酶)、ORF26(DNA连接酶)、ORF20(DNA结合蛋白)、ORF25(DNA解旋酶)等。同时,经过对比分析并没有发现噬菌体SWH-Ab-3中存在毒力因子的编码及转移基因,表明该噬菌体具有较好的生物安全性。
|
| 1:100~200 bp;2:>200~300 bp;3:>300~400 bp;4:>400~500 bp;5:>500~600 bp;6:>600~700 bp;7:>700~800 bp;8:>800~900 bp;9:>900~1 000 bp;10:>1 000~2 000 bp;11:>2 000 bp 图 6 噬菌体SWH-Ab-3推定功能基因的长度分布 |
| ORF | 相关蛋白描述 | 长度/bp | E值 | 登录号 |
| 1 | Putative holin [Acinetobacter phage vB_AbaP_D2] | 336 | 5.00E-73 | AVP40473.1 |
| 2 | Endolysin [Acinetobacter phage vB_AbaP_B5] | 558 | 1.00E-128 | ASN73457.1 |
| 3 | DNA maturase A[Acinetobacter phage vB_ApiP_P1] | 309 | 2.00E-66 | ASN73507.1 |
| 4 | Putative DNA maturase B [Acinetobacter phage Abp1] | 1 938 | 0 | YP_008058244.1 |
| 5 | Hypothetical protein [Acinetobacter phage IME200] | 135 | 3.00E-22 | YP_009216494.1 |
| 6 | Hypothetical protein Abp1_0054 [Acinetobacter phage Abp1] | 204 | 2.00E-36 | YP_008058246.1 |
| 7 | Hypothetical protein Abp1_001 [Acinetobacter phage Abp1] | 207 | 1.00E-43 | YP_008058193.1 |
| 8 | Hypothetical protein Abp1_002 [Acinetobacter phage Abp1] | 282 | 9.00E-61 | YP_008058194.1 |
| 9 | Hypothetical protein Abp1_003 [Acinetobacter phage Abp1] | 510 | 6.00E-120 | YP_008058195.1 |
| 10 | Hypothetical protein Abp1_004 [Acinetobacter phage Abp1] | 375 | 4.00E-87 | YP_008058196.1 |
| 11 | Hypothetical protein vBAbaPPDAB9_45 [Acinetobacter phage vB_AbaP_PD-AB9] | 234 | 2.00E-49 | YP_009189868.1 |
| 12 | Hypothetical protein Abp1_006 [Acinetobacter phage Abp1] | 597 | 1.00E-147 | YP_008058198.1 |
| 13 | Hypothetical protein Abp1_007 [Acinetobacter phage Abp1] | 180 | 4.00E-37 | YP_008058199.1 |
| 14 | Hypothetical protein Abp1_008 [Acinetobacter phage Abp1] | 117 | 7.00E-18 | YP_008058200.1 |
| 15 | Hypothetical protein Abp1_009 [Acinetobacter phage Abp1] | 399 | 3.00E-93 | YP_008058201.1 |
| 16 | Hypothetical protein Abp1_0010 [Acinetobacter phage Abp1] | 489 | 1.00E-116 | YP_008058202.1 |
| 17 | Hypothetical protein vBAbaPPDAB9_40 [Acinetobacter phage vB_AbaP_PD-AB9] | 435 | 4.00E-103 | YP_009189863.1 |
| 18 | Hypothetical protein vBAbaPPDAB9_39 [Acinetobacter phage vB_AbaP_PD-AB9] | 168 | 3.00E-30 | YP_009189862.1 |
| 19 | Hypothetical protein Abp1_0014 [Acinetobacter phage Abp1] | 219 | 2.00E-44 | YP_008058206.1 |
| 20 | DNA binding protein [Acinetobacter phage vB_AbaP_AS11] | 210 | 3.00E-36 | AQN32666.1 |
| 21 | HNH endonuclease [Acinetobacter phage vB_AbaP_B1] | 450 | 2.00E-86 | ASN73324.1 |
| 22 | DNA primase/helicase [Acinetobacter phage vB_AbaP_B5] | 708 | 4.00E-176 | ASN73427.1 |
| 23 | Hypothetical protein Abp1_0018 [Acinetobacter phage Abp1] | 318 | 2.00E-70 | YP_008058210.1 |
| 24 | Hypothetical protein Abp1_0019 [Acinetobacter phage Abp1] | 237 | 3.00E-51 | YP_008058211.1 |
| 25 | Putative DNA helicase [Acinetobacter phage Abp1] | 1 299 | 0 | YP_008058212.1 |
| 26 | Putative DNA ligase [Acinetobacter phage WCHABP5] | 981 | 0 | ARQ94892.1 |
| 27 | Putative HNH endonuclease [Acinetobacter phage phiAB1] | 447 | 5.00E-104 | YP_009189356.1 |
| 28 | DNA polymerase Ⅰ [Acinetobacter phage Abp1] | 861 | 0 | YP_008058215.1 |
| 29 | Putative HNH endonuclease [Acinetobacter phage WCHABP5] | 438 | 1.00E-98 | ARQ94894.1 |
| 30 | Putative DNA polymerase [Acinetobacter phage WCHABP5] | 1 461 | 0 | ARQ94895.1 |
| 31 | Putative HNH endonuclease [Acinetobacter phage WCHABP5] | 480 | 4.00E-117 | ARQ94896.1 |
| 32 | Hypothetical protein vBAbaPPDAB9_27 [Acinetobacter phage vB_AbaP_PD-AB9] | 891 | 0 | YP_009189850.1 |
| 33 | Zn-ribbon domain containing protein [Acinetobacter phage vB_AbaP_AS12] | 120 | 7.00E-20 | APW79810.1 |
| 34 | Putative DNA exonuclease [Acinetobacter phage Abp1] | 957 | 0 | YP_008058221.1 |
| 35 | Phage-associated homing endonuclease [Acinetobacter phage Abp1] | 438 | 5.00E-105 | YP_008058222.1 |
| 36 | DNA endonuclease Ⅶ [Acinetobacter phage vB_AbaP_B1] | 441 | 4.00E-105 | ASN73337.1 |
| 37 | Phosphoestherase with HTH domain [Acinetobacter phage vB_AbaP_AS11] | 936 | 0 | AQN32682.1 |
| 38 | Putative dNMP kinase [Acinetobacter phage vB_AbaP_AS12] | 651 | 6.00E-156 | APW79816.1 |
| 39 | Phage-associated RNA polymerase [Acinetobacter phage Abp1] | 2 418 | 0 | YP_008058226.1 |
| 40 | Hypothetical protein Abp1_0035 [Acinetobacter phage Abp1] | 198 | 2.00E-40 | YP_008058227.1 |
| 41 | Structural protein [Acinetobacter phage phiAB1] | 252 | 3.00E-53 | YP_009189369.1 |
| 42 | Head-to-tail joining protein [Acinetobacter phage Abp1] | 1 557 | 0 | YP_008058229.1 |
| 43 | Putative scaffolding protein [Acinetobacter phage AB3] | 798 | 4.00E-177 | YP_008060144.1 |
| 44 | Scaffolding protein [Acinetobacter phage vB_AbaP_B5] | 141 | 3.00E-24 | ASN73446.1 |
| 45 | Phage capsid and scaffold protein [Acinetobacter phage Abp1] | 1 032 | 0 | YP_008058231.1 |
| 46 | Hypothetical protein Abp1_0040 [Acinetobacter phage Abp1] | 186 | 2.00E-33 | YP_008058232.1 |
| 47 | Tail needle protein [Acinetobacter phage vB_AbaP_AS11] | 294 | 1.00E-50 | AQN32691.1 |
| 48 | Putative tail tubular protein A [Acinetobacter phage Abp1] | 561 | 9.00E-134 | YP_008058234.1 |
| 49 | Tail tube [Acinetobacter phage Abp1] | 2 292 | 0 | YP_008058235.1 |
| 50 | Putative internal virion protein B [Acinetobacter phage SH-Ab 15519] | 672 | 4.00E-158 | APD19443.1 |
| 51 | Internal virion protein B [Acinetobacter phage vB_AbaP_AS11] | 2 886 | 0 | AQN32695.1 |
| 52 | Putative internal virion protein [Acinetobacter phage Abp1] | 3 099 | 0 | YP_008058238.1 |
| 53 | Tail fiber [Acinetobacter phage phiAB6] | 2 100 | 0 | YP_009288671.1 |
3 讨论
近年来,鲍曼不动杆菌的耐药性日趋严重,多耐药甚至泛耐药菌株不断增多,已成为院内感染的主要条件致病菌而备受高度关注[1, 11]。目前,鲍曼不动杆菌对亚胺培南等碳青霉烯类抗菌药物的耐药率已达70%左右,临床抗感染治疗时可供选择的抗菌药物十分有限[12],尤其是ICU病房的重症感染患者,往往无药可用或抗感染治疗效果不佳,患者病死率高[13]。因此,开发和建立新的抗菌药物、制剂或方法成为全球抗感染领域关注的焦点。
裂解性噬菌体是一类能感染、杀灭细菌的天然病毒。其对宿主菌的感染和裂解作用具有特异性,较少出现耐药问题,也不会造成机体的微生态失调,开发周期短且成本低,使得噬菌体疗法(phage therapy)成为抗感染治疗的研究热点。近年来,噬菌体制剂在农牧业生产、食品安全和抗感染治疗方面的研究十分活跃[14]。如比利时批准了应用噬菌体混合制剂(鸡尾酒疗法)治疗铜绿假单胞菌、金黄色葡萄球菌所致的烧伤创面混合感染[15]。但噬菌体对细菌的裂解作用具有严格的宿主特异性,导致其裂菌谱较窄,单一噬菌体制剂可能不满足临床治疗的要求,这就需要分离获得针对某种病原菌不同亚型、不同亚种的特异性噬菌体,制备成“鸡尾酒”的混合制剂,以拓宽噬菌体的杀菌谱。
本研究从医院污水中成功分离1株鲍曼不动杆菌的特异裂解性噬菌体SWH-Ab-3,其头部呈典型的正多面体对称结构,头部直径(约70 nm)与其尾部长度(约80 nm)相当。由于本研究未对噬菌体DNA的单双链性质进行验证和系统发育分析,按照噬菌体分类学标准[16],初步判断该噬菌体应归属于肌尾噬菌体科(Myoviridae,尾部长度与头部直径相等),明显不同于本室[9]和其他学者[17]分离的鲍曼不动杆菌长尾噬菌体(Siphoviridae,尾部长度是头部直径的2倍左右)。结果表明,在自然界中存在针对同种细菌的不同种类和形态的噬菌体,正是由于噬菌体的多样性,增加了噬菌体分离筛选的复杂性,但也为“鸡尾酒”型混合噬菌体制剂的制备提供了可能性。
生物学特性是评价噬菌体裂解细菌效能的重要参考指标,对噬菌体的分离纯化、结构蛋白制备、基因组提取等具有指导意义。噬菌体SWH-Ab-3按照最佳感染复数(0.1)行一步生长曲线试验,测得其潜伏期为40 min,爆发时间为50 min,裂解量为31.4 PFU/cell,裂菌能力较强,但与文献[18-19]分离的鲍曼不动杆菌噬菌体比较,其裂解能力较弱。裂解谱测定发现噬菌体SWH-Ab-3在随机选择的42株鲍曼不动杆菌中仅能裂解6株临床菌株,裂解谱较窄,表明该株噬菌体对宿主菌具有高度的特异性。但临床分离的鲍曼不动杆菌具有多种亚型,单一噬菌体制剂的抗菌作用可能有限,提示多种噬菌体混合的“鸡尾酒”型噬菌体制剂是未来噬菌体临床抗感染治疗应用的趋势[20]。另外,通过温度和pH耐受实验发现,噬菌体SWH-Ab-3具有良好的酸碱稳定性及较强的温度适应能力,为噬菌体制剂的制备、保存及临床应用提供了可靠保证。
目前,GenBank收录的鲍曼不动杆菌噬菌体的基因组从30 kb到170 kb,表明鲍曼不动杆菌噬菌体存在多样性。通过对噬菌体SWH-Ab-3基因组进行DNA测序和同源性比对分析发现,噬菌体SWH-Ab-3基因组全长为41 730 bp,G+C%含量为39.38%,其长度与同属肌尾噬菌体的YMC-13-01-C62(44 844 bp,NC_024785)的基因组大小相似。BLASTn比对分析发现,噬菌体SWH-Ab-3与Abp1和vB_AbaP_B3的序列相似性在94.0%以上,但Abp1和vB_AbaP_B3的基因组长度、G+C%含量及形态特征均与SWH-Ab-3不同。因此,噬菌体SWH-Ab-3是1株新分离的裂解性鲍曼不动杆菌噬菌体。
通过对噬菌体SWH-Ab-3的全基因组进行ORF预测和BLASTp比对分析,预测出33个具有已知功能的基因,主要包括噬菌体结构蛋白基因、裂解相关基因及核酸复制表达相关基因。SWH-Ab-3基因组中存在假定的裂解酶(endolysin)及穿孔蛋白(holin)编码基因,分别由ORF02和ORF01编码表达。裂解酶作用于细菌细胞壁的肽聚糖,是噬菌体可接触细菌的主要功能分子,但缺乏引导其转移的信号肽,不能透过宿主菌的胞膜直接作用于细胞壁。穿孔蛋白是触发细菌裂解的“分子定时器”,在特定时间点能在细胞质膜上形成跨膜通道,诱导裂解酶到达作用靶位发挥裂解作用。由此可见,噬菌体SWH-Ab-3可能通过上述高效、特异性的“穿孔蛋白-裂解酶”二元系统的协调作用,实现对宿主菌的裂解[21]。后续可对其裂解酶基因进行基因工程表达的研究,制备裂解酶相关生物制剂,为抗感染治疗提供新的思路[22]。同时,对比分析并没有发现该噬菌体存在tRNA、毒力因子的生成及转移基因,说明噬菌体在翻译过程中利用的是宿主菌的tRNA,充分证明了其生物安全性,为开发噬菌体制剂奠定了一定基础。
| [1] | DOI Y, MURRAY G L, PELEG A Y. Acinetobacter baumannii: evolution of antimicrobial resistance-treatment options[J]. Semin Respir Crit Care Med, 2015, 36(1): 85–98. DOI:10.1055/s-0034-1398388 |
| [2] | KARAGEORGOPOULOS D E, FALAGAS M E. Current control and treatment of multidrug-resistant Acinetobacter baumannii infections[J]. Lancet Infect Dis, 2008, 8(12): 751–762. DOI:10.1016/S1473-3099(08)70279-2 |
| [3] |
朱育玮, 李玉保, 王守荣, 等. 噬菌体及其治疗细菌感染的研究进展[J].
中国畜牧兽医, 2015, 42(3): 769–773.
ZHU Y W, LI Y B, WANG S R, et al. Research progress on bacteriophage and the therapy in bacterial infection[J]. China Anim Husband Vet Med, 2015, 42(3): 769–773. DOI:10.11843/j.issn.1671-7236.2015.03.041 |
| [4] |
王盛, 童贻刚. 噬菌体治疗研究进展[J].
微生物学通报, 2009, 36(7): 1019–1024.
WANG S, TONG Y G. Recent advance in bacteriophage therapy[J]. Microbiol China, 2009, 36(7): 1019–1024. DOI:10.3969/j.issn.1671-6027.2002.04.004 |
| [5] | MATSUZAKI S, RASHEL M, UCHIYAMA J, et al. Bacteriophage therapy: A revitalized therapy against bacterial infectious diseases[J]. J Infect Chemother, 2005, 11(5): 211–219. DOI:10.1007/s10156-005-0408-9 |
| [6] | VIERTEL T M, RITTER K, HORZ H P. Viruses versus bacteria-novel approaches to phage therapy as a tool against multidrug-resistant pathogens[J]. J Antimicrob Chemother, 2014, 69(9): 2326–2336. DOI:10.1093/jac/dku173 |
| [7] | KUTTER E, SULAKVELIDZE A. Bacteriophages: biology and applications[M]. Boca Raton: CRC Press, 2004. |
| [8] | MERABISHVILI M, VANDENHEUVEL D, KROPINSKI A M, et al. Characterization of newly isolated lytic bacteriophages active against Acinetobacter baumannii[J]. PLoS ONE, 2014, 9(8): e104853. DOI:10.1371/journal.pone.0104853.eCollection2014 |
| [9] |
罗娟, 张然, 吴柳, 等. 鲍曼不动杆菌噬菌体SWH-Ab-1分离鉴定及其重要功能基因的生物信息学分析[J].
第三军医大学学报, 2018, 40(1): 23–30.
LUO J, ZHANG R, WU L, et al. Isolation and identification of phage SWH-Ab-1 against Acinetobacter baumannii and bioinformatic analysis of its major functional genes[J]. J Third Mil Med Univ, 2018, 40(1): 23–30. DOI:10.16016/j.1000-5404.201708010 |
| [10] |
邢少贞, 张湘莉兰, 舒鹏, 等. 粪肠球菌噬菌体vB_E.faecalis_IME196的生物学特性及其全基因组分析[J].
微生物学通报, 2016, 43(9): 2040–2048.
XING S Z, ZHANG X L L, SHU P, et al. Characterization and complete genome sequence analysis of Enterococcus faecalis bacteriophage VBE.faecalisIME196[J]. Microbiol China, 2016, 43(9): 2040–2048. DOI:10.13344/j.microbiol.china.151053 |
| [11] | HUANG H, DONG Y, YANG Z L, et al. Complete sequence of pABTJ2, a plasmid from Acinetobacter baumannii MDR-TJ, carrying many phage-like elements[J]. Genom Proteom Bioinf, 2014, 12(4): 172–177. DOI:10.1016/j.gpb.2014.05.001 |
| [12] | MENDES J J, LEANDRO C, MOTTOLA C, et al. In vitro design of a novel lytic bacteriophage cocktail with therapeutic potential against organisms causing diabetic foot infections[J]. J Med Microbiol, 2014, 63(Pt_8): 1055–1065. DOI:10.1099/jmm.0.071753-0 |
| [13] | JIN J, LI Z J, WANG S W, et al. Isolation and characterization of ZZ1, a novel lytic phage that infects Acinetobacter baumannii clinical isolates[J]. BMC Microbiol, 2012, 12: 156. DOI:10.1186/1471-2180-12-156 |
| [14] | LU T K, KOERIS M S. The next generation of bacteriophage therapy[J]. Curr Opin Microbiol, 2011, 14(5): 524–531. DOI:10.1016/j.mib.2011.07.028 |
| [15] | VERBEKEN G, DE VOS D, VANEECHOUTTE M, et al. European regulatory conundrum of phage therapy[J]. Future Microbiol, 2007, 2(5): 485–491. DOI:10.2217/17460913.2.5.485 |
| [16] | MATSUZAKI S, RASHEL M, UCHIYAMA J, et al. Bacteriophage therapy: a revitalized therapy against bacterial infectious diseases[J]. J Infect Chemother, 2005, 11(5): 211–219. DOI:10.1007/s10156-005-0408-9 |
| [17] | JEON J, D'SOUZA R, PINTO N, et al. Complete genome sequence of the siphoviral bacteriophage Βϕ-R3177, which lyses an OXA-66-producing carbapenem-resistant Acinetobacter baumannii isolate[J]. Arch Virol, 2015, 160(12): 3157–3160. DOI:10.1007/s00705-015-2604-y |
| [18] |
余丹静, 史红艳, 王丹, 等. 鲍曼不动杆菌噬菌体生物学特性的研究[J].
微生物学杂志, 2013, 33(2): 17–23.
YU D J, SI H Y, WANG D, et al. Biologic characteristics of Bacteriophages specific to Acinetobacter baumannii[J]. J Microbiol, 2013, 33(2): 17–23. DOI:10.3969/j.issn.1005-7021.2013.02.004 |
| [19] |
彭帆, 童贻刚, 柏长青. 多耐药鲍曼不动杆菌噬菌体IME-AB6的分离、生物特性及保存方法研究[J].
生物技术通讯, 2013, 24(5): 641–644.
PENG F, TONG Y G, BO C Q. Isolation, biological characteristics and preservation methods of multidrug-resistant Acinetobacter baumannii bacteriophage IME-AB6[J]. Lett Biotech, 2013, 24(5): 641–644. DOI:10.3969/j.issn.1009-0002.2013.05.010 |
| [20] |
马翔宇, 熊鸿燕, 朱才众, 等. 新型多价大肠埃希菌噬菌体285P生物学特性研究[J].
微生物学杂志, 2008, 28(5): 12–15.
MA X Y, XIONG H Y, ZHU C Z, et al. Biological characteristics of a neo-polyvalent e.coli phage 285P[J]. J Microbiol, 2008, 28(5): 12–15. DOI:10.3969/j.issn.1005-7021.2008.05.003 |
| [21] | WON G, HAJAM I A, LEE J H. Improved lysis efficiency and immunogenicity of Salmonella ghosts mediated by co-expression of λ phage holin-endolysin and φX174 gene E[J]. Sci Rep, 2017, 7: 45139. DOI:10.1038/srep45139 |
| [22] | CUI Z L, SHEN W B, WANG Z, et al. Complete genome sequence of Klebsiella pneumoniae phage JD001[J]. J Virol, 2012, 86(24): 13843. DOI:10.1128/JVI.02435-12 |



