2. 400037 重庆,陆军军医大学(第三军医大学)第二附属医院口腔科
2. Department of Stomatology, Second Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400037, China
口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)是口腔最常见的恶性肿瘤,在全世界发病率最高的癌症中排名第6位[1]。由于OSCC进展快,且在进展过程中通常不会出现疼痛或其他明显症状,常常在转移到颈部淋巴结时才被发现[2]。尽管近年关于OSCC的治疗手段不断取得进展,但患者的长期生存率却没有显著改善[3],该病对人类健康构成了极大的威胁。
OSCC患者最容易发生淋巴结转移,约50%的患者在诊断疾病时即可检测到淋巴结受累[4]。同时,发生淋巴转移的患者预后状况明显差于无淋巴转移者,淋巴转移患者中能达到5年生存的仅25%~40%,而无淋巴转移的患者可达近90%[5]。目前OSCC淋巴转移的分子机制尚不确切,极大地限制了对该病的诊治能力。而肿瘤淋巴转移的过程是一个多基因协同的过程,涉及多个分子和信号通路[6]。借助高通量的基因表达谱芯片能够同时对万级及以上的基因水平进行检测,完成对多个基因表达情况的分析研究[7],但该方法在OSCC淋巴转移中的研究应用还相对较少。本研究利用OSCC患者的表达谱芯片GSE2280,运用生物信息学方法筛选及分析和淋巴转移发生有关的基因,并基于肿瘤基因图谱数据库(the cancer genome atlas,TCGA)进行基因突变的生存分析比较,为OSCC发生淋巴转移及其与预后关联的分子机制研究,以及OSCC的临床诊治方案的深入研发提供参考。
1 资料与方法 1.1 基因芯片数据从基因表达数据库(gene expression omnibus,GEO, http://www.ncbi.nlm.nih.gov/geo/)中下载获取本研究中mRNA表达谱芯片GSE2280[8],包含22例OSCC患者肿瘤原发部位标本的芯片数据,其中有5个病例发生淋巴转移。基于GPL96平台(美国Affymetrix公司人类基因组U133A基因芯片)进行检测。
1.2 筛选差异表达基因使用GEO2R(http://www.ncbi.nlm.nih.gov/geo/geo2r)筛选OSCC患者中发生淋巴转移与未发生淋巴转移在肿瘤原发部位标本的芯片数据之间的表达差异基因(differentially expressed genes,DEGs)。GEO2R是基于limma R软件包的对1个GEO系列中2个或多个数据集进行比较以识别出DEGs的在线分析工具。本研究在筛选DEGs过程中,筛选条件为|log2(FC)|>1(FC指表达改变倍数)。
1.3 芯片数据生物信息学分析及筛选使用DAVID在线数据库(https://david.ncifcrf.gov/)对DEGs进行基因本体(gene ontology,GO)注释及京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)通路富集分析,其中GO注释分析包括生物过程、细胞组成和分子功能3个部分。在GO注释和KEGG通路分析中,均取数据库中结果P < 0.05为显著性阈值。利用STRING在线工具[9]以综合评分>0.15评估蛋白质之间的相互作用,并采用Cytoscape软件(3.7.1版)[10]进行蛋白相互作用网络可视化处理。采用CytoHubba插件[11]进行DEGs中上调基因和下调基因的枢纽基因鉴定,根据Maximal clique centrality(MCC)的拓扑分析方法,在符合MCC得分≥10的前提下,选取了上调和下调的DEGs中MCC分数最高的前10位基因,确定为候选的枢纽基因[12]。并将筛选出的枢纽基因用于TCGA数据库口腔肿瘤病例的生存分析。
1.4 统计学分析运用GEO2R工具,使用Benjamini & Hochberg法分析GSE2280表达谱芯片数据,采用SAM法筛选芯片数据中DEGs。在DAVID数据库中,通过Bonferroni校正法和Bootstrap法等进行GO分析的检验,利用Fisher精确概率检验和基因富集分析等方法进行KEGG分析。在Cytoscape中,选取CytoHubba插件中MCC拓扑分析算法进行数据分析。在TCGA数据库网站使用在线分析工具,采用Kaplan-Meier法进行生存分析,以P < 0.05为差异有统计学意义。
2 结果 2.1 DEGs的筛选根据|log2(FC)|>1的筛选条件,筛选出OSCC发生淋巴转移患者表达谱芯片的DEGs。结果共有131个DEGs,其中上调基因有74个,下调基因有57个。
2.2 GO分析及KEGG通路分析基于DAVID数据库对DEGs进行GO分析与KEGG通路分析。在表达上调和下调基因中,满足P < 0.05前提下,筛选出P值最小的前10项GO注释结果(图 1),其中横坐标为P值取-log10的值。上调基因富集在消化降解(GO:0007586)、中间丝细胞骨架(GO:0045111)和受损的DNA结合(GO:0003684)等,下调基因富集在细胞外基质组成(GO:0030198)、胶原三聚体(GO:0005581)和细胞外基质结构成分(GO:0005201)等。
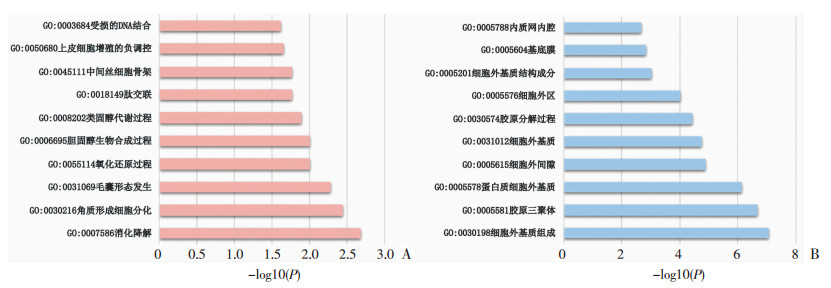
|
| A:上调基因;B:下调基因 图 1 DEGs的GO注释分析 |
在KEGG通路分析中,根据P < 0.05筛选出的结果见图 2,提示上调基因主要富集于胰腺分泌(hsa04972)和代谢途径(hsa01100)中,下调基因主要富集于细胞外基质受体相互作用(hsa04512)及蛋白质消化吸收(hsa04974)中。
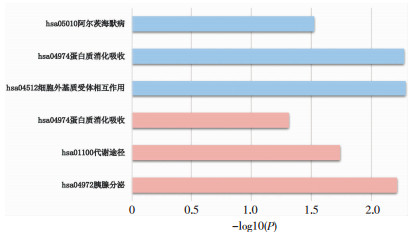
|
| 红色:上调基因;蓝色:下调基因 图 2 DEGs的KEGG通道富集分析 |
2.3 蛋白质互作网络分析及枢纽基因筛选
运用STRING分析131个DEGs的蛋白相互作用(图 3A),共有122个节点及390条关系满足条件,其中每1个节点代表DEGs对应的蛋白,节点间连线代表蛋白之间相互作用关系,连线颜色由橙至蓝,越接近蓝色表示蛋白间联系越强。74个上调基因的蛋白相互作用结果(图 3B)显示有70个节点及109条关系;57个下调基因的蛋白相互作用结果(图 3C)显示有52个节点及142条关系。
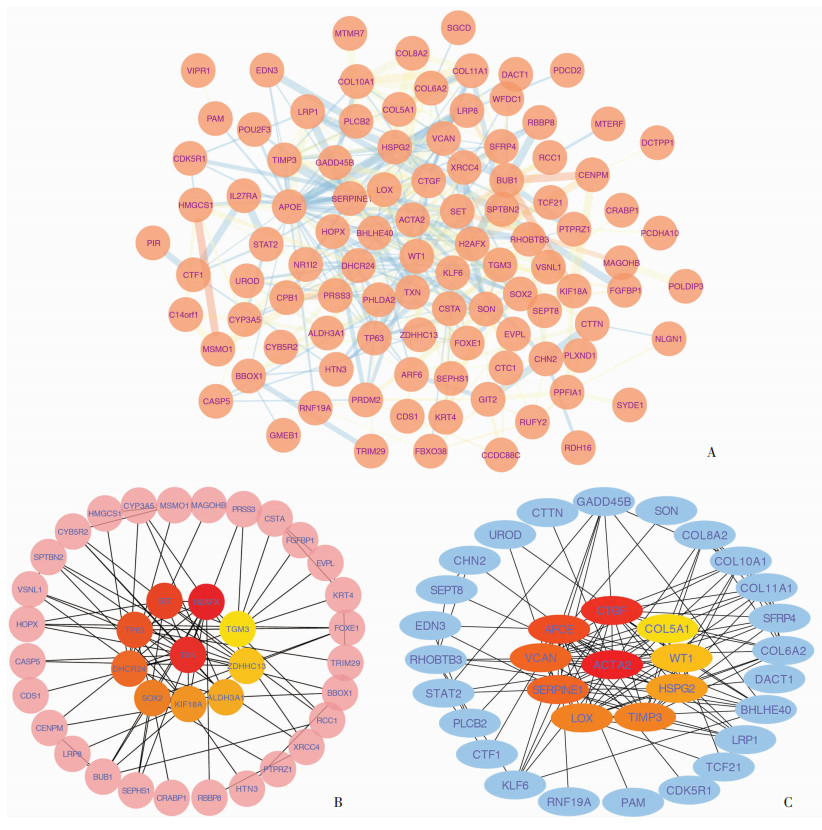
|
| A:所有DEGs;B:上调的DEGs;C:下调的DEGs 图 3 差异基因的蛋白相互作用网络 |
基于蛋白质相互作用网络分析结果,在Cytoscape中使用CytoHubba插件,根据筛选条件确定出DEGs中的枢纽基因。MCC得分最高的前10个基因由黄色至红色表示,颜色越红代表MCC分数越高,更倾向于是关键蛋白。因此,在上调基因中(图 3B)确定的枢纽基因分别是:H2AFX、TXN、SET、TP63、DHCR24、SOX2、KIF18A、ALDH3A1、ZDHHC13、TGM3,其中DHCR24和ALDH3A1参与到代谢途径的通路中。在下调基因中(图 3C)确定的枢纽基因分别是:ACTA2、CTGF、APOE、SERPINE1、VCAN、LOX、TIMP3、HSPG2、WT1、COL5A1,其中HSPG2和COL5A1参与到细胞外基质受体相互作用通路中,COL5A1还参与了蛋白质消化吸收的通路,APOE参与到阿尔茨海默病的通路。
2.4 TCGA数据库中验证枢纽基因的临床意义在TCGA数据库中选取口腔肿瘤病例,将筛选出来的枢纽基因用于生存分析。仅TP63和VCAN两个基因突变有相对足够的病例数量进行生存分析比较。结果显示:存在TP63突变的患者其生存时间较其他患者更短(P=0.717),而VCAN突变患者的生存时间较其他患者长(P=0.568),但差异均不具有统计学意义(图 4)。
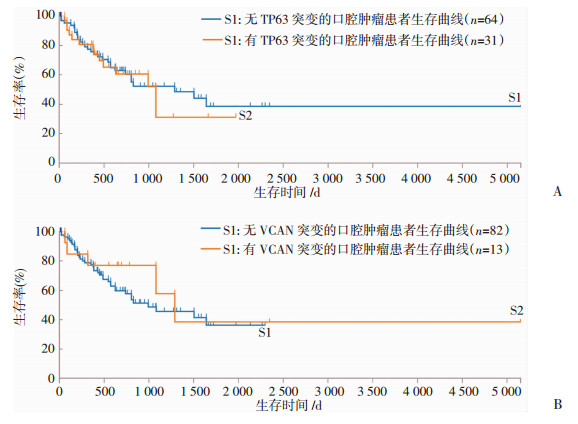
|
| 图 4 TP63(A)和VCAN(B)基因突变对口腔肿瘤患者生存情况的影响 |
3 讨论
肿瘤转移是指肿瘤细胞通过血流或淋巴管扩散到体内的新区域。OSCC的特点在于通过引流淋巴管转移到局部淋巴结中。临床中OSCC患者发生淋巴转移的比例较高,有些患者即便在出现原发灶时没有淋巴结转移,在以后也会发生。并且OSCC的淋巴转移与不良预后的关系密切[8]。这给OSCC的临床治疗带来了很大难度,也加大了对患者预后评估的难度。本研究基于OSCC患者肿瘤原发部位标本的芯片数据,分析其中发生淋巴转移的病例和未发生淋巴转移的病例在基因表达上的差异,得到131个符合筛选条件的差异表达基因,并分别对其中上调基因和下调基因进行了生物信息学分析及枢纽基因鉴定。
在表达上调的枢纽基因中,DHCR24和ALDH3A1参与到代谢途径的通路,但目前鲜有这两个基因与OSCC淋巴转移有关的报道。其中,DHCR24可编码一种黄素腺嘌呤二核苷酸依赖性氧化还原酶,在胆固醇生物合成过程中催化甾醇中间体的还原。PEDRO等[13]通过对不同的氧化应激相关基因表达情况的分析,报道了该基因是OSCC发病的关键基因。DAI等[14]发现DHCR24可加重子宫内膜癌的癌症侵袭。ALDH3A1编码的酶形成胞质同二聚体,优先氧化芳香族和中链醛底物。YAN等[15]通过开展前列腺肿瘤异种移植的动物实验发现ALDH3A1可以促进前列腺肿瘤细胞的肺转移的发生。上述文献报道结合本研究结果,提示这两种基因在OSCC发生淋巴转移机制的探讨中具有潜在的研究价值。
在表达下调的枢纽基因中,COL5A1参与到细胞外基质受体相互作用通路及蛋白质消化吸收的通路中,HSPG2参与到细胞外基质受体相互作用通路中,APOE参与到阿尔茨海默病的通路中。COL5A1基因的产物与Ⅺ胶原蛋白密切相关。LI等[16]利用GEO数据库中的芯片数据分析了正常组织与OSCC组织中的DEGs,评估了COL5A1可能是OSCC发生的关键基因之一。HSPG2是编码串珠素蛋白,在多种生物活动中发挥重要作用,可以促进生长因子活动、刺激内皮细胞生长和再生,能调节肿瘤的血管生成、增殖、分化和侵袭[17]。因该基因编码的串珠素在肿瘤浸润和转移过程中发生降解,被应用于检测前列腺癌组织浸润情况的研究[18]。APOE编码的蛋白质是乳糜微粒的主要载脂蛋白。结合特定的肝脏和外周细胞受体,对富含甘油三酯的脂蛋白成分的正常分解代谢至关重要。JAYAKAR等[19]发现APOE可以影响细胞胆固醇以及随后对细胞信号传导,引起侵袭相关蛋白的表达发生变化,在OSCC肿瘤侵袭中起着重要作用。提示上述基因在OSCC患者中可能作为潜在的临床预测淋巴转移发生的标志物,值得临床结合基础更进一步进行研究。
基于OSCC发生淋巴转移与不良预后具有直接关联[8],本研究利用TCGA公共数据库资源,对候选枢纽基因与口腔肿瘤患者生存率情况进行了探讨,以在临床标本中间接验证与OSCC淋巴转移具有潜在关联的候选基因。在TCGA数据库对候选枢纽基因进行检验,只有上调基因TP63和下调基因VCAN有足够突变患者做出生存曲线分析。其中TP63在上皮细胞发育和各种癌症的进展中起着主要调节作用。ZHUANG等[20]发现在OSCC组织和细胞系中,TP63的主要亚型明显高于正常对照组,其表达与OSCC患者的病理分化、淋巴结转移和临床分期密切相关。ABRAM等[21]应用自体荧光成像和细胞学芯片技术对范可尼贫血患者口腔潜在恶性疾病进行风险分层研究,发现TP63突变是口腔恶性疾病的重要危险因素。在对TP63突变进行生存分析的研究中,有31例口腔肿瘤患者发生TP63突变,64例无TP63突变,尽管两组间生存率的差异不具有统计学意义,但TP63突变患者出现了生存率降低的趋势。我们推测这可能与TP63影响口腔肿瘤的转移有关,而发生肿瘤转移患者的生存情况普遍较差,可以解释生存率降低的趋势;又鉴于TP63与生存情况之间只存在潜在的间接关系,可以解释TP63突变导致的生存率差异并不显著。故后续研究有必要进一步探讨TP63与OSCC淋巴转移的直接关联及具体作用机制。另一个基因VCAN,其编码的蛋白质是细胞外基质的主要成分。该蛋白参与细胞粘附、增殖、迁移和血管生成,并在组织形态发生和维持中起到中心作用。近年的研究发现VCAN可以促进多种肿瘤,如乳腺癌[22]、卵巢癌[23]等的侵袭、生长及迁移。并且在肝细胞肝癌[24]和肾脏肿瘤[25]中与不良预后有较大关系。在本研究中,VCAN突变的患者生存率与未发生突变者的差异无统计学意义,其中发生VCAN突变的口腔肿瘤仅13例,未发生突变者82例。我们推测是因为VCAN突变组病例较少,且两组间病例数据量差距较大,故生存分析结果并不理想,提示在后续研究一方面要扩大病例样本量进行生存分析,以得到更具科学性的结果;另一方面还需要结合OSCC临床标本完成进一步的验证实验,明确其与淋巴转移发生的关联。
综上所述,本研究应用生物信息学的方法对OSCC淋巴转移进行了综合分析,并对筛选基因进行了初步的验证。从分子水平层面为OSCC发生淋巴转移及其与预后关联的机制提供了理论参考,有助于OSCC淋巴转移机制的进一步研究。并为OSCC的靶向治疗和预后判断等提供了潜在的研究方向,有利于临床诊治方案的深入研发和探讨。
| [1] | WARNAKULASURIYA S. Living with oral cancer: epidemi-ology with particular reference to prevalence and life-style changes that influence survival[J]. Oral Oncol, 2010, 46(6): 407–410. DOI:10.1016/j.oraloncology.2010.02.015 |
| [2] | SEVERINO P, OLIVEIRA L S, ANDREGHETTO F M, et al. Small RNAs in metastatic and non-metastatic oral squamous cell carcinoma[J]. BMC Med Genomics, 2015, 8: 31. DOI:10.1186/s12920-015-0102-4 |
| [3] | LAIMER K, BLASSNIG N, SPIZZO G, et al. Prognostic significance of 14-3-3sigma expression in oral squamous cell carcinoma (OSCC)[J]. Oral Oncol, 2009, 45(2): 127–134. DOI:10.1016/j.oraloncology.2008.04.006 |
| [4] | TORUNER G A, ULGER C, ALKAN M, et al. Association between gene expression profile and tumor invasion in oral squamous cell carcinoma[J]. Cancer Genet Cytogenet, 2004, 154(1): 27–35. DOI:10.1016/j.cancergencyto.2004.01.026 |
| [5] | HSIA K T, LIU C J, MAR K, et al. Impact of apurinic/apyrimidinic endonuclease 1/redox factor-1 on treatment response and survival in oral squamous cell carcinoma[J]. Head Neck, 2016, 38(4): 550–559. DOI:10.1002/hed.23927 |
| [6] | TATANGELO F, DI MAURO A, SCOGNAMIGLIO G, et al. Posterior HOX genes and HOTAIR expression in the proximal and distal colon cancer pathogenesis[J]. J Transl Med, 2018, 16(1): 350. DOI:10.1186/s12967-018-1725-y |
| [7] | BELACEL N, WANG Q, CUPERLOVIC-CULF M. Clustering methods for microarray gene expression data[J]. OMICS, 2006, 10(4): 507–531. DOI:10.1089/omi.2006.10.507 |
| [8] | O'DONNELL R K, KUPFERMAN M, WEI S J, et al. Gene expression signature predicts lymphatic metastasis in squamous cell carcinoma of the oral cavity[J]. Oncogene, 2005, 24(7): 1244–1251. DOI:10.1038/sj.onc.1208285 |
| [9] | SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017: quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Res, 2017, 45(D1): D362–D368. DOI:10.1093/nar/gkw937 |
| [10] | KOHL M, WIESE S, WARSCHEID B. Cytoscape: software for visualization and analysis of biological networks[J]. Methods Mol Biol, 2011, 696: 291–303. DOI:10.1007/978-1-60761-987-1_18 |
| [11] | CHIN C H, CHEN S H, WU H H, et al. CytoHubba: identifying hub objects and sub-networks from complex interactome[J]. BMC Syst Biol, 2014, 8(Suppl 4): S11. DOI:10.1186/1752-0509-8-S4-S11 |
| [12] | LIU H, LI J J, YANG Y, et al. Alterations of 63 hub genes during lingual carcinogenesis in C57BL/6J mice[J]. Sci Rep, 2018, 8(1): 12626. DOI:10.1038/s41598-018-31103-3 |
| [13] | PEDRO N F, BISELLI J M, MANIGLIA J V, et al. Candidate biomarkers for oral squamous cell carcinoma: differential expression of oxidative stress-related genes[J]. Asian Pac J Cancer Prev, 2018, 19(5): 1343–1349. DOI:10.22034/APJCP.2018.19.5.1343 |
| [14] | DAI M, ZHU X L, LIU F, et al. Cholesterol synthetase DHCR24 induced by insulin aggravates cancer invasion and progesterone resistance in endometrial carcinoma[J]. Sci Rep, 2017, 7: 41404. DOI:10.1038/srep41404 |
| [15] | YAN J, DE MELO J, CUTZ J C, et al. Aldehyde dehydrogenase 3A1 associates with prostate tumorigenesis[J]. Br J Cancer, 2014, 110(10): 2593–2603. DOI:10.1038/bjc.2014.201 |
| [16] | LI G, LI X, YANG M, et al. Prediction of biomarkers of oral squamous cell carcinoma using microarray technology[J]. Sci Rep, 2017, 7: 42105. DOI:10.1038/srep42105 |
| [17] | GRINDEL B J, MARTINEZ J R, TELLMAN T V, et al. Matrilysin/MMP-7 cleavage of Perlecan/HSPG2 complexed with semaphorin 3A supports FAK-mediated stromal invasion by prostate cancer cells[J]. Sci Rep, 2018, 8(1): 7262. DOI:10.1038/s41598-018-25435-3 |
| [18] | GRINDEL B, LI Q L, ARNOLD R, et al. Correction: Perlecan/HSPG2 and matrilysin/MMP-7 as indices of tissue invasion: tissue localization and circulating perlecan fragments in a cohort of 288 radical prostatectomy patients[J]. Oncotarget, 2016, 7(37): 60775. DOI:10.18632/oncotarget.11976 |
| [19] | JAYAKAR S K, LOUDIG O, BRANDWEIN-GENSLER M, et al. Apolipoprotein E promotes invasion in oral squamous cell carcinoma[J]. Am J Pathol, 2017, 187(10): 2259–2272. DOI:10.1016/j.ajpath.2017.06.016 |
| [20] | ZHUANG Z, XIE N, HU J, et al. Interplay between ΔNp63 and miR-138-5p regulates growth, metastasis and stemness of oral squamous cell carcinoma[J]. Oncotarget, 2017, 8(13): 21954–21973. DOI:10.18632/oncotarget.15752 |
| [21] | ABRAM T J, PICKERING C R, LANG A K, et al. Risk stratification of oral potentially malignant disorders in fanconi anemia patients using autofluorescence imaging and cytology-on-a chip assay[J]. Transl Oncol, 2018, 11(2): 477–486. DOI:10.1016/j.tranon.2018.01.014 |
| [22] | ZHANG Y, ZOU X, QIAN W, et al. Enhanced PAPSS2/VCAN sulfation axis is essential for Snail-mediated breast cancer cell migration and metastasis[J]. Cell Death Differ, 2019, 26(3): 565–579. DOI:10.1038/s41418-018-0147-y |
| [23] | SALEM M, O'BRIEN J A, BERNAUDO S, et al. miR-590-3p promotes ovarian cancer growth and metastasis via a novel FOXA2-versican pathway[J]. Cancer Res, 2018, 78(15): 4175–4190. DOI:10.1158/0008-5472.CAN-17-3014 |
| [24] | LIU X G, HAN C Y, LIAO X W, et al. Genetic variants in the exon region of versican predict survival of patients with resected early-stage hepatitis B virus-associated hepatocellular carcinoma[J]. Cancer Manag Res, 2018, 10: 1027–1036. DOI:10.2147/CMAR.S161906 |
| [25] | MITSUI Y, SHIINA H, KATO T, et al. Versican promotes tumor progression, metastasis and predicts poor prognosis in renal carcinoma[J]. Mol Cancer Res, 2017, 15(7): 884–895. DOI:10.1158/1541-7786.MCR-16-0444 |



