2. 400010 重庆,重庆医科大学附属第二医院肝胆外科
2. Department of Hepatobiliary Surgery, the Second Affiliated Hospital of Chongqing Medical University, Chongqing, 400010, China
目前,不孕症已成为继肿瘤和心脑血管疾病外的第3大疾病,是全世界范围内的重要医学及社会问题。越来越多的不孕症患者需要通过辅助生殖技术(assisted reproductive technology, ART)获得妊娠。据统计,在发达国家,1.7%~4.0%的儿童是通过ART获得的[1]。ART能否成功妊娠与其每个步骤密切相关。尽管近年来ART助孕技术迅速发展,但仍有10%的患者会发生反复着床失败(recurrent implantation failure,RIF)。RIF是指女性至少经历连续3次体外受精(in vitro fertilization,IVF),且每次均有1~2个优质胚胎移植或多次移植总胚胎数≥10枚仍未获临床妊娠[2-3]。RIF与胚胎质量、子宫内膜容受性及免疫因素等有着密切的联系,其中子宫内膜容受性的损害是关键原因,子宫内膜容受性受多种因素调控,宫内膜的形态学改变和胞饮突的出现、白血病抑制因子、白介素-1、整合素、同源框基因(HOA基因)等蛋白及基因表达水平的变化会影响子宫内膜容受性,但目前临床上仍不能通过监测相关因素来准确估计子宫内膜着床窗。因此,进一步筛选出影响子宫内膜容受性的关键基因,明确RIF的发病机制显得十分重要。近年来生物信息学技术快速发展,为研究RIF的发病机制提供了新的方法。本研究通过运用生物信息学知识,挖掘RIF相关基因芯片,筛选出差异表达基因并进行分析,为RIF分子机制的研究提供新的线索。
1 资料与方法 1.1 资料来源从美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)公共数据平台GEO(Gene Expression Om-nibus)中搜索“recurrent implantation failure”,选择种属为人体组织,下载基因芯片数据集GSE111974。GSE111974包含24个RIF标本及24个正常对照组标本,数据为人类mRNA表达芯片的原始数据。
1.2 研究方法 1.2.1 基因芯片的数据处理利用R语言“marray”包对GSE111974数据中的每个样本探针表达值进行背景校正归一化处理。通过“limma”包对RIF组织和正常对照组织中每个探针进行t检验。差异表达基因(differently expressed genes, DEGs)以log2(fold change,FC)的绝对值≥2.5和调整的P < 0.05为筛选标准,其中log2FC≥log22.5的为上调,log2FC≤-log22.5的为下调。最后通过GPL17077平台将探针转换成基因名称。
1.2.2 GO分析和KEGG分析DAVID是一个生物信息数据库,其包含生物数据及分析工具。使用DAVID Bioinformatics Resources 6.8对筛选出的DEGs进行GO(Gene Ontology)富集分析和KEGG信号通路分析,GO富集分析分为分子功能(molecular function)、生物过程(biological process)和细胞组成(cellular component)3个部分。利用R语言“ggplot2”包对GO和KEGG分析进行可视化操作。
1.2.3 蛋白相互作用网络的构建及分析STRING数据库是一个搜寻已知蛋白质之间和预测蛋白质之间相互作用的系统。将DEGs输入STRING(version10.5)网站,设置相互作用可信区间为中度(medium confidence 0.400),得出蛋白质相互作用网络(protein-proteininteraction,PPI)图。再将网络图信息导入生物信息分析软件Cytoscape(版本3.6.1)。利用Cytoscape-hubba插件把网络调控中心的基因定义为关键基因(hub gene)。最后运用Cytoscape中的iRegulon插件,设置标准化富集分数(normalized enrichment score,NES)>3[4],寻找关键基因的转录因子。
1.3 统计学分析GSE111974原始数据利用R语言“marray”包进行背景校正归一化处理, 通过“limma”包进行t检验, 通过设定阈值筛选具有统计学显著性的DEGs。GO分析和KEGG分析通过DAVID数据库自带统计学工具分析。Cytoscape中的hubba插件通过自身Degree拓扑分析方法分析,iRegulon插件通过自身计算motif富集分析来预测转录因子。
2 结果 2.1 确定DEGs根据GEO数据库平台中获得符合标准的基因芯片数据集GSE111974,运用R语言筛选反复着床失败有关的DEGs,得到170个差异表达基因,其中上调的基因127个,下调的基因43个(图 1A)。上调和下调最显著的前10个基因见表 1,并对表达差异最明显的20个基因进行聚类热图分析(图 1B)。
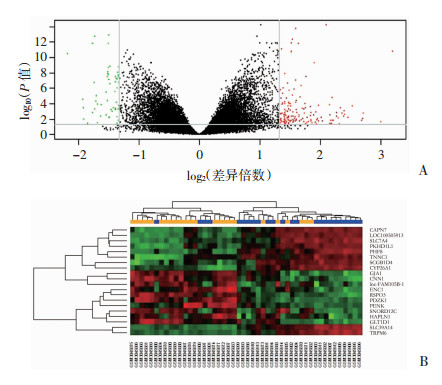
|
| A:火山图红色点表示表达量上调的DEGs;绿色点表示表达量下调的DEGs;B:聚类热图橘色表示正常组织样本(n=24);蓝色为RIF样本(n=24);红色表示mRNAs高表达;绿色表示mRNAs低表达 图 1 反复着床失败子宫内膜组织与正常组织中差异表达的基因 |
| 基因代码 | 基因名称 | log2FC | P值 | 校正后P值 |
| A_24_P130865 | PHF8 | 3.196867 | 8.63E-15 | 1.67E-11 |
| A_23_P138655 | CYP26A1 | 2.996310 | 0.003675 | 0.021971 |
| A_23_P120794 | SLC7A4 | 2.711293 | 0.000144 | 0.001856 |
| A_23_P217570 | CAPN6 | 2.703317 | 0.001019 | 0.008298 |
| A_21_P0000697 | LOC100505912 | 2.684249 | 0.002037 | 0.014073 |
| A_24_P383356 | SLC39A14 | 2.531248 | 8.16E-06 | 0.000196 |
| A_33_P3254136 | PKHD1L1 | 2.457114 | 0.000694 | 0.006225 |
| A_24_P26792 | TRPM6 | 2.402216 | 0.000213 | 0.002516 |
| A_23_P166823 | TNNC1 | 2.392614 | 4.94E-05 | 0.000811 |
| A_33_P3228014 | SCGB1D4 | 2.383097 | 0.000397 | 0.004075 |
| 基因代码 | 基因名称 | log2FC | P值 | 校正后P值 |
| A_24_P55295 | GJA1 | -1.70454 | 1.93E-07 | 1.04E-05 |
| A_23_P111402 | RSPO3 | -1.71108 | 1.49E-06 | 5.24E-05 |
| A_19_P00809682 | lnc-FAM105B-1 | -1.72602 | 2.51E-17 | 1.78E-13 |
| A_23_P7727 | HAPLN1 | -1.72643 | 2.73E-05 | 0.000511 |
| A_32_P19294 | GLT1D1 | -1.76600 | 0.000101 | 0.001424 |
| A_23_P417918 | PENK | -1.84236 | 0.007446 | 0.037073 |
| A_23_P52121 | PDZK1 | -1.90412 | 0.001593 | 0.011670 |
| A_23_P125233 | CNN1 | -1.91692 | 1.75E-05 | 0.000358 |
| A_24_P69095 | ENC1 | -1.92980 | 7.37E-07 | 2.98E-05 |
| A_21_P0000238 | SNORD12C | -2.17751 | 2.84E-14 | 3.66E-11 |
2.2 DEGs的GO富集分析
通过对差异表达基因GO富集分析显示:在生物学过程层面 DEGs主要与氧化还原过程、细胞周期阻滞、脂质代谢过程等10个功能相关(图 2A);在细胞组成层面主要涉及细胞膜、内质网、细胞内膜结合的细胞器和顶端质膜等10个方面(图 2B);在分子功能层面主要与血红素结合、维甲酸结合、信号传递器活性、过氧化物酶活性等10个功能簇相关(图 2C)。
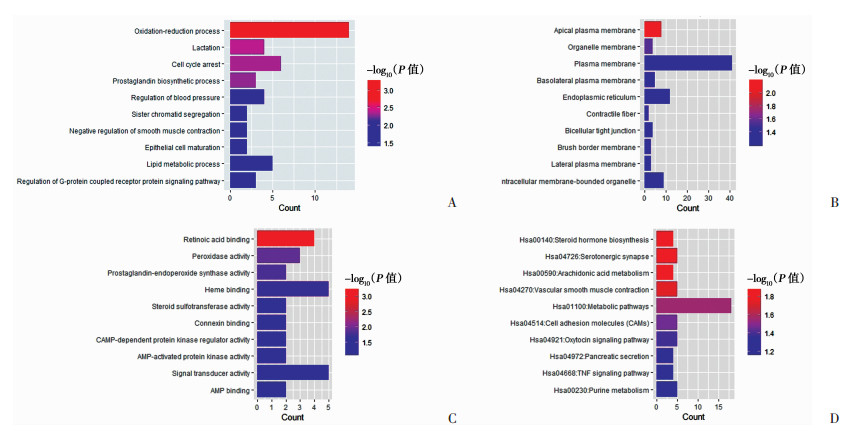
|
| A:生物过程;B:细胞组成;C:分子功能;D:KEGG信号通路 图 2 差异表达基因的GO富集分析和KEGG通路分析 |
2.3 KEGG通路分析
KEGG通路分析显示:主要富集的通路包括代谢途径信号通路、催产素信号通路、肿瘤坏死因子信号通路以及5羟色胺突触、血管平滑肌收缩、细胞黏附分子等(图 2D)。其中代谢途径相关的通路最为显著富集。
2.4 编码蛋白相互作用网络分析为了进一步了解反复着床失败患者内膜组织与正常对照组织中的关键差异基因及其蛋白调控关系,构建了DEGs蛋白互作网络。结果显示,根据interactionscore 0.4为阈值,STRING数据库构建的PPI网络包括144个节点蛋白的84条关系,大部分蛋白间都存在相互作用。利用Cytoscape软件对蛋白互作网络进行可视化分析,再用Cytoscape-hubba插件根据Degree筛选出前10位关键基因(hub基因):PTGS2、TOP2A、ACTA2、ALDH1A1、MUC1、LATS1、PRKAG2、PLCB1、GNG4、FOLR1(图 3)。
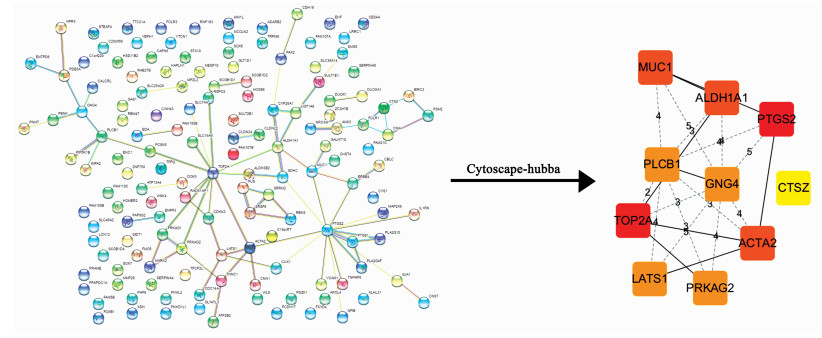
|
| 图 3 差异表达基因的蛋白-蛋白相互作用及hub基因筛选 |
2.5 10个hub基因相关的转录因子分析
运用生物信息分析学软件Cytoscape中的iRegulon插件预测DEGs中hub基因的转录因子,并分析这些转录因子所调节的DEGs。发现与hub基因相关的转录因子有HES2、CEBPB、CDX2、ESR1、IL24、HSF1、E2F6、OTX2、SPI1、PAX5等39个,其前10位转录因子(NES>3)见表 2。转录因子的靶基因作用见图 4。
| TF | NES | 靶基因数目 | 转录调控元件数目 |
| HES2 | 6.938 | 5 | 7 |
| CEBPB | 6.542 | 4 | 28 |
| CDX2 | 6.095 | 5 | 3 |
| ESR1 | 5.266 | 4 | 1 |
| IL24 | 5.171 | 5 | 6 |
| HSF1 | 5.160 | 4 | 3 |
| E2F6 | 5.125 | 4 | 4 |
| OTX2 | 4.966 | 3 | 2 |
| SPI1 | 4.963 | 3 | 3 |
| PAX5 | 4.900 | 7 | 2 |
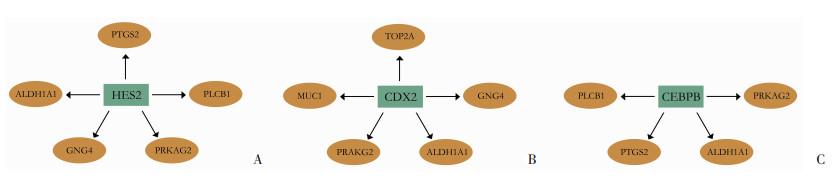
|
| A:作用于HES2的转录因子相互作用网络;B:作用于CDX2的转录因子相互作用网络;C:作用于CEBPB的转录因子相互作用网络;绿色方格代表转录因子,黄色椭圆代表转录因子的靶基因 图 4 hub基因转录因子的靶基因作用 |
3 讨论
IVF治疗成功的关键在于成功的胚胎植入,若反复着床失败,尽管不孕夫妇有数个胚胎移植,但仍不能受孕。RIF有许多危险因素,包括高龄、吸烟状况、体质量指数升高和压力等[5],但排除移植技术优劣、胚胎遗传或发育异常、特定自身抗体等免疫因素存在,输卵管积水,子宫病变如子宫黏膜下肌瘤、子宫内膜息肉、子宫内膜炎及先天性解剖异常等因素后,子宫内膜容受性是决定胚胎植入成功与否的重要因素。子宫内膜容受性的降低将直接导致RIF,其变化可能与众多基因、基因参与的信号通路及其转录因子相关。既往此类研究多基于宫内膜的组织学和细胞学,缺乏整体性和系统性。随着高通量技术的飞速提升,近年来生物信息学发展迅速。本研究采用生物信息学方法挖掘出RIF及正常对照组织中的差异表达基因共170个,其中上调的基因127个,为PHF8、CYP26A1、SLC7A4、CAPN6、LOC100505912等;下调的基因43个,为SNORD12C、ENC1、CNN1、PDZK1、PENK等。PHD锌指蛋白8(PHD finger protein 8,PHF8)是一组蛋白去甲基化酶,在细胞周期进展和调控rRNA转录方面起关键作用[6-7]。研究表明,PHF8是内皮细胞迁移所必需,且PHF8 siRNA减弱了毛细血管样结构的迁移和发展能力[8], 推测PHF8与子宫内膜蜕膜化及子宫螺旋动脉重铸相关。钙蛋白酶(calpains, CAPNs)是一类参与多种生物学过程的细胞内半胱氨酸蛋白酶[9],CAPN6是该家族的成员之一。有研究证明,CAPN6可抑制细胞凋亡,促进细胞增殖[10]。在乳腺癌细胞中,雌激素受体α(estrogen receptor,ER-α)通过胰岛素样生长因子1受体(insulinlike growth factor 1 receptor,IGF-1R)间接调节PDZK1的表达,PDZK1可以促进细胞增殖[11]。本研究显示,PDZK1在RIF患者子宫内膜组织中显著低表达,推测RIF患者内膜早期增殖受损,影响其后续子宫内膜分化、蜕膜化及胚胎着床。
自20世纪70年代以来胚胎着床过程中子宫内膜代谢的改变就引起了人们的注意,子宫内膜容受性与各种糖基化的变化有关,包括子宫内膜上皮中糖蛋白、蛋白多糖和糖脂[12-13]。另有研究表明在冻融胚胎移植时,测定宫内氧化还原电位,发现氧化还原电位较低者ART成功率高[14]。本研究通过GO功能富集分析发现:RIF的DEGs主要富集于细胞膜、内质网等区域,主要参与氧化还原过程、细胞周期阻滞、脂质代谢过程等。KEGG通路分析显示,主要富集的通路包括代谢途径信号通路、催产素信号通路、肿瘤坏死因子信号通路等,其中代谢途径相关的通路富集最显著。胚胎植入和发育需要子宫内膜基质细胞蜕膜化,其分化过程需要葡萄糖的利用。除了葡萄糖,许多细胞依赖脂肪酸作为能源。TSAI等[15]的研究发现脂肪酸β氧化途径对人和小鼠子宫内膜基质细胞的蜕膜化具有重要作用。这些代谢途径相关的通路与子宫内膜的变化密切相关,在RIF的发生中起着重要作用,与我们的预测结果一致。
运用STRING数据库进一步分析差异基因并使用Cytoscape-hubba插件筛选出前10位与RIF相关的hub基因,其中PTGS2、MUC1、LATS1、PRKAG2、PLCB1、FOLR1在RIF患者子宫内膜组织中高表达,TOP2A、ALDH1A1、ACTA2、GNG4低表达。前列腺素内过氧化物合酶2(prostaglandin-endoperoxide synthase 2,PTGS2)的上调与细胞黏附增加、表型改变、抗凋亡和肿瘤血管生成有关。PTGS2的多态性可导致结直肠癌发生[16]。拓扑异构酶2A(topoisomeraseⅡα, TOP2A)为已知的致癌基因,TOP2A在正常机体的组织和细胞内处于低水平状态,在乳腺癌中高表达,与肿瘤进展、复发和预后不良密切相关[17]。平滑肌肌动蛋白α(actinalpha 2,ACTA2)与细胞活动力相关,ACTA2的异常表达加速了乳腺癌细胞的侵袭和转移[18]。文献[19]报道乙醛脱氢酶1(aldehyde dehydrogenase 1,ALDH1)高表达与子宫内膜样腺癌细胞侵袭性增加相关。ALDH1A1是ALDH1家族中的一种重要亚型。本研究显示,ALDH1A1呈低表达,可能与内膜雌激素受体不足有关。黏蛋白1(mucin 1,MUC1)为显著差异表达上调的基因;但在一项包含14例RIF患者、25例复发性流产患者、20例可育对照者的研究中[20],显示RIF患者管腔上皮和腺上皮MUC1的表达均明显低于复发性流产患者和可育对照组,这个结果尚需进一步验证。大规模肿瘤抑制因子1(large tumor suppressor 1,LATS1)是Hippo信号通路的核心成员,而Hippo信号通路在抑制子宫内膜纤维化、宫腔粘连中起重要作用[21]。PRKAG2是AMP激活蛋白激酶(AMP-activated protein kinase,AMPK)的3个调节亚基之一,AMPK的激活已被证明是在应激条件下小鼠滋养层干细胞分化的关键步骤[22],证实该通路在胚胎着床中是有功能的。磷脂酶Cβ1(phospholipaseCbeta 1, PLCB1)参与了药物依赖的易感性[23]。鸟嘌呤核苷酸结合蛋白γ亚基4(guanine nucleotide binding-protein gamma subunit 4,GNG4)是多种跨膜信号传导系统的调节或转导蛋白,GNG4可能与正常老年人的认知能力下降有关[24]。在病理情况下,叶酸受体1(folate receptor 1, FOLR1)可能过表达以增加叶酸的摄取,以便在加速的细胞生长期间应对核酸合成和修复的增加[25],FOLR1在肾癌、胰腺癌、子宫内膜癌、宫颈鳞癌和卵巢癌中均有表达[26]。上述研究结果显示,此10个hub基因的表达可参与细胞的增殖、凋亡、迁移、侵袭及蛋白质的合成等,因此可能与子宫内膜增殖、蜕膜化及容受性相关,但均需进一步深入验证其在RIF发病中的作用。
运用Cytoscape中的iRegulon插件预测DEGs中hub基因的转录因子,发现与其相关的转录因子有HES2、CEBPB、CDX2、ESR1等39个;部分转录因子在子宫内膜中的作用已经得到证实。蜕膜化人子宫内膜基质细胞(human endometrial stromal cells,HESCs)的染色质免疫沉淀分析证实,BMP信号蛋白及SMAD1/5通过结合该基因的3′UTR中特异调节序列直接调节CEBPB的表达,而CEBPB又调节孕激素受体的表达[27]。人类基因表达子宫内膜受体数据库(human gene expression endometrial receptivity database,HGEx-ERdb)显示,大多数上调的受体相关基因(receptivity associated genes,RAGs)具有CDX2等结合位点,因此提示此转录因子在受体阶段激活的可能性,且对转录因子结合位点的分析显示,CDX2结合位点在上调的RAGs中过度表达[28]。雌激素受体-α(estrogen receptor-α,ESR1)可以通过调节细胞周期调节因子的表达来抑制人HESCs的增殖,也可通过诱导蜕膜化过程中重要调控因子FOXO1和WNT4的表达,促进HESCs的分化[29]。我们猜测这些转录因子对RIF的发生有调控作用。
综上,胚胎着床是一个极为复杂的过程,任何一个小问题都可能导致反复着床失败。由于医学发展的局限,医师只能尽量减少由于移植技术、子宫内膜病变等可控因素造成的RIF,但仍有许多未知的致病因素等待我们去挖掘和克服。通过生物信息学的方法分析GEO数据库中的数据,成功筛选出RIF患者子宫内膜组织的差异表达基因,对其进行GO分析和KEGG通路分析,初步了解了它们的功能和相关的信号通路。筛选出的关键基因PTGS2、TOP2A、ACTA2等和转录因子CEBPB、CDX2、ESR1等可能在RIF的发病机制中起重要作用,可能与子宫内膜的微环境如内膜的增殖与分泌、基质的修复及血管形成等密切相关,并调节子宫内膜容受性,影响胚胎着床及IVF的临床成功率。它们可能成为RIF研究和治疗的新靶点,为进一步运用体内外实验研究其病理生理机制提供新的思路。
| [1] | TALAULIKAR V S, ARULKUMARAN S. Reproductive outcomes after assisted conception[J]. Obstet Gynecol Surv, 2012, 67(9): 566–583. DOI:10.1097/ogx.0b013e31826a5d4a |
| [2] | THORNHILL A R, DEDIE-SMULDERS C E, GERAEDTS J P, et al. ESHRE PGD Consortium 'Best practice guidelines for clinical preimplantation genetic diagnosis (PGD) and preimplantation genetic screening (PGS)'[J]. Hum Reprod, 2005, 20(1): 35–48. DOI:10.1093/humrep/deh579 |
| [3] | SIMON A, LAUFER N. Repeated implantation failure: clinical approach[J]. Fertil Steril, 2012, 97(5): 1039–1043. DOI:10.1016/j.fertnstert.2012.03.010 |
| [4] | JANKY R, VERFAILLIE A, IMRICHOVÁ H, et al. IRegulon: from a gene list to a gene regulatory network using large motif and track collections[J]. PLoS Comput Biol, 2014, 10(7): e1003731. DOI:10.1371/journal.pcbi.1003731 |
| [5] | MARTINI A E, JASULAITIS S, FOGG L F, et al. Evaluating the utility of intralipid infusion to improve live birth rates in patients with recurrent pregnancy loss or recurrent implantation failure[J]. J Hum Reprod Sci, 2018, 11(3): 261–268. DOI:10.4103/jhrs.JHRS_28_18 |
| [6] | LEE S, LEE J W, LEE S K. UTX, a histone H3-lysine 27 demethylase, acts as a critical switch to activate the cardiac developmental program[J]. Dev Cell, 2012, 22(1): 25–37. DOI:10.1016/j.devcel.2011.11.009 |
| [7] | ZHU Z Q, WANG Y R, LI X, et al. PHF8 is a histone H3K9me2 demethylase regulating rRNA synthesis[J]. Cell Res, 2010, 20(7): 794–801. DOI:10.1038/cr.2010.75 |
| [8] | GU L D, HITZEL J, MOLL F, et al. The histone demethylase PHF8 is essential for endothelial cell migration[J]. PLoS ONE, 2016, 11(1): e0146645. DOI:10.1371/journal.pone.0146645 |
| [9] | XIANG Y Z, LI F, WANG L, et al. Decreased calpain 6 expression is associated with tumorigenesis and poor prognosis in HNSCC[J]. Oncol Lett, 2017, 13(4): 2237–2243. DOI:10.3892/ol.2017.5687 |
| [10] | ANDRIQUE C, MORARDET L, LINARES L K, et al. Calpain-6 controls the fate of sarcoma stem cells by promoting autophagy and preventing senescence[J]. JCI Insight, 2018, 3(17): 121225. DOI:10.1172/jci.insight.121225 |
| [11] | KIM H, ABD ELMAGEED Z Y, JU J H, et al. PDZK1 is a novel factor in breast cancer that is indirectly regulated by estrogen through IGF-1R and promotes estrogen-mediated growth[J]. Mol Med, 2013, 19: 253–262. DOI:10.2119/molmed.2011.00001 |
| [12] | KUBUSHIRO K, KOJIMA K, MIKAMI M, et al. Menstrual cycle-associated alteration of sulfogalactosylceramide in human uterine endometrium: possible induction of glycolipid sulfation by sex steroid hormones[J]. Arch Biochem Biophys, 1989, 268(1): 129–136. DOI:10.1016/0003-9861(89)90573-0 |
| [13] | ZHU Z, DENG H, FENDERSON B A, et al. Glycosphingolipids of human myometrium and endometrium and their changes during the menstrual cycle, pregnancy and ageing[J]. J Reprod Fertil, 1990, 88(1): 71–79. DOI:10.1530/jrf.0.0880071 |
| [14] | NAKAMURA H, HOSONO T, TANIGUCHI T, et al. Prediction of pregnancy after frozen-thawed embryo transfer via in vivo intrauterine oxidation-reduction potential measurements: A pilot study[J]. Reprod Med Biol, 2018, 17(3): 255–261. DOI:10.1002/rmb2.12098 |
| [15] | TSAI J H, CHI M M, SCHULTE M B, et al. The fatty acid beta-oxidation pathway is important for decidualization of endometrial stromal cells in both humans and mice[J]. Biol Reprod, 2014, 90(2): 34. DOI:10.1095/biolreprod.113.113217 |
| [16] | VOGEL L K, SÆBØ M, HØYER H, et al. Intestinal PTGS2 mRNA levels, PTGS2 gene polymorphisms, and colorectal carcinogenesis[J]. PLoS ONE, 2014, 9(8): e105254. DOI:10.1371/journal.pone.0105254 |
| [17] | FRITZ P, CABRERA C M, DIPPON J, et al. C-ErbB2 and topoisomerase IIalpha protein expression independently predict poor survival in primary human breast cancer: A retrospective study[J]. Breast Cancer Res, 2005, 7(3): R374–R384. DOI:10.1186/bcr1012 |
| [18] | JEON M, YOU D, BAE S Y, et al. Dimerization of EGFR and HER2 induces breast cancer cell motility through STAT1-dependent ACTA2 induction[J]. Oncotarget, 2017, 8(31): 50570–50581. DOI:10.18632/oncotarget.10843 |
| [19] | RAHADIANI N, IKEDA J, MAMAT S, et al. Expression of aldehyde dehydrogenase 1 (ALDH1) in endometrioid adenocarcinoma and its clinical implications[J]. Cancer Sci, 2011, 102(4): 903–908. DOI:10.1111/j.1349-7006.2011.01864.x |
| [20] | WU F R, CHEN X Y, LIU Y Y, et al. Decreased MUC1 in endometrium is an independent receptivity marker in recurrent implantation failure during implantation window[J]. Reprod Biol Endocrinol, 2018, 16(1): 60. DOI:10.1186/s12958-018-0379-1 |
| [21] | ZHU H Y, GE T X, PAN Y B, et al. Advanced role of hippo signaling in endometrial fibrosis: implications for intrauterine adhesion[J]. Chin Med J, 2017, 130(22): 2732–2737. DOI:10.4103/0366-6999.218013 |
| [22] | ZHONG W, XIE Y, ABDALLAH M, et al. Cellular stress causes reversible, PRKAA1/2-, and proteasome-dependent ID2 protein loss in trophoblast stem cells[J]. Reproduction, 2010, 140(6): 921–930. DOI:10.1530/REP-10-0268 |
| [23] | CABANA-DOMÍNGUEZ J, RONCERO C, PINEDA-CIRERA L, et al. Association of the PLCB1 gene with drug dependence[J]. Sci Rep, 2017, 7(1): 10110. DOI:10.1038/s41598-017-10207-2 |
| [24] | BONHAM L W, EVANS D S, LIU Y M, et al. Neurotransmitter pathway genes in cognitive decline during aging: evidence for GNG4 and KCNQ2 genes[J]. Am J Alzheimers Dis Other Demen, 2018, 33(3): 153–165. DOI:10.1177/1533317517739384 |
| [25] | KELEMEN L E, SELLERS T A, KEENEY G L, et al. Multivitamin and alcohol intake and folate receptor alpha expression in ovarian cancer[J]. Cancer Epidemiol Biomarkers Prev, 2005, 14(9): 2168–2172. DOI:10.1158/1055-9965.EPI-05-0260 |
| [26] | PILLAI M R, CHACKO P, KESARI L A, et al. Expression of folate receptors and heterogeneous nuclear ribonucleoprotein E1 in women with human papillomavirus mediated transformation of cervical tissue to cancer[J]. J Clin Pathol, 2003, 56(8): 569–574. DOI:10.1136/jcp.56.8.569 |
| [27] | CLEMENTI C, TRIPURANI S K, LARGE M J, et al. Activin-like kinase 2 functions in peri-implantation uterine signaling in mice and humans[J]. PLoS Genet, 2013, 9(11): e1003863. DOI:10.1371/journal.pgen.1003863 |
| [28] | BHAGWAT S R, CHANDRASHEKAR D S, KAKAR R, et al. Endometrial receptivity: a revisit to functional genomics studies on human endometrium and creation of HGEx-ERdb[J]. PLoS ONE, 2013, 8(3): e58419. DOI:10.1371/journal.pone.0058419 |
| [29] | KAYA OKUR H S, DAS A, TAYLOR R N, et al. Roles of estrogen receptor-α and the coactivator MED1 during human endometrial decidualization[J]. Mol Endocrinol, 2016, 30(3): 302–313. DOI:10.1210/me.2015-1274 |



