2. 400038 重庆, 陆军军医大学(第三军医大学):第一附属医院感染科
2. Department of Infectious Disease, First Affiliated Hospital, Army Medical Universtiy (Third Military Medical university), Chongqing, 400038, China
乙型肝炎病毒(hepatitis B virus,HBV)感染可引起急、慢性病毒性肝炎,是世界重要的公共卫生问题之一。据WHO数据显示:全世界尚有2.57亿乙肝病毒表面抗原阳性感染者,而在中国,依然有8 000余万表面抗原阳性感染者,感染率达到6.1%[1]。在我国西藏地区,长期世居藏族人相对隔离,HBsAg携带率高达12%以上[2]。有研究显示:汉族人HBV感染以B、C基因型为主,B型多见于南方,C型多见于北方[3],而藏族感染者除了有常见的基因型,还有C/D重组型,且以重组型为优势毒株[4]。此种特殊的C/D重组型乙肝病毒,其感染所致的肝炎病程特征、预后、病毒学特征等,目前研究得不多。
HBV为全长3.2 kb的部分环状双链DNA病毒,其自身复制以pgRNA为模板通过逆转录实现,由于逆转录酶缺乏3′- 5′外切酶活性,缺乏校正功能,故易错配产生一系列序列不同的病毒DNA,称之为准种。HBV准种的存在与肝炎的转归、肝癌的发生、免疫逃逸、耐药等密切相关[5],故引起了人们越来越多的关注。一直以来有关HBV准种的研究多采用片段扩增、克隆测序的方式进行[6-8],此种方法成本较高,获得的病毒克隆数有限,难以发现低丰度的准种突变。有研究者采用二代高通量测序的方式进行准种研究[9-10],获得了大量DNA数据,但二代测序读长较短,需要进行拼接得到目的片段,难以获得单条HBV基因组序列。近年来美国Pacific Biosciences(PacBio)公司推出的基于单分子实时测序(single molecule realtime,SMRT)的PacBio RSⅡ测序平台,是第三代测序技术的代表之一,其文库构建是将双链DNA分子两端加上接头序列形成发卡结构,同时在直径100 nm的零模波导孔(zero-mode waveguide,ZMW)底部固定DNA聚合酶,在ZMW中DNA双链打开形成环状DNA, 进而实现连续的合成测序。SMRT测序技术可以实现高通量、长读长的单条DNA分子测序[11-12],平均读长可达10 kb,完全可覆盖HBV基因组,免除了克隆、拼接的步骤,极大简化了准种分析,是理想的准种分析工具。
本研究采用PacBio RSⅡ测序平台对HBV基因组进行单分子测序,成功获得HBV全长序列,进而对藏族乙肝感染者的HBV准种特征进行了深入探讨,旨在为藏族人群的乙肝诊治提供新的依据。
1 资料与方法 1.1 研究对象本科室于2016年5月至2017年7月在藏南地区(包括察隅、类乌齐、米林、墨脱4个县)进行乙型肝炎病毒流行病学调查,调查对象签署《关于参加自然疫源性疾病病原谱调查研究的知情同意书》后,详细收集人口学资料和血清样本,共采集到样本508例,其中高原世居者样本209例、高原移居者299例。血清学检出乙型肝炎病毒表面抗原(HBsAg)阳性样本24例:HBsAg强阳性(HBsAg>250 IU/mL)14例,均为高原世居藏族人; HBsAg弱阳性(0.05 IU/mL < HBsAg < 0.5 IU/mL)10例,其中5例为高原移居汉族、2例为高原世居藏族、3例为高原世居其他少数民族。24例HBsAg阳性样本作为高原世居藏组(Tibet,T)纳入本研究,其中男性15例(62.5%),女性9例(37.5%),年龄(36.6±13.2)岁。同时,按照性别、年龄、生化指标匹配的原则,选择2017年在我科门诊就诊的HBsAg阳性汉族人24例,留取临床检测后剩余血清样本作为平原汉族对照组样本(Control,C),其中男性15例(62.5%),女性9例(37.5%),年龄(36.5±12.1)岁,患者均签署《临床检测剩余血保存及研究知情同意书》。本研究于2014年7月通过陆军军医大学(第三军医大学)第一附属医院伦理委员会审批[2014年科研第(35)号]。
1.2 HBV DNA提取及全长扩增用美国Omega Bio-Tek公司的磁珠总核酸提取试剂盒,从100 μL血清中提取HBV DNA,遵照厂家说明严格操作。参照GVNTHER等[13]的方法设计引物,并在引物上设计16 nt的随机碱基作为标签以标记样本,正向引物位于1 821~1 841 nt(5′-barcod-TTTTTCACCTCTGCCTARTCA-3′),反向引物位于1 825~1 806 nt (5′-barcod-AAAAAGTTGCATGGTGCTGG-3′)。采用50 μL PCR反应体系,包括5 μL 10×PCR Buffer,5 μL MgCl2(25 mmol/L),8 μL dNTP(2.5 mmol/L),3 μL正反向引物(10 mmol/L),0.5 μL LA Taq(5 U/μL), HBV DNA 5 μL, 用ddH2O补足体积; PCR反应条件为:变性94 ℃/5 min; 循环(40次)94 ℃/25 s, 55 ℃/30 s, 72 ℃/210 s; 延伸72 ℃/10 min。PCR产物在1%琼脂糖凝胶中进行电泳,验证是否成功扩增。
1.3 测序及数据处理将PCR产物进行磁珠纯化,通过Nanodrop、Qubit检测DNA浓度,再将每个样本等量混合,随后在武汉未来组生物科技有限公司PacBio RSⅡ平台进行建库及测序,依据厂家官方步骤操作。获得原始下机数据3.5 GB,经过质控去除低质量reads,再根据序列比对、标签序列拆分获得412 MB有效数据,选择测序读长≥9.6 kb(即≥3倍HBV全长)的reads,通过SMRT Tools生成共有序列(circular consensus sequence,CCS),即准种序列。用ClustalW 2.1、BioEdit 7.0分别完成序列比对、编辑; 选取3条序列作为参考序列,即B型(AB033554)、C型(AY123041)、D型(X65259),对插入缺失错误的indel进行必要的手动校正; 用Modeltest 3.7分析得出适用的核酸替换模型为Tamura-Nei model+G(Tamura-Nei模型+伽马分布),氨基酸替换模型用Jones-Taylor-Thornton (JTT)+G。准种复杂度和多样性分别用标准化的香农熵(Shannon entropy,Sn)和平均遗传距离(mean genetic distance,d)来表示,前者计算公式为:Sn=-Σi(piInpi)/InN,pi为第i个准种序列出现的频率,N为总的准种数量。同时选用校正的Nei-Gojobori method (Jukes-Cantor)+G模型,计算同义突变率(synonymous mutation,dS)、非同义突变率(nonsynonymous mutation, dN); 同时计算非同义突变率/同义突变率比值用于反映阳性选择压力的大小。重组分析用SimPlot 3.5完成,遗传距离、同义突变、非同义突变分析,以及系统发育树构建用MEGA 7.0完成,突变检测的阈值定为1%。
1.4 统计学分析采用SPSS 22.0统计软件,均值的比较用Student’s t检验,突变率的比较用Fisher精确概率检验,统计结果采用双侧检验结果,检验水准:α=0.05。
2 结果 2.1 样本概况高原世居藏族组24例样本中仅有9例HBsAg强阳性样本HBV全长扩增成功并测序,5例HBsAg强阳性样本及10例HBsAg弱阳性样本多次扩增失败。9例扩增成功的样本均获得良好的测序结果,均为高原世居藏族人,纳入本研究后续分析。平原汉族对照样本全部HBV全长扩增成功并测序,但有4例样本获得CCS较少(CCS < 18条),不纳入后续分析,将20例对照样本纳入后续分析。高原世居藏族组平均65条CCS/样本(18~229条),平原汉族对照组平均124条CCS/样本(24~408条)。两组样本基本情况见表 1。
| 组别 | n | 男/女 | 年龄/岁 | ALT/U·L-1 | ALB/g·L-1 | TBIL/μmol·L-1 | HBV DNAa/lg10 U·mL-1 | HBsAg/IU·mL-1 | 三代测序获得准种数量 |
| 高原世居藏族组 | 9 | 4/5 | 39.9±14.2 | 53.2±29.7 | 43.7±3.6 | 13.7±6.6 | - | >250 | 65±63 |
| 平原汉族对照组 | 20 | 14/6 | 35.5±12.8 | 45.3±19.4 | 44.7±6.1 | 13.7±5.3 | 7.8±0.7 | >250 | 124±110 |
| a:因采集的样本量有限,高原世居藏族样本只进行了HBsAg滴度检测,未检测HBV DNA载量 | |||||||||
2.2 系统发育分析及重组鉴定
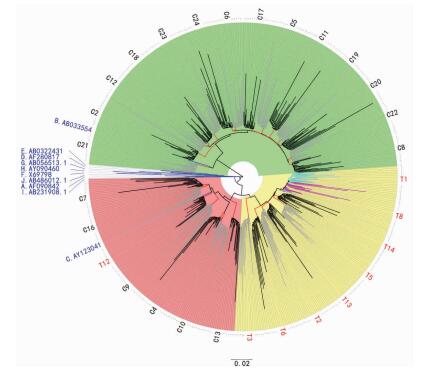
将29例样本(9例高原世居藏族样本,20例平原汉族对照样本)各选择18条准种序列,和基因型A~I共9条HBV参考序列比对后,用NJ法构建系统发育树(图 1)。发现8例藏族样本同属一个进化支,与C型参考株距离最近(0.04~0.05),其中1例藏族样本(T3)与其他7例样本遗传距离稍远; T1与T8样本进化支相互交错,提示它们遗传距离很近,可能有共同的传染源; 此外,还有1例藏族样本(T12)紧邻C型参考株,为C基因型; 汉族对照样本则分别有6例C基因型,14例B基因型。
|
| 红色分支表示1例样本的主进化支,蓝色分支表示HBV参考株,青色分支表示T1,紫色分支表示T8;绿色扇区代表B基因型,红色扇区代表C基因型,黄色扇区代表藏族样本自成一个进化支; 比例尺表示遗传距离(替换/碱基) 图 1 29例样本与HBV参考株系统发育树 |
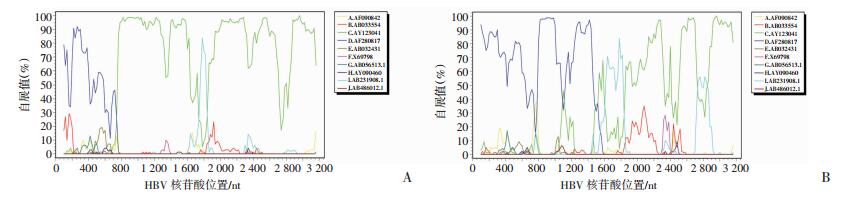
进一步用Simplot进行重组分析,发现7例藏族样本在750 nt发生重组(图 2A),1~750 nt是D型,而751~3 215 nt是C型,为C/D1重组型; 1例藏族样本在1 500 nt发生重组(图 2B),1~1 500 nt是D型,1 501~3 215 nt是C型,为C/D2重组型。剩下1例藏族样本(T12)及20例对照样本则无重组现象。
|
| A:藏族样本T2为C/D1重组型,重组发生在750 nt; B:藏族样本T3为C/D2重组型,重组发生在1 500 nt; 参考序列为A(AF090842)、B(AB033554)、C(AY123041)、D(X65259)、E(AB032431)、F(X69798)、H(AY090460) 图 2 藏族HBV基因组Simplot重组分析 |
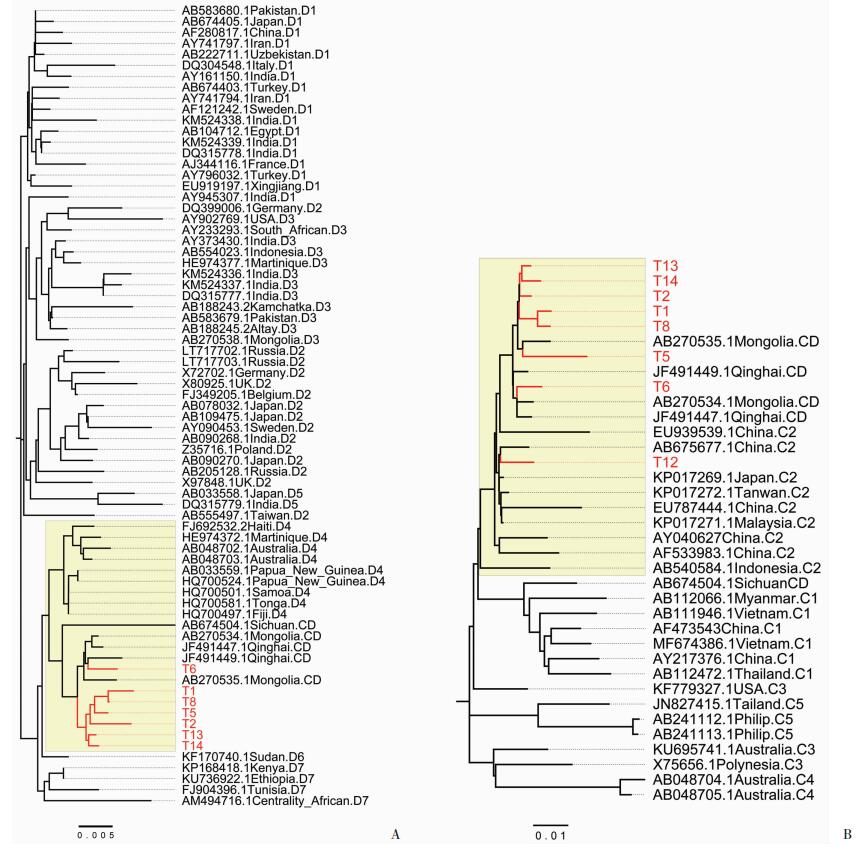
为进一步探讨HBV重组可能的来源,从GenBank选取23例C基因型参考株、61例D基因型参考株,5例C/D重组型参考株,对藏族样本HBV的2个重组片段进行系统发育分析。C/D1型样本D型片段(1~750 nt)除了与青海、蒙古、四川C/D型HBV较接近外,还与D4亚型HBV较接近(图 3A); 其C型片段(751~3 215 nt)除与青海、蒙古C/D型HBV接近,与C2亚型较接近(图 3B)。对1例C/D2型HBV做同样的系统发育分析,发现其D型片段(1~1 500 nt)与D3亚型较接近,尤其与蒙古的1株D3型HBV距离较近; 而其C型片段(1 501~3 215 nt)同样与C2型较接近。此外,1例C型藏族样本T12属C2亚型。
|
| A:C/D1型HBV 1~750 nt片段系统发育分析; B:C/D1型HBV 751~3 215 nt片段系统发育分析 图 3 C/D1型HBV重组片段的系统发育分析 |
2.3 准种复杂度和多样性分析
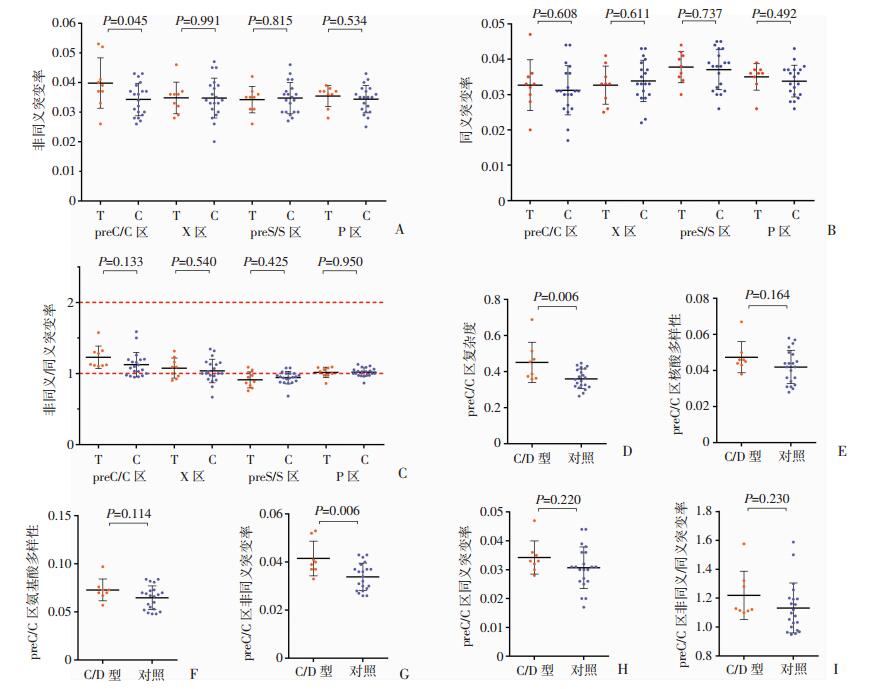
高原世居藏族组与平原汉族对照组在HBV 4个编码区(preC/C、X、preS/S、P)内比较,准种复杂度、多样性差异无统计学意义; 但藏族组在preC/C区的非同义突变率高于对照组(图 4A,P=0.045),而在其他3个编码区差异无统计学意义; 同义突变率组间差异也无统计学意义(图 4B)。全部的藏族样本以及75%(15/20)的对照样本preC/C区dN/dS>1(图 4C),而X、preS/S、P区未出现此种特征,提示preC/C区相比其他3个编码区受到更大的阳性选择压力。
|
| A:藏族样本(T)与对照样本(C)在各编码区非同义突变率的比较; B:T与C在各编码区同义突变率的比较; C:T与C在各编码区非同义突变率/同义突变率的比值比较; D:C/D型HBV与对照基因型HBV在preC/C编码区准种复杂度比较; E:C/D型与对照基因型HBV在preC/C编码区核酸水平多样性比较; F:C/D型与对照基因型HBV在preC/C编码区氨基酸水平多样性比较; G:C/D型与对照基因型HBV在preC/C编码区非同义突变率比较; H: C/D型与对照基因型HBV在preC/C编码区同义突变率比较; I: C/D型与对照基因型HBV在preC/C编码区非同义突变率/同义突变率比值的比较每个点代表 1个样本该编码区得出的平均值,黑色短横线代表均数±标准差 图 4 29例样本HBV准种复杂度、多样性、同义/非同义突变率的比较 |
为进一步了解C/D型HBV在preC/C的准种特征,将8例C/D型样本与21例B、C型样本(包括T12)进行比较发现,C/D型HBV的复杂度(图 4D)、多样性(图 4E、F)、非同义突变率(图 4G)、同义突变率(图 4H)都高于对照样本,且复杂度和非同义突变率最为显著(P=0.006)。
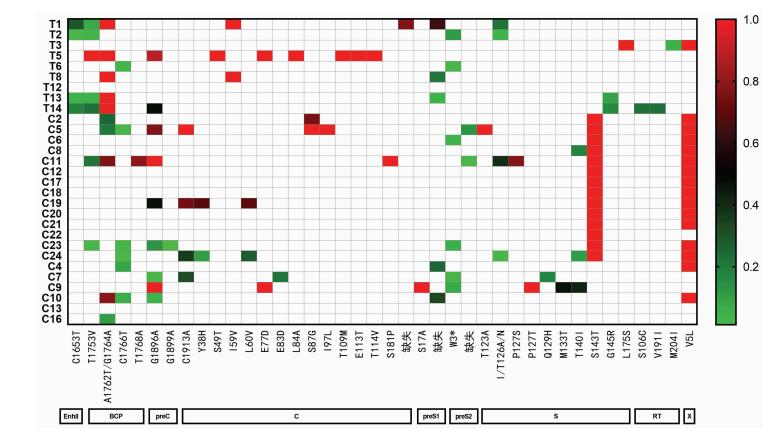
2.4 准种突变检测对所有样本进行突变检测发现(图 5):高原世居藏族样本在基本核心启动子区(basal core promoter,BCP)比平原汉族对照样本有更多的增强子Ⅱ(EnhⅡ)突变C1653T(P=0.005),和T1753V/A1762T/G1764A三突变(P=0.022)。C基因区的变异各有不同,在T细胞表位区,C1913A、Y38H、L60V只在对照组中出现,而I59V突变只在藏族样本中出现; 在B细胞表位区,发现1例藏族样本(T5)同时带有E77D、L84A、T109M、E113T、T114V突变,对照样本E77D、E83D、S87G突变分别在不同样本间散在分布。另外,只在1例藏族样本中观察到C基因的缺失突变。两组样本在preS1区都发现有缺失突变,preS2都发现有W3*突变,而preS2的缺失突变只在2例对照样本中发现。在S基因的“a”决定区(aa124~147),藏族样本只发现2例低频率的I/T126A/N突变,2例低频率G145R突变,非“a”决定区有1例L175S突变; 对照样本则发现较多的“a”决定区突变,除有I/T126A/N外,还有P127S/T、Q129H、M133T、T140I、S143T等突变,其中S143T显著多于藏族样本(P=0.001);RT区耐药突变只在2例藏族样本中低频率出现,对照样本未发现耐药突变; X基因变异V5L对照组显著多于藏族组(P=0.030)。其他报道过的突变如S区免疫逃逸突变A128V、Q129H/R、G130N、M133L/T、Y134F/H, RT区耐药突变I169L、V173L、L180M、A181V, S202C/I、N236T、P237H等在所有样本中未发现。
|
| 纵坐标为样本编号,横坐标为各功能区的突变,将频率>1%的突变纳入分析,突变频率用相应的颜色表示,白色表示没有突变; *:终止密码子 图 5 29例样本HBV突变筛查结果 |
3 讨论
本研究借助PacBio RSⅡ第三代测序平台,成功对29例样本进行了HBV全长测序,两组样本各获得了平均65条、135条准种序列。结果表明第三代测序相比一代、二代测序,有长读长、操作简便、成本较低的优势,适用于HBV基因组全长测序,在HBV全长准种分析上有优势。但此次测序下机数据为3.5 GB,处理后最终得到的有效数据为421 MB,拆分率仅有12%,其原因可能是PCR扩增不完全、杂质干扰、DNA降解断裂等,以致文库中存在部分短片段DNA,拟在以后的研究中运用DNA凝胶回收的方法加以改进。此外,样本收集条件受限,样本量较少,HBV DNA、HBeAg定量等数据未能获得,也是本研究的不足之处。
2002年就有研究者发现青海、西藏地区乙肝感染者HBV存在着大量的C/D重组现象,且C/D重组型是西藏地区的优势毒株[4, 14]。实际上,有关HBV重组现象时有报道,例如印度发现的A/D型[15],非洲发现的D/E型[16],甚至东南亚发现的Ⅰ型毒株也有人认为是基于C型HBV的重组株[17]。而C/D重组型则主要在中国青藏高原发现,可能因为青藏高原具有独特的地理环境和宗教信仰,藏族人群长期世居,与外界鲜有交流,故此种重组型HBV毒株仅在此地域流行。C/D型HBV与C型参考株基因组序列仅有4%~5%的差异,而全基因序列差异>8%可列为新的基因型,差异>4%可列为新的基因亚型[18],此种重组型HBV是否可以划定为一个新的C基因型亚型,目前尚无定论。本研究在9例高原世居藏族样本中,鉴定出7例C/D1型HBV,重组发生在750 nt,1~750 nt为D型,其他部分为C型,此种类型占绝大多数; 此外鉴定出1例C/D2型HBV,重组发生在1 500 nt,1~1 500 nt为D型,其他部分为C型; 两种重组型的发现与文献报道的重组方式基本一致[18-19]。C/D1型HBV分别由D4亚型与C2亚型HBV相应的片段组成,C/D2型则由D3亚型与C2亚型组成,两者都是在C2亚型的基础上,加入了短片段的D型HBV序列; D4亚型见于南太平洋的澳大利亚、汤加、斐济以及加勒比海域等地,而D3型在印度、印尼等地多有报道,C2型则流行于东亚大陆[20]。C/D型HBV重组的发生,一个可能的解释是,上述地域传入的D型HBV, 与C2型HBV共同感染人体,在免疫系统选择压力下发生了重组。有趣的是,藏族人C/D型HBV流行株在核酸水平除了与地缘关系较近的青海流行的C/D型较接近外, 还与在蒙古分离出的两株C/D型HBV也十分接近,提示西藏、青海、蒙古三地C/D型HBV的传播有着紧密的联系。
藏族人C/D型HBV在preC/C基因区复杂度和非同义突变率显著高于对照组,并且preC/C区相比其他编码区受到更大的阳性选择压力,提示在藏族人的遗传背景下,HBV preC/C基因区面临更大的选择压力。此外,本研究发现在C/D重组型HBV准种变异主要集中在EnhⅡ区(1 636~1 744 nt)、BCP区(1 742~ 1 849 nt)和preS区(2 848~3 215 nt,1~154 nt),例如C1653T突变、T1753V/A1762T/G1764A三联突变、preS的缺失突变等,SHEN等[21]曾报道过C/D型HBV有较多A1762T/G1764A双突变,与本研究的结果较一致。对照组准种变异则主要在BCP、preC(1 814~1 900 nt)、preS、S(155~835 nt)、X(1 374~1 838 nt)等功能区,例如G1896A、preS缺失突变、sT140I、sS143T、xV5L等变异。A1762T/G1764A双突变会抑制preC蛋白的合成,进而抑制HBeAg的合成,此突变被认为与肝硬化、肝癌(HCC)的进展有关[22-23];另一项研究显示:preS突变、C1653T、T1753V、A1762T/G1764A的存在会增加HCC患病风险[24]。A1762T/G1764A双突变同时会导致X基因的K130M/V131I双突变,而有研究显示:X基因V5L突变和K130M/V131I双突变会随着年龄的增加而增多,同时会增加HCC的患病风险[25]。藏族人C/D型HBV存在较多EnhⅡ、BCP、preS区变异,而S基因区表位变异少,X基因V5L变异少,呈现出与汉族人不同的变异特点,其是否会有较大的HCC患病风险,有待进一步研究。
本研究采用了第三代测序技术对藏族人HBV准种进行了深入探讨,第三代测序技术(单分子测序)具有长读长、操作简便、成本较低等特点,用于HBV全长准种分析有其独特的优势。本研究在藏族人中鉴定了C/D1与C/D2两种HBV重组型,且C/D1为优势毒株; 继而发现C/D重组型HBV准种突变集中在EnhⅡ区、BCP区、preS区,而S基因、X基因突变较少; HBV重组的发生机制及其与肝炎发展、肝癌发生的关系还有待进一步研究。
| [1] | Polaris Observatory Collaborators. Global prevalence, treatment, and prevention of hepatitis B virus infection in 2016: a modelling study[J]. Lancet Gastroenterol Hepatol, 2018, 3(6): 383–403. DOI:10.1016/S2468-1253(18)30056-6 |
| [2] |
达娃桑姆, 达瓦, 姜倩, 等. 西藏自治区2202名藏族人群乙型肝炎病毒感染影响因素研究[J].
中华流行病学杂志, 2011, 32(4): 427–428.
DAWA S M, DA W, JIANG Q, et al. Study on the risk factors of Hepatitis B virus infection of 2202 Tibetans in Tibet Autonomous Region[J]. Chin J Epidemiol, 2011, 32(4): 427–428. DOI:10.3760/cma.j.issn.0254-6450.2011.04.024 |
| [3] | LI H M, WANG J Q, WANG R, et al. Hepatitis B virus genotypes and genome characteristics in China[J]. World J Gastroenterol, 2015, 21(21): 6684–6697. DOI:10.3748/wjg.v21.i21.6684 |
| [4] | CUI C Y, SHI J X, HUI L J, et al. The dominant hepatitis B virus genotype identified in Tibet is a C/D hybrid[J]. J Gen Virol, 2002, 83(Pt 11): 2773–2777. DOI:10.1099/0022-1317-83-11-2773 |
| [5] |
刘辉. 乙型肝炎病毒准种特点及实验诊断[J].
医学综述, 2012, 18(10): 1539–1542.
LIU H. Characteristics and laboratory diagnosis of hepatitis B virus quasispecies[J]. Med Recapitulate, 2012, 18(10): 1539–1542. |
| [6] | LIU F, CHEN L, YU D M, et al. Evolutionary patterns of hepatitis B virus quasispecies under different selective pressures: correlation with antiviral efficacy[J]. Gut, 2011, 60(9): 1269–1277. DOI:10.1136/gut.2010.226225 |
| [7] | XUE Y, WANG M J, YANG Z T, et al. Clinical features and viral quasispecies characteristics associated with infection by the hepatitis B virus G145R immune escape mutant[J]. Emerg Microbes Infect, 2017, 6(3): e15. DOI:10.1038/emi.2017.2 |
| [8] | KIM H, JEE Y M, SONG B C, et al. Analysis of hepatitis B virus quasispecies distribution in a Korean chronic patient based on the full genome sequences[J]. J Med Virol, 2007, 79(3): 212–219. DOI:10.1002/jmv.20789 |
| [9] | YANG G F, LIU Z H, YANG J C, et al. Quasispecies characteristics in mother-to-child transmission of hepatitis B virus by next-generation sequencing[J]. J Infect, 2017, 75(1): 48–58. DOI:10.1016/j.jinf.2017.04.012 |
| [10] | WANG X, DENG W Y, QIAN K L, et al. Quasispecies characters of hepatitis B virus in immunoprophylaxis failure infants[J]. Eur J Clin Microbiol Infect Dis, 2018, 37(6): 1153–1162. DOI:10.1007/s10096-018-3235-5 |
| [11] | NAKANO K, SHIROMA A, SHIMOJI M, et al. Advantages of genome sequencing by long-read sequencer using SMRT technology in medical area[J]. Hum Cell, 2017, 30(3): 149–161. DOI:10.1007/s13577-017-0168-8 |
| [12] | BETZ-STABLEIN B D, TÖPFER A, LITTLEJOHN M, et al. Single-molecule sequencing reveals complex genome variation of hepatitis B virus during 15 years of chronic infection following liver transplantation[J]. J Virol, 2016, 90(16): 7171–7183. DOI:10.1128/JVI.00243-16 |
| [13] | GÜNTHER S, LI B C, MISKA S, et al. A novel method for efficient amplification of whole hepatitis B virus genomes permits rapid functional analysis and reveals deletion mutants in immunosuppressed patients[J]. J Virol, 1995, 69(9): 5437–5444. |
| [14] | ZHOU B, XIAO L, WANG Z H, et al. Geographical and ethnic distribution of the HBV C/D recombinant on the Qinghai-Tibet Plateau[J]. PLoS ONE, 2011, 6(4): e18708. DOI:10.1371/journal.pone.0018708 |
| [15] | CHAUHAN R, KAZIM S N, KUMAR M, et al. Identification and characterization of genotype A and D recombinant hepatitis B virus from Indian chronic HBV isolates[J]. World J Gastroenterol, 2008, 14(40): 6228–6236. DOI:10.3748/wjg.14.6228 |
| [16] | ABDOU CHEKARAOU M, BRICHLER S, MANSOUR W, et al. A novel hepatitis B virus (HBV) subgenotype D (D8) strain, resulting from recombination between genotypes D and E, is circulating in Niger along with HBV/E strains[J]. J Gen Virol, 2010, 91(Pt 6): 1609–1620. DOI:10.1099/vir.0.018127-0 |
| [17] | TRAN T T, TRINH T N, ABE K. New complex recombinant genotype of hepatitis B virus identified in Vietnam[J]. J Virol, 2008, 82(11): 5657–5663. DOI:10.1128/JVI.02556-07 |
| [18] | LIU T Z, WANG F Z, ZHANG S, et al. Whole-gene analysis of two groups of hepatitis B virus C/D inter-genotype recombinant strains isolated in Tibet, China[J]. PLoS ONE, 2017, 12(6): e0179846. DOI:10.1371/journal.pone.0179846 |
| [19] | WANG Z H, LIU Z H, ZENG G B, et al. A new intertype recombinant between genotypes C and D of hepatitis B virus identified in China[J]. J Gen Virol, 2005, 86(Pt 4)): 985–990. DOI:10.1099/vir.0.80771-0 |
| [20] | SUNBUL M. Hepatitis B virus genotypes: global distribution and clinical importance[J]. World J Gastroenterol, 2014, 20(18): 5427–5434. DOI:10.3748/wjg.v20.i18.5427 |
| [21] | SHEN L P, YIN W J, ZHENG H, et al. Molecular epidemiological study of hepatitis B virus genotypes in Southwest, China[J]. J Med Virol, 2014, 86(8): 1307–1313. DOI:10.1002/jmv.23965 |
| [22] | TU W H, LV Y, ZHANG Y M, et al. Precore/basal core promoter mutants quantification throughout phases of hepatitis B virus infection by Simpleprobe[J]. World J Gastroenterol, 2015, 21(21): 6639–6648. DOI:10.3748/wjg.v21.i21.6639 |
| [23] | RAJORIYA N, COMBET C, ZOULIM F, et al. How viral genetic variants and genotypes influence disease and treatment outcome of chronic hepatitis B. time for an individualised approach?[J]. J Hepatol, 2017, 67(6): 1281–1297. DOI:10.1016/j.jhep.2017.07.011 |
| [24] | LIU S J, ZHANG H W, GU C Y, et al. Associations between hepatitis B virus mutations and the risk of hepatocellular carcinoma: a meta-analysis[J]. J Natl Cancer Inst, 2009, 101(15): 1066–1082. DOI:10.1093/jnci/djp180 |
| [25] | LEE J H, HAN K H, LEE J M, et al. Impact of hepatitis B virus (HBV) x gene mutations on hepatocellular carcinoma development in chronic HBV infection[J]. Clin Vaccine Immunol, 2011, 18(6): 914–921. DOI:10.1128/CVI.00474-10 |



